È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sviluppo di linee cellulari muscolari knock-out utilizzando l'editing genetico CRISPR/Cas9 mediato da lentivirus
In questo articolo
Riepilogo
Il protocollo descrive come generare mioblasti knock-out utilizzando CRISPR/Cas9, partendo dalla progettazione di RNA guida alla clonazione cellulare e alla caratterizzazione dei cloni knock-out.
Abstract
Un'importante applicazione delle ripetizioni palindrome brevi intervallate regolatorie raggruppate (CRISPR)/Cas 9 è lo sviluppo di linee cellulari knock-out, in particolare per studiare la funzione di nuovi geni/proteine associati a una malattia, identificati durante la diagnosi genetica. Per lo sviluppo di tali linee cellulari, due questioni principali devono essere districate: l'inserimento degli strumenti CRISPR (il Cas9 e l'RNA guida) ad alta efficienza nelle cellule scelte e la restrizione dell'attività Cas9 alla delezione specifica del gene scelto. Il protocollo qui descritto è dedicato all'inserimento degli strumenti CRISPR in cellule difficili da trasfettare, come le cellule muscolari. Questo protocollo si basa sull'uso di lentivirus, prodotti con plasmidi pubblicamente disponibili, per i quali vengono descritte tutte le fasi di clonazione per colpire un gene di interesse. Il controllo dell'attività di Cas9 è stato effettuato utilizzando un adattamento di un sistema precedentemente descritto chiamato KamiCas9, in cui la trasduzione delle cellule con un lentivirus che codifica un RNA guida mirato al Cas9 consente la progressiva abolizione dell'espressione di Cas9. Questo protocollo è stato applicato allo sviluppo di una linea cellulare muscolare umana RYR1-knock out, che è stata ulteriormente caratterizzata a livello proteico e funzionale, per confermare il knockout di questo importante canale del calcio coinvolto nel rilascio intracellulare di calcio muscolare e nell'accoppiamento eccitazione-contrazione. La procedura qui descritta può essere facilmente applicata ad altri geni nelle cellule muscolari o in altre cellule difficili da trasfettare e produrre strumenti preziosi per studiare questi geni nelle cellule umane.
Introduzione
Con il progredire del sequenziamento genico e l'identificazione di mutazioni in geni di funzioni sconosciute in un tessuto specifico, lo sviluppo di modelli cellulari rilevanti per comprendere la funzione di un nuovo gene bersaglio e confermare il suo coinvolgimento nei relativi meccanismi fisiopatologici costituisce uno strumento essenziale. Inoltre, questi modelli sono di grande importanza per i futuri sviluppi terapeutici 1,2 e costituiscono un'interessante alternativa allo sviluppo di modelli animali knock-out in linea retta con le raccomandazioni internazionali per la riduzione dell'uso di animali nella sperimentazione. L'editing genetico con CRISPR/Cas9 è tra gli strumenti più potenti attualmente disponibili, che ha permesso lo sviluppo di molti modelli knock-out/knock-in, e la validazione genica mirata con CRISPR/Cas9 è tra le applicazioni più utilizzate di CRISPR/Cas93. Il successo dell'editing genetico si basa sulla capacità di introdurre gli strumenti CRISPR (gli RNA guida e la nucleasi Cas9) nel modello cellulare target, che può essere una sfida in molte cellule difficili da trasfettare, come le cellule muscolari4. Questa sfida può essere superata con l'uso del virus, di solito lentivirus, che ha il grande vantaggio di trasdurre in modo efficiente molti tipi di cellule e fornire il suo transgene. Ma il suo principale svantaggio è l'integrazione del transgene nel genoma della cellula ospite, che porta a una potenziale alterazione dei geni localizzati nel sito di integrazione e all'espressione permanente del transgene, che nel caso della nucleasi Cas9 comporterebbe conseguenze dannose5. Merienne e colleghi6 hanno proposto una soluzione intelligente, che consiste nell'introduzione nelle cellule di un RNA guida mirato al gene Cas9 stesso, portando all'inattivazione di Cas9. Un adattamento di questa strategia è presentato qui come un protocollo facile da usare e versatile che consente di eliminare praticamente qualsiasi gene in cellule difficili da trasfettare.
L'obiettivo del protocollo qui presentato è quello di indurre l'inattivazione di un gene di interesse nelle cellule muscolari immortalizzate. Può essere usato per mettere fuori uso qualsiasi gene di interesse, in diversi tipi di cellule immortalizzate. Il protocollo qui descritto contiene passaggi per progettare gli RNA guida e la loro clonazione in plasmidi lentivirali, per produrre gli strumenti CRISPR in vettori lentivirali, per trasdurre le cellule con i diversi lentivirus e per clonare le cellule per produrre una linea cellulare omogenea modificata.
Utilizzando questo protocollo, sono state sviluppate cellule muscolari scheletriche umane immortalizzate con delezione del recettore della rianodina di tipo 1 (RyR1), un canale del calcio essenziale coinvolto nel rilascio intracellulare del calcio e nella contrazione muscolare7. Il knock-out (KO) del gene è stato confermato a livello proteico utilizzando Western blot e a livello funzionale utilizzando l'imaging del calcio.
Protocollo
Le biopsie muscolari sono state ottenute dalla Banca dei tessuti per la ricerca (Myobank, partner della rete UE EuroBioBank, Parigi, Francia) in conformità con le raccomandazioni europee e la legislazione francese. Il consenso informato scritto è stato ottenuto da tutti gli individui. I mioblasti immortalati sono stati gentilmente prodotti dal Dr. V. Mouly (Istituto di Miologia, Parigi, Francia) e i protocolli sono stati approvati dal comitato etico dell'Istituto di Miologia (MESRI, n AC-2019-3502).
1. Progettazione della guida CRISPR
- Identificare la regione del gene da eliminare. Cerca la sua sequenza genomica utilizzando strumenti di browser del genoma come ensembl.org o genome.ucsc.edu e determina le coordinate cromosomiche delle due regioni per cercare l'RNA guida (gRNA), su entrambi i lati della regione da eliminare.
- Per il gene qui utilizzato, ottenere la sequenza FASTA del gene RYR1 e la sequenza dell'esone 101 come segue. In ensembl, cerca RYR1 nella versione più recente del genoma umano, seleziona la prima voce e fai clic sulla trascrizione della sequenza di codifica della proteina. Quindi, fare clic su Esoni per reindirizzare all'elenco degli esoni del gene.
- Fare clic su Scarica sequenza e selezionare Solo sequenza genomica per scaricare la sequenza di consenso completo dell'intero gene. Scorri verso il basso l'elenco degli esoni e degli introni del gene e seleziona quello o quelli mirati.
- Trova la sequenza nucleotidica corrispondente nel gene. Selezionare le sequenze nucleotidiche degli introni immediatamente a monte e a valle dell'esone da eliminare, che verranno utilizzate per cercare i gRNA.
- Progettare i due gRNA, chiamati guida 1 e guida 2 qui, nelle regioni identificate nella fase 1.1 (introni a monte e a valle della regione da eliminare) utilizzando strumenti online come Crispor.tefor.net8. Scegli i due gRNA separati da alcune centinaia di coppie di basi (bp), la sequenza di ciascun gRNA è esattamente lunga 20 nucleotidi senza il motivo adiacente del protospazio (PAM). Scegli le migliori guide disponibili per limitare l'off-target. Vedere la Figura 1 per un esempio di progettazione della guida.
- La sequenza tra le due guide dovrebbe essere cancellata e comportare il knock-out del gene di interesse, quindi scegliere la posizione delle due guide in modo tale da eliminare una sequenza essenziale o un esone essenziale nel gene di interesse. Assicurarsi che la sequenza/esone eliminato non sia presente esclusivamente in una trascrizione alternativa del gene e/o che codifichi una parte importante della proteina, quindi la sua delezione comporterà un knock-out funzionale.
NOTA: Sebbene si preveda che il sito di scissione Cas9 si verifichi 3 bp a monte del motivo adiacente del protospacer (PAM), può verificarsi anche la scissione a una distanza maggiore, quindi una buona soluzione non è quella di dipendere dalla localizzazione precisa del sito di scissione, come la scissione nell'introne.
- La sequenza tra le due guide dovrebbe essere cancellata e comportare il knock-out del gene di interesse, quindi scegliere la posizione delle due guide in modo tale da eliminare una sequenza essenziale o un esone essenziale nel gene di interesse. Assicurarsi che la sequenza/esone eliminato non sia presente esclusivamente in una trascrizione alternativa del gene e/o che codifichi una parte importante della proteina, quindi la sua delezione comporterà un knock-out funzionale.
- Determinare la sequenza del complemento inverso (RC) per ogni gRNA, senza il PAM, in modo da avere le seguenti sequenze: Guida 1 e Guida 1-RC, Guida 2 e Guida 2-RC.
- Ordinare i primer presentati nella Tabella 1 per eseguire la clonazione dei plasmidi. Durante tutto il protocollo, utilizzare primer ad una concentrazione di 10 nM in H2O sterile.
NOTA: Le sequenze aggiunte ai gRNA in questi primer (grassetto e sottolineato) corrispondono alla sequenza del plasmide prima e dopo la guida, il promotore e l'RNA trans-attivante-Crispr (tracrRNA), rispettivamente e non devono essere modificate al fine di garantire una buona sovrapposizione tra i primer e il plasmide.
2. Clonazione di plasmidi
NOTA: In questa fase, i gRNA verranno inseriti nella spina dorsale del plasmide per la produzione di lentivirus. Una cassetta che codifica i due gRNA viene prima prodotta da successive reazioni a catena della polimerasi (PCR), utilizzando i primer sovrapposti. La nuova cassetta viene quindi inserita nel plasmide della spina dorsale lentivirale #87919.
- Ottenere i seguenti plasmidi: plasmide #87919, codifica per RNA guida CRISPR in un vettore lentivirale e plasmide #87904 che codifica per la sequenza SpCas9 in un vettore lentivirale.
- Costruzione di cassette
NOTA: il protocollo di clonazione è riassunto in Figura 2.- Eseguire una reazione PCR (A), con 2 μL di plasmide #87919, 2 μL di primer_XmaIF, 2 μL di primer_Guide1R, 25 μL di miscela di polimerasi e 19 μL di H2O. Eseguire il seguente programma PCR (programma 1): denaturazione iniziale 5 min a 98 °C, seguita da 30 cicli di: 30 s a 98 °C, 30 s a 60 °C, 1 min 45 s a 72 °C e un allungamento finale di 7 min a 72 °C. Il Tm dei primer descritti al punto 1.4 è di 60 °C.
NOTA: Sebbene il tempo di allungamento nel programma 1 appaia piuttosto lungo, questo tempo di allungamento è stato scelto per garantire la produzione di materiale sufficiente delle giuste dimensioni. Infatti, a causa delle sequenze ripetute nel plasmide (la sequenza di tracrRNA dopo le guide dell'RNA viene ripetuta tre volte nel plasmide #87919) l'amplificazione PCR del DNA atteso è difficile e ulteriori bande più piccole sono prodotte nelle successive PCR. Pertanto, a causa della concorrenza tra i diversi prodotti PCR, o il tempo di allungamento è stato aumentato (per favorire quello più lungo e per avere abbastanza materiale purificato alla fine), o una PCR touch down (programma 2) è stata utilizzata per il frammento lungo (come descritto per la PCR (finale) nel passaggio 2.2.6). - Separare i prodotti PCR su un gel di agarosio all'1% in tampone Tris-Borato-EDTA (TBE), asportare e purificare il frammento da 300 bp. Eseguire la purificazione utilizzando un kit dedicato seguendo le istruzioni del fabbricante, con eluizione in un volume finale di 20 μL. Utilizzare direttamente il frammento purificato o conservare a -20 °C per il punto 2.2.5.
- Eseguire una reazione PCR (B), con 2 μL di plasmide #87919, 2 μL di primer_Guide1F, 2 μL di primer_Guide2R, 25 μL di miscela di polimerasi e 19 μL di H2O utilizzando il programma PCR 1. Separare i prodotti PCR su un gel di agarosio all'1% in TBE, asportare e purificare il frammento da 400 bp in 20 μL di tampone di eluizione. Utilizzare direttamente il frammento purificato o conservare a -20 °C per il passaggio 2.2.5.
- Eseguire una reazione PCR (C), con 2 μL di plasmide #87919, 2 μL di primer_Guide2F, 2 μL di primer_BlpIR, 25 μL di miscela di polimerasi e 19 μL di H2O utilizzando il programma PCR 1. Separare i prodotti PCR su un gel di agarosio all'1% in tampone TBE. Accisare e purificare il frammento da 600 bp in 20 μL di tampone di eluizione. Utilizzare direttamente il frammento purificato o conservare a -20 °C per il passaggio 2.2.6.
NOTA: Un altro frammento a 900 bp può essere visibile, a causa dell'ibridazione del primer sulle regioni ripetute nel plasmide, come descritto nella NOTA del passo 2.2.1. Se presente, questa banda dovrebbe essere scartata. - Eseguire una reazione PCR (D), con 2 μL di PCR di eluizione A (dallo step 2.2.2), 2 μL di PCR di eluizione B (dal step 2.2.3), 2 μL di primer_XmaIF, 2 μL di primer_Guide2R, 25 μL di miscela di polimerasi e 19 μL di H2O, con programma PCR 1. Separare i prodotti PCR su un gel di agarosio all'1% in tampone TBE; asportare e purificare il frammento da 700 bp in 20 μL di tampone di eluizione. Utilizzare direttamente il frammento purificato o conservare a -20 °C per il passaggio 2.2.6.
NOTA: Altre bande possono essere visibili a >1.000 bp, 400 bp e 300 bp, a causa dell'ibridazione dei primer sulle regioni ripetute e devono essere scartate. - Eseguire una reazione PCR (finale), con 4 μL di PCR di eluizione C (dallo stadio 2.2.4), 4 μL di PCR D di eluizione (dallo stadio 2.2.5), 4 μL di primer_XmaIF, 4 μL di primer_BlpIR, 50 μL di miscela di polimerasi e 34 μL di H2O. Il programma utilizzato è il seguente (programma PCR 2): denaturazione iniziale per 5 min a 98 °C; sei cicli di: 30 s a 98 °C, 30 s a 66 °C (riduzione della temperatura di ibridazione di 1 °C per ciclo), 1 min 45 a 72 °C; 35 cicli di: 30 s a 98 °C, 30 s a 60 °C, 1 min 45 a 72 °C e un allungamento finale di 5 min a 72 °C.
NOTA: Il programma PCR per questa amplificazione finale è una PCR touch down, diversa dalla precedente, a causa delle grandi dimensioni dell'amplificazione finale che contiene due sequenze di tracrRNA ripetute subito dopo ogni guida. - Separare i prodotti PCR su un gel di agarosio all'1% in tampone TBE; asportare e purificare la cassetta finale, che migra a circa 1.300 bp in 20 μL di tampone di eluizione. Quantificare il prodotto eluito. Utilizzare direttamente il frammento purificato o conservare a -20 °C per il passaggio 2.3.2.1. Questo è il prodotto finale che verrà inserito nel plasmide lentivirale.
NOTA: Altri frammenti possono essere visibili a >1000 bp, 400 bp e 300 bp, corrispondenti a frammenti di PCR incompleti che devono essere scartati.
- Eseguire una reazione PCR (A), con 2 μL di plasmide #87919, 2 μL di primer_XmaIF, 2 μL di primer_Guide1R, 25 μL di miscela di polimerasi e 19 μL di H2O. Eseguire il seguente programma PCR (programma 1): denaturazione iniziale 5 min a 98 °C, seguita da 30 cicli di: 30 s a 98 °C, 30 s a 60 °C, 1 min 45 s a 72 °C e un allungamento finale di 7 min a 72 °C. Il Tm dei primer descritti al punto 1.4 è di 60 °C.
- Inserimento della cassetta gRNA nella spina dorsale del plasmide lentivirale.
- Linearizzare il plasmide mediante doppia digestione del plasmide #87919 con gli enzimi di restrizione XmaI e BlpI.
- Preparare la reazione con 15 μL di tampone raccomandato, 15 μL di plasmide (1μg/μL), 7,5 μL di enzima BlpI (a 10 U/μL), 7,5 μL di enzima XmaI (a 10 U/μL) e 112,5 μL di H2O. Incubare per 1 ora a 37 °C, e poi per 20 minuti a 65 °C. Caricare la quantità totale su un gel di agarosio all'1%, ritagliare e purificare il plasmide ~10 kb con un kit appropriato. L'eluizione viene eseguita in 20 μL di tampone e il prodotto eluito viene quantificato utilizzando la misurazione ottica della densità.
NOTA: utilizzare un protocollo di purificazione appropriato per frammenti di DNA di grandi dimensioni, come il protocollo descritto da Sun e coll9.
- Preparare la reazione con 15 μL di tampone raccomandato, 15 μL di plasmide (1μg/μL), 7,5 μL di enzima BlpI (a 10 U/μL), 7,5 μL di enzima XmaI (a 10 U/μL) e 112,5 μL di H2O. Incubare per 1 ora a 37 °C, e poi per 20 minuti a 65 °C. Caricare la quantità totale su un gel di agarosio all'1%, ritagliare e purificare il plasmide ~10 kb con un kit appropriato. L'eluizione viene eseguita in 20 μL di tampone e il prodotto eluito viene quantificato utilizzando la misurazione ottica della densità.
- Ligate la cassetta gRNA e il plasmide. Preparare la miscela di reazione con la cassetta di gRNA dal passo 2.2.7 e il plasmide linearizzato dal passo 2.3.1.1, aggiungere 2 μL di enzima e H2O per avere un volume finale di 10 μL. Incubare per 15 minuti a 50 °C per produrre il plasmide finale chiamato p_guides.
NOTA: La quantità di cassetta del DNA deve essere compresa tra 50-100 ng e la quantità di plasmide tra 100-200 ng, con un rapporto molare 2: 1.
- Linearizzare il plasmide mediante doppia digestione del plasmide #87919 con gli enzimi di restrizione XmaI e BlpI.
- Utilizzare 2 μL del plasmide appena preparato per trasformare E.Coli chimicamente competenti come Stbl3 (50 μL) o XL10-Gold e distribuire su piastra di agar LB con ampicillina da 100 μg/mL dopo 1 ora di crescita a 37 °C senza antibiotico. Incubare a 37 °C durante la notte. Scegli alcune colonie ed esegui una mini preparazione utilizzando un kit commerciale seguendo le istruzioni del produttore.
- Eseguire un'amplificazione PCR sul DNA miniprep con Primer_XmaI e Primer_BlpR utilizzando il programma PCR 1 (vedere Figura 2). Separare i prodotti PCR su un gel di agarosio all'1% in tampone TBE. Selezionare alcune colonie (~5) con una banda alla dimensione prevista di circa 1300 bp.
- Eseguire il sequenziamento del DNA delle colonie selezionate, utilizzando il Primer_XmaI o Primer_BlpR, per confermare il corretto inserimento della cassetta gRNA.
NOTA: Una delle colonie verificate in sequenza viene ulteriormente utilizzata nello studio e il plasmide è chiamato p_guides. - Ripeti dal passaggio 2.2 (costruzione della cassetta) con i Primers-Killer F e R e i Primers-mCherry F e R. Usa una colonia verificata dalla sequenza per ulteriori analisi. Il plasmide è chiamato p_Killer.
3. Produzione di lentivirus
- Produrre e purificare una grande quantità di tutti i plasmidi necessari utilizzando un kit di preparazione maxi-prep privo di endotossine seguendo le istruzioni del produttore. Preparare aliquote a 2 μg/μL. Conservare a -20 °C
- Preparazione delle cellule (Giorno 1)
- Preparare 18 piastre da 145 cm seminate con 1 x 106 heK293 cellule per piastra in 16 mL di mezzo composto da piruvato ad alto contenuto di glucosio ad alto contenuto di glucosio dmEM (DMEM) di Dulbecco, integrato con il 10% di siero bovino fetale (FBS) e l'1% di penicillina/streptomicina. Amplificare le celle a 37 °C, in un incubatore a CO2 al 5% per 3 giorni.
NOTA: La produzione di lentivirus deve essere eseguita con cautela in un laboratorio di livello di biosicurezza 2, utilizzando dispositivi di protezione adattati, tra cui tuta protettiva monouso, cappuccio protettivo e guanti. Tutti gli esperimenti devono essere fatti sotto una cappa a flusso laminare (armadio di sicurezza BSLII) con punte filtranti. Tutte le soluzioni contenenti lentivirus e tutti i rifiuti di plastica/vetro usati devono essere inattivati con etanolo al 70% o altro inattivatore virale.
- Preparare 18 piastre da 145 cm seminate con 1 x 106 heK293 cellule per piastra in 16 mL di mezzo composto da piruvato ad alto contenuto di glucosio ad alto contenuto di glucosio dmEM (DMEM) di Dulbecco, integrato con il 10% di siero bovino fetale (FBS) e l'1% di penicillina/streptomicina. Amplificare le celle a 37 °C, in un incubatore a CO2 al 5% per 3 giorni.
- Trasfezione delle cellule (Giorno 4)
- Controllare la confluenza delle cellule, per essere sicuri che abbiano raggiunto il 60% -65% di confluenza.
- Preparare la soluzione di trasfezione contenente per ogni piastra: 20,8 μg del plasmide di interesse (p-guides, o p-Killer, o pCas9 #87904), 4,8 μg del plasmide che codifica l'involucro (VSV-G, #8454), 20,8 μg del plasmide psPAX2 (#12260) per il confezionamento lentivirale, 136 μL di fosfato di calcio e regolare con H2O ad un volume finale di 1.000 μL. Aggiungere questa soluzione a goccia e sotto agitazione a 1 mL di 2x soluzione salina tamponata HEPES (HBS).
NOTA: Non preparare una miscela per tutte le piastre contemporaneamente per garantire una preparazione ottimale dei reagenti; preparare un mix per sei piatti contemporaneamente, ad esempio, per 18 piatti, preparare tre volte un mix per sei piatti. - Incubare a temperatura ambiente (RT) per almeno 10 minuti e aggiungere 2 ml di soluzione a goccia alle cellule. Omogeneizzare il reagente di trasfezione con una delicata agitazione della piastra all'indietro, in avanti, su e giù, incubare a 37 °C, 5% CO2 per almeno 5 ore.
NOTA: Da questo punto e fino alla fine della produzione di lentivirus, indossare dispositivi di protezione aggiuntivi, tra cui un secondo paio di guanti, maniche di protezione e un piastrone monouso. - Cinque ore dopo la trasfezione, rimuovere il mezzo dalle piastre e risciacquare con PBS per eliminare i reagenti di trasfezione. Aggiungere 12 mL di terreno fresco e incubare 48 h a 37 °C, 5% CO2.
- Raccolta delle particelle virali (Giorno 6)
- Raccogli e raggruppa il mezzo da tutti i piatti. Centrifugare a 800 x g per 5 min a 4 °C, per pellettizzare i detriti cellulari. Filtrare il surnatante utilizzando un filtro da 0,45 μm (sono necessari più filtri).
- Centrifuga a 68.300 x g per 2 ore a 4 °C in un rotore a benna oscillante. Rimuovere il surnatante e lasciare i tubi capovolti sotto l'armadio di sicurezza su una carta per 5-10 minuti per rimuovere più liquido possibile, quindi aggiungere 100 μL di mezzo di proliferazione HEK per pellet. Dopo almeno 2 ore a 4 °C, risospese il pellet mediante pipettaggio su e giù. Metti in comune tutti i pellet risospesi.
NOTA: Il pellet può essere lasciato nel mezzo durante la notte a 4 °C prima dell'aliquotazione. - Aliquota lentivirus in 10 μL o 25 μL di dimensione del campione (a seconda dell'uso) e congelamento a scatto con azoto liquido. Conservare a -80 °C. Non congelare un'aliquota che è stata scongelata.
- Ripetere i passaggi da 3.2 a 3.4 con altri plasmidi di interesse per produrre guide LV, LV-Killer e LV-Cas9.
NOTA: In alternativa, i lentivirus possono essere acquistati da un'azienda o da una struttura antivirus.
4. Titolazione del lentivirus
NOTA: la titolazione del virus viene eseguita su cellule HEK293. La titolazione è importante per incorporare nei passaggi successivi un numero preciso di lentivirus per cellula (qualunque sia il lotto di lentivirus), per le cellule di interesse. Il numero di particelle virali che trasducono in modo efficiente una cellula è chiamato molteplicità di infezione (MOI): MOI 10 corrisponde quindi all'introduzione di 10 particelle virali per cellula. Poiché il ciclo di congelamento/scongelamento influisce sulla vitalità del lentivirus, la titolazione viene eseguita con un'aliquota di lentivirus congelata e ogni esperimento successivo verrà eseguito con una nuova aliquota dello stesso pool. Un metodo di titolazione è descritto qui, ma altri metodi possono essere utilizzati.
- Il giorno 1, seme 1 x 105 celle per piastra in cinque lastre da 35 mm con coperchi di vetro sul fondo e due lastre da 35 mm senza coverslips.
- Il giorno 2, dalle due piastre senza coverslips, raccogliere e contare il numero di cellule dopo la tripsinizzazione e determinare la quantità media di cellule per piastra (N).
- Preparare 100 μL di lentivirus diluito a 1/10 nel mezzo di proliferazione. Trasdurre le cinque piastre coltivate con diversi volumi di virus diluito, da 1 a 50 μL di virus diluito. Per questo protocollo, utilizzare i seguenti volumi per trasdurre le cinque piastre: 1 μL, 5 μL, 10 μL, 20 μL, 50 μL. Incubare per 48 h a 37 °C, in un 5% CO2.
- Il giorno 4, fissare le cellule mediante incubazione dei coverslip a RT per 20-30 minuti in paraformaldeide al 4%. Per il LV-Cas9, permeabilizzare le cellule con 0,1% Triton X100 in Phosphate Buffer Saline (PBS) per 10 min a RT, saturare in PBS-0,1% Triton X100-2% Goat Serum- 0,5% Bovine Serum (BSA) per 20 min a RT, ed etichettare per 45 min a RT con anticorpo primario anti-V5 (diluizione 1/400) seguito da 30 min di incubazione a RT con anticorpo secondario fluorescente. Etichettare i nuclei con Hoescht (10 μg/mL in PBS) per 10 min a RT.
- Montare i coverslip su un vetrino e osservare utilizzando un microscopio fluorescente dotato di un obiettivo 20x. Per le guide LV e LV-Killer, dopo la fissazione procedere direttamente all'etichettatura dei nuclei e montare i coverslip. Per ogni coverslip, contare il numero totale di nuclei (numero totale di celle) nel campo visivo e il numero di celle etichettate (con V5 o mCherry) e determinare il rapporto tra le cellule etichettate per ogni coverslip.
- Selezionare un coverslip in cui il rapporto trasdotto delle cellule sia almeno del 10% e non superiore al 50%. Determinare l'efficienza di trasduzione (X) per questo coverslip e notare il volume di virus diluito (V, in μL) utilizzato per ottenere questa efficienza di trasduzione.
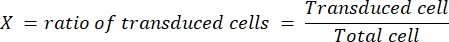
- Determinare il titolo (in particelle infettive, rappresentate come ip/mL) del virus secondo la seguente formula:

Il fattore di diluizione è la diluizione del lentivirus eseguita nel passaggio 4.3. Abbiamo regolarmente ottenuto un titolo finale di 1 x 109 ip / mL per LV-Cas9 e 1 x 1010 ip / mL per LV-guide, determinato in celle HEK.
5. Trasduzione dei mioblasti
NOTA: I mioblasti immortalizzati vengono successivamente trasdotti con i tre lentivirus precedentemente prodotti. Sono mantenuti ad una densità inferiore al 50% in un terreno di proliferazione composto da F10 di Ham integrato con il 20% di FBS, il 2% di penicillina / streptomicina, il 2% di Ultroser G e coltivato a 37 ° C, 5% CO2.
- Determinare il volume di lentivirus necessario per trattare il numero scelto di cellule con MOI 10 per LV-Cas9 e LV-guide e MOI 20 per LV-killer, secondo la seguente formula:
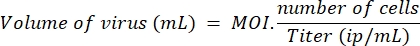
NOTA: In un esperimento parallelo, l'efficienza di trasduzione è stata confrontata su mioblasti e HEK, utilizzando un lentivirus di controllo (lenti-GFP), e abbiamo determinato che sono necessari cinque volte più lentivirus per trasdurre un mioblasto in modo efficiente rispetto a una cellula HEK. Pertanto, MOI 10 misurato su HEK corrisponde a due particelle virali per mioblasto. I MOI utilizzati qui sono calcolati su celle HEK. - Il giorno 1, seminare piastre a 96 pozzetti con 10.000 cellule in 100 μL di terreno di proliferazione per pozzetto. Il giorno 2, trasdurre le celle sotto l'armadio di sicurezza aggiungendo il volume appropriato di guide LV e LV-Cas9 calcolato nel passaggio 5.1. Restituire le cellule all'incubatrice fino al giorno 7.
NOTA: L'uso di MOI superiore a 25, in particolare per LV-Cas9, potrebbe non migliorare il numero di cellule modificate a causa della maggiore morte cellulare ad un'alta concentrazione di lentivirus. - Il giorno 7, eseguire la tripsinizzazione e contare le cellule. Semina le cellule a una confluenza dal 40% al 50% in una nuova piastra e restituisci le cellule all'incubatrice. Cinque ore dopo, trasdurre le cellule con LV-Killer ad un MOI di 20 (volume calcolato nel passaggio 5.1). Amplificare le cellule per 5-10 giorni dopo la trasfezione, per almeno due passaggi, e mantenerle sempre ad una bassa confluenza di <50%. Le cellule sono pronte per il passo successivo, la clonazione cellulare, quando la loro crescita viene riportata alla normalità (stimata dal tempo di divisione).
NOTA: La proliferazione potrebbe essere un po' più lenta dopo la trasduzione. La normale crescita cellulare può essere determinata prima di questa procedura sperimentale stimando il tempo di divisione delle cellule.
6. Clonazione cellulare
NOTA: Poiché la trasduzione dei mioblasti è difficile e non raggiunge mai il 100% di efficienza, anche quando si utilizza il lentivirus, è necessaria la clonazione cellulare per ottenere una linea cellulare completamente corretta. Questo è possibile solo con cellule immortalizzate, o cellule che possono essere coltivate e amplificate durante poche settimane / mesi.
- Tripsinizzare e contare le cellule. Diluire le cellule in mezzo di proliferazione a 10 cellule/mL e seminare le cellule a 1 cellula/pozzetto in piastre a 96 pozzetti contenenti 100 μL/pozzetto di mezzo.
NOTA: Il numero di piastre da seminare dipende dalla probabilità dell'editing genetico previsto, da 2 a 10 piastre vengono utilizzate di routine. - Monitorare la crescita delle cellule e amplificare progressivamente ogni pozzetto in una piastra più grande fino a raggiungere almeno una piastra di 35 mm, mantenendo la confluenza dei mioblasti al di sotto del 50%. Questo passaggio può durare 2-6 settimane, a seconda delle cellule utilizzate e della loro capacità di crescere una volta isolate in un pozzo.
7. Selezione del clone
NOTA: questo passaggio viene eseguito per identificare quali dei cloni in crescita sono stati opportunamente modificati.
- Progettare un set di primer composto da un primer situato prima della prima guida (Primer_BeforeGuide1F) e un altro primer situato dopo la seconda guida (Primer_AfterGuide2R) al fine di amplificare la regione che racchiude la sequenza presumibilmente modificata. Vedere la Tabella 1 per i primer utilizzati qui.
- Raccogli le cellule da ogni clone, risparmia almeno 300.000 cellule per l'amplificazione futura ed estrai il DNA genomico utilizzando qualsiasi protocollo standard sulle cellule rimanenti.
- Se un gran numero di cloni sta crescendo, per scartare quelli non modificati, eseguire un test rapido raggruppando le cellule di cinque cloni nella stessa provetta per estrarre il DNA da questo pool e testare con PCR. Ripeti con tutti i cloni necessari. Quindi, separare ulteriormente i pool che contenevano celle modificate per eseguire analisi individuali.
- Controlla l'editing tramite PCR come segue. Preparare la reazione PCR con 1 μL di Primer_BeforeGuide1F, 1 μL di Primer_AfterGuide2R, 12,5 μL di miscela di polimerasi, 3 μL di DNA genomico e 7,5 μL di H2O. Amplificare in un termociclatore secondo le istruzioni del produttore e i parametri dei primer. Esegui su un gel di agarosio all'1% per identificare i cloni modificati.
- Controllare la modifica sequenziando come segue. Eseguire il sequenziamento Sanger dei cloni selezionati per confermare l'eliminazione e identificare come è stata eseguita la modifica in ciascun clone. Mantenere più di un clone modificato per assicurarsi che solo il gene bersaglio sia stato modificato ed è responsabile dell'effetto fisiologico osservato e mantenere un clone non modificato che verrà utilizzato come clone di controllo (CTRL) negli esperimenti successivi.
- Espandere i cloni selezionati. Una volta che la confluenza di ciascun clone ha raggiunto circa il 50%, tripsinizzare le cellule e piallare le cellule in un piatto più grande, fino a quando non sono state prodotte abbastanza cellule per eseguire le caratterizzazioni biochimiche e funzionali (di solito più di 1 x 106 per clone) e conservare aliquote congelate di ciascun clone per un uso futuro.
8. Caratterizzazione dei cloni modificati
NOTA: Una volta che alcuni cloni sono stati scelti e confermati dal sequenziamento del DNA, la delezione del gene bersaglio può essere confermata a livello proteico utilizzando Western blot e a livello funzionale se è disponibile un test cellulare funzionale per questo gene. Nel caso di RYR1-KO, poiché RyR1 è un canale del calcio, la caratterizzazione funzionale è stata eseguita utilizzando l'imaging del calcio su cellule in coltura.
- Espressione proteica in cloni modificati
NOTA: RyR1 è espresso solo in miotubi differenziati10. La sua espressione è stata valutata nei miotubi utilizzando Western blot, per confermare la delezione a livello proteico di RyR1, così come la delezione della proteina Cas9.- Piastra 200.000 cellule in mezzo di proliferazione (descritto sopra, fase 5) su una superficie di circa 1,76 cm2 in una piastra da 35 mm rivestita di laminina (superficie corrispondente a una goccia di laminina di 200 μL a 10 mg/mL in PBS con calcio). Una volta che le cellule sono attaccate alla piastra dopo essere state incubate per 2-3 ore a 37 ° C, 5% CO2, spostare il terreno di coltura su un mezzo di differenziazione composto da DMEM basso glucosio + 10% siero di cavallo + 1% penicillina / streptomicina e restituire le cellule all'incubatrice per 6 giorni.
- Dopo 6 giorni di differenziazione, raccogliere e lisare le cellule con 200 μL di RIPA integrati con inibitori della proteasi. Determinare la concentrazione proteica utilizzando il metodo Folin Lowry11.
- Caricare 15 μg di proteine, dopo denaturazione per 30 min a RT nel tampone di denaturazione Laemmli, su un gel di acrilammide sfumato al 5%-15%. Dopo la separazione elettroforetica, trasferire le proteine su Immobilon P a 0,8 V per 4 h11.
- Dopo la saturazione della membrana per 30 minuti a RT in PBS contenente 0,1% Tween 20 e 5% di latte secco non grasso, incubare la membrana con gli anticorpi primari diluiti nello stesso tampone per 2 ore a RT o durante la notte a 4 °C, lavare la membrana 5x per 5 min con PBS-0,1% Tween 20 e incubare la membrana con gli anticorpi secondari per 1 ora a RT. Gli anticorpi primari utilizzati sono: anticorpi contro V5-tag (diluizione: 1/5000) per rilevare Cas9, anti-GAPDH (diluizione: 1/1000) come controllo del carico, anticorpo anti-RyR112,13 (diluizione: 1/10.000), anticorpo contro la subunità alfa 1 di DHPR (diluizione: 1/1000) e anticorpo contro la catena pesante della miosina MF20 (diluizione: 1/1000).
- Lavare la membrana 5x per 5 min con PBS-0.1% Tween 20, asciugare l'eccesso di liquido e aggiungere il substrato chemiluminescente. Procedere come raccomandato dal fornitore del substrato per rilevare il segnale chemiluminescente.
- Caratterizzazione funzionale dei cloni modificati
NOTA: La funzione di RyR1 è stata valutata utilizzando l'imaging del calcio in miotubi differenziati, prodotti da cloni CTRL o KO14.- Piastra 50.000 cellule su una superficie di 0,2 cm2 al centro di piatti da 35 mm rivestiti di laminina (superficie coperta da una goccia di laminina da 50 μL, a 10 mg / mL in PBS con calcio) e indurre la differenziazione per 6 giorni come descritto nel passaggio 8.1.1. Preparare tre piastre per ogni stimolazione, per avere un triplice biologico.
- Caricare i miotubi con 50 μL di fluo 4-diretto, diluito 1:1 in mezzo di differenziazione e incubato per 30 min a 37 °C. Risciacquare le cellule due volte con tampone KREBS integrato con glucosio a 1 mg/ml.
- Misurare le variazioni di fluorescenza con un microscopio fluorescente invertito o un microscopio confocale utilizzando un obiettivo 10x. Installare la piastra sul palco del microscopio e avviare l'acquisizione a 1 fotogramma al secondo per 90 s.
- Rimuovere il KREBS rimanente e stimolare le cellule al fotogramma 25 con l'aggiunta di 2 mL di KCl per la depolarizzazione della membrana (concentrazione finale di 140 mM) o 2 mL di 4 CmC (concentrazione finale di 500 μM) per la stimolazione diretta di RyR1. Assicurarsi che almeno 10 miotubi siano presenti nel campo registrato.
- Quantificare la variazione di fluorescenza in ogni miotubo, utilizzando un software dedicato. Selezionare per l'analisi almeno 10 miotubi per piatto (idealmente 20-30 miotubi per piatto), tracciare una linea (o una regione di interesse (ROI)) sull'asse lungo di ciascun miotubo e raccogliere la fluorescenza F lungo questa linea per tutti i fotogrammi.
- Determinare il valore fluorescente iniziale, F0, corrispondente ai fotogrammi da 1 a 24. Traccia la variazione fluorescente (F-F0)/F0 in funzione del tempo da 0 a 90 s. Ripeti l'esperimento tre volte per ottenere la variazione di fluorescenza da almeno 90 miotubi di tre diverse colture. Raggruppa tutti i risultati per i 90 miotubi e calcola il SEM medio ± di (F-F0)/F0 in ogni intervallo di tempo. Quantificare l'ampiezza di picco del rilascio di calcio per ogni stimolazione e ogni clone.
Risultati
Questo protocollo è stato applicato a mioblasti immortalizzati da un soggetto sano15 (le cosiddette cellule HM, per i mioblasti umani), in cui il RyR1 è stato precedentemente caratterizzato16, al fine di eliminare il gene RYR1 che codifica per la proteina RyR1. Il design dell'RNA delle guide è stato fatto per eliminare la sequenza che comprende parte dell'esone 101 e dell'introne 101 del gene. Si prevede che la cancellazione di parte dell'esone 101 provocherà l...
Discussione
Un passo importante sulla strada verso la caratterizzazione di geni di funzione sconosciuta coinvolti in patologie è lo sviluppo di modelli cellulari rilevanti per studiare la funzione di questi geni. L'uso dell'editing genetico utilizzando CRISPR / Cas9 è un campo di ricerca in crescita esponenziale e lo sviluppo di modelli knock-out come presentato qui è tra le sue applicazioni più utilizzate. In questo contesto, proponiamo qui un protocollo versatile per sviluppare un knock-out della linea cellulare umana in quals...
Divulgazioni
Gli autori non hanno conflitti di interesse da divulgare.
Riconoscimenti
Questo lavoro è stato finanziato da sovvenzioni dell'Association Française contre les myopathies (AFM-Téléthon) e dell'Auvergne-Rhône Alpes Région (AURA).
Materiali
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
Riferimenti
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon