A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
الشاشات الوراثية المجمعة المستندة إلى كريسبر في خلايا الثدييات
In This Article
Summary
توفر تقنية CRISPR-Cas9 طريقة فعالة لتحرير الجينوم الثدييات بدقة في أي نوع من أنواع الخلايا وتمثل وسيلة جديدة لأداء الشاشات الوراثية على نطاق الجينوم. ويرد هنا بروتوكول مفصل يناقش الخطوات اللازمة للنجاح في أداء شاشات كريسبر-كاس9 المجمعة على نطاق الجينوم.
Abstract
وقد أدى تحرير الجينوم باستخدام نظام كريسبر-كاس إلى تقدم كبير في القدرة على تحرير الجينومات من مختلف الكائنات الحية بدقة. وفي سياق خلايا الثدييات، تمثل هذه التكنولوجيا وسيلة جديدة لإجراء شاشات جينية على نطاق الجينوم من أجل دراسات علم الجينوم الوظيفية. تسمح مكتبات الإرشاد RNAs (sgRNA) التي تستهدف جميع إطارات القراءة المفتوحة بتوليد الآلاف من الاضطرابات الوراثية السهلة في مجموعة واحدة من الخلايا التي يمكن فحصها لأنواع ظاهرية محددة لتوريط وظيفة الجينات والعمليات الخلوية في بطريقة غير متحيزة ومنهجية. توفر شاشات CRISPR-Cas للباحثين طريقة بسيطة وفعالة وغير مكلفة للكشف عن المخططات الوراثية للأنماط الظاهرية الخلوية. وعلاوة على ذلك، فإن التحليل التفاضلي للشاشات التي يتم إجراؤها في مختلف خطوط الخلايا ومن أنواع السرطان المختلفة يمكن أن يحدد الجينات الضرورية من حيث السياق في الخلايا السرطانية، مما يكشف عن الأهداف المحتملة لعلاجات محددة مضادة للسرطان. ويمكن أن يكون أداء الشاشات على نطاق الجينوم في الخلايا البشرية أمراً شاقاً، لأن ذلك ينطوي على معالجة عشرات الملايين من الخلايا ويتطلب تحليلاً لمجموعات كبيرة من البيانات. غالباً ما يتم تجاهل تفاصيل هذه الشاشات، مثل توصيف خط الخلية، واعتبارات مكتبة CRISPR، وفهم حدود وقدرات تقنية CRISPR أثناء التحليل. ويرد هنا بروتوكول مفصل للأداء الناجح للشاشات المجمعة على نطاق الجينوم CRISPR-Cas9.
Introduction
كريسبر-كاس، قصيرة لتتجمع بانتظام بين المسافات قصيرة باليندروميك وتكرار المرتبطة كريسبر، يتكون من بروتين nuclease واحد (على سبيل المثال، Cas9) في مجمع مع الحمض النووي الريبي دليل الاصطناعية (sgRNA). يستهدف هذا المركب الريبونوكليوبروتين إنزيم Cas9 للحث على فواصل الحمض النووي المزدوج ة في موضع جيني محدد1. يمكن إصلاح الفواصل المزدوجة التي تقطعت بها السبل عن طريق إصلاح موجه ة (HDR) أو، بشكل أكثر شيوعًا، من خلال الانضمام النهائي غير المتجانس (NHEJ)، وهي آلية إصلاح عرضة للخطأ تؤدي إلى الإدراج و/أو الحذف (INDELS) التي تعطل في كثير من الأحيان وظيفة الجينات 1.كفاءة وبساطة كريسبر تمكن من مستوى لا يمكن تحقيقه من قبل من استهداف الجينوم الذي يتجاوز بكثير تقنيات تحرير الجينوم السابقة [أي، الزنك الاصبع nucleases (ZNF) أو الفاعل المنشط النسخ مثل nucleases ( TALENS)، وكلاهما يعاني من تعقيد التصميم المتزايد، وانخفاض كفاءة التغوط، والقيود في تحرير الجينات متعددة2].
وقد أتاح التطبيق البحثي الأساسي لتحرير الجينوم القائم على الحمض النووي الريبي من كريسبر بكفاءة وبتكلفة زهيدة للعلماء أن يستجوبوا بكفاءة وبتكلفة غير مكلفة وظائف الجينات الفردية وطبولوجيا شبكات التفاعل الجيني. وقد تعززت القدرة على أداء الشاشات الوظيفية على نطاق الجينوم إلى حد كبير باستخدام نظام كريسبر-كاس، لا سيما عند مقارنتها بتكنولوجيات الاضطراب الجيني السابقة مثل تداخل الحمض النووي الريبي (RNAi) وموت فخ الجينات. على وجه الخصوص، RNAi يعاني من آثار عالية خارج الهدف وضربة قاضية غير مكتملة، مما أدى إلى انخفاض الحساسية والخصوصية بالمقارنة مع كريسبر3،4،5،في حين أن أساليب فخ الجينات هي ممكنة فقط في haploid خلايا لشاشات فقدان وظيفة، والحد من نطاق نماذج الخلية التي يمكن استجوابها6. قدرة كريسبر على توليد الجينات كاملة خروج المغلوب يوفر نظام أكثر قوة بيولوجيا لاستجواب الأنماط الظاهرية متحولة، مع انخفاض مستوى الضجيج، والحد الأدنى من الآثار خارج الهدف والنشاط المستمر عبر الكواشف5. تتوفر الآن على نطاق واسع مكتبات CRISPR-Cas9 sgRNA التي تستهدف الجينوم البشري بأكمله، مما يسمح بتوليد الآلاف من الضربات الذاتية للجين في تجربةواحدة3و7و8و9 .
لقد قمنا بتطوير مكتبات فريدة من نوعها على نطاق الجينوم كريسبر-Cas9 sgRNA lentiviral تسمى مكتبات تورونتو خروج المغلوب (TKO) (المتاحة من خلال Addgene) التي هي المدمجة وتسلسل الأمثل لتسهيل شاشات الجينوم وظيفية عالية الدقة. أحدث مكتبة، TKOv3، تستهدف ~ 18،000 الجينات الإنسان البروتين الترميز مع 71،090 أدلة الأمثل لكفاءة التحرير باستخدام البيانات التجريبية10. بالإضافة إلى ذلك، TKOv3 متاح كمكتبة مكون واحد (LCV2::TKOv3، معرف Addgene #90294) التعبير عن Cas9 وsgRNAs على متجه واحد، مما يخفف من الحاجة إلى توليد خلايا ثابتة التعبير عن Cas9، مما يتيح ضرب على نطاق الجينوم عبر مجموعة واسعة من أنواع الخلايا الثدييات. TKOv3 هو متاح أيضا في ناقلات دون Cas9 (pLCKO2::TKOv3, معرف Addgene # 125517) ويمكن استخدامها في الخلايا التي تعبر Cas911.
يمكن أن يتعرض عدد الخلايا المحررة من CRISPR-Cas9 على نطاق الجينوم لظروف نمو مختلفة، مع وفرة sgRNAs مع مرور الوقت كمياً من خلال تسلسل الجيل التالي، مما يوفر قراءة لتقييم التسرب أو إثراء الخلايا ذات الجينات الوراثية التي يمكن تتبعها الاضطرابات. يمكن تسخير مكتبات كريسبر للخروج من الباب لتحديد الجينات التي تسبب، عند الاضطراب، عيوب ًا في اللياقة البدنية الخلوية، أو حساسية معتدلة للأدوية (مثل الجينات الحساسة أو المقاومة)، أو تنظم التعبير عن البروتين (مراسل، على سبيل المثال)، أو تكون مطلوبة لمعين وظيفة المسار والدولة الخلوية12،13،14. على سبيل المثال، شاشات اللياقة البدنية التفاضلية في خط الخلايا السرطانية يمكن تحديد كلمن استنفاد أو الحد من الأنكوجينيات والإثراء أو زيادة الجينات مثبطات الورم3،14،15. وبالمثل، يمكن استخدام جرعات وسيطة من الأدوية العلاجية تكشف عن كل من مقاومة المخدرات وجينات التوعية16،17.
وترد هنا بروتوكول فحص مفصل لفحص فقدان الوظائف على نطاق الجينوم CRISPR-Cas9 باستخدام مكتبات تورونتو للخروج من الباب العام (TKOv1 أو v3) في خلايا الثدييات من توليد المكتبات، وأداء الفرز إلى تحليل البيانات. على الرغم من أن هذا البروتوكول قد تم تحسينه للفرز باستخدام مكتبات تورونتو خروج المغلوب، فإنه يمكن تطبيقه وتصبح قابلة للتحجيم لجميع المكتبات تجمع كريسبر sgRNA.
Access restricted. Please log in or start a trial to view this content.
Protocol
وينبغي أن تتبع التجارب المبينة أدناه المبادئ التوجيهية لمكتب الصحة والسلامة البيئيين التابع للمعهد.
1. مجمعة كريسبر sgRNA مكتبة لينفيروسيت تضخيم بلازميد
- تمييع مكتبة الحمض النووي البلازميد البلازميد الجاهز ة كريسبر إلى 50 نانوغرام/ميكرولتر في TE (على سبيل المثال، TKOv3).
- الكهربائي المكتبة باستخدام الخلايا الكهربائية المختصة. إعداد ما مجموعه أربعة تفاعلات الكهربائي كما هو موضح أدناه.
- إضافة 2 ميكرولتر من 50 نانوغرام /ميكرولتر مكتبة TKO إلى 25 ميكرولتر من الخلايا الكهربائية المذابة إلى cuvettes المبردة مسبقا (1.0 ملم) على الجليد.
- الكهربائي باستخدام الإعدادات المثلى المقترحة من قبل بروتوكول الشركة المصنعة. في غضون 10 ثانية من النبض، إضافة 975 درجة مئوية من الانتعاش المتوسطة (أو SOC المتوسطة) إلى كوفيت.
- نقل الخلايا الكهربائية إلى أنبوب الثقافة وإضافة 1 مل من الانتعاش المتوسطة. أنابيب الحضانة في حاضنة تهتز في 250 دورة في الدقيقة لمدة 1 ساعة في 37 درجة مئوية.
- إعداد لوحة تخفيف لtiter المكتبة وتقدير كفاءة التحول.
- تجمع جميع 8 مل من الخلايا المستردة ومزيج جيدا. نقل 10 ميكرولتر من الخلايا المجمعة إلى 990 ميكرولتر من الانتعاش المتوسطة لتخفيف 800 أضعاف وتخلط جيدا.
- لوحة 20 درجة مئوية من التخفيف على 10 سم مسخن مسبقا LB + carbenicillin (100 ميكروغرام / لتر) لوحة أجار. وهذا يؤدي إلى تخفيف 40،000 أضعاف من التحولات التي سيتم استخدامها لحساب كفاءة التحول.
- لوحة 400 درجة مئوية من الخلايا المستردة على كل لوحة عبر ما مجموعه 20 قبل تسخينها 15 سم LB + carbenicillin لوحات أجار. احتضان لوحات لمدة 14-16 ساعة عند 30 درجة مئوية.
ملاحظة: النمو في هذه درجة الحرارة المنخفضة يقلل من إعادة التركيب بين تكرار المحطة الطرفية الطويلة (LTR)18. - لحساب كفاءة التحويل، عد عدد المستعمرات على لوحة التخفيف 40،000 أضعاف (الخطوة 1.3.2). ضرب عدد المستعمرات التي تم عدها بـ 40,000 للحصول على العدد الإجمالي للمستعمرات على جميع اللوحات. المضي قدما إذا كان العدد الإجمالي للمستعمرات يمثل تغطية مكتبة تعادل الحد الأدنى من المستعمرات 200x لكل sgRNA (الأكثر الأمثل هو 500-1000x).
- على سبيل المثال، الحد الأدنى لعدد المستعمرة لمكتبة TKOv3 (71,090 sgRNA) هو 1.4 × 107، وهو ما يعادل 200x المستعمرات لكل sgRNA. إذا كان تمثيل المستعمرة غير كاف، قم بزيادة عدد البُرَسِّة في الخطوة 1.2 استناداً إلى عدد المستعمرات على لوحة التخفيف لتحقيق الحد الأدنى من تغطية المكتبة.
- حصاد المستعمرات كما هو موضح أدناه
- إلى كل لوحة 15 سم، إضافة 7 مل من LB + carbenicillin (100 ميكروغرام / لتر) المتوسطة، ثم كشط المستعمرات قبالة مع موزع الخلية. مع ماصة 10 مل، نقل الخلايا كشط في قارورة مخروطية معقمة 1 L أو زجاجة.
- مرة أخرى شطف لوحة مع 5 مل من LB + carbenicillin المتوسطة ونقل الحل إلى زجاجة.
- كرر لجميع لوحات لتجميع الخلايا من 20 لوحات في زجاجة معقمة.
- خلط الخلايا التي تم جمعها مع شريط تحريك لمدة 1 ساعة في درجة حرارة الغرفة (RT) لتفريق كتل الخلية. نقل الخلايا إلى زجاجات الطرد المركزي وأجهزة الطرد المركزي تزن مسبقا في 7000 × ز إلى البكتيريا بيليه، ثم تجاهل وسائل الإعلام.
- وزن بيليه الخلية الرطبة وطرح وزن زجاجة الطرد المركزي لتحديد الوزن النهائي للبيليه الرطب. تنقية الحمض النووي بلازميد باستخدام مجموعة تنقية بلازميد ماكسي أو ميجا النطاق اعتمادا على كمية من الكريات البكتيرية كل عمود يمكن معالجة.
2. على نطاق واسع كريسبر sgRNA مكتبة إنتاج فيروس لينتي
ملاحظة: يتم تنفيذ كافة الخطوات في هذا القسم من البروتوكول في مرفق BSL2+ في خزانة السلامة الأحيائية من النوع A2 من الفئة الثانية.
- حساب عدد لوحات 15 سم المطلوبة لإنتاج الفيروس استناداً إلى تقدير أن 18 مل من الفيروس يتم حصادها عادة من لوحة واحدة 15 سم.
- إعداد الخلايا للمتدخلة عن طريق زرع خلايا التعبئة والتغليف HEK-293T في وسائل الإعلام نمو منخفض المضادات الحيوية (DMEM + 10٪ FBS + اختياري: 0.1X القلم / العقديات) في 8 × 106 خلايا لكل لوحة 15 سم في 20 مل من وسائل الإعلام. حضانة الخلايا بين عشية وضحاها في 37 درجة مئوية، 5٪ CO2. تأكد من أن الخلايا المطلية هي 70٪ -80٪ مترافقة وتنتشر بالتساوي في لحظة من التغوط.
- في اليوم التالي، قم بإعداد ثلاثة خليط بلازميدات الانفعلى النحو المبين في الجدول 1 للوحات 15 سم. حساب كمية البلازميد اللازمة للمغيرات واحد وجعل مزيج من بلازميدات لعدد من لوحات، بالإضافة إلى واحد ليتم نقلها.
- إعداد كاشف الانف القابل للتغوط القائم على الدهون لكل عملية نقل على النحو المبين في الجدول 2. خفضت Aliquot وسائل الإعلام المصل في أنابيب الطرد المركزي الصغيرة 1.5 مل الفردية لعدد من لوحات ليتم نقلها. يُضاف كاشف الإنفذيب، ويُمزج برفق، ويُحضن لمدة 5 دقائق في RT.
- بعد 5 دقائق الحضانة، إضافة كمية الحمض النووي المطلوبة لالتغوط واحد إلى كاشف التغوط لنسبة 3:1 من الكاشف transfection إلى ميكروغرام من مجمع الحمض النووي. يُمزج المزيج برفق والحضانة لمدّة 30 دقيقة في RT.
ملاحظة: يمكن إعداد عمليات التغوط اللاحقة في مجموعات من خمسة أو أقل، مع فواصل 5 دقائق لتحسين الوقت وتجنب الإفراط في الحضانة. - بعد 30 دقيقة من الحضانة، نقل بعناية كل مزيج transfection إلى كل لوحة من خلايا التعبئة والتغليف. أضف المزيج بأكمله باستخدام طرف ماصة 1 مل في حركة دائرية متعرجة دون إزعاج الطبقة الأحادية للخلية. حضانة الخلايا في 37 درجة مئوية لمدة 18 ساعة في 5٪ CO2.
- إعداد وسائل الإعلام الحصاد الفيروسي: 500 مل من DMEM المتوسطة + 32 مل من مخزون BSA (20 غرام / 100 مل، حل في DMEM، وتصفيتها مع فلتر 0.22 م) + 5 مل من 100X القلم / العقدية.
- بعد 18 ساعة، إزالة وسائل الإعلام (استخدام المناولة السليمة من النفايات لينتيفيروس مثل الحضانة في 1٪ هيبوكلوريت الصوديوم لمدة 30 دقيقة قبل التخلص منها). استبدال بلطف مع 18 مل من وسائل الإعلام الحصاد الفيروسي إلى كل لوحة. حضانة الخلايا في 37 درجة مئوية لمدة 18 ساعة في 5٪ CO2.
- بعد 24 ساعة، تحقق من خلايا التعبئة والتغليف للمورفولوجيا غير طبيعية وتنصهر كمؤشر على إنتاج الفيروس جيدة. ثم، حصاد الفيروس اللين عن طريق جمع كل supernatant ونقل هافين إلى أنبوب الطرد المركزي المخروطية العقيمة.
- تدور وسائل الإعلام التي تحتوي على الفيروس في 300 × ز لمدة 5 دقائق وبيليه خلايا التعبئة والتغليف. Aliquot supernatant في أنبوب البولي بروبلين المعقمة دون إزعاج بيليه.
- تخزين الفيروس في 4 درجة مئوية لفترات قصيرة (أقل من 1 أسبوع) أو على الفور في -80 درجة مئوية للتخزين على المدى الطويل. Aliquot فيروس على نطاق واسع تستعد لأحجام استخدام واحد للتخزين على المدى الطويل لتجنب تجميد / ذوبان.
3- توصيف خط الخلية للفحص
- حدد خط الخلية المطلوب.
- قياس وتسجيل الوقت تقريبا مضاعفة من الخلايا.
- تحديد كثافة طلاء الخلايا المثلى لخلايا الزراعة كل 3-4 خلايا تتضاعف في وعاء زراعة الأنسجة المفضل (على سبيل المثال، لوحات زراعة الأنسجة 15 سم).
- تحديد تركيز البورومسين لاستخدامه في خط الخلية المطلوب لاختيار مكتبات TKO التي تحتوي على علامة مقاومة البوروميسين على النحو التالي:
- خلايا البذور في لوحة بئر 12 في الكثافة المطلوبة للوصول إلى التقاء بعد 72 ساعة، ثم حضانة بين عشية وضحاها (37 درجة مئوية، 5٪ CO2).
- في اليوم التالي، تغيير إلى وسائل الإعلام التي تحتوي على مجموعة تخفيف تركيزات البورومسين من 0 ميكروغرام / مل إلى 10 ميكروغرام / مل، في زيادات 0.5 ميكروغرام / مل. حضانة الخلايا لمدة 48 ساعة.
- بعد 48 ساعة، قياس صلاحية الخلية عن طريق عد الخلايا أو تلطيخ alamarBlue.
- تحديد أدنى تركيز يقتل 100% من الخلايا في 48 ح. استخدم هذا التركيز لتحديد مجموعات الخلايا المستحثة لمكتبة كريسبر في الخطوتين 4-6 و5-2-6.
ملاحظة: بالنسبة لخطوط الخلايا مع أوقات مضاعفة أطول، يمكن التسامح مع الحضانة أطول مع البوروميسين. في هذه الحالات، حدد منحنى القتل لوقت الحضانة المطلوب لمضاعفة الخلية <3. تقليل وقت الاختيار لتجنب تسرب الجينات الأساسية قبل بدء الفحص.
- فحص الخلايا للتأكد من حساسيتها لبروميد سداسي الديميثرين (حتى 8 ميكروغرام/مل) عن طريق إجراء منحنى استجابة للجرعة بنفس الطريقة المستخدمة لقياس حساسية البورومسين (الخطوة 3-2). إذا لوحظت سمية مع <8 ميكروغرام/مل من بروميد سداسي الديمثيرين، لا تستخدم.
4. المعايرة الوظيفية لمكتبة فيروس لينتي كريسبر المجمعة لتحديد وزارة الداخلية
- قم بذوبان مجموعة جديدة من الـ 100 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000 000
- تصميم سلسلة من وحدات تخزين الفيروس لاختبار ما بين نطاق 0-2 مل (أي 0 مل، 0.25 مل، 0.5 مل، 1 مل، و 2 مل).
- حصاد الخلايا المستهدفة وخلايا البذور في لوحات 15 سم في الكثافة المطلوبة للوصول إلى التقاء في 72 ساعة.
- لكل حجم فيروس ليتم اختبارها، وإعداد لوحات مكررة. إضافة الخلايا، والفيروس، وبروميد سداسي الديمثيرين (8 ميكروغرام/مل)، والوسائط إلى حجم نهائي قدره 20 مل. لوحات مزيج تماما، والجلوس مستوى لوحات في حاضنة وحضانة لمدة 24 ساعة (37 درجة مئوية، 5٪ CO2).
- بعد 24 ساعة، إزالة الفيروس الذي يحتوي على وسائل الإعلام والتخلص (استخدام احتياطات السلامة البيولوجية للتعامل مع النفايات lentivirus). اختياريا، غسل بلطف لوحة مع PBS الدافئة لإزالة الفيروس الدخيل.
- لكل حالة فيروس، يستعاض عن 20 مل من الوسائط التي تحتوي على البورومسين باستخدام التركيز المحدد لقتل الخلايا في القسم 3، إلى لوحة واحدة تكرار. إلى لوحة أخرى، إضافة 20 مل من وسائل الإعلام الطازجة دون البورومسين. حضانة لمدة 48 ساعة (37 درجة مئوية، 5٪ CO2).
- بعد 48 ساعة، تحقق من أن جميع الخلايا غير المصابة (0 مل حالة الفيروس) تعامل مع البورومسين قد لقوا حتفهم. حصاد جميع لوحات على حدة وتفريق الخلايا عن طريق الأنابيب لطيف المتكررة.
- عد الخلايا من جميع اللوحات وحساب وزارة الداخلية لكل حجم الفيروس عن طريق مقارنة عدد الخلايا مع اختيار البورومسين إلى عدد الخلايا دون البوروميسين (أي + / - بورومسين).
- نتائج الرسم البياني لتحديد حجم الفيروس الذي يؤدي إلى 30٪ -40٪ بقاء الخلية مع اختيار البوروميسين مقابل دون البوروميسين. استخدم حجم الفيروس هذا لتحقيق وزارة الداخلية من 0.3-0.4 أثناء الشاشة تحت نفس ظروف زراعة الأنسجة.
5. عدوى الشاشة الأساسية، واختيار، وتمرير الخلية
- حدد تغطية مكتبة كريسبر sgRNA ليتم الاحتفاظ بها في جميع أنحاء الشاشة (يوصى بالحد الأدنى 200 ضعف).
- استناداً إلى تغطية المكتبة، حدد عدد الخلايا المطلوبة للحفاظ على هذه التغطية لكل sgRNA وعدد الخلايا المطلوبة للعدوى في وزارة الداخلية 0.3(الجدول 3).
- تحديد عدد اللوحات المطلوبة لإعداد العدوى(الجدول 4).
- إصابة الخلايا بمكتبة كريسبر
- حصاد الخلايا والبذور رقم الخلية المطلوبة لكل لوحة 15 سم.
- يُضاف بروميد سداسي الديمثيرين (8 ميكروغرام/مل) إلى جميع الأطباق.
- إضافة الفيروس في وحدة التخزين المطلوبة لMOI 0.3 إلى الفرز ولوحات التحكم 2. للتحكم 1 لا تقم بإضافة فيروس واستبدال وحدة التخزين هذه مع الوسائط.
- مزيج لوحات جيدا عن طريق إمالة. وضع لوحات في حاضنة، والتأكد من أنها على مستوى.
ملاحظة: يمكن أن يتم العدوى دفعة عن طريق الجمع بين مزيج رئيسي من الفيروس، وسائل الإعلام، وبروميد هيكساديميثيرين إلى الخلايا في تعليق قبل الطلاء. - إزالة وسائل الإعلام واستبدالها مع وسائل الإعلام الجديدة التي تحتوي على البورومسين في التركيز المحدد في الخطوة 3.2.4 إلى فحص والسيطرة 1 لوحات 24 ساعة بعد العدوى بالفيروس. إضافة وسائل الإعلام الطازجة مع عدم وجود البورومسين إلى لوحة التحكم 2. حضانة الخلايا لمدة 48 ساعة (37 درجة مئوية، 5٪ CO2).
- 48 ح بعد إضافة البورومسين، تأكد من أن جميع الخلايا غير المصابة ميتة (السيطرة 1) لتأكيد نشاط البورومسين، ثم حصاد الخلايا المصابة.
- حصاد خلايا مصابة وتمرير الخلايا
- حصاد الخلايا التي اختارها البورومسين من جميع لوحات الفحص في حاوية معقمة واحدة. جمع الخلايا من كل لوحة التحكم على حدة. تفريق الخلايا عن طريق الأنابيب المتكررة لطيف.
- عد الخلايا من خلايا الفحص المجمعة، والتحكم 1، والتحكم في 2 بشكل منفصل وحساب عدد الخلايا لكل 1 مل.
- حساب وزارة الداخلية وأضعاف التغطية التي تحققت على النحو التالي:
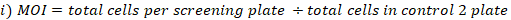
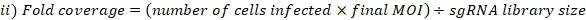
- جمع ثلاثة نسخ من الكريات الخلية من الخلايا المجمعة في تغطية المكتبة المختارة لاستخراج الحمض النووي الجينومي. الطرد المركزي الخلايا في 500 × ز لمدة 5 دقائق. تسمية الأنابيب وتجميد الجافة الكريات الخلية في -80 درجة مئوية (هذه هي عينات مرجعية T0).
- تقسيم مجموعة الخلايا المصابة إلى ثلاث مجموعات نسخ متماثل (على سبيل المثال، نسخ A، نسخ B، نسخ متماثل C)، مع الحفاظ على تغطية المكتبة داخل كل تكرار. خلايا البذور في نفس كثافة البذر كما يمكن استخدامها عادة عند توسيعها. استخدم نفس عدد الخلايا لكل لوحة نسخ متماثل ونفس العدد الإجمالي للخلايا بين النسخ المتماثل.
- الاستمرار في مرور الخلايا وحصاد ثلاثة نسخ من الكريات الخلية من كل تكرار للخلايا المصابة مجمعة على النحو الوارد أعلاه، كل 3-8 أيام اعتمادا على خط الخلية، لمدة تصل إلى 15-20 الخلية مضاعفة. في كل ممر، حصاد الخلايا من جميع لوحات في كل مجموعة تكرار مع بعضها البعض (أي، يتم إعادة خلط جميع الخلايا من لوحات A تكرار معا، يتم إعادة خلط جميع الخلايا من لوحات B تكرار معا، وما إلى ذلك).
- تسمية كل بيليه مع الوقت (T) وتكرار تعيين. وهذا يتوافق مع عدد الأيام بعد T0 يتم جمع بيليه (على سبيل المثال، T3_A، T3_B، T3_C، الخ).
- لاختيار شاشات المخدرات السلبية، والسماح للخلايا لاسترداد لمرور واحد على الأقل بعد T0 قبل العلاج. في T3 أو T6، تقسيم الخلايا من كل مجموعة تكرار (A، B، C) إلى العلاج من المخدرات والسيطرة على السكان، وذلك باستخدام نفس كثافة البذر المستخدمة في الخطوة 5.3.5.
- بشكل منفصل تجميع عدد الخلايا المطلوبة لتغطية المكتبة لكل تكرار في مجموعة العلاج من المخدرات. إضافة الدواء في تركيزات وسيطة (IC20-IC50). بذور الخلايا والحضانة (37 درجة مئوية، 5٪ CO2)حتى مرور المقبل.
- بشكل منفصل تجمع عدد الخلايا المطلوبة لتغطية المكتبة لكل تكرار في مجموعة التحكم في السيارة. إضافة التحكم في السيارة باستخدام نفس حجم المخدرات (<0.5% v/v). بذور الخلايا والحضانة (37 درجة مئوية، 5٪ CO2)حتى الممر التالي.
- الاستمرار في مرور الخلايا وحصاد الكريات الخلية للحمض النووي الجينومي كل 3 أيام كما هو موضح في الخطوة 5.3.5، في حين تحديث الدواء أو السيارة في كل ممر.
- لاختيار إيجابي أو شاشات مقاومة المخدرات، وتقسيم كل مجموعة تكرار وفقا لعدد الخلايا المطلوبة لتغطية المكتبة. إضافة IC90 تركيزات المخدرات إلى كل تكرار. في IC90، سيتم قتل غالبية الخلايا. السماح للسكان المقاومين بالنمو وجمع كريات الخلايا (1-2 × 107 خلايا) لاستخراج الحمض النووي الجينومي.
6. كريسبر إعداد العينة وتسلسل
- تنقية الحمض النووي الجينومي
- احتضان الكريات الخلية المجمدة لمدة 5-10 دقيقة في RT للذوبان.
- إضافة 1.4 مل من PBS إلى أنبوب الطرد المركزي 50 مل تحتوي على بيليه الخلية. دوامة لمدة 20 ق لإعادة تعليق الخلايا والراحة لمدة 1 دقيقة. إذا لزم الأمر، ماصة 15X مع P1000 لتفريق كتل الخلية المتبقية. إذا نقل الخلايا من أنبوب 15 مل أو 1.5 مل، إعادة تعليق الخلايا مع 1 مل من PBS، ثم نقل الخلايا إلى أنبوب 50 مل وشطف الأنبوب الأصلي مع 400 درجة مئوية من PBS.
- إضافة 5 مل من محلول الليسيس النووي إلى الخلايا التي تم تعليقها. باستخدام ماصة 10 مل، مزيج العينة عن طريق الأنابيب صعودا وهبوطا 5X.
- إضافة 32 ميكرولتر من RNase A (20 ملغ/مل؛ للحصول على تركيز نهائي قدره 100 ميكروغرام/مل) إلى الليسات النووية وخلط العينة عن طريق عكس الأنبوب 5X. تحضن الخليط في 37 درجة مئوية لمدة 15 دقيقة والسماح للعينة لتبرد لمدة 10 دقائق في RT.
- إضافة 1.67 مل من محلول هطول الأمطار البروتين إلى lysate ودوامة بقوة لمدة 20 ق. كتل البروتين الصغيرة قد تكون مرئية بعد الخلط.
- الطرد المركزي في 4500 × ز لمدة 10 دقائق في RT.
- باستخدام ماصة 10 مل، نقل supernatant إلى أنبوب الطرد المركزي 50 مل تحتوي على 5 مل من isopropanol. خلط بلطف الحل 10X عن طريق الانعكاس حتى يتم ملاحظة الحمض النووي.
ملاحظة: يمكن ملاحظة الحمض النووي كخيوط بيضاء تشبه الخيط تشكل كتلة مرئية. - الطرد المركزي في 4500 × ز لمدة 5 دقائق في RT لبيليه الحمض النووي.
- باستخدام ماصة 10 مل، وإزالة بعناية supernatant وتجنب خلع بيليه الحمض النووي. إضافة 5 مل من الإيثانول 70٪ في RT إلى الحمض النووي. تدوير بلطف أنبوب لغسل بيليه الحمض النووي والجانبين من أنبوب الطرد المركزي.
- الطرد المركزي في 4500 × ز لمدة 5 دقائق في RT.
- باستخدام ماصة 10 مل، وإزالة بعناية الإيثانول 70٪ وتجنب خلع بيليه الحمض النووي. الحمض النووي الجينومي الجاف لمدة 10 دقائق في RT.
- إضافة 400 درجة مئوية من حل TE إلى الأنبوب والسماح للحمض النووي تذوب عن طريق الحضانة في 65 درجة مئوية لمدة 1 ساعة. إذا كان الحمض النووي لا يذوب تماما، وحضانة أنبوب في 65 درجة مئوية لمدة 1 ساعة إضافية في حين نفض الغبار بلطف الأنبوب كل 15 دقيقة، وتركها في 4 درجة مئوية بين عشية وضحاها.
- الطرد المركزي في 4500 × ز لمدة دقيقة واحدة في RT ونقل الحمض النووي الجيني إلى أنبوب 1.5 مل منخفضة ملزمة.
- قياس وقياس نقاء الحمض النووي الجينومي على كل من مقياس الطيف الضوئي (لإجمالي محتوى الحمض النووي) ومقياس الفلور (لمحتوى الحمض النووي المزدوج الذين تقطعت بهم السبل).
- اختياريا، يعجل الحمض النووي الجينومي إذا كان هناك مشاكل مع تضخيم PCR المصب من sgRNA على النحو التالي.
- نقل 400 ميكرولتر الحمض النووي الجينومي في أنبوب الطرد المركزي الصغير 1.5 مل.
- إضافة 18 ميكرولتر من 5 M NaCl (التركيز النهائي من 0.2 M) و 900 درجة مئوية من الإيثانول 95٪.
- عكس أنبوب 10X حتى مختلطة تماما، ثم الطرد المركزي في 16،000 × ز لمدة 10 دقيقة في RT.
- إزالة بعناية supernatant وتجنب خلع بيليه الحمض النووي. غسل بيليه الحمض النووي مع 500 درجة مئوية من الإيثانول 70٪. تدوير بلطف أنبوب لغسل بيليه الحمض النووي.
- الطرد المركزي في 16،000 × ز لمدة 5 دقائق في RT.
- إزالة بعناية supernatant وتجنب خلع بيليه الحمض النووي. الحمض النووي الجينومي الجاف لمدة 10 دقائق في RT.
- إضافة 300 درجة مئوية من TE لإذابة الحمض النووي كما هو موضح في الخطوات 6.1.12.
- تحديد وقياس نقاء الحمض النووي الجينومي على النحو المبين في الخطوة 6-1-14.
- إعداد مكتبة تسلسل كريسبر
- إعداد PCR 1 على النحو المبين في الجدول 5 باستخدام ما مجموعه 100 ميكروغرام من الحمض النووي الجينومي. إضافة 3.5 ميكروغرام من الحمض النووي الجينومي لكل رد فعل 50 ميكرولتر وإعداد ردود فعل متطابقة 50 ميكرولتر لتحقيق التغطية المطلوبة. يسرد الجدول 6 أمثلة للتسلسلات التمهيدية لتضخيم مكتبات التسلسل LCV2::TKOv3. يسرد الجدول 7 أمثلة للتسلسلات التمهيدية لتضخيم مكتبات التسلسل pLCKO2::TKOv3.
- تضخيم تفاعلات PCR 1 في دراجة حرارية باستخدام البرنامج المبين في الجدول 8.
- تحقق من تضخيم PCR 1 عن طريق تشغيل 2 ميكرولتر من منتج PCR على هلام أغاروز 1٪. PCR 1 ينتج منتج من 600 نقطة أساس.
- تجمع جميع ردود الفعل الفردية 50 درجة مئوية لكل عينة الحمض النووي الجينومي ومزيج من دوامة.
- إعداد تفاعل PCR 2 واحد (50 ميكرولتر) لكل عينة على النحو المبين في الجدول 9 باستخدام 5 ميكرولتر من منتج PCR 1 المجمع كقالب. استخدم تركيبات التمهيدي ة الفريدة لكل عينة فردية للسماح بتجميع عينات مكتبة التسلسل.
- تضخيم تفاعل PCR 2 في دراجة حرارية باستخدام البرنامج المبين في الجدول 10.
- تنظيف معدات هلام أغاروز لتنقية المنتجات تضخيم مع 0.1 N حمض الهيدروكلوريك لمدة 10 دقيقة قبل صب هلام. إعداد هلام أغاروز 2٪ تحتوي على وصمة عار الحمض النووي لتنقية PCR 2 تضخيم المنتجات.
- تشغيل المنتج PCR 2 على هلام أجاروز 2٪ في الجهد المنخفض (1.0-1.5 ساعة تشغيل). PCR 2 ينتج منتج من 200 نقطة أساس.
- تصور منتجات PCR على مضيء الضوء الأزرق. قم باستئصال النطاق 200 نقطة أساس وتنقية الحمض النووي من شريحة هلام أغاروز باستخدام مجموعة استخراج الجل. تحديد وقياس نقاء مكتبة التسلسل على كل من مقياس الطيف الضوئي ومقياس الفلور.
ملاحظة: يتراوح تركيز مكتبة التسلسل المنقّي للجل النموذجي من 5-10 نانوغرام/ميكرولتر وغلة إجمالية تتراوح بين 150 و300 نانوغرام.
- تسلسل عالي الإنتاجية
- تسلسل مكتبات تسلسل CRISPR على أجهزة التسلسل من الجيل التالي.
- تسلسل مرجع عينات T0 في عمق القراءة أعلى من 400-500 أضعاف تغطية المكتبة. تسلسل عينات الوقت التجريبية للشاشات المنسدلة في عمق القراءة على الأقل من 200 أضعاف. بالنسبة لشاشات الاختيار الإيجابية القوية، يكفي الحد الأدنى من عمق القراءة من تغطية 50 ضعفًا لتحديد sgRNAs المخصب.
ملاحظة: من الضروري تسلسل نموذج T0 لتحديد تمثيل المكتبة لشاشة معينة و تكون مرجعاً لتحديد التغييرات أضعاف sgRNA مع مرور الوقت.
7 - تحليل البيانات
- محاذاة التسلسل باستخدام برامج مثل Bowtie لتعيين تسلسل يقرأ إلى المكتبة المرجعية باستخدام المعلمات التالية: -v2 (السماح اثنين من عدم التطابق) و -m1 (تجاهل أي قراءة التي تم تعيينها إلى أكثر من تسلسل واحد في المكتبة).
- تطبيع عدد القراءات المعينة بشكل فريد لكل sgRNA لعينة معينة إلى 10 مليون قراءة لكل عينة كما يلي:
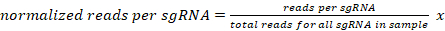 107
107
- حساب تغيير أضعاف log2 من كل sgRNA لكل تكرار في كل point timepoint (Tn) مقارنة مع نموذج T0 (Tn/T0). إضافة عدد زائف من 0.5 يقرأ إلى كافة عمليات قراءة عمليات التوقف لمنع الانقطاع عن الأصفار. استبعاد sgRNAs مع <30 يقرأ الخام في عينة T0 من حساب تغيير الطي وتحليل المصب.
- تحليل التغييرات أضعاف مع تحليل بايزي للجينات الأساسية (BAGEL) خوارزمية , باستخدام مجموعات التدريب الأساسية وغير الأساسية المحددة سابقا19 لشاشات أساسيات الجينات ( الجدول التكميلي S1) أو DrugZ لشاشات المخدرات20.
- حساب الدقة والاستدعاء لتقييم أداء الشاشة باستخدام درجات BF. استخدم المجموعة الأساسية من الخطوة 7.4 كقائمة إيجابية حقيقية لدالة precision_recall_curve مكتبة Scikit-learn لPython، جنباً إلى جنب مع المجموعة الفرعية درجة BF أعلاه. بدلاً من ذلك، تنفيذ نفس باستخدام حزمة PRROC في R.
- حساب متوسط تغيير أضعاف من جميع الأدلة لكل جين. توليد قطع أرض الكثافة للجينات الأساسية وغير الأساسية (انظر الخطوة 7.4) في R أو ما يعادلها من البرامج. في R، إذا كان x.ess هو متجه يحتوي على قيم تغيير أضعاف السجل للجينات الأساسية وx.nonEss تحتوي على جينات غير أساسية، رسم باستخدام الأمر التالي:
مؤامرة (كثافة (x.ess)، xlab = "يعني logFC"، كول = "الأحمر"، lwd = 2 )
خطوط (كثافة (x.nonEss)، كول = "الأزرق"، lwd = 2 )
ملاحظة: للاطلاع على تفاصيل إصدار Python والحزم المستخدمة، راجع scikit-learn v0.19.1: (نشرت من قبل Pedregosa et al.21).
Access restricted. Please log in or start a trial to view this content.
النتائج
نظرة عامة على سير عمل فحص كريسبر على نطاق الجينوم
يوضح الشكل 1 لمحة عامة عن تدفق أعمال فحص كريسبر المجمع، بدءاً من إصابة الخلايا المستهدفة بفيروس لينتي في مكتبة كريسبر في محطة وزارة الداخلية المنخفضة لضمان أحداث ...
Access restricted. Please log in or start a trial to view this content.
Discussion
نظرًا لبساطتها في الاستخدام والموثوقية العالية، فقد تم اعتماد تقنية CRISPR على نطاق واسع كأداة اختيار لتحرير الجينوم بدقة. يوفر فحص كريسبر المجمع طريقة لاستجواب الآلاف من الاضطرابات الوراثية في تجربة واحدة. في الشاشات المجمعة، تعمل مكتبات sgRNA كالباركود الجزيئي، حيث أن كل تسلسل فريد ويتم تع?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
ولا يعلن صاحبا البلاغ عن أي مصالح مالية متنافسة.
Acknowledgements
وقد دعم هذا العمل جينوم كندا، وصندوق أونتاريو للبحوث، والمعاهد الكندية للبحوث الصحية (MOP-142375, PJT-148802).
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 0.22 micron filter | |||
| 30°C plate incubator | |||
| 37°C shaking incubator | |||
| 37°C, 5% CO2 incubator | |||
| 5 M NaCl | Promega | V4221 | |
| 50X TAE buffer | BioShop | TAE222.4 | |
| 6 N Hydrochloric acid solution | BioShop | HCL666.500 | |
| 95% Ethanol | |||
| Alamar blue | ThermoFisher Scientific | DAL1025 | |
| Blue-light transilluminator | ThermoFisher Scientific | G6600 | |
| Bovine Serum Albumin,Heat Shock Isolation, Fraction V. Min. 98%, Biotechnology grade | Bioshop | ALB001.250 | |
| Dulbecco's Modification of Eagles Medium | Life Technologies | 11995-065 | Cel culture media |
| Electroporation cuvettes | BTX | 45-0134 | |
| Electroporator | BTX | 45-0651 | |
| Endura electrocompetent cells | Lucigen | 90293 | |
| Fetal Bovine Serum | GIBCO | 12483-020 | |
| HEK293T packaging cells | ATCC | CRL-3216 | recommend passage number <15 |
| Hexadimethrine Bromide (Polybrene) | Sigma | H9268 | Cationic polymer to enhance transduction efficiency |
| Hexadimethrine Bromide (Polybrene) | |||
| LB agar plates with carbenicillin | |||
| LB medium with carbenicillin | |||
| Low molecular weight DNA ladder | New England Biolabs | N3233S | |
| Nanodrop spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| NEBNext Ultra II Q5 Master Mix | New England Biolabs | M0544L | |
| Opti-MEM | Life Technologies | 31985-070 | Reduced serum media |
| Plasmid maxi purification kit | Qiagen | 12963 | |
| pMD2.G (envelope plasmid) | Addgene | Plasmid #12259 | lentiviral system |
| psPAX2 (packaging plasmid) | Addgene | Plasmid #12260 | lentiviral system |
| Puromycin | Wisent | 400-160-UG | |
| QIAquick gel extraction kit | Qiagen | 28704 | |
| Qubit dsDNA BR assay | ThermoFisher Scientific | Q32853 | |
| Qubit fluorometer | ThermoFisher Scientific | Q33226 | |
| RNAse A | Invitrogen | 12091021 | |
| S.O.C recovery medium | Invitrogen | 15544034 | |
| SYRB Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| Toronto KnockOut CRIPSR library (TKOv3) - Cas9 included | Addgene | Addgene ID #90203 | Genome-wide CRISPR library , includes Cas9, 71,090 sgRNA |
| Toronto KnockOut CRIPSR library (TKOv3) - non-cas9 | Addgene | Addgene ID #125517 | Genome-wide CRISPR library, non-Cas9, 71,090 sgRNA |
| Tris-EDTA (TE) solution, pH8.0 | |||
| UltraPure agarose | ThermoFisher Scientific | 16500500 | |
| Wizard genomic DNA purification kit | Promega | A1120 | |
| X-tremeGENE 9 DNA transfection reagent | Roche | 06 365 809 001 | Lipid based transfection reagent |
References
- Jiang, F., Doudna, J. A. CRISPR-Cas9 Structures and Mechanisms. Annual Review of Biophysics. 46, 505-529 (2017).
- Baliou, S., et al. CRISPR therapeutic tools for complex genetic disorders and cancer (Review). International Journal of Oncology. 53 (2), 443-468 (2018).
- Hart, T., et al. High-Resolution CRISPR Screens Reveal Fitness Genes and Genotype-Specific Cancer Liabilities. Cell. 163 (6), 1515-1526 (2015).
- Morgens, D. W., Deans, R. M., Li, A., Bassik, M. C. Systematic comparison of CRISPR/Cas9 and RNAi screens for essential genes. Nature Biotechnology. 34 (6), 634-636 (2016).
- Evers, B., et al. CRISPR knock-out screening outperforms shRNA and CRISPRi in identifying essential genes. Nature Biotechnology. 34 (6), 631-633 (2016).
- Miles, L. A., Garippa, R. J., Poirier, J. T. Design, execution, and analysis of pooled in vitro CRISPR/Cas9 screens. The FEBS Journal. 283 (17), 3170-3180 (2016).
- Wang, T., Wei, J. J., Sabatini, D. M., Lander, E. S. Genetic screens in human cells using the CRISPR-Cas9 system. Science. 343 (6166), 80-84 (2014).
- Wang, T., et al. Identification and characterization of essential genes in the human genome. Science. 350 (6264), 1096-1101 (2015).
- Sanson, K. R., et al. Optimized libraries for CRISPR-Cas9 genetic screens with multiple modalities. Nature Communications. 9 (1), 5416(2018).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knock-out Screens. G3: Genes|Genomes|Genetics. 7 (8), 2719-2727 (2017).
- Mair, B., Tomic, J., et al. Essential gene profiles for human pluripotent stem cells identify uncharacterized genes and substrate dependencies. Cell Reports. 27 (2), 599-615 (2019).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knock-out screening in human cells. Science. 343 (6166), 84-87 (2014).
- Sharma, S., Petsalaki, E. Application of CRISPR-Cas9 Based Genome-Wide Screening Approaches to Study Cellular Signalling Mechanisms. International Journal of Molecular Sciences. 19 (4), (2018).
- Steinhart, Z., et al. Genome-wide CRISPR screens reveal a Wnt-FZD5 signaling circuit as a druggable vulnerability of RNF43-mutant pancreatic tumors. Nature Medicine. 23 (1), 60-68 (2017).
- Wang, T., et al. Gene Essentiality Profiling Reveals Gene Networks and Synthetic Lethal Interactions with Oncogenic Ras. Cell. 168 (5), 890-903 (2017).
- Zimmermann, M., et al. CRISPR screens identify genomic ribonucleotides as a source of PARP-trapping lesions. Nature. 559 (7713), 285-289 (2018).
- Deans, R. M., et al. Parallel shRNA and CRISPR-Cas9 screens enable antiviral drug target identification. Nature Chemical Biology. 12 (5), 361-366 (2016).
- Trinh, T. J. J., Bloom, F., Hirsch, V. STBL2: an Escherichia coli strain for the stable propagation of retroviral clones and direct repeat sequences. Focus. 16, 78-80 (1994).
- Hart, T., Moffat, J. BAGEL: a computational framework for identifying essential genes from pooled library screens. BMC Bioinformatics. 17, 164(2016).
- Wang, G. Z. M., et al. Identifying drug-gene interactions from CRISPR knock-out screens with drugZ. bioRxiv. , Available from: https://doi.org/10.1101/232736 (2017).
- Pedregosa, F. V., G,, et al. Scikit-learn: Machine Learning in Python. Journal of Machine Learning Research. 12, 2825-2830 (2011).
- Ketela, T., et al. A comprehensive platform for highly multiplexed mammalian functional genetic screens. BMC Genomics. 12, 213(2011).
- Doench, J. G. Am I ready for CRISPR? A user's guide to genetic screens. Nature Review Genetics. 19 (2), 67-80 (2018).
- Hartenian, E., Doench, J. G. Genetic screens and functional genomics using CRISPR/Cas9 technology. FEBS Journal. 282 (8), 1383-1393 (2015).
- Li, W., et al. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knock-out screens. Genome Biology. 15 (12), 554(2014).
- Sheel, A., Xue, W. Genomic Amplifications Cause False Positives in CRISPR Screens. Cancer Discovery. 6 (8), 824-826 (2016).
- Meyers, R. M., et al. Computational correction of copy number effect improves specificity of CRISPR-Cas9 essentiality screens in cancer cells. Nature Genetics. 49 (12), 1779-1784 (2017).
- Henser-Brownhill, T., Monserrat, J., Scaffidi, P. Generation of an arrayed CRISPR-Cas9 library targeting epigenetic regulators: from high-content screens to in vivo assays. Epigenetics. 12 (12), 1065-1075 (2017).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved