Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Pantallas genéticas basadas en CRISPR en células de mamíferos
En este artículo
Resumen
La tecnología CRISPR-Cas9 proporciona un método eficiente para editar con precisión el genoma de mamíferos en cualquier tipo de célula y representa un medio novedoso para realizar pantallas genéticas en todo el genoma. Aquí se proporciona un protocolo detallado que analiza los pasos necesarios para el buen rendimiento de las pantallas CRISPR-Cas9 agrupadas para todo el genoma.
Resumen
La edición del genoma utilizando el sistema CRISPR-Cas ha avanzado enormemente la capacidad de editar con precisión los genomas de varios organismos. En el contexto de las células de los mamíferos, esta tecnología representa un medio novedoso para realizar pantallas genéticas en todo el genoma para estudios genómicos funcionales. Las bibliotecas de ARN guía (sgRNA) dirigidas a todos los marcos de lectura abiertos permiten la generación fácil de miles de perturbaciones genéticas en un solo grupo de células que se pueden analizar para fenotipos específicos para implicar la función génica y los procesos celulares en un manera imparcial y sistemática. Las pantallas CRISPR-Cas proporcionan a los investigadores un método simple, eficiente y económico para descubrir los planos genéticos de los fenotipos celulares. Además, el análisis diferencial de las pantallas realizadas en varias líneas celulares y de diferentes tipos de cáncer puede identificar genes que son esenciales desde el punto de vista contextual en las células tumorales, revelando objetivos potenciales para terapias específicas contra el cáncer. Realizar pantallas de todo el genoma en células humanas puede ser desalentador, ya que esto implica el manejo de decenas de millones de células y requiere el análisis de grandes conjuntos de datos. Los detalles de estas pantallas, como la caracterización de líneas celulares, las consideraciones de la biblioteca CRISPR y la comprensión de las limitaciones y capacidades de la tecnología CRISPR durante el análisis, a menudo se pasan por alto. Aquí se proporciona un protocolo detallado para el rendimiento exitoso de las pantallas basadas en CRISPR-Cas9 en todo el genoma agrupado.
Introducción
CRISPR-Cas, abreviatura de repeticiones palindrómicas cortas interespaciadas y nucleasas asociadas a CRISPR, consiste en una única proteína de nucleasa (por ejemplo, Cas9) en complejo con un ARN guía sintético (sgRNA). Este complejo de ribonucleoproteínas se dirige a la enzima Cas9 para inducir roturas de ADN de doble cadena en un locus genómico específico1. Los cortes de doble cadena se pueden reparar mediante reparación dirigida por homología (HDR) o, más comúnmente, a través de unión final no homóloga (NHEJ), un mecanismo de reparación propenso a errores que da como resultado la inserción y/o deleciones (INDELS) que con frecuencia interrumpen la función génica 1. La eficiencia y simplicidad de CRISPR permite un nivel de segmentación genómica hasta ahora inalcanzable que supera con creces las tecnologías de edición del genoma anteriores [es decir, nucleasas de dedos de zinc (ZNF) o nucleasas de efectos similares a los activadores de transcripción ( TALENS), ambos sufren de mayor complejidad de diseño, menor eficiencia de transfección y limitaciones en la edición de genes multiplex2].
La aplicación básica de investigación de la edición del genoma basado en ARN de una sola guía de CRISPR ha permitido a los científicos interrogar de manera eficiente y económica las funciones de los genes individuales y la topología de las redes de interacción genética. La capacidad de realizar pantallas funcionales de todo el genoma se ha visto muy mejorada por el uso del sistema CRISPR-Cas, particularmente en comparación con tecnologías de perturbación genética anteriores, como la interferencia del ARN (ARN) y la mutagénesis de las trampas genéticas. En particular, el ARNi sufre de altos efectos fuera del objetivo y derribos incompletos, lo que resulta en una menor sensibilidad y especificidad en comparación con CRISPR3,4,5, mientras que los métodos de captura genética sólo son factibles en haploides células para pantallas de pérdida de funciones, limitando el alcance de los modelos de celda que se pueden interrogar6. La capacidad de CRISPR para generar un knock-out genético completo proporciona un sistema biológicamente más robusto para interrogar fenotipos mutantes, con bajo ruido, efectos mínimos fuera del objetivo y actividad constante a través de los reactivos5. Las bibliotecas de sgRNA CRISPR-Cas9 que se dirigen a todo el genoma humano están ahora ampliamente disponibles, permitiendo la generación simultánea de miles de noqueos genéticos en un solo experimento3,7,8,9 .
Hemos desarrollado bibliotecas lentivirales de sgRNA únicas para todo el genoma CRISPR-Cas9 llamadas las bibliotecas Toronto Knock-out (TKO) (disponibles a través de Addgene) que son compactas y están optimizadas para secuencias para facilitar pantallas genómicas funcionales de alta resolución. La última biblioteca, TKOv3, se dirige a 18.000 genes de codificación de proteínas humanas con 71.090 guías optimizadas para la eficiencia de edición utilizando datos empíricos10. Además, TKOv3 está disponible como una biblioteca de un componente (LCV2::TKOv3, Addgene ID #90294) que expresa Cas9 y sgRNAs en un solo vector, aliviando la necesidad de generar células estables de expresión de Cas9, lo que permite el desconexión del genoma en una amplia gama de tipos de células de mamíferos. TKOv3 también está disponible en un vector sin Cas9 (pLCKO2::TKOv3, Addgene ID 125517) y se puede utilizar en celdas que expresan Cas911.
Una población celular editada por CRISPR-Cas9 en todo el genoma puede estar expuesta a diferentes condiciones de crecimiento, con la abundancia de sgRNA a lo largo del tiempo cuantificada por la secuenciación de próxima generación, proporcionando una lectura para evaluar la deserción o el enriquecimiento de células con Perturbaciones. Las bibliotecas CRISPR knock-out se pueden aprovechar para identificar genes que, tras la perturbación, causan defectos de aptitud celular, sensibilidad moderada a los medicamentos (por ejemplo, genes sensibles o resistentes), regulan la expresión de proteínas (por ejemplo, reportero), o son necesarios para un determinado función de la vía y estado celular12,13,14. Por ejemplo, las pantallas de aptitud diferencial en una línea celular cancerosa pueden identificar tanto el agotamiento o la reducción de oncogenes como el enriquecimiento o un aumento de los genes supresores tumorales3,14,15. Del mismo modo, el uso de dosis intermedias de fármacos terapéuticos puede revelar tanto la resistencia a los fármacos como los genes de sensibilización16,17.
Aquí se proporciona un protocolo de cribado detallado para la detección de la pérdida de función CRISPR-Cas9 a escala del genoma utilizando las bibliotecas Knock-out de Toronto (TKOv1 o v3) en células de mamíferos desde la generación de bibliotecas, el rendimiento de cribado hasta el análisis de datos. Aunque este protocolo se ha optimizado para su detección con las bibliotecas Knock-out de Toronto, se puede aplicar y convertirse en escalable para todas las bibliotecas agrupadas de SgRNA de CRISPR.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Los experimentos descritos a continuación deben seguir las directrices de la Oficina de Seguridad y Salud Ambiental del instituto.
1. Amplificación de plásmido de plásmido de biblioteca lentiviral CRISPR sgRNA agrupada
- Diluir la biblioteca de ADN de plásmido CRISPR sgRNA ya hecha a 50 ng/L en TE (por ejemplo, TKOv3).
- Electroportar la biblioteca utilizando células electrocompetentes. Configure un total de cuatro reacciones de electroporación como se describe a continuación.
- Añadir 2 l de biblioteca de tko de 50 ng/L a 25 l de células electrocompetentes desaligadas a las cubetas preenfriadas (1,0 mm) en hielo.
- Electroporato utilizando ajustes óptimos sugeridos por el protocolo del fabricante. Dentro de 10 s del pulso, añadir 975 s de medio de recuperación (o medio SOC) a la cubeta.
- Transfiera las células electroporadas a un tubo de cultivo y agregue 1 ml de medio de recuperación. Incubar tubos en una incubadora de agitación a 250 rpm durante 1 h a 37oC.
- Configure una placa de dilución para iterar la biblioteca y estimar la eficiencia de la transformación.
- Piscina de los 8 ml de células recuperadas y mezcle bien. Transfiera 10 l de las células agrupadas a 990 s de medio de recuperación para una dilución de 800 veces y mezcle bien.
- Placa de 20 ml de la dilución en una placa de agar precalentada de 10 cm LB + carbenicilina (100 g/L). Esto da como resultado una dilución de 40.000 veces de los transformadores que se utilizarán para calcular la eficiencia de transformación.
- Placa 400 l de células recuperadas en cada placa a través de un total de 20 placas de agar precalentadas de 15 cm LB + carenicilina. Incubar las placas durante 14-16 h a 30oC.
NOTA: El crecimiento a esta temperatura más baja minimiza la recombinación entre repeticiones de terminales largos (LTR)18. - Para calcular la eficiencia de transformación, cuente el número de colonias en la placa de dilución de 40.000 veces (paso 1.3.2). Multiplicar el número de colonias contadas por 40.000 para obtener el número total de colonias en todas las placas. Proceda si el número total de colonias representa una cobertura de biblioteca equivalente a un mínimo de 200x colonias por sgRNA (la más óptima es 500-1000x).
- Por ejemplo, el número mínimo de colonia para la biblioteca TKOv3 (71.090 sgRNA) es 1,4 x 107,lo que equivale a 200 colonias por sgRNA. Si la representación de colonias es insuficiente, aumente el número de electroporaciones en el paso 1.2 en función del número de colonias en la placa de dilución para lograr la cobertura mínima de la biblioteca.
- Cosecha las colonias como se describe a continuación
- A cada placa de 15 cm, agregue 7 ml de lb + carbenicilina (100 g/l) medio, luego raspe las colonias con un esparcidor celular. Con una pipeta de 10 ml, transfiera las células raspadas a un matraz o botella cónico estéril de 1 L.
- Enjuagar una vez más la placa con 5 ml de medio de carbenicilina LB + y transferir la solución al frasco.
- Repita el procedimiento para que todas las placas a las células de la piscina de 20 placas en una botella estéril.
- Mezclar las células recogidas con una barra de agitación durante 1 h a temperatura ambiente (RT) para romper los grumos celulares. Transfiera las células a botellas de centrífuga prepesadas y centrífuga a 7.000 x g a bacterias pellets y, a continuación, deseche los medios.
- Pesar el pellet de celda húmeda y restar el peso de la botella de centrífuga para determinar el peso final del pellet húmedo. Purifica el ADN plásmido usando un kit de purificación de plásmido a escala maxi o mega escala dependiendo de la cantidad de pellet bacteriano que cada columna pueda procesar.
2. Producción de lentivirus de la biblioteca CRISPR sgRNA a gran escala
NOTA: Todos los pasos de esta sección del protocolo se realizan en una instalación BSL2+ en un gabinete de bioseguridad Clase II, Tipo A2.
- Calcular el número de placas de 15 cm necesarias para la producción de virus en función de la estimación de que 18 ml de virus se cosechan típicamente a partir de una placa de 15 cm.
- Preparar las células para la transfección mediante la sembración de células de envasado HEK-293T en medios de crecimiento con bajo contenido de antibióticos (DMEM + 10% FBS + opcional: 0,1x pluma/estreptococo) a 8 x 106 células por placa de 15 cm en 20 ml de medios. Incubar células durante la noche a 37oC, 5%CO2. Asegúrese de que las células chapadas sean de 70%–80% de confluente y se propaguen uniformemente en el momento de la transfección.
- Al día siguiente, preparar tres mezclas de plásmidos de transfección como se describe en la Tabla 1 para placas de 15 cm. Calcular la cantidad de plásmido necesario para una transfección y hacer una mezcla de plásmidos para el número de placas, más uno para ser transinfectados.
- Preparar un reactivo de transfección a base de lípidos para cada transfección como se describe en la Tabla 2. Aliquot redujo los medios séricos en tubos de microcentrífuga individuales de 1,5 ml para el número de placas que se van a transpirar. Agregue el reactivo de transfección, mezcle suavemente e incubar durante 5 min a RT.
- Después de 5 min de incubación, agregue la cantidad de ADN necesaria para un transfección al reactivo de transfección para una proporción de 3:1 de reactivo de transfección a g de complejo de ADN. Mezclar suavemente e incubar durante 30 min a RT.
NOTA: Las transfecciones posteriores se pueden preparar en conjuntos de cinco o menos, con intervalos de 5 minutos para optimizar el tiempo y evitar la sobreincubación. - Después de 30 minutos de incubación, transfiera cuidadosamente cada mezcla de transfección a cada placa de células de embalaje. Agregue toda la mezcla usando una punta de pipeta de 1 ml con gota en un movimiento circular en zigzag sin molestar a la monocapa de la celda. Incubar células a 37oC durante 18 h a 5% co2.
- Preparar medios de cosecha viral: 500 mL de medio DMEM + 32 ml de stock de BSA (20 g/100 ml, disuelto en DMEM, filtro esterilizado con filtro de 0,22 m) + 5 ml de pluma/estreptococo de 100x.
- Después de 18 h, retire los medios (utilice el manejo adecuado de los residuos de lentivirus, como la incubación en hipoclorito de sodio al 1% durante 30 minutos antes de su eliminación). Reemplace suavemente con 18 ml de medios de cosecha virales en cada placa. Incubar células a 37oC durante 18 h a 5% co2.
- Después de las 24 horas, compruebe si las células de envasado son morfológicas anormales y fusionadas como una indicación de una buena producción de virus. Luego, cosecha el lentivirus recogiendo todo el sobrenadante y transfiriéndolo a un tubo de centrífuga cónica estéril.
- Gire el medio que contiene el virus a 300 x g durante 5 min y pelete las células de embalaje. Alíspe el sobrenadante en un tubo de polipropileno estéril sin molestar el pellet.
- Almacene el virus a 4 oC durante períodos cortos (menos de 1 semana) o inmediatamente a -80 oC para el almacenamiento a largo plazo. El virus a gran escala de Aliquot se prepara para volúmenes de un solo uso para el almacenamiento a largo plazo para evitar la congelación/descongelación.
3. Caracterización de la línea celular para el cribado
- Seleccione la línea de celda deseada.
- Mida y registre el tiempo aproximado de duplicación de las celdas.
- Determinar la densidad óptima de chapado celular para el cultivo de células cada 3-4 duplicaciones celulares en un recipiente de cultivo de tejido de elección (por ejemplo, placas de cultivo de tejido de 15 cm).
- Determine la concentración de puromicina que se utilizará en la línea celular deseada para la selección de bibliotecas de tko que contengan marcador de resistencia a la puromicina de la siguiente manera:
- Células de semilla en una placa de 12 pocillos a la densidad necesaria para alcanzar la confluencia después de 72 h, luego incubar durante la noche (37 oC, 5% CO2).
- Al día siguiente, cambie a un medio que contenga un rango de dilución de concentraciones de puromicina de 0 g/ml a 10 g/ml, en incrementos de 0,5 g/ml. Incubar las células durante 48 h.
- Después de 48 h, mida la viabilidad celular contando celularmente o manchando alamarBlue.
- Determinar la concentración más baja que mata el 100% de las células en 48 h. Utilice esta concentración para seleccionar las poblaciones de células transducidas de la biblioteca CRISPR en los pasos 4.6 y 5.2.6.
NOTA: Para líneas celulares con tiempos de duplicación más largos, se pueden tolerar incubaciones más largas con puromicina. En estas situaciones, determine la curva de muerte para el tiempo de incubación necesario para las duplicaciones de celdas <3. Minimice el tiempo de selección para evitar la deserción de genes esenciales antes del inicio del cribado.
- Compruebe la sensibilidad de las células al bromuro de hexadictrina (hasta 8 g/ml) realizando una curva de respuesta a la dosis en el mismo método que se utiliza para medir la sensibilidad a la puromicina (paso 3.2). Si se observa toxicidad con <8 g/ml de bromuro de hexadimetrina, no lo use.
4. Valoración funcional de la biblioteca de lentivirus CRISPR agrupada para la determinación del MOI
- Descongelar una nueva alícuota de lentivirus de la biblioteca CRISPR sgRNA agrupado (por ejemplo, LCV2::TKOv3) y mantener en hielo.
- Diseñar una serie de volúmenes de virus para probar entre el rango de 0-2 mL (es decir, 0 mL, 0,25 ml, 0,5 ml, 1 ml y 2 ml).
- Cosecha células diana y células de semilla en placas de 15 cm a la densidad necesaria para alcanzar la confluencia en 72 h.
- Para que se pruebe cada volumen de virus, prepare placas duplicadas. Agregue las células, el virus, el bromuro de hexadimetrina (8 g/ml) y los medios a un volumen final de 20 ml. Mezclar bien las placas, las placas de la coltura se nivelan en la incubadora y se incuban durante 24 h (37 oC, 5% CO2).
- Después de las 24 h, eliminar el virus que contiene los medios y desechar (utilizar las precauciones de bioseguridad para la manipulación de residuos de lentivirus). Opcionalmente, lave suavemente la placa con PBS caliente para eliminar el virus extraño.
- Para cada condición del virus, reemplace con 20 ml de medios que contengan puromicina utilizando la concentración determinada a matar células en la sección 3, a una placa de réplica. Al otro plato, añadir 20 ml de medios frescos sin puromicina. Incubar durante 48 h (37oC, 5% CO2).
- Después de 48 h, compruebe que todas las células no infectadas (condición del virus 0 ml) tratadas con puromicina estén muertas. Cosecha todas las placas individualmente y dispersa las células mediante pipeteo suave repetido.
- Cuente las células de todas las placas y calcule el MOI para cada volumen de virus comparando el recuento celular con la selección de puromicina con el recuento celular sin puromicina (es decir, +/- puromicina).
- Gráfico de resultados para determinar el volumen del virus que conduce a 30%-40% de supervivencia celular con selección de puromicina en comparación con sin puromicina. Utilice este volumen de virus para lograr un MOI de 0.3–0.4 durante la pantalla bajo las mismas condiciones de cultivo de tejido.
5. Infección primaria de la pantalla, selección y passaging celular
- Seleccione la cobertura de la biblioteca CRISPR sgRNA que se mantendrá en toda la pantalla (mínimo recomendado de 200 veces).
- Sobre la base de la cobertura de la biblioteca, determine el número de células necesarias para mantener esta cobertura por sgRNA y el número de células necesarias para la infección en MOI 0.3(Tabla 3).
- Determine el número de placas necesarias para configurar la infección (Tabla 4).
- Infectar las celdas con la biblioteca CRISPR
- Cosecha las células y la semilla del número de celda requerido para cada placa de 15 cm.
- Añadir bromuro de hexadimetrina (8 g/ml) a todas las placas.
- Agregue el virus al volumen requerido para MOI 0.3 al cribado y las placas Control 2. Para el Control 1, no agregue virus y reemplace ese volumen con medios.
- Mezcle bien las placas inclinando. Coloque las placas en la incubadora, asegurándose de que estén niveladas.
NOTA: Las infecciones por lotes se pueden hacer combinando una mezcla maestra de virus, medios de comunicación y bromuro de hexadimetrina a las células en suspensión antes del enchapado. - Retire los medios y sustitúyalos por medios frescos que contengan puromicina en la concentración determinada en el paso 3.2.4 hasta el cribado y controle 1 placas 24 h después de la infección por virus. Añadir medios frescos sin puromicina a la placa de control 2. Incubar células durante 48 h (37oC, 5% CO2).
- 48 h después de la adición de puromicina, asegúrese de que todas las células no infectadas están muertas (control 1) para confirmar la actividad de la puromicina, luego cosecha las células infectadas.
- Cosecha de población celular infectada y paso celular
- Cosecha las células seleccionadas con puromicina de todas las placas de cribado en un recipiente estéril. Recoja las celdas de cada placa de control por separado. Disperse las células mediante pipeteo suave y repetido.
- Cuente las células de las células de detección agrupadas, controle 1, y controle 2 por separado y calcule el número de celdas por 1 mL.
- Calcule el MOI y la cobertura de plegado logradas de la siguiente manera:
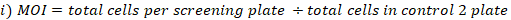
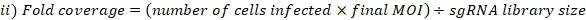
- Recoger tres réplicas de pellets celulares de las células agrupadas en la cobertura de la biblioteca seleccionada para la extracción de ADN genómico. Centrifugar las células a 500 x g durante 5 min. Etiquetar los tubos y congelar los gránulos celulares a -80 oC (se trata de muestras de referencia T0).
- Divida el grupo de celdas infectadas en tres grupos de réplica (por ejemplo, replicar A, replicar B, replicar C), mientras mantiene la cobertura de la biblioteca dentro de cada réplica. Células de semilla sin la misma densidad de semilla que normalmente se utilizaría al expandirlas. Utilice el mismo número de celdas para cada placa de réplica y el mismo número total de celdas entre réplicas.
- Continúe pasando células y cosechar tres réplicas de pellets celulares de cada réplica de células infectadas en conjunto como arriba, cada 3-8 días dependiendo de la línea celular, para hasta 15-20 duplicaciones celulares. En cada paso, cosechar las células de todas las placas de cada grupo de réplica entre sí (es decir, todas las células de las placas de réplica A se remezclan, todas las células de las placas de réplica B se remezclan entre sí, etc.).
- Etiquete cada pellet con un tiempo (T) y replique la designación. Esto corresponde al número de días después de T0 se recoge el pellet (por ejemplo, T3_A, T3_B, T3_C, etc.).
- Para las pantallas de drogas de selección negativa, permita que las células se recuperen durante al menos un pasaje después de T0 antes del tratamiento. En T3 o T6, divida las células de cada grupo de réplica (A, B, C) en poblaciones de tratamiento y control de medicamentos, utilizando la misma densidad de semilla utilizada en el paso 5.3.5.
- Agrupado por separado el número de celdas necesarias para la cobertura de la biblioteca para cada réplica en el grupo de tratamiento de medicamentos. Añadir el fármaco a concentraciones intermedias (IC20-IC50). Sembrar las células e incubar (37oC, 5% CO2)hasta el siguiente pasaje.
- Agrupa por separado el número de celdas necesarias para la cobertura de la biblioteca para cada réplica en el grupo de control del vehículo. Añadir el control del vehículo utilizando el mismo volumen que la droga (<0.5% v/v). Sembrar las células e incubar (37oC, 5% CO2)hasta el siguiente pasaje.
- Continúe enpaso las células y cosechar los pellets celulares para ADN genómico cada 3 días como se describe en el paso 5.3.5, mientras se refresca la droga o el vehículo en cada pasaje.
- Para las pantallas de selección positiva o resistencia a fármacos, divida cada grupo de réplica según el número de celdas necesarias para la cobertura de la biblioteca. Agregue concentraciones de fármacos IC90 a cada réplica. En IC90,la mayoría de las células serán asesinadas. Permitir que las poblaciones resistentes crezcan y recojan pellets celulares (1-2 x 107 células) para la extracción genómica de ADN.
6. Preparación y secuenciación de muestras CRISPR
- Purificación de ADN genómico
- Incubar los gránulos de células congeladas durante 5-10 min a RT para descongelar.
- Añadir 1,4 ml de PBS a un tubo centrífugo de 50 ml que contenga un pellet celular. Vórtice durante 20 s para resuspender las células y descansar durante 1 min. Si es necesario, pipetee 15x con P1000 para romper los grumos de células restantes. Si transfiere células de un tubo de 15 ml o 1,5 ml, resuspenda las celdas con 1 ml de PBS, luego transfiera las células a un tubo de 50 ml y enjuague el tubo original con 400 ml de PBS.
- Añadir 5 ml de solución de nuclei lisis a las células resuspendidas. Con una pipeta de 10 ml, mezcle la muestra pipeteando hacia arriba y hacia abajo 5 veces.
- Añadir 32 l de RNase A (20 mg/ml; para obtener una concentración final de 100 g/ml) al lisado nuclear y mezclar la muestra invirtiendo el tubo 5x. Incubar la mezcla a 37oC durante 15 min y dejar que la muestra se enfríe durante 10 min a RT.
- Añadir 1,67 ml de solución de precipitación de proteínas al lisado y al vórtice vigorosamente durante 20 s. Los pequeños grumos de proteínas pueden ser visibles después de la mezcla.
- Centrífuga a 4.500 x g durante 10 min a RT.
- Con una pipeta de 10 ml, transfiera el sobrenadante a un tubo centrífugo de 50 ml que contenga 5 ml de isopropanol. Mezcle suavemente la solución 10 veces por inversión hasta que se observe el ADN.
NOTA: El ADN se puede observar como hebras blancas, similares a hilos que forman una masa visible. - Centrífuga a 4.500 x g durante 5 min a RT para peletizar el ADN.
- Con una pipeta de 10 ml, retire cuidadosamente el sobrenadante y evite desalojar el pellet de ADN. Añadir 5 ml de 70% de etanol a RT al ADN. Gire suavemente el tubo para lavar el pellet de ADN y los lados del tubo centrífugo.
- Centrífuga a 4.500 x g durante 5 min a RT.
- Con una pipeta de 10 ml, retire cuidadosamente el 70% de etanol y evite desalojar el pellet de ADN. ADN genómico secado al aire durante 10 min a RT.
- Añadir 400 sl de solución TE al tubo y dejar que el ADN se disuelva incubando a 65 oC durante 1 h. Mezclar el ADN moviendo suavemente el tubo cada 15 minutos. Si el ADN no se disuelve por completo, incubar el tubo a 65 oC durante 1 h adicional mientras mueve suavemente el tubo cada 15 minutos, y déjelo a 4 oC durante la noche.
- Centrifugar a 4.500 x g durante 1 min a RT y transferir ADN genómico a un tubo de unión baja de 1,5 ml.
- Cuantificar y medir la pureza del ADN genómico tanto en el espectrofotómetro (para el contenido total de ácido nucleico) como en el fluorómetro (para contenido de ADN de doble cadena).
- Opcionalmente, precipita el ADN genómico si hay problemas con la amplificación de PCR aguas abajo del sgRNA de la siguiente manera.
- Transfiera el ADN genómico de 400 l a un tubo de microcentrífuga de 1,5 ml.
- Añadir 18 ml de 5 M de NaCl (concentración final de 0,2 M) y 900 ml de etanol al 95%.
- Invierta el tubo 10x hasta que se mezcle completamente, luego centrifugar a 16.000 x g durante 10 min a RT.
- Retire con cuidado el sobrenadante y evite desalojar el pellet de ADN. Lave el pellet de ADN con 500 ml de etanol al 70%. Gire suavemente el tubo para lavar el pellet de ADN.
- Centrífuga a 16.000 x g durante 5 min a RT.
- Retire con cuidado el sobrenadante y evite desalojar el pellet de ADN. ADN genómico secado al aire durante 10 min a RT.
- Añadir 300 s de TE para disolver el ADN como se describe en los pasos 6.1.12.
- Cuantifique y mida la pureza del ADN genómico como se describe en el paso 6.1.14.
- Preparación de la biblioteca de secuenciación CRISPR
- Configurar la PCR 1 como se describe en la Tabla 5 utilizando un total de 100 g de ADN genómico. Añadir 3,5 g de ADN genómico por reacción de 50 s y configurar reacciones idénticas de 50 l para lograr la cobertura deseada. En el Cuadro 6 se enumeran ejemplos de secuencias de imprimación para la amplificación de bibliotecas de secuenciación LCV2::TKOv3. En la Tabla 7 se enumeran ejemplos de secuencias de imprimación para la amplificación de bibliotecas de secuenciación pLCKO2::TKOv3.
- Amplificar las reacciones de PCR 1 en un termociclador utilizando el programa descrito en la Tabla 8.
- Compruebe la amplificación de PCR 1 ejecutando 2 ml del producto PCR en un gel de agarosa del 1%. PCR 1 produce un producto de 600 bp.
- Agrupa todas las reacciones individuales de 50 l para cada muestra de ADN genómico y mezcla por vórtice.
- Configure una reacción PCR 2 (50 l) para cada muestra, tal como se describe en la Tabla 9, utilizando 5 l del producto PCR 1 agrupado como plantilla. Utilice combinaciones de imprimación de índice únicas para cada muestra individual para permitir la agrupación de muestras de biblioteca de secuenciación.
- Amplificar la reacción PCR 2 en un termociclador utilizando el programa descrito en la Tabla 10.
- Limpie el equipo de gel de agarosa para purificar los productos amplificados con 0,1 N HCl durante 10 minutos antes de lanzar un gel. Preparar un gel de agarosa del 2% que contenga manchas de ADN para purificar productos amplificados PCR 2.
- Ejecute el producto PCR 2 en el gel de agarosa del 2% a baja tensión (1,0–1,5 h de funcionamiento). PCR 2 produce un producto de 200 bp.
- Visualice los productos PCR en un transiluminador de luz azul. Exbobe la banda de 200 bp y purifica el ADN de la rodaja de gel de agarosa usando un kit de extracción de gel. Cuantifique y mida la pureza de la biblioteca de secuenciación tanto en el espectrofotómetro como en el fluorómetro.
NOTA: Una concentración típica de la biblioteca de secuenciación purificada por gel oscila entre 5-10 ng/L y un rendimiento total de 150–300 ng.
- Secuenciación de alto rendimiento
- Secuenciar las bibliotecas de secuenciación CRISPR en secuenciadores de próxima generación.
- Muestras de referencia de secuencia T0 a mayor profundidad de lectura de 400 a 500 veces la cobertura de la biblioteca. Secuenciar muestras experimentales de puntos de tiempo para pantallas desplegables a una profundidad de lectura mínima de 200 veces. Para las pantallas de selección positivas fuertes, un mínimo de profundidad de lectura de 50 veces la cobertura es suficiente para la identificación de los sgRNA enriquecidos.
NOTA: Es fundamental secuenciar la muestra T0 para determinar la representación de la biblioteca para una pantalla determinada y servir como referencia para los cambios de pliegue sgRNA determinante a lo largo del tiempo.
7. Análisis de datos
- Alinee la secuencia utilizando programas como Bowtie para asignar lecturas de secuencia a la biblioteca de referencia utilizando los siguientes parámetros: -v2 (permitiendo dos discrepancias) y -m1 (descartando cualquier lectura que se asigne a más de una secuencia de la biblioteca).
- Normalice el número de lecturas asignadas de forma única para cada sgRNA para una muestra determinada a 10 millones de lecturas por muestra de la siguiente manera:
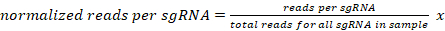 107
107
- Calcule el cambio de plegado log2 de cada sgRNA para cada réplica en cada punto de tiempo (Tn) en comparación con la muestra T0 (Tn/T0). Agregue un pseudorecuento de 0,5 lecturas a todos los recuentos de lectura para evitar discontinuidades de ceros. Excluya los sgRNAs con <30 lecturas sin procesar en la muestra T0 del cálculo de cambio silnlegado y el análisis descendente.
- Analizar los cambios de pliegue con el algoritmo de Análisis Bayesiano de La Esencialidad Genética (BAGEL) , utilizando los conjuntos de entrenamiento esenciales y no esenciales definidos anteriormente19 para las pantallas de esencialidad genética ( Tabla Suplementaria S1) o DrugZ para pantallas de drogas20.
- Calcule la precisión y la recuperación para la evaluación del rendimiento de la pantalla utilizando puntuaciones BF. Utilice el conjunto esencial del paso 7.4 como lista positiva verdadera para la función precision_recall_curve de la biblioteca Scikit-learn para Python, junto con el subconjunto de puntuación BF anterior. Alternativamente, realice lo mismo utilizando el paquete PRROC en R.
- Calcule el cambio de pliegue medio de todas las guías para cada gen. Generar gráficas de densidad para los genes esenciales y no esenciales (ver paso 7.4) en R o software equivalente. En R, si x.ess es un vector que contiene los valores de cambio de plegado de tronco de genes esenciales y x.nonEss contienen genes no esenciales, trace utilizando el siguiente comando:
plot( density( x.ess ), xlab-"mean logFC",col-"red",lwd-2 )
lines( density( x.nonEss ), col-"blue",lwd-2 )
NOTA: Para los detalles de la versión de Python y los paquetes utilizados, consulte scikit-learn v0.19.1: (publicado por Pedregosa et al.21).
Access restricted. Please log in or start a trial to view this content.
Resultados
Descripción general del flujo de trabajo de cribado CRISPR a escala del genoma
La Figura 1 ilustra una visión general del flujo de trabajo de cribado CRISPR agrupado, comenzando con la infección de las células diana con lentivirus de la biblioteca CRISPR a un MOI bajo para garantizar eventos de integración única y una representación adecuada de la biblioteca (normalmente de 2...
Access restricted. Please log in or start a trial to view this content.
Discusión
Debido a su simplicidad de uso y alta flexibilidad, la tecnología CRISPR ha sido ampliamente adoptada como la herramienta de elección para la edición precisa del genoma. El cribado CRISPR agrupado proporciona un método para interrogar miles de perturbaciones genéticas en un solo experimento. En las pantallas agrupadas, las bibliotecas sgRNA sirven como códigos de barras moleculares, ya que cada secuencia es única y se asigna al gen objetivo. Al aislar el ADN genómico de la población celular, los genes que causan...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no declaran intereses financieros en competencia.
Agradecimientos
Este trabajo fue apoyado por el Genoma Canadá, el Fondo de Investigación de Ontario y los Institutos Canadienses para la Investigación Sanitaria (MOP-142375, PJT-148802).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 micron filter | |||
| 30°C plate incubator | |||
| 37°C shaking incubator | |||
| 37°C, 5% CO2 incubator | |||
| 5 M NaCl | Promega | V4221 | |
| 50X TAE buffer | BioShop | TAE222.4 | |
| 6 N Hydrochloric acid solution | BioShop | HCL666.500 | |
| 95% Ethanol | |||
| Alamar blue | ThermoFisher Scientific | DAL1025 | |
| Blue-light transilluminator | ThermoFisher Scientific | G6600 | |
| Bovine Serum Albumin,Heat Shock Isolation, Fraction V. Min. 98%, Biotechnology grade | Bioshop | ALB001.250 | |
| Dulbecco's Modification of Eagles Medium | Life Technologies | 11995-065 | Cel culture media |
| Electroporation cuvettes | BTX | 45-0134 | |
| Electroporator | BTX | 45-0651 | |
| Endura electrocompetent cells | Lucigen | 90293 | |
| Fetal Bovine Serum | GIBCO | 12483-020 | |
| HEK293T packaging cells | ATCC | CRL-3216 | recommend passage number <15 |
| Hexadimethrine Bromide (Polybrene) | Sigma | H9268 | Cationic polymer to enhance transduction efficiency |
| Hexadimethrine Bromide (Polybrene) | |||
| LB agar plates with carbenicillin | |||
| LB medium with carbenicillin | |||
| Low molecular weight DNA ladder | New England Biolabs | N3233S | |
| Nanodrop spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| NEBNext Ultra II Q5 Master Mix | New England Biolabs | M0544L | |
| Opti-MEM | Life Technologies | 31985-070 | Reduced serum media |
| Plasmid maxi purification kit | Qiagen | 12963 | |
| pMD2.G (envelope plasmid) | Addgene | Plasmid #12259 | lentiviral system |
| psPAX2 (packaging plasmid) | Addgene | Plasmid #12260 | lentiviral system |
| Puromycin | Wisent | 400-160-UG | |
| QIAquick gel extraction kit | Qiagen | 28704 | |
| Qubit dsDNA BR assay | ThermoFisher Scientific | Q32853 | |
| Qubit fluorometer | ThermoFisher Scientific | Q33226 | |
| RNAse A | Invitrogen | 12091021 | |
| S.O.C recovery medium | Invitrogen | 15544034 | |
| SYRB Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| Toronto KnockOut CRIPSR library (TKOv3) - Cas9 included | Addgene | Addgene ID #90203 | Genome-wide CRISPR library , includes Cas9, 71,090 sgRNA |
| Toronto KnockOut CRIPSR library (TKOv3) - non-cas9 | Addgene | Addgene ID #125517 | Genome-wide CRISPR library, non-Cas9, 71,090 sgRNA |
| Tris-EDTA (TE) solution, pH8.0 | |||
| UltraPure agarose | ThermoFisher Scientific | 16500500 | |
| Wizard genomic DNA purification kit | Promega | A1120 | |
| X-tremeGENE 9 DNA transfection reagent | Roche | 06 365 809 001 | Lipid based transfection reagent |
Referencias
- Jiang, F., Doudna, J. A. CRISPR-Cas9 Structures and Mechanisms. Annual Review of Biophysics. 46, 505-529 (2017).
- Baliou, S., et al. CRISPR therapeutic tools for complex genetic disorders and cancer (Review). International Journal of Oncology. 53 (2), 443-468 (2018).
- Hart, T., et al. High-Resolution CRISPR Screens Reveal Fitness Genes and Genotype-Specific Cancer Liabilities. Cell. 163 (6), 1515-1526 (2015).
- Morgens, D. W., Deans, R. M., Li, A., Bassik, M. C. Systematic comparison of CRISPR/Cas9 and RNAi screens for essential genes. Nature Biotechnology. 34 (6), 634-636 (2016).
- Evers, B., et al. CRISPR knock-out screening outperforms shRNA and CRISPRi in identifying essential genes. Nature Biotechnology. 34 (6), 631-633 (2016).
- Miles, L. A., Garippa, R. J., Poirier, J. T. Design, execution, and analysis of pooled in vitro CRISPR/Cas9 screens. The FEBS Journal. 283 (17), 3170-3180 (2016).
- Wang, T., Wei, J. J., Sabatini, D. M., Lander, E. S. Genetic screens in human cells using the CRISPR-Cas9 system. Science. 343 (6166), 80-84 (2014).
- Wang, T., et al. Identification and characterization of essential genes in the human genome. Science. 350 (6264), 1096-1101 (2015).
- Sanson, K. R., et al. Optimized libraries for CRISPR-Cas9 genetic screens with multiple modalities. Nature Communications. 9 (1), 5416(2018).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knock-out Screens. G3: Genes|Genomes|Genetics. 7 (8), 2719-2727 (2017).
- Mair, B., Tomic, J., et al. Essential gene profiles for human pluripotent stem cells identify uncharacterized genes and substrate dependencies. Cell Reports. 27 (2), 599-615 (2019).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knock-out screening in human cells. Science. 343 (6166), 84-87 (2014).
- Sharma, S., Petsalaki, E. Application of CRISPR-Cas9 Based Genome-Wide Screening Approaches to Study Cellular Signalling Mechanisms. International Journal of Molecular Sciences. 19 (4), (2018).
- Steinhart, Z., et al. Genome-wide CRISPR screens reveal a Wnt-FZD5 signaling circuit as a druggable vulnerability of RNF43-mutant pancreatic tumors. Nature Medicine. 23 (1), 60-68 (2017).
- Wang, T., et al. Gene Essentiality Profiling Reveals Gene Networks and Synthetic Lethal Interactions with Oncogenic Ras. Cell. 168 (5), 890-903 (2017).
- Zimmermann, M., et al. CRISPR screens identify genomic ribonucleotides as a source of PARP-trapping lesions. Nature. 559 (7713), 285-289 (2018).
- Deans, R. M., et al. Parallel shRNA and CRISPR-Cas9 screens enable antiviral drug target identification. Nature Chemical Biology. 12 (5), 361-366 (2016).
- Trinh, T. J. J., Bloom, F., Hirsch, V. STBL2: an Escherichia coli strain for the stable propagation of retroviral clones and direct repeat sequences. Focus. 16, 78-80 (1994).
- Hart, T., Moffat, J. BAGEL: a computational framework for identifying essential genes from pooled library screens. BMC Bioinformatics. 17, 164(2016).
- Wang, G. Z. M., et al. Identifying drug-gene interactions from CRISPR knock-out screens with drugZ. bioRxiv. , Available from: https://doi.org/10.1101/232736 (2017).
- Pedregosa, F. V., G,, et al. Scikit-learn: Machine Learning in Python. Journal of Machine Learning Research. 12, 2825-2830 (2011).
- Ketela, T., et al. A comprehensive platform for highly multiplexed mammalian functional genetic screens. BMC Genomics. 12, 213(2011).
- Doench, J. G. Am I ready for CRISPR? A user's guide to genetic screens. Nature Review Genetics. 19 (2), 67-80 (2018).
- Hartenian, E., Doench, J. G. Genetic screens and functional genomics using CRISPR/Cas9 technology. FEBS Journal. 282 (8), 1383-1393 (2015).
- Li, W., et al. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knock-out screens. Genome Biology. 15 (12), 554(2014).
- Sheel, A., Xue, W. Genomic Amplifications Cause False Positives in CRISPR Screens. Cancer Discovery. 6 (8), 824-826 (2016).
- Meyers, R. M., et al. Computational correction of copy number effect improves specificity of CRISPR-Cas9 essentiality screens in cancer cells. Nature Genetics. 49 (12), 1779-1784 (2017).
- Henser-Brownhill, T., Monserrat, J., Scaffidi, P. Generation of an arrayed CRISPR-Cas9 library targeting epigenetic regulators: from high-content screens to in vivo assays. Epigenetics. 12 (12), 1065-1075 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados