È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Schermi genetici basati su CRISPR in cellule mammaliane
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
La tecnologia CRISPR-Cas9 fornisce un metodo efficiente per modificare con precisione il genoma dei mammiferi in qualsiasi tipo di cellula e rappresenta un nuovo mezzo per eseguire schermi genetici a livello di genoma. Qui viene fornito un protocollo dettagliato che illustra i passaggi necessari per il successo delle prestazioni degli schermi CRISPR-Cas9 a livello di genoma raggruppato.
Abstract
L'editing del genoma utilizzando il sistema CRISPR-Cas ha notevolmente avanzato la capacità di modificare con precisione i genomi di vari organismi. Nel contesto delle cellule dei mammiferi, questa tecnologia rappresenta un nuovo mezzo per eseguire schermi genetici a livello di genoma per studi di genomica funzionale. Le librerie di RNA guida (sgRNA) rivolti a tutti i frame di lettura aperti consentono la facile generazione di migliaia di perturbazioni genetiche in un singolo pool di cellule che possono essere sottoposte a screening per fenotipi specifici per implicare la funzione genica e i processi cellulari in un modo imparziale e sistematico. Gli schermi CRISPR-Cas forniscono ai ricercatori un metodo semplice, efficiente ed economico per scoprire i progetti genetici per i fenotipi cellulari. Inoltre, l'analisi differenziale degli schermi eseguiti in varie linee cellulari e da diversi tipi di cancro può identificare geni che sono contestualmente essenziali nelle cellule tumorali, rivelando potenziali bersagli per specifiche terapie anticancro. L'esecuzione di schermi a livello di genoma nelle cellule umane può essere scoraggiante, in quanto ciò comporta la gestione di decine di milioni di cellule e richiede l'analisi di grandi insiemi di dati. I dettagli di queste schermate, come la caratterizzazione della cella, le considerazioni sulla libreria CRISPR e la comprensione delle limitazioni e delle funzionalità della tecnologia CRISPR durante l'analisi, sono spesso trascurati. Qui è fornito un protocollo dettagliato per il successo delle prestazioni di successo di schermi basati su genomica in pool CRISPR-Cas9.
Introduzione
CRISPR-Cas, abbreviazione per brevi ripetizioni palindromiche raggruppate regolarmente interspaziate e nuclease associate a CRISPR, è costituito da una singola proteina nucleasi (ad esempio Cas9) in complesso con una guida sintetica RNA (sgRNA). Questo complesso di ribonucleoproteina si rivolge all'enzima Cas9 per indurre rotture di DNA a doppio filamento in uno specifico locus genomico1. Le rotture a doppio filamento possono essere riparate tramite riparazione diretta homologia (HDR) o, più comunemente, attraverso l'unione finale non omologa (NHEJ), un meccanismo di riparazione soggetto a errori che si traduce in inserimento e/o delezioni (INDELS) che spesso interrompono la funzione genica 1. L'efficienza e la semplicità di CRISPR consentono un livello di target genomico precedentemente irraggiungibile che supera di gran lunga le precedenti tecnologie di editing del genoma [ad esempio, nucleasi di dita di zinco o trascrittiviere nuosi effettore simili a TALENS), entrambi affetti da una maggiore complessità progettuale, da una minore efficienza di trasfezione e da limitazioni nell'editing genico multiplex2].
L'applicazione di ricerca di base dell'editing del genoma basato sull'RNA CRISPR ha permesso agli scienziati di interrogare in modo efficiente ed economico le funzioni dei singoli geni e la topologia delle reti di interazione genetica. La capacità di eseguire schermi funzionali ad ampio raggio su genoma è stata notevolmente migliorata dall'uso del sistema CRISPR-Cas, in particolare se confrontato con le precedenti tecnologie di perturbazione genetica come l'interferenza dell'RNA (RNAi) e la mutagenesi della trappola genica. In particolare, l'RNAi soffre di elevati effetti fuori bersaglio e knockdown incompleto, con conseguente minore sensibilità e specificità rispetto al CRISPR3,4,5, mentre i metodi di trappola genica sono fattibili solo in aploide celle per le schermate di perdita della funzione, limitando l'ambito dei modelli cellulari che possono essere interrogati6. La capacità di CRISPR di generare un sistema completo di eliminazione genica fornisce un sistema più biologicamente robusto per interrogare i fenotipi mutanti, con basso rumore, effetti minimi fuori bersaglio e un'attività costante tra i reagenti5. Le librerie di sgRNA CRISPR-Cas9 che si rivolgono all'intero genoma umano sono ora ampiamente disponibili, consentendo la generazione simultanea di migliaia di eliminazioni geniche in un singolo esperimento3,7,8,9 .
Abbiamo sviluppato uniche librerie di lentivirali sgRNAvirale a livello genomico CRISPR-Cas9 chiamate le librerie di Toronto Knock-out (TKO) (disponibili tramite Addgene) compatte e ottimizzate per la sequenza per facilitare schermi genomici funzionali ad alta risoluzione. L'ultima libreria, TKOv3, si rivolge a 18.000 geni di codifica delle proteine umane con 71.090 guide ottimizzate per l'editing dell'efficienza utilizzando dati empirici10. Inoltre, TKOv3 è disponibile come libreria a un componente (LCV2::TKOv3, Addgene ID #90294) che esprime Cas9 e sgRNA su un singolo vettore, alleviando la necessità di generare celle stabili e che esprimono Cas9, consentendo l'eliminazione a livello di genoma in un'ampia gamma di tipi di cellule dei mammiferi. TKOv3 è disponibile anche in un vettore senza Cas9 (pLCKO2::TKOv3, Addgene ID 125517) e può essere utilizzato in celle che esprimono Cas911.
Una popolazione cellulare modificata CRISPR-Cas9 a livello di genoma può essere esposta a diverse condizioni di crescita, con l'abbondanza di sgRNA nel tempo quantificati dal sequenziamento di prossima generazione, fornendo una lettura per valutare l'abbandono o l'arricchimento delle cellule con genetica tracciabile Perturbazioni. Le librerie knock-out CRISPR possono essere sfruttate per identificare i geni che, dopo la perturbazione, causano difetti di forma fisica cellulare, sensibilità moderata ai farmaci (ad esempio, geni sensibili o resistenti), regolano l'espressione proteica (ad esempio, reporter) o sono necessari per un certo funzione del percorso e lo stato cellulare12,13,14. Ad esempio, schermi di fitness differenziale in una linea cellulare del cancro in grado di identificare sia l'esaurimento o la riduzione di oncogeni e arricchimento o un aumento dei geni soppressori tumorali3,14,15. Allo stesso modo, l'uso di dosi intermedie di farmaci terapeutici può rivelare sia la resistenza ai farmaci che i geni di sensibilizzazione16,17.
Fornito qui è un protocollo di screening dettagliato per lo screening della perdita di perdita CRISPR-Cas9 su scala genomica utilizzando le librerie Knock-out di Toronto (TKOv1 o v3) nelle cellule dei mammiferi dalla generazione di librerie, dalle prestazioni di screening all'analisi dei dati. Anche se questo protocollo è stato ottimizzato per lo screening utilizzando le librerie Knock-out di Toronto, può essere applicato e diventare scalabile a tutte le librerie in pool di sgRNA CRISPR.
Protocollo
Gli esperimenti descritti di seguito devono seguire le linee guida dell'Ufficio per la salute e la sicurezza ambientale dell'istituto.
1. Amplificazione plasmid della libreria lentivirale CRISPR in pool
- Diluire la libreria di DNA plasmid di sgRNA CRISPR ready-made a 50 ng/L in TE (ad esempio, TKOv3).
- Elettroporare la libreria utilizzando cellule elettrocompetenti. Impostare un totale di quattro reazioni di elettroporazione come descritto di seguito.
- Aggiungete su ghiaccio 2 -L di 50 ng/-L nella libreria TKO a 25 -L di cellule elettrocompetenti scongelate.
- Elettroporate utilizzando le impostazioni ottimali suggerite dal protocollo del produttore. Entro 10 s dell'impulso, aggiungere 975 l di Recovery Medium (o SOC medium) alla cuvette.
- Trasferire le cellule elettroporate in un tubo di coltura e aggiungere 1 mL di Recovery Medium. Incubare i tubi in un'incubatrice acquosa a 250 giri per 1 h a 37 gradi centigradi.
- Impostare una piastra di diluizione per il titer della libreria e stimare l'efficienza della trasformazione.
- Piscina tutti 8 mL di cellule recuperate e mescolare bene. Trasferire 10 l delle celle raggruppate a 990 o L di Recovery Medium per una diluizione di 800 volte e mescolare bene.
- Piastra 20 -L della diluizione su una piastra di agar preriscaldata di 10 cm LB e carbenicillin (100 g/L). Ciò si traduce in una diluizione di 40.000 volte dei trasformatori che verranno utilizzati per calcolare l'efficienza di trasformazione.
- Piastra di 400 litri di cellule recuperate su ogni piastra su un totale di 20 piastre di agar da 15 cm preriscaldate e piastre di agar carbenicillin. Incubare le piastre per 14-16 h a 30 gradi centigradi.
NOTA: la crescita a questa temperatura più bassa riduce al minimo la ricombinazione tra ripetizioni a terminale lungo (LTR)18. - Per calcolare l'efficienza della trasformazione, contare il numero di colonie sulla piastra di diluizione di 40.000 volte (passaggio 1.3.2). Moltiplicare il numero di colonie contate per 40.000 per ottenere il numero totale di colonie su tutte le piastre. Procedere se il numero totale di colonie rappresenta una copertura biblioteca equivalente al minimo di 200 colonie x per sgRNA (la più ottimale è 500-1000x).
- Ad esempio, il numero minimo di colonia per la libreria TKOv3 (71.090 sgRNA) è 1,4 x 107, che equivale a 200 colonie x per sgRNA. Se la rappresentazione della colonia è insufficiente, aumentare il numero di elettroporazioni nel passaggio 1.2 in base al numero di colonie sulla piastra di diluizione per ottenere la copertura minima della biblioteca.
- Raccogliere le colonie come descritto di seguito
- Ad ogni lastra da 15 cm, aggiungete 7 mL di lbecillano (100 g/L), quindi raschiete le colonie con uno spalmatore di cellule. Con una pipetta da 10 mL, trasferire le cellule raschiate in una bottiglia o una bottiglia conica sterile da 1 L.
- Ancora una volta risciacquare la piastra con 5 mL di LB - mezzi carbenicillin e trasferire la soluzione alla bottiglia.
- Ripetere per tutti i piatti di mettere in comune le celle da 20 piatti in una bottiglia sterile.
- Mescolare le celle raccolte con una barra di agitazione per 1 h a temperatura ambiente (RT) per rompere i grumi delle cellule. Trasferire le cellule in bottiglie di centrifuga pre-pesate e centrifugare a 7.000 x g ai batteri pellet, quindi scartare i media.
- Pesare il pellet a cellule bagnate e sottrarre il peso della bottiglia di centrifuga per determinare il peso finale del pellet bagnato. Purificare il DNA plasmide utilizzando un kit di purificazione plasmida maxi o mega a seconda della quantità di pellet batterico che ogni colonna può elaborare.
2. Produzione di lentivirus della libreria di sgRNA CRISPR su larga scala
NOTA: tutti i passaggi in questa sezione del protocollo vengono eseguiti in una struttura BSL2 in un mobile di biosicurezza di classe II di tipo A2.
- Calcolare il numero di piastre da 15 cm necessarie per la produzione di virus sulla base della stima che 18 mL di virus viene in genere raccolto da una piastra di 15 cm.
- Preparare le cellule per la trasfezione seminando cellule di imballaggio HEK-293T in supporti a bassa crescita antibiotica (DMEM - 10% FBS - opzionale: 0,1x penna / strep) a 8 x 106 cellule per 15 cm piastra in 20 mL di supporti. Incubare le cellule durante la notte a 37 gradi centigradi, 5% DI CO2. Assicurarsi che le cellule placcate siano 70-80% confluenti e si diffondano uniformemente al momento della trasfezione.
- Il giorno successivo, preparare tre plasmidi trasfezione come descritto nella tabella 1 per piastre da 15 cm. Calcolare la quantità di plasmide necessaria per una trasfezione e fare un mix di plasmidi per il numero di piastre, più uno da trasfetare.
- Preparare un reagente di trasfezione a base di lipidi per ogni trasfezione, come descritto nella tabella 2. È stato ridotto il supporto del siero in singoli tubi di microcentrifuga da 1,5 ml per il numero di piastre da trafetare. Aggiungere il reagente di trasfezione, mescolare delicatamente e incubare per 5 min a RT.
- Dopo l'incubazione di 5 min, aggiungere la quantità di DNA necessaria per una trasfezione al reagente di trasfezione per un rapporto 3:1 di reagente di trasfezione-g di complesso di DNA. Mescolare delicatamente e incubare per 30 min a RT.
NOTA: Le trasfetazioni successive possono essere preparate in serie di cinque o meno, con intervalli di 5 minuti per ottimizzare il tempo ed evitare un'eccessiva incubazione. - Dopo 30 min di incubazione, trasferire attentamente ogni miscela di trasfezione ad ogni piatto di cellule di imballaggio. Aggiungere l'intero mix utilizzando una punta di pipette da 1 mL dropwise in un movimento circolare a zig-zag senza disturbare il monostrato cellulare. Incubare le cellule a 37 gradi centigradi per 18 h al 5% di CO2.
- Preparare i mezzi di raccolta virale: 500 mL di mezzo DMEM , 32 mL di brodo BSA (20 g/100 mL, disciolto nel DMEM, filtro sterilizzato con filtro da 0,22 m) - 5 mL di penna/strep.
- Dopo 18 h, rimuovere i supporti (utilizzare una corretta gestione dei rifiuti di lentivirus come l'incubazione in ipocloreto di sodio 1% per 30 min prima dello smaltimento). Sostituire delicatamente con 18 mL di supporti di raccolta virale per ogni piatto. Incubare le cellule a 37 gradi centigradi per 18 h al 5% di CO2.
- Dopo 24 h, controllare le cellule di imballaggio per morfologia anormale e fusa come indicazione di buona produzione di virus. Quindi, raccogliere il lentivirus raccogliendo tutti i supernatali e trasferendosi in uno sterile tubo di centrifuga conico.
- Girare il supporto contenente virus a 300 x g per 5 min e pellet le cellule di imballaggio. Aliquota del supernatante in un tubo sterile di polipropilene senza disturbare il pellet.
- Conservare il virus a 4 gradi centigradi per brevi periodi (meno di 1 settimana) o immediatamente a -80 gradi centigradi per lo stoccaggio a lungo termine. Aliquota virus prep a volumi monouso per l'archiviazione a lungo termine per evitare il congelamento / scongelamento.
3. Caratterizzazione della riga di cella per lo screening
- Selezionare la linea di celle desiderata.
- Misurare e registrare il tempo approssimativo di raddoppio delle cellule.
- Determinare la densità di placcatura cellulare ottimale per la coltura delle cellule ogni 3-4 raddoppi cellulari in un recipiente di coltura dei tessuti di scelta (ad esempio, piastre di coltura tissutale di 15 cm).
- Determinare la concentrazione di puromycina da utilizzare nella linea cellulare desiderata per la selezione di librerie TKO contenenti marcatore di resistenza pari alla formacina come segue:
- Le cellule di semi in una piastra di 12 pozzi alla densità necessaria per raggiungere la confluenza dopo 72 h, poi incubano durante la notte (37 gradi centigradi, 5% di CO2).
- Il giorno successivo, passare a un supporto contenente un intervallo di diluizione di concentrazioni di puromicina da 0 g/mL a 10 g/mL, in incrementi di 0,5 g/mL. Incubare le cellule per 48 h.
- Dopo 48 h, misurare la vitalità cellulare mediante il conteggio cellulare o la colorazione alamarBlue.
- Determinare la concentrazione più bassa che uccide il 100% delle cellule in 48 h. Utilizzare questa concentrazione per selezionare per le popolazioni di cellule trasdotte della libreria CRISPR nei passaggi 4.6 e 5.2.6.
NOTA: Per le linee cellulari con tempi di raddoppio più lunghi, possono essere tollerate incubazioni più lunghe con puromycin. In queste situazioni, determinare la curva di uccisione per il tempo di incubazione richiesto per i raddoppi delle cellule <3. Ridurre al minimo il tempo di selezione per evitare l'abbandono dei geni essenziali prima dell'inizio dello screening.
- Controllare le cellule per la sensibilità al bromuro di esadimethrine (fino a 8 g/mL) eseguendo una curva di risposta alla dose nello stesso metodo utilizzato per misurare la sensibilità della mastracina (passaggio 3.2). Se si osserva tossicità con <8 g/mL di bromuro di esadimettoria, non utilizzare.
4. Titore funzionale della libreria di lentivirus CRISPR in pool per la determinazione di MOI
- Scongelare una nuova aliquota di lentivirus della libreria di sgRNA CRISPR in pool (ad esempio, LCV2::TKOv3) e continuare a pattinare.
- Progettare una serie di volumi di virus da testare tra l'intervallo di 0-2 mL (ad esempio, 0 mL, 0,25 mL, 0,5 mL, 1 mL e 2 mL).
- Raccogliere cellule bersaglio e cellule di semi in piastre da 15 cm alla densità necessaria per raggiungere la confluenza in 72 h.
- Per ogni volume di virus da testare, preparare piastre duplicate. Aggiungere cellule, virus, bromuro di esadimethrine (8 g/mL) e supporti ad un volume finale di 20 mL. Mescolare accuratamente le piastre, sedersi a livello di piastre in incubatrice e incubare per 24 h (37 gradi centigradi, 5% CO2).
- Dopo 24 h, rimuovere il virus contenente i supporti e smaltire (utilizzare precauzioni di biosicurezza per la manipolazione dei rifiuti di lentivirus). Facoltativamente, lavare delicatamente la piastra con PBS caldo per rimuovere il virus estraneo.
- Per ogni condizione di virus, sostituire con 20 mL di supporti contenenti puromycina utilizzando la concentrazione determinata a uccidere le cellule nella sezione 3, in una piastra di replica. All'altra piastra, aggiungere 20 mL di supporti freschi senza puromycina. Incubare per 48 h (37 gradi centigradi, 5% CO2).
- Dopo 48 h, verificare che tutte le cellule non infette (condizione del virus 0 mL) trattate con panminacina siano morte. Raccogliere tutte le piastre singolarmente e disperdere le cellule da ripetuti pipettaggio delicato.
- Contare le cellule di tutte le piastre e calcolare il MOI per ogni volume di virus confrontando i conteggi delle cellule con la selezione di puromicina con i conteggi delle cellule senza puromycina (ad es.
- Risultati del grafico per determinare il volume del virus che porta al 30%-40% di sopravvivenza cellulare con selezione della pancina contro senza puromicina. Utilizzare questo volume di virus per ottenere un MOI di 0.3–0.4 durante lo schermo nelle stesse condizioni di coltura dei tessuti.
5. Infezione dello schermo primario, selezione e passaggio cellulare
- Selezionare la copertura della libreria di sgRNA CRISPR da mantenere in tutto lo schermo (minimo consigliato di 200 volte).
- In base alla copertura della biblioteca, determinare il numero di cellule necessarie per mantenere questa copertura per sgRNA e il numero di cellule necessarie per l'infezione al MOI 0.3(Tabella 3).
- Determinare il numero di piastre necessarie per impostare l'infezione (Tabella 4).
- Infettare le cellule con la libreria CRISPR
- Raccogliere le cellule e sedare il numero di cella richiesto per ogni piastra di 15 cm.
- Aggiungere il bromuro di esadimethrine (8 g/mL) a tutte le piastre.
- Aggiungere il virus al volume richiesto per MOI 0.3 per lo screening e le piastre Control 2. Per il controllo 1, non aggiungere virus e sostituire tale volume con i supporti.
- Mescolare accuratamente le piastre inclinando. Mettere le piastre in incubatrice, assicurandosi che siano livellate.
NOTA: Le infezioni da lotti possono essere eseguite combinando un mix master di virus, supporti ed esadimetofine bromuro alle cellule in sospensione prima della placcatura. - Rimuovere i supporti e sostituirli con supporti freschi contenenti la puromicina alla concentrazione determinata al punto 3.2.4 allo screening e controllare 1 piastre 24 h dopo l'infezione da virus. Aggiungere supporti freschi senza puromycin alla piastra di controllo 2. Incubare le cellule per 48 h (37 oC, 5% CO2).
- 48 h dopo puromycin aggiungere, assicurarsi che tutte le cellule non infette sono morti (controllo 1) per confermare l'attività puromycina, quindi raccogliere le cellule infette.
- Raccolta della popolazione cellulare infetta e passaggio cellulare
- Raccogliere le cellule selezionate da puromycina da tutte le piastre di screening in un contenitore sterile. Raccogliere le celle da ogni piastra di controllo separatamente. Disperdere le cellule con una leggera pipettatura ripetuta.
- Contare le celle da celle di screening in pool, controllare 1 e controllare 2 separatamente e calcolare il numero di celle per 1 mL.
- Calcolare la copertura MOI e piegatura ottenuta come segue:
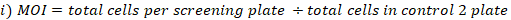
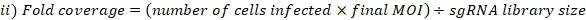
- Raccogli tre repliche di pellet cellulari dalle cellule raggruppate alla copertura della libreria selezionata per l'estrazione genomica del DNA. Centrifugare le cellule a 500 x g per 5 min. Lavare con PBS. Etichettare i tubi e asciugare a secco i pellet delle cellule a -80 gradi centigradi (questi sono campioni di riferimento T0).
- Suddividere il pool di celle infette in tre gruppi di replica (ad esempio, replicare A, replicare B, replicare C), mantenendo la copertura della libreria all'interno di ogni replica. Cellule di semi alla stessa densità di semina normalmente utilizzate per l'espansione. Utilizzare lo stesso numero di celle per ogni piastra di replica e lo stesso numero totale di celle tra le repliche.
- Continuare a passare le cellule e raccogliere tre repliche di pellet cellulari da ogni replica di cellule infettate in pool come sopra, ogni 3-8 giorni a seconda della linea cellulare, per un massimo di 15-20 raddoppi cellulari. Ad ogni passaggio, raccogliere le cellule da tutte le piastre in ogni gruppo di replica tra loro (cioè, tutte le cellule da piastre di replica Vengono rimescolate insieme, tutte le cellule da piastre di replica B vengono ri-mescolate insieme, ecc.).
- Etichettare ogni pellet con un tempo (T) e replicare la designazione. Corrisponde al numero di giorni dopo la raccolta del pellet (ad esempio, T3_A, T3_B, T3_C e così via).
- Per gli schermi di farmaci a selezione negativa, consentire alle cellule di recuperare per almeno un passaggio dopo T0 prima del trattamento. A T3 o T6, dividere le cellule da ciascun gruppo di replica (A, B, C) in popolazioni di trattamento e controllo farmacologiche, utilizzando la stessa densità di semina utilizzata nel passaggio 5.3.5.
- Raggruppare separatamente il numero di cellule necessarie per la copertura della biblioteca per ogni replica nel gruppo di trattamento farmacologico. Aggiungere il farmaco a concentrazioni intermedie (IC20-IC50). Semina le cellule e incubano (37 gradi centigradi, 5% CO2) fino al passaggio successivo.
- Raggruppare separatamente il numero di celle necessarie per la copertura della libreria per ogni replica nel gruppo di controllo del veicolo. Aggiungere il controllo del veicolo utilizzando lo stesso volume del farmaco (<0.5% v/v). Semina le cellule e incubano (37 gradi centigradi, 5% CO2) fino al passaggio successivo.
- Continuare a passare le cellule e raccogliere i pellet cellulari per il DNA genomico ogni 3 giorni, come descritto al passaggio 5.3.5, rinfrescando il farmaco o il veicolo ad ogni passaggio.
- Per gli schermi di selezione positiva o resistenza ai farmaci, dividere ogni gruppo di replica in base al numero di cellule necessarie per la copertura della biblioteca. Aggiungere le concentrazioni di droga IC90 a ogni replica. All'IC90,la maggior parte delle cellule sarà uccisa. Consentire alle popolazioni resistenti di crescere e raccogliere pellet cellulari (1-2 x 107 cellule) per l'estrazione genomica del DNA.
6. Preparazione e sequenziamento dei campioni CRISPR
- Purificazione genomica del DNA
- Incubare i pellet a celle congelate per 5-10 min a RT per lo scongelamento.
- Aggiungere 1,4 mL di PBS a un tubo di centrifuga da 50 mL contenente un pellet cellulare. Vortice per 20 s per risospendere le cellule e riposare per 1 min. Se necessario, pipetta 15x con P1000 per rompere i grumi di cellule rimanenti. Se si trasferiscono le cellule da un tubo da 15 mL o da 1,5 mL, sospendere le celle con 1 mL di PBS, quindi trasferire le celle a un tubo da 50 mL e risciacquare il tubo originale con 400 .
- Aggiungere 5 mL di Nuclei Lysis Solution alle cellule risospese. Utilizzando una pipetta da 10 mL, mescolare il campione pipetting su e giù 5x.
- Aggiungete al lisate nucleare 32 gradi di RNase A (20 mg/mL; per ottenere una concentrazione finale di 100 g/mL) al lisato nucleare e mescolate il campione invertendo il tubo 5x. Incubare la miscela a 37oC per 15 min e lasciare raffreddare il campione per 10 min a RT.
- Aggiungere 1,67 mL di Protein Precipitation Solution al lisato e vortice vigorosamente per 20 s. Piccoli grumi proteici possono essere visibili dopo la miscelazione.
- Centrifuga a 4.500 x g per 10 min a RT.
- Utilizzando una pipetta da 10 mL, trasferire il supernatante in un tubo centrifuga di 50 mL contenente 5 mL di isopropanolo. Mescolare delicatamente la soluzione 10x per inversione fino a quando non viene osservato il DNA.
NOTA: Il DNA può essere osservato come fili bianchi simili a fili che formano una massa visibile. - Centrifuga a 4.500 x g per 5 min a RT per pellet il DNA.
- Utilizzando una pipetta da 10 mL, rimuovere con cura il supernatante ed evitare di slocare il pellet di DNA. Aggiungere al DNA 5 mL di 70% di etanolo al DNA. Ruotare delicatamente il tubo per lavare il pellet del DNA e i lati del tubo di centrifuga.
- Centrifuga a 4.500 x g per 5 min a RT.
- Utilizzando una pipetta da 10 mL, rimuovere con cura il 70% di etanolo ed evitare di slocare il pellet di DNA. DNA genomico asciutto per 10 min alla RT.
- Aggiungere al tubo 400 l di TE e lasciare sciogliere il DNA incubando a 65 gradi centigradi per 1 h. Mescolare il DNA facendo scorrere delicatamente il tubo ogni 15 min. Se il DNA non si scioglie completamente, incubano il tubo a 65 gradi centigradi per un ulteriore 1 h mentre si snoda delicatamente il tubo ogni 15 min, e lasciarlo a 4 gradi durante la notte.
- Centrifuga a 4.500 x g per 1 min a RT e trasferimento del DNA genomico a un tubo a bassa legatura da 1,5 mL.
- Quantificare e misurare la purezza del DNA genomico sia sullo spettrofotometro (per il contenuto totale di acido nucleico) che sul fluorometro (per il contenuto di DNA a doppio filamento).
- Facoltativamente, precipitare il DNA genomico se ci sono problemi con l'amplificazione PCR a valle dello sgRNA come segue.
- Trasferire il DNA genomico di 400 .L in un tubo di microcentrifuga da 1,5 mL.
- Aggiungere 18 luna di 5 M NaCl (concentrazione finale di 0,2 M) e 900 -L del 95% di etanolo.
- Invertire il tubo 10x fino a quando accuratamente mescolato, quindi centrifugare a 16.000 x g per 10 min a RT.
- Rimuovere con cura il supernatante ed evitare di dislocare il pellet di DNA. Lavare il pellet di DNA con 500 lun del 70% di etanolo. Ruotare delicatamente il tubo per lavare il pellet di DNA.
- Centrifuga a 16.000 x g per 5 min a RT.
- Rimuovere con attenzione il supernatante ed evitare di dislocare il pellet di DNA. DNA genomico asciutto per 10 min alla RT.
- Aggiungere 300 l di TE per sciogliere il DNA come descritto nei passaggi 6.1.12.
- Quantificare e misurare la purezza del DNA genomico come descritto al punto 6.1.14.
- Preparazione della libreria di sequenziamento CRISPR
- Impostare la PCR 1 come indicato nella tabella 5 utilizzando un totale di 100 g di DNA genomico. Aggiungere 3,5 g di DNA genomico per una reazione 50 a Ello e impostare reazioni identiche di 50 ll per ottenere la copertura desiderata. Nella tabella 6 sono elencati esempi di sequenze di primer per l'amplificazione delle librerie di sequenziamento LCV2::TKOv3. Nella tabella 7 sono elencati esempi di sequenze di primer per l'amplificazione delle librerie di sequenziamento pLCKO2::TKOv3.
- Amplificare le reazioni PCR 1 in un termociclore utilizzando il programma descritto nella tabella 8.
- Controllare l'amplificazione PCR 1 eseguendo 2 ll l del prodotto PCR su un gel di agarose dell'1%. PCR 1 produce un prodotto di 600 bp.
- Mettere in comune tutte le singole 50 reazioni di 50 -L per ogni campione genomico di DNA e mescolare mediante vortice.
- Impostare una reazione PCR 2 (50 gradi centigradi) per ogni campione, come descritto nella tabella 9 utilizzando come modello 5 L del prodotto PCR 1 in pool. Usare combinazioni di primer dell'indice univoche per ogni singolo campione per consentire il pooling di esempi di libreria di sequenziamento.
- Amplificare la reazione PCR 2 in un termociclore utilizzando il programma descritto nella tabella 10.
- Pulire l'attrezzatura gel ad agarose per purificare i prodotti amplificati con 0,1 N HCl per 10 minuti prima di lanciare un gel. Preparare un gel di agarose del 2% contenente macchie di DNA per purificare i prodotti amplificati PCR 2.
- Eseguire il prodotto PCR 2 sul gel di agarose 2% a bassa tensione (1.0–1.5 h run). PCR 2 produce un prodotto di 200 bp.
- Visualizza i prodotti PCR su un transilluminatore a luce blu. Accise la fascia di 200 bp e purificano il DNA dalla fetta di gel di agarose utilizzando un kit di estrazione gel. Quantificare e misurare la purezza della libreria di sequenziamento sia sullo spettrofotometro che sul fluorometro.
NOTA: una tipica concentrazione della libreria di sequenziamento ad esempio in gel varia da 5 a 10 ng/L e una resa totale di 150-300 ng.
- Sequenza ad alta velocità effettiva
- Sequenziare le librerie di sequenziamento CRISPR nei sequencer di nuova generazione.
- Riferimento sequenza T0 campioni ad una maggiore profondità di lettura di 400-500 volte la copertura della libreria. Sequenziare campioni sperimentali di timepoint per schermi drop-out ad una profondità di lettura minima di 200 volte. Per schermi di selezione positivi forti, una profondità minima di lettura di 50 volte la copertura è sufficiente per l'identificazione di sgRNA arricchiti.
NOTA: È fondamentale sequenziare il campione T0 per determinare la rappresentazione della libreria per un particolare schermo e fungere da riferimento per le modifiche di piegatura dello sgRNA determinante nel tempo.
7. Analisi dei dati
- Allineare la sequenza utilizzando programmi come Bowtie per mappare le letture di sequenza nella libreria di riferimento utilizzando i seguenti parametri: -v2 (consentendo due mancate corrispondenze) e -m1 (ignorando qualsiasi lettura mappata a più di una sequenza nella libreria).
- Normalizzare il numero di letture mappate in modo univoco per ogni sgRNA per un dato campione a 10 milioni di letture per campione come segue:
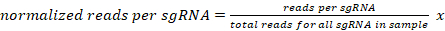 107 del sistema
107 del sistema
- Calcolare la modifica della piega log2 di ogni sgRNA per ogni replica in ogni punto temporale (Tn) rispetto al campione T0 (Tn/T0). Aggiungere uno pseudo conteggio di 0,5 letture a tutti i conteggi di lettura per evitare discontinuità da zeri. Escludere gli sgRNA con <30 letture non elaborate nel campione T0 dal calcolo della modifica di piegatura e dall'analisi a valle.
- Analizzare i cambiamenti di piegatura con l'algoritmo Bayesian Analysis of Gene Essentiality (BAGEL) , utilizzando i set di allenamento essenziali e non essenziali di base definiti in precedenza19 per gli schermi di essenzialità del gene ( La tabella supplementare S1) o Drug per gli schermi di droga20.
- Calcolare la precisione e il richiamo per la valutazione delle prestazioni dello schermo utilizzando i punteggi BF. Utilizzare il set essenziale del passaggio 7.4 come vero elenco positivo per la funzione precision_recall_curve della libreria Scikit-learn per Python, insieme al sottoinsieme di punteggiBF sopra riportato. In alternativa, eseguire la stessa operazione utilizzando il pacchetto PRROC in R.
- Calcolare il cambio media di tutte le guide per ogni gene. Generare grafici di densità per i geni essenziali e non essenziali (vedere il passaggio 7.4) in R o in software equivalente. In R, se x.ess è un vettore contenente i valori di modifica della piega del tronco dei geni essenziali e x.nonEscontengono geni non essenziali, tracciare usando il seguente comando:
plot( densità( x.ess ), xlab , "medio logFC",col , "rosso", lwd , 2 )
lines( densità( x.nonEss ), col
NOTA: per i dettagli della versione Python e i pacchetti utilizzati, vedere scikit-learn v0.19.1: (pubblicato da Pedregosa et al.21).
Risultati
Panoramica del flusso di lavoro di screening CRISPR su scala genomica
La figura 1 illustra una panoramica del flusso di lavoro di screening CRISPR in pool, a partire dall'infezione delle cellule bersaglio con lentivirus della libreria CRISPR con un basso MOI per garantire singoli eventi di integrazione e un'adeguata rappresentazione della libreria (in genere da 200 a 1000 -fold). Do...
Discussione
Grazie alla sua semplicità d'uso e all'elevata flessibilità, la tecnologia CRISPR è stata ampiamente adottata come strumento di scelta per un montaggio preciso del genoma. Lo screening CRISPR in pool fornisce un metodo per interrogare migliaia di perturbazioni genetiche in un singolo esperimento. Negli schermi in pool, le librerie di sgRNA fungono da codici a barre molecolari, poiché ogni sequenza è unica ed è mappata al gene mirato. Isolando il DNA genomico della popolazione cellulare, i geni che causano il fenoti...
Divulgazioni
Gli autori non dichiarano interessi finanziari concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto da Genome Canada, Ontario Research Fund e Canadian Institutes for Health Research (MOP-142375, PJT-148802).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 micron filter | |||
| 30°C plate incubator | |||
| 37°C shaking incubator | |||
| 37°C, 5% CO2 incubator | |||
| 5 M NaCl | Promega | V4221 | |
| 50X TAE buffer | BioShop | TAE222.4 | |
| 6 N Hydrochloric acid solution | BioShop | HCL666.500 | |
| 95% Ethanol | |||
| Alamar blue | ThermoFisher Scientific | DAL1025 | |
| Blue-light transilluminator | ThermoFisher Scientific | G6600 | |
| Bovine Serum Albumin,Heat Shock Isolation, Fraction V. Min. 98%, Biotechnology grade | Bioshop | ALB001.250 | |
| Dulbecco's Modification of Eagles Medium | Life Technologies | 11995-065 | Cel culture media |
| Electroporation cuvettes | BTX | 45-0134 | |
| Electroporator | BTX | 45-0651 | |
| Endura electrocompetent cells | Lucigen | 90293 | |
| Fetal Bovine Serum | GIBCO | 12483-020 | |
| HEK293T packaging cells | ATCC | CRL-3216 | recommend passage number <15 |
| Hexadimethrine Bromide (Polybrene) | Sigma | H9268 | Cationic polymer to enhance transduction efficiency |
| Hexadimethrine Bromide (Polybrene) | |||
| LB agar plates with carbenicillin | |||
| LB medium with carbenicillin | |||
| Low molecular weight DNA ladder | New England Biolabs | N3233S | |
| Nanodrop spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| NEBNext Ultra II Q5 Master Mix | New England Biolabs | M0544L | |
| Opti-MEM | Life Technologies | 31985-070 | Reduced serum media |
| Plasmid maxi purification kit | Qiagen | 12963 | |
| pMD2.G (envelope plasmid) | Addgene | Plasmid #12259 | lentiviral system |
| psPAX2 (packaging plasmid) | Addgene | Plasmid #12260 | lentiviral system |
| Puromycin | Wisent | 400-160-UG | |
| QIAquick gel extraction kit | Qiagen | 28704 | |
| Qubit dsDNA BR assay | ThermoFisher Scientific | Q32853 | |
| Qubit fluorometer | ThermoFisher Scientific | Q33226 | |
| RNAse A | Invitrogen | 12091021 | |
| S.O.C recovery medium | Invitrogen | 15544034 | |
| SYRB Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| Toronto KnockOut CRIPSR library (TKOv3) - Cas9 included | Addgene | Addgene ID #90203 | Genome-wide CRISPR library , includes Cas9, 71,090 sgRNA |
| Toronto KnockOut CRIPSR library (TKOv3) - non-cas9 | Addgene | Addgene ID #125517 | Genome-wide CRISPR library, non-Cas9, 71,090 sgRNA |
| Tris-EDTA (TE) solution, pH8.0 | |||
| UltraPure agarose | ThermoFisher Scientific | 16500500 | |
| Wizard genomic DNA purification kit | Promega | A1120 | |
| X-tremeGENE 9 DNA transfection reagent | Roche | 06 365 809 001 | Lipid based transfection reagent |
Riferimenti
- Jiang, F., Doudna, J. A. CRISPR-Cas9 Structures and Mechanisms. Annual Review of Biophysics. 46, 505-529 (2017).
- Baliou, S., et al. CRISPR therapeutic tools for complex genetic disorders and cancer (Review). International Journal of Oncology. 53 (2), 443-468 (2018).
- Hart, T., et al. High-Resolution CRISPR Screens Reveal Fitness Genes and Genotype-Specific Cancer Liabilities. Cell. 163 (6), 1515-1526 (2015).
- Morgens, D. W., Deans, R. M., Li, A., Bassik, M. C. Systematic comparison of CRISPR/Cas9 and RNAi screens for essential genes. Nature Biotechnology. 34 (6), 634-636 (2016).
- Evers, B., et al. CRISPR knock-out screening outperforms shRNA and CRISPRi in identifying essential genes. Nature Biotechnology. 34 (6), 631-633 (2016).
- Miles, L. A., Garippa, R. J., Poirier, J. T. Design, execution, and analysis of pooled in vitro CRISPR/Cas9 screens. The FEBS Journal. 283 (17), 3170-3180 (2016).
- Wang, T., Wei, J. J., Sabatini, D. M., Lander, E. S. Genetic screens in human cells using the CRISPR-Cas9 system. Science. 343 (6166), 80-84 (2014).
- Wang, T., et al. Identification and characterization of essential genes in the human genome. Science. 350 (6264), 1096-1101 (2015).
- Sanson, K. R., et al. Optimized libraries for CRISPR-Cas9 genetic screens with multiple modalities. Nature Communications. 9 (1), 5416 (2018).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knock-out Screens. G3: Genes|Genomes|Genetics. 7 (8), 2719-2727 (2017).
- Mair, B., Tomic, J., et al. Essential gene profiles for human pluripotent stem cells identify uncharacterized genes and substrate dependencies. Cell Reports. 27 (2), 599-615 (2019).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knock-out screening in human cells. Science. 343 (6166), 84-87 (2014).
- Sharma, S., Petsalaki, E. Application of CRISPR-Cas9 Based Genome-Wide Screening Approaches to Study Cellular Signalling Mechanisms. International Journal of Molecular Sciences. 19 (4), (2018).
- Steinhart, Z., et al. Genome-wide CRISPR screens reveal a Wnt-FZD5 signaling circuit as a druggable vulnerability of RNF43-mutant pancreatic tumors. Nature Medicine. 23 (1), 60-68 (2017).
- Wang, T., et al. Gene Essentiality Profiling Reveals Gene Networks and Synthetic Lethal Interactions with Oncogenic Ras. Cell. 168 (5), 890-903 (2017).
- Zimmermann, M., et al. CRISPR screens identify genomic ribonucleotides as a source of PARP-trapping lesions. Nature. 559 (7713), 285-289 (2018).
- Deans, R. M., et al. Parallel shRNA and CRISPR-Cas9 screens enable antiviral drug target identification. Nature Chemical Biology. 12 (5), 361-366 (2016).
- Trinh, T. J. J., Bloom, F., Hirsch, V. STBL2: an Escherichia coli strain for the stable propagation of retroviral clones and direct repeat sequences. Focus. 16, 78-80 (1994).
- Hart, T., Moffat, J. BAGEL: a computational framework for identifying essential genes from pooled library screens. BMC Bioinformatics. 17, 164 (2016).
- Wang, G. Z. M., et al. Identifying drug-gene interactions from CRISPR knock-out screens with drugZ. bioRxiv. , (2017).
- Pedregosa, F. V., G, , et al. Scikit-learn: Machine Learning in Python. Journal of Machine Learning Research. 12, 2825-2830 (2011).
- Ketela, T., et al. A comprehensive platform for highly multiplexed mammalian functional genetic screens. BMC Genomics. 12, 213 (2011).
- Doench, J. G. Am I ready for CRISPR? A user's guide to genetic screens. Nature Review Genetics. 19 (2), 67-80 (2018).
- Hartenian, E., Doench, J. G. Genetic screens and functional genomics using CRISPR/Cas9 technology. FEBS Journal. 282 (8), 1383-1393 (2015).
- Li, W., et al. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knock-out screens. Genome Biology. 15 (12), 554 (2014).
- Sheel, A., Xue, W. Genomic Amplifications Cause False Positives in CRISPR Screens. Cancer Discovery. 6 (8), 824-826 (2016).
- Meyers, R. M., et al. Computational correction of copy number effect improves specificity of CRISPR-Cas9 essentiality screens in cancer cells. Nature Genetics. 49 (12), 1779-1784 (2017).
- Henser-Brownhill, T., Monserrat, J., Scaffidi, P. Generation of an arrayed CRISPR-Cas9 library targeting epigenetic regulators: from high-content screens to in vivo assays. Epigenetics. 12 (12), 1065-1075 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon