É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Telas genéticas com base em CRISPR agrupadas em células de mamíferos
Neste Artigo
Resumo
A tecnologia CRISPR-Cas9 fornece um método eficiente para editar precisamente o genoma dos mamíferos em qualquer tipo de célula e representa um novo meio para realizar telas genéticas de todo o genoma. Um protocolo detalhado que discute as etapas necessárias para o desempenho bem-sucedido de telas de CRISPR-Cas9 agrupadas em todo o genoma é fornecido aqui.
Resumo
A edição do genoma usando o sistema CRISPR-CAS avançou enormente a capacidade de editar precisamente os genomas de vários organismos. No contexto das células de mamíferos, esta tecnologia representa um novo meio para realizar telas genéticas de todo o genoma para estudos de genômica funcional. Bibliotecas de guia RNAs (sgRNA) visando todos os quadros de leitura aberta permitem a geração facile de milhares de perturbações genéticas em um único pool de células que podem ser rastreados para fenótipos específicos para implicar a função gênica e processos celulares em um forma imparcial e sistemática. As telas CRISPR-CAS fornecem aos pesquisadores um método simples, eficiente e barato para descobrir os esquemas genéticos para fenótipos celulares. Além disso, a análise diferencial de telas realizadas em várias linhagens celulares e de diferentes tipos de câncer pode identificar genes que são contextualmente essenciais nas células tumorais, revelando potenciais alvos para terapias anticâncer específicas. A realização de telas de todo o genoma em células humanas pode ser assustadora, pois isso envolve o manuseio de dezenas de milhões de células e requer a análise de grandes conjuntos de dados. Os detalhes dessas telas, como a caracterização da linha celular, as considerações da biblioteca CRISPR e a compreensão das limitações e capacidades da tecnologia CRISPR durante a análise, são muitas vezes negligenciadas. Aqui é fornecido um protocolo detalhado para o desempenho bem-sucedido de telas baseadas em agregados de CRISPR-Cas9 em todo o genoma.
Introdução
CRISPR-CAS, abreviação de repetições palindrômicas curtas interespaçadas regularmente e nuclease associada a CRISPR, consiste em uma única proteína nuclease (por exemplo, Cas9) em complexo com um RNA guia sintético (sgRNA). Este complexo de ribonucleoproteína tem como alvo a enzima Cas9 para induzir quebras de DNA de dupla cadeia em um locus genómico específico1. As rupturas dobro-encalhado podem ser reparadas através da reparação dirigida homologia (HDR) ou, mais geralmente, com a junção não-homóloga da extremidade (nhej), um mecanismo de reparo propenso ao erro que resulte na inserção e/ou nos deleções (indels) que interrompem freqüentemente a função do gene a eficiência 1. The e a simplicidade de crispr permitem um nível previamente inatingível da segmentação genomic que ultrapassa distante tecnologias precedentes da edição do genoma [isto é, nucleases do dedo do zinco (ZNF) ou a transcrição ativador-como nucleases do efetoras ( TALENS), ambos sofrem de complexidade de design aumentada, menor eficiência de transfecção e limitações na edição de genes multiplex2].
A aplicação básica da pesquisa da edição do genoma RNA-baseada do único-guia de CRISPR permitiu que os cientistas interrogue eficientemente e barata as funções de genes individuais e topologia de redes genéticas da interação. A habilidade de executar telas funcionais do genoma-largo foi aumentada extremamente pelo uso do sistema de crispr-CAS, particular quando comparado às tecnologias genéticas mais adiantadas do perturbação tais como a interferência do RNA (RNAi) e a mutagenese da armadilha do gene. Em particular, RNAi sofre de efeitos off-Target elevados e knockdown incompleto, resultando em menor sensibilidade e especificidade em comparação com crispr3,4,5, enquanto os métodos de armadilha genética só são viáveis em haplóides células para telas de perda de função, limitando o escopo de modelos de células que podem ser interrogados6. A capacidade de CRISPR para gerar o knock-out completo do gene fornece um sistema mais biologicamente robusto para interrogar fenótipos mutantes, com baixo ruído, efeitos mínimos fora do alvo e atividade consistente em todos os reagentes5. As bibliotecas de crispr-Cas9 sgrna que visam todo o genoma humano estão agora amplamente disponíveis, permitindo a geração simultânea de milhares de knock-outs de genesem um único experimento3,7,8,9 .
Desenvolvemos bibliotecas de lentivirais sgRNA em todo o genoma CRISPR-Cas9, denominadas bibliotecas de Toronto knock-out (TKO) (disponíveis através do Addgene) que são compactas e otimizadas para sequências para facilitar telas de genômica funcional de alta resolução. A mais recente biblioteca, TKOv3, alvos ~ 18.000 genes de codificação de proteínas humanas com 71.090 guias otimizados para a eficiência de edição usando dados empíricos10. Além disso, TKOv3 está disponível como uma biblioteca de um componente (LCV2:: TKOv3, ID de Addgene #90294) expressando Cas9 e sgRNAs em um único vetor, aliviando a necessidade de gerar células de expressão de Cas9 estáveis, permitindo knock-out em todo o genoma em uma ampla gama de tipos de células de mamíferos. TKOv3 também está disponível em um vetor sem Cas9 (pLCKO2:: TKOv3, Addgene ID # 125517) e pode ser utilizado em células que expressam Cas911.
Uma população de células editada em todo o genoma CRISPR-Cas9 pode ser exposta a diferentes condições de crescimento, com a abundância de sgRNAs ao longo do tempo quantificada por sequenciamento de próxima geração, proporcionando uma leitura para avaliar o abandono ou enriquecimento de células com genética rastreável Perturbações. As bibliotecas de knock-out CRISPR podem ser aproveitadas para identificar genes que, após a perturbação, causam defeitos de aptidão celular, sensibilidade moderada à droga (por exemplo, genes sensíveis ou resistentes), regulam a expressão protéica (por exemplo, repórter), ou são necessários para um certo função do caminho e estado celular12,13,14. Por exemplo, as telas diferenciais de aptidão em uma linha celular cancerosa podem identificar tanto depleção ou redução de oncogenes e enriquecimento ou um aumento dos genes supressores de tumor3,14,15. Da mesma forma, o uso de doses intermediárias de fármacos terapêuticos pode revelargenes de resistênciae sensibilização dos fármacos16,17.
Aqui é fornecido um protocolo detalhado de triagem para a triagem de perda de função de CRISPR-Cas9 em escala de genoma usando as bibliotecas de Toronto knock-out (TKOv1 ou V3) em células de mamíferos de geração de biblioteca, desempenho de triagem para análise de dados. Embora esse protocolo tenha sido otimizado para triagem usando as bibliotecas de knock-out de Toronto, ele pode ser aplicado e tornar-se escalável para todas as bibliotecas agrupadas do sgRNA CRISPR.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Os experimentos descritos abaixo devem seguir as diretrizes do escritório de saúde e segurança ambiental do Instituto.
1. amplificação do plasmídeo da biblioteca de Lentivirus CRISPR sgRNA
- Diluir a biblioteca de DNA de plasmídeo CRISPR sgRNA pronta para 50 ng/μL em TE (por exemplo, TKOv3).
- Electroporate a biblioteca usando pilhas electrocompetentes. Configure um total de quatro reações de electroporação, conforme descrito abaixo.
- Adicionar 2 μL de 50 ng/μL de biblioteca TKO a 25 μL de células electrocompetentes descongelados para as cobetas pré-refrigeradas (1,0 mm) no gelo.
- Electroporate usando ajustes ideais sugeridos pelo protocolo do fabricante. Dentro de 10 s do pulso, adicione 975 μL de meio de recuperação (ou meio SOC) à cubeta.
- Transfira pilhas em a um tubo da cultura e adicione 1 ml do meio da recuperação. Incubar tubos em uma incubadora de agitação em 250 rpm para 1 h em 37 ° c.
- Configurar uma placa de diluição para Titer a biblioteca e estimar a eficiência de transformação.
- Piscina todos os 8 mL de células recuperadas e misture bem. Transfira 10 μL das células agrupadas para 990 μL de meio de recuperação para uma diluição de 800 vezes e misture bem.
- Placa 20 μL da diluição para uma placa de agar de 10 cm LB + carbenicilina (100 μg/L) pré-aquecida. Isto conduz a uma diluição 40.000-fold dos transformantes que serão usados para calcular a eficiência da transformação.
- Placa 400 μL de células recuperadas em cada placa em um total de 20 placas de agar de 15 cm LB + carbenicilina pré-aquecido. Incubar as placas por 14 – 16 h a 30 ° c.
Nota: o crescimento nesta temperatura mais baixa minimiza a recombinação entre as repetições de longo-terminal (LTR)18. - Para calcular a eficiência da transformação, conte o número de colônias na placa de diluição de 40.000 vezes (passo 1.3.2). Multiplique o número de colônias contados por 40.000 para obter o número total de colônias em todas as placas. Prossiga se o número total de colônias representa uma cobertura de biblioteca equivalente ao mínimo de colônias de 200x por sgRNA (a maioria de ideal é 500-1000x).
- Por exemplo, o número de colônia mínimo para TKOv3 biblioteca (71.090 sgRNA) é 1,4 x 107, que é equivalente a 200x colônias por sgrna. Se a representação da colônia for insuficiente, aumente o número de eletroporações na etapa 1,2 com base no número de colônias na placa de diluição para alcançar a cobertura mínima da biblioteca.
- Colha as colônias como descrito abaixo
- Para cada placa de 15 cm, adicionar 7 mL de LB + carbenicilina (100 μg/L) médio, em seguida, raspar as colônias fora com um espalhador de células. Com uma pipeta de 10 mL, transfira as células raspadas para um frasco ou garrafa de 1 L cônico estéril.
- Enxague mais uma vez a placa com 5 mL de LB + meio de carbenicilina e transfira a solução para a garrafa.
- Repita para todas as placas para as células da piscina de 20 placas em uma garrafa estéril.
- Misture as células coletadas com uma barra de agitação para 1 h à temperatura ambiente (RT) para quebrar os aglomerantes de células. Transfira as células para garrafas de centrifugação pré-pesadas e Centrifugue a 7.000 x g a bactérias da pelota e descarte a mídia.
- Pesar o pellet célula molhada e subtrair o peso do frasco de centrífuga para determinar o peso final da pelota molhada. Purify o ADN do plasmídeo usando um Maxi-ou o jogo Mega-Scale da purificação do plasmídeo dependendo da quantidade de pelota bacteriana cada coluna pode processar.
2. grande-escala CRISPR sgRNA biblioteca Lentivirus produção
Observação: todas as etapas nesta seção do protocolo são executadas em uma instalação BSL2 + em uma classe II, tipo a2 gabinete de biossegurança.
- Calcule o número de placas de 15 cm necessárias para a produção de vírus com base na estimativa de que 18 mL de vírus é normalmente colhidas a partir de 1 15 cm placa.
- Prepare as células para a transfecção semeando as células de empacotamento HEK-293T em meios de crescimento de baixo antibiótico (DMEM + 10% FBS + opcional: 0.1 x Pen/Strep) a 8 x 106 células por placa de 15 cm em 20 ml de mídia. Incubar células durante a noite a 37 ° c, 5% CO2. Assegure-se de que as pilhas chapeadas sejam 70%-80% confluente e espalhe uniformente no momento do transfection.
- No dia seguinte, prepare três misturas de plasmídeos de transfecção conforme descrito na tabela 1 para placas de 15 cm. Calcule a quantidade de plasmídeo necessária para um transfection e faça uma mistura dos plasmídeos para o número de placas, mais um a ser transfected.
- Prepare um reagente de transfecção à base de lipídios para cada transfecção, conforme descrito na tabela 2. Alíquota reduziu a mídia sérica em tubos de microcentrífuga individuais de 1,5 mL para o número de placas a serem transfectadas. Adicione o reagente do transfection, misture delicadamente, e incubar por 5 minutos em RT.
- Após a incubação de 5 minutos, adicione a quantidade de DNA exigida para uma transfection ao reagente do transfection para uma relação 3:1 do reagente-à-μg do transfection do complexo do ADN. Misture suavemente e incubar por 30 min em RT.
Nota: as transfecções subsequentes podem ser preparadas em conjuntos de cinco ou menos, com intervalos de 5 min para otimizar para o tempo e evitar a incubação. - Após 30 min de incubação, transfira cuidadosamente cada mistura de transfecção para cada placa de células de embalagem. Adicione a mistura inteira usando um gota gota da ponta da pipeta de 1 ml em um movimento circular, do ziguezague sem perturbar o monolayer da pilha. Incubar células a 37 ° c por 18 h a 5% CO2.
- Prepare a colheita viral Media: 500 mL de DMEM médio + 32 mL de BSA estoque (20 g/100 mL, dissolvido em DMEM, filtro esterilizado com 0,22 μm filtro) + 5 mL de 100x Pen/Strep.
- Após 18 h, remova a mídia (use o manuseio adequado de resíduos de Lentivirus, como incubação em 1% de hipoclorito de sódio por 30 minutos antes da eliminação). Substitua suavemente por 18 mL de meios de colheita virais a cada placa. Incubar células a 37 ° c por 18 h a 5% CO2.
- Após 24 h, verifique as células de empacotamento para a morfologia anormal e fundida como uma indicação da boa produção do vírus. Então, colha o Lentivirus coletando todo o sobrenadante e Transferindo em um tubo de centrifugação cônico estéril.
- Gire a mídia contendo vírus em 300 x g por 5 min e pellet as células de embalagem. Aliquot o sobrenadante em um tubo de polipropileno estéril sem perturbar o pellet.
- Armazene o vírus a 4 ° c por curtos períodos (menos de 1 semana) ou imediatamente a-80 ° c para armazenamento de longo prazo. Aliquot o vírus em grande escala Preps aos volumes do uso único para o armazenamento a longo prazo para evitar congelar-se/thawing.
3. Caracterização da linha celular para triagem
- Selecione a linha celular desejada.
- Meça e registre o tempo de duplicação aproximado das células.
- Determine a densidade óptima do chapeamento de pilha para cultivar pilhas cada 3 – 4 duplicações da pilha em uma embarcação da cultura do tecido da escolha (por exemplo, placas da cultura do tecido de 15 cm).
- Determine a concentração de puromicina para usar na linha celular desejada para a seleção de bibliotecas TKO contendo marcador de resistência da puromicina como segue:
- Células de semente em uma placa de 12 poços na densidade necessária para alcançar a confluência após 72 h, em seguida, incubar durante a noite (37 ° c, 5% CO2).
- No dia seguinte, mude para uma mídia contendo uma faixa de diluição de concentrações de puromicina de 0 μg/mL a 10 μg/mL, em incrementos de 0,5 μg/mL. Incubar as células para 48 h.
- Após 48 h, medir a viabilidade celular por contagem de células ou alamarBlue coloração.
- Determine a menor concentração que mata 100% das células em 48 h. Use esta concentração para selecionar para populações de células transadas da biblioteca CRISPR nas etapas 4,6 e 5.2.6.
Nota: para linhas celulares com tempos de duplicação mais longos, incubações mais longas com puromicina podem ser toleradas. Nessas situações, determine a curva de morte para o tempo de incubação necessário para as duagações de < 3 células. Minimize o tempo de seleção para evitar o abandono de genes essenciais antes do início da triagem.
- Verifique as células de sensibilidade ao brometo de hexadimethrine (até 8 μg/mL) realizando uma curva de resposta da dose no mesmo método utilizado para medir a sensibilidade da puromicina (passo 3,2). Se a toxicidade for observada com < 8 μg/mL de brometo de hexadimetrina, não utilize.
4. titulação funcional da biblioteca de Lentivirus CRISPR agrupada para determinação de MOI
- Descongele uma alíquota fresca de Lentivirus da biblioteca de CRISPR sgRNA agrupada (por exemplo, LCV2:: TKOv3) e mantenha no gelo.
- Projetar uma série de volumes de vírus para testar entre o intervalo de 0 – 2 mL (i.e., 0 mL, 0,25 mL, 0,5 mL, 1 mL e 2 mL).
- Colha células-alvo e células de semente em placas de 15 cm na densidade necessária para alcançar a confluência em 72 h.
- Para cada volume de vírus a ser testado, prepare placas duplicadas. Adicionar células, vírus, brometo de hexadimethrine (8 μg/mL), e meios a um volume final de 20 mL. Misture as placas completamente, sentar-se nível de placas na incubadora e incubar para 24 h (37 ° c, 5% CO2).
- Após 24 h, remova o vírus contendo meios de comunicação e descarte (use precauções de biossegurança para o manuseio de resíduos de Lentivirus). Opcionalmente, lave suavemente a placa com PBS quente para remover vírus estranhos.
- Para cada condição de vírus, substitua por 20 mL de mídia contendo puromicina usando a concentração determinada para matar células na seção 3, para uma placa de repetição. Para a outra placa, adicione 20 mL de mídia fresca sem puromicina. Incubar para 48 h (37 ° c, 5% CO2).
- Após 48 h, verifique se todas as células não infectadas (0 mL de condição de vírus) tratadas com puromicina estão mortas. Colha todas as chapas individualmente e dispersar as células por pipetagem suave repetida.
- Contagem de células de todas as placas e calcular o MOI para cada volume de vírus, comparando contagens de células com a seleção de puromicina para contagens de células sem puromicina (ou seja, +/-puromicina).
- Resultados do gráfico para determinar o volume de vírus que leva a 30% – 40% de sobrevida celular com seleção de puromicina versus sem puromicina. Use este volume do vírus para conseguir um MOI de 0.3-0.4 durante a tela as mesmas condições da cultura do tecido.
5. infecção preliminar da tela, seleção, e de da pilha
- Selecione a cobertura da biblioteca CRISPR sgRNA a ser mantida em toda a tela (mínimo recomendado de 200 vezes).
- Com base na cobertura da biblioteca, determine o número de células necessárias para manter essa cobertura por sgRNA e o número de células necessárias para a infecção no MOI 0,3 (tabela 3).
- Determine o número de placas necessárias para configurar a infecção (tabela 4).
- Infectando as células com a biblioteca CRISPR
- Colha células e semente o número de célula necessário para cada placa de 15 cm.
- Adicionar brometo de hexadimethrine (8 μg/mL) a todas as placas.
- Adicione o vírus no volume necessário para MOI 0,3 para triagem e as placas de controle 2. Para o controle 1, não adicione vírus e substitua esse volume pela mídia.
- Misture cuidadosamente as placas inclinando. Coloque as placas na incubadora, certificando-se de que estão nivelados.
Nota: as infecções do grupo podem ser feitas combinando uma mistura mestra do vírus, da mídia, e do brometo do hexadimethrine às pilhas na suspensão antes de chapeamento. - Retire a mídia e substitua com a mídia fresca contendo puromicina na concentração determinada na etapa 3.2.4 para a triagem e controle 1 placas 24 h após a infecção pelo vírus. Adicione meios frescos sem o puromicina à placa do controle 2. Incubar células para 48 h (37 ° c, 5% CO2).
- 48 h após a adição da puromicina, assegure-se de que todas as pilhas não infectadas estejam inoperantes (controle 1) para confirmar a atividade do puromicina, a seguir colhem as pilhas contaminadas.
- Colhendo população de células infectadas e de celular
- Colha as células selecionadas por puromicina de todas as placas de triagem em um recipiente estéril. Colete as células de cada placa de controle separadamente. Dispersar células por pipetagem repetida suave.
- Conte células de células de triagem agrupadas, controle 1 e controle 2 separadamente e calcule o número de células por 1 mL.
- Calcule MOI e cobertura de dobra alcançada da seguinte forma:
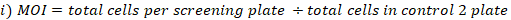
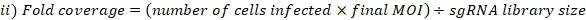
- Colete três repetições de pelotas da pilha das pilhas agrupadas na cobertura selecionada da biblioteca para a extração genomic do ADN. Centrifugue as células a 500 x g durante 5 min. Lave com PBS. Etiquetar os tubos e congelar-secar as pelotas de células a-80 ° c (Estas são amostras de referência T0).
- Divida o pool de células infectadas em três grupos replicados (por exemplo, replicar A, replicar B, replicar C), mantendo a cobertura da biblioteca dentro de cada replicação. Células de semente na mesma densidade de semeadura que normalmente seriam usadas ao expandirem-los. Use o mesmo número de células para cada placa replicadas e o mesmo número total de células entre réplicas.
- Continue a passagem de células e colher três repetições de pelotas de células de cada repetição de células infectadas em pool como acima, a cada 3 – 8 dias, dependendo da linha celular, para até 15 – 20 doublings celular. Em cada passagem, colher as células de todas as placas em cada grupo replicar uns com os outros (ou seja, todas as células de replicar as placas são re-misturados juntos, todas as células de replicar placas B são re-misturado juntos, etc.).
- Rotule cada pellet com um tempo (T) e replique a designação. Isto corresponde ao número de dias pós-T0 o pellet é coletado (por exemplo, T3_A, T3_B, T3_C, etc).
- Para as telas de drogas de seleção negativa, permita que as células se recuperem por pelo menos uma passagem após T0 antes do tratamento. Em T3 ou T6, divida as células de cada grupo replicado (A, B, C) em populações de tratamento e controle de drogas, usando a mesma densidade de semeadura usada na etapa 5.3.5.
- Agrupe separadamente o número de células necessárias para a cobertura da biblioteca para cada repetição no grupo de tratamento medicamentoso. Adicione a droga em concentrações intermediárias (IC20-IC50). Semeie as células e incubar (37 ° c, 5% CO2) até a próxima passagem.
- Agrupe separadamente o número de células necessárias para a cobertura da biblioteca para cada repetição no grupo de controle do veículo. Adicione o controle do veículo usando o mesmo volume que a droga (< 0.5% v/v). Semeie as células e incubar (37 ° c, 5% CO2) até a próxima passagem.
- Continue a passar as células e colher as pelotas de células para o DNA genómico a cada 3 dias, conforme descrito na etapa 5.3.5, enquanto atualizando a droga ou veículo em cada passagem.
- Para as telas positivas de seleção ou resistência a medicamentos, divida cada grupo replicada de acordo com o número de células necessárias para a cobertura da biblioteca. Adicionar IC90 concentrações de drogas para cada repetição. Na IC90, a maioria das células será morta. Permitir que populações resistentes cresçam e coletem pelotas de células (1 – 2 x 107 células) para extração de DNA genômica.
6. preparação e sequenciamento da amostra CRISPR
- Purificação do ADN de Genomic
- Incubar as pastilhas de células congeladas por 5 – 10 min em RT para descongelamento.
- Adicionar 1,4 mL de PBS a um tubo de centrifugação de 50 mL contendo um pellet de células. Vortex para 20 s para Ressuspender as células e descansar por 1 min. Se necessário, pipeta 15x com P1000 para separar os aglomerantes restantes da pilha. Se a transferência de células de um tubo de 15 mL ou 1,5 mL, suspender as células com 1 mL de PBS, em seguida, transferir as células para um tubo de 50 mL e enxaguar o tubo original com 400 μL de PBS.
- Adicionar 5 mL de solução de lise de núcleos às células ressoadas. Usando uma pipeta de 10 mL, misture a amostra pipetando para cima e para baixo 5x.
- Adicionar 32 μL de RNase A (20 mg/mL; para obter uma concentração final de 100 μg/mL) para o lisado nuclear e misturar a amostra invertendo o tubo 5x. Incubar a mistura a 37 ° c durante 15 min e permitir que a amostra esfrie por 10 min em RT.
- Adicionar 1,67 mL de solução de precipitação proteica ao lisado e ao vórtice vigorosamente durante 20 s. pequenos aglomerados de proteínas podem ser visíveis após a mistura.
- Centrífuga a 4.500 x g por 10 min em RT.
- Utilizando uma pipeta de 10 mL, transfira o sobrenadante para um tubo de centrifugação de 50 mL contendo 5 mL de isopropanol. Misture suavemente a solução 10x por inversão até que o DNA seja observado.
Nota: o DNA pode ser observado como linhas brancas, como fios que formam uma massa visível. - Centrifugador em 4.500 x g por 5 min em RT para pellet o DNA.
- Utilizando uma pipeta de 10 mL, Retire cuidadosamente o sobrenadante e evite desalojar o pellet de ADN. Adicione 5 mL de 70% de etanol em RT ao DNA. Gire delicadamente o tubo para lavar a pelota do ADN e os lados do tubo de centrifugação.
- Centrifugador a 4.500 x g por 5 min em RT.
- Usando uma pipeta de 10 mL, Retire cuidadosamente o etanol 70% e evite desalojar o pellet de DNA. DNA genómico a ar seco por 10 min em RT.
- Adicione 400 μL de solução TE ao tubo e deixe que o DNA se dissolva incubando a 65 ° c por 1 h. Misture o DNA gentilmente mexendo o tubo a cada 15 min. Se o DNA não se dissolver completamente, incubar tubo a 65 ° c por um adicional de 1 h enquanto suavemente flicking o tubo a cada 15 min, e deixá-lo a 4 ° c durante a noite.
- Centrifugador em 4.500 x g para 1 minuto no RT e transfira o ADN genomic a um tubo Low-binding de 1,5 ml.
- Quantificar e medir a pureza do DNA genómico no espectrofotômetro (para o teor de ácido nucleico total) e no fluorômetro (para o conteúdo de DNA de dupla fita).
- Opcionalmente, precipitar o ADN genomic se há umas edições com a amplificação a jusante do PCR do sgrna como segue.
- Transfira 400 μL de ADN genómico para um tubo de microcentrifugação de 1,5 mL.
- Adicionar 18 μL de NaCl de 5 M (concentração final de 0,2 M) e 900 μL de 95% de etanol.
- Inverta o tubo 10x até misturado completamente, a seguir Centrifugue em 16.000 x g por 10 minutos em RT.
- Retire cuidadosamente o sobrenadante e evite desalojar o pellet de ADN. Lave o pellet de DNA com 500 μL de 70% de etanol. Gire suavemente o tubo para lavar o pellet de DNA.
- Centrifugador a 16.000 x g por 5 min em RT.
- Remova cuidadosamente o sobrenadante e evite desalojar o pellet de DNA. DNA genómico a ar seco por 10 min em RT.
- Adicionar 300 μL de TE para dissolver o DNA conforme descrito nas etapas 6.1.12.
- Quantificar e medir a pureza do DNA genómico, conforme descrito na etapa 6.1.14.
- Preparação da biblioteca de sequenciamento CRISPR
- Configurar o PCR 1 conforme descrito na tabela 5 usando um total de 100 ΜG de DNA genómico. Adicionar 3,5 μg de ADN genómico por reacção de 50 μL e configurar reações idênticas de 50 μL para atingir a cobertura desejada. A tabela 6 lista exemplos de sequências de primer para a amplificação das bibliotecas de sequenciamento LCV2:: TKOv3. A tabela 7 lista exemplos de sequências de primer para amplificação de bibliotecas de sequenciamento pLCKO2:: TKOv3.
- Amplify as reações do PCR 1 em um termociclador usando o programa esboçado na tabela 8.
- Verifique a amplificação do PCR 1 executando 2 μL do produto do PCR em um gel de agarose de 1%. PCR 1 produz um produto de 600 BP.
- Pool todas as reações individuais de 50 μL para cada amostra de DNA genômica e misture por vortexing.
- Configure uma reação de PCR 2 (50 μL) para cada amostra, conforme descrito na tabela 9 usando 5 μl do produto PCR 1 agrupado como modelo. Use combinações exclusivas de primer de índice para cada amostra individual para permitir o agrupamento de amostras de biblioteca de sequenciamento.
- Amplificar a reação de PCR 2 em um termocicer usando o programa delineado na tabela 10.
- Limpe o equipamento do gel do agarose para purificante produtos amplificados com HCl 0,1 N por 10 minutos antes de moldar um gel. Prepare um gel do agarose de 2% que contem a mancha do ADN para purificante produtos amplificados do PCR 2.
- Funcione o produto do PCR 2 no gel do agarose de 2% na baixa tensão (1.0-1.5 h funcionado). PCR 2 produz um produto de 200 BP.
- Visualize os produtos do PCR em um transilluminator claro azul. Extirpar a banda de 200 BP e purificar o DNA da fatia de gel de agarose usando um kit de extração de gel. Quantificar e medir a pureza da biblioteca de sequenciamento tanto no espectrofotômetro quanto no fluorômetro.
Nota: uma concentração de biblioteca de sequenciamento gel-purificada típica varia de 5 – 10 ng/μL e um rendimento total de 150 – 300 ng.
- Sequenciamento de alta taxa de transferência
- Sequenciar as bibliotecas de sequenciamento CRISPR em seqüenciadores de próxima geração.
- Referência de sequência T0 amostras em maior profundidade de leitura de 400-para 500-dobra biblioteca cobertura. Sequenciar amostras de ponto de tempo experimentais para telas suspensas com uma profundidade de leitura mínima de 200 vezes. Para telas de seleção positivas fortes, um mínimo de profundidade de leitura de cobertura de 50 vezes é suficiente para a identificação de sgRNAs enriquecidas.
Nota: é crítico para sequenciar a amostra T0 para determinar a representação da biblioteca para uma tela específica e servir como uma referência para as alterações de dobra sgRNA determinando ao longo do tempo.
7. análise de dados
- Alinhe a sequência usando programas como o bowtie para mapear leituras de sequência para a biblioteca de referência usando os seguintes parâmetros:-v2 (permitindo duas incombinações) e-M1 (descartando qualquer leitura que mapeou para mais de uma seqüência na biblioteca).
- Normalize o número de leituras mapeadas exclusivamente para cada sgRNA para um determinado exemplo para 10 milhões leituras por exemplo da seguinte maneira:
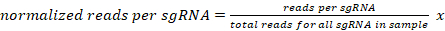 107 anos
107 anos
- Calcule a mudança log2 fold de cada sgRNA para cada repetição em cada ponto de tempo (TN) em comparação com a amostra T0 (TN/T0). Adicione uma contagem pseudo de 0,5 leituras para todas as contagens de leitura para evitar descontinuidades de zeros. Exclua sgRNAs com < 30 leituras brutas na amostra T0 do cálculo da dobra-mudança e da análise a jusante.
- Analise as alterações de dobra com a análise Bayesiana do algoritmo de essencialidade gênica (BAGEL) < https://github.com/Hart-Lab/bagel >, usando os conjuntos de treinamento essenciais e não essenciais definidos anteriormente19 para telas de essencialidade genética ( Tabela complementar S1) ou droz < https://github.com/Hart-Lab/Droz > para telas de drogas20.
- Calcule a precisão e a recordação para a avaliação de desempenho da tela usando contagens BF. Use o conjunto essencial da etapa 7,4 como a verdadeira lista positiva para a função precision_recall_curve da biblioteca Scikit-Learn para Python, juntamente com o subconjunto de Pontuação BF acima. Alternativamente, execute o mesmo usando o pacote PRROC em R.
- Calcule a mudança média da dobra de todas as guias para cada gene. Gere parcelas de densidade para os genes essenciais e não essenciais (ver passo 7,4) em R ou software equivalente. Em R, se x. ESS é um vetor que contém os valores de mudança de dobra de log de genes essenciais e x. nonEss contêm genes não essenciais, plotar usando o seguinte comando:
Plot (densidade (x. ESS), XLAB = "Mean logFC", Col = "Red", LWD = 2)
linhas (densidade (x. nonEss), Col = "azul", LWD = 2)
Observação: para obter detalhes e pacotes de versão do Python usados, consulte scikit-Learn v 0.19.1: (publicado por pedregosa et al.21).
Access restricted. Please log in or start a trial to view this content.
Resultados
Visão geral do fluxo de trabalho de triagem CRISPR em escala de genoma
A Figura 1 ilustra uma visão geral do fluxo de trabalho de triagem crispr agrupado, começando com a infecção de células-alvo com Lentivirus da biblioteca crispr em um moi baixo para garantir eventos de integração única e representação de biblioteca adequada (tipicamente 200-para 1000 -fold). Após a in...
Access restricted. Please log in or start a trial to view this content.
Discussão
Devido à sua simplicidade de uso e alta maleabilidade, a tecnologia CRISPR tem sido amplamente adotada como a ferramenta de escolha para a edição precisa do genoma. A triagem de CRISPR agrupada fornece um método para interrogar milhares de perturbações genéticas em um único experimento. Em telas agrupadas, as bibliotecas sgRNA servem como códigos de barras moleculares, pois cada sequência é única e é mapeada para o gene alvo. Isolando o ADN genomic da população da pilha, os genes que causam o phenotype do ...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não declaram interesses financeiros concorrentes.
Agradecimentos
Este trabalho foi apoiado pelo genoma do Canadá, o fundo de investigação de Ontário, e os institutos canadenses de pesquisa em saúde (MOP-142375, PJT-148802).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.22 micron filter | |||
| 30°C plate incubator | |||
| 37°C shaking incubator | |||
| 37°C, 5% CO2 incubator | |||
| 5 M NaCl | Promega | V4221 | |
| 50X TAE buffer | BioShop | TAE222.4 | |
| 6 N Hydrochloric acid solution | BioShop | HCL666.500 | |
| 95% Ethanol | |||
| Alamar blue | ThermoFisher Scientific | DAL1025 | |
| Blue-light transilluminator | ThermoFisher Scientific | G6600 | |
| Bovine Serum Albumin,Heat Shock Isolation, Fraction V. Min. 98%, Biotechnology grade | Bioshop | ALB001.250 | |
| Dulbecco's Modification of Eagles Medium | Life Technologies | 11995-065 | Cel culture media |
| Electroporation cuvettes | BTX | 45-0134 | |
| Electroporator | BTX | 45-0651 | |
| Endura electrocompetent cells | Lucigen | 90293 | |
| Fetal Bovine Serum | GIBCO | 12483-020 | |
| HEK293T packaging cells | ATCC | CRL-3216 | recommend passage number <15 |
| Hexadimethrine Bromide (Polybrene) | Sigma | H9268 | Cationic polymer to enhance transduction efficiency |
| Hexadimethrine Bromide (Polybrene) | |||
| LB agar plates with carbenicillin | |||
| LB medium with carbenicillin | |||
| Low molecular weight DNA ladder | New England Biolabs | N3233S | |
| Nanodrop spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| NEBNext Ultra II Q5 Master Mix | New England Biolabs | M0544L | |
| Opti-MEM | Life Technologies | 31985-070 | Reduced serum media |
| Plasmid maxi purification kit | Qiagen | 12963 | |
| pMD2.G (envelope plasmid) | Addgene | Plasmid #12259 | lentiviral system |
| psPAX2 (packaging plasmid) | Addgene | Plasmid #12260 | lentiviral system |
| Puromycin | Wisent | 400-160-UG | |
| QIAquick gel extraction kit | Qiagen | 28704 | |
| Qubit dsDNA BR assay | ThermoFisher Scientific | Q32853 | |
| Qubit fluorometer | ThermoFisher Scientific | Q33226 | |
| RNAse A | Invitrogen | 12091021 | |
| S.O.C recovery medium | Invitrogen | 15544034 | |
| SYRB Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| Toronto KnockOut CRIPSR library (TKOv3) - Cas9 included | Addgene | Addgene ID #90203 | Genome-wide CRISPR library , includes Cas9, 71,090 sgRNA |
| Toronto KnockOut CRIPSR library (TKOv3) - non-cas9 | Addgene | Addgene ID #125517 | Genome-wide CRISPR library, non-Cas9, 71,090 sgRNA |
| Tris-EDTA (TE) solution, pH8.0 | |||
| UltraPure agarose | ThermoFisher Scientific | 16500500 | |
| Wizard genomic DNA purification kit | Promega | A1120 | |
| X-tremeGENE 9 DNA transfection reagent | Roche | 06 365 809 001 | Lipid based transfection reagent |
Referências
- Jiang, F., Doudna, J. A. CRISPR-Cas9 Structures and Mechanisms. Annual Review of Biophysics. 46, 505-529 (2017).
- Baliou, S., et al. CRISPR therapeutic tools for complex genetic disorders and cancer (Review). International Journal of Oncology. 53 (2), 443-468 (2018).
- Hart, T., et al. High-Resolution CRISPR Screens Reveal Fitness Genes and Genotype-Specific Cancer Liabilities. Cell. 163 (6), 1515-1526 (2015).
- Morgens, D. W., Deans, R. M., Li, A., Bassik, M. C. Systematic comparison of CRISPR/Cas9 and RNAi screens for essential genes. Nature Biotechnology. 34 (6), 634-636 (2016).
- Evers, B., et al. CRISPR knock-out screening outperforms shRNA and CRISPRi in identifying essential genes. Nature Biotechnology. 34 (6), 631-633 (2016).
- Miles, L. A., Garippa, R. J., Poirier, J. T. Design, execution, and analysis of pooled in vitro CRISPR/Cas9 screens. The FEBS Journal. 283 (17), 3170-3180 (2016).
- Wang, T., Wei, J. J., Sabatini, D. M., Lander, E. S. Genetic screens in human cells using the CRISPR-Cas9 system. Science. 343 (6166), 80-84 (2014).
- Wang, T., et al. Identification and characterization of essential genes in the human genome. Science. 350 (6264), 1096-1101 (2015).
- Sanson, K. R., et al. Optimized libraries for CRISPR-Cas9 genetic screens with multiple modalities. Nature Communications. 9 (1), 5416(2018).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knock-out Screens. G3: Genes|Genomes|Genetics. 7 (8), 2719-2727 (2017).
- Mair, B., Tomic, J., et al. Essential gene profiles for human pluripotent stem cells identify uncharacterized genes and substrate dependencies. Cell Reports. 27 (2), 599-615 (2019).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knock-out screening in human cells. Science. 343 (6166), 84-87 (2014).
- Sharma, S., Petsalaki, E. Application of CRISPR-Cas9 Based Genome-Wide Screening Approaches to Study Cellular Signalling Mechanisms. International Journal of Molecular Sciences. 19 (4), (2018).
- Steinhart, Z., et al. Genome-wide CRISPR screens reveal a Wnt-FZD5 signaling circuit as a druggable vulnerability of RNF43-mutant pancreatic tumors. Nature Medicine. 23 (1), 60-68 (2017).
- Wang, T., et al. Gene Essentiality Profiling Reveals Gene Networks and Synthetic Lethal Interactions with Oncogenic Ras. Cell. 168 (5), 890-903 (2017).
- Zimmermann, M., et al. CRISPR screens identify genomic ribonucleotides as a source of PARP-trapping lesions. Nature. 559 (7713), 285-289 (2018).
- Deans, R. M., et al. Parallel shRNA and CRISPR-Cas9 screens enable antiviral drug target identification. Nature Chemical Biology. 12 (5), 361-366 (2016).
- Trinh, T. J. J., Bloom, F., Hirsch, V. STBL2: an Escherichia coli strain for the stable propagation of retroviral clones and direct repeat sequences. Focus. 16, 78-80 (1994).
- Hart, T., Moffat, J. BAGEL: a computational framework for identifying essential genes from pooled library screens. BMC Bioinformatics. 17, 164(2016).
- Wang, G. Z. M., et al. Identifying drug-gene interactions from CRISPR knock-out screens with drugZ. bioRxiv. , Available from: https://doi.org/10.1101/232736 (2017).
- Pedregosa, F. V., G,, et al. Scikit-learn: Machine Learning in Python. Journal of Machine Learning Research. 12, 2825-2830 (2011).
- Ketela, T., et al. A comprehensive platform for highly multiplexed mammalian functional genetic screens. BMC Genomics. 12, 213(2011).
- Doench, J. G. Am I ready for CRISPR? A user's guide to genetic screens. Nature Review Genetics. 19 (2), 67-80 (2018).
- Hartenian, E., Doench, J. G. Genetic screens and functional genomics using CRISPR/Cas9 technology. FEBS Journal. 282 (8), 1383-1393 (2015).
- Li, W., et al. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knock-out screens. Genome Biology. 15 (12), 554(2014).
- Sheel, A., Xue, W. Genomic Amplifications Cause False Positives in CRISPR Screens. Cancer Discovery. 6 (8), 824-826 (2016).
- Meyers, R. M., et al. Computational correction of copy number effect improves specificity of CRISPR-Cas9 essentiality screens in cancer cells. Nature Genetics. 49 (12), 1779-1784 (2017).
- Henser-Brownhill, T., Monserrat, J., Scaffidi, P. Generation of an arrayed CRISPR-Cas9 library targeting epigenetic regulators: from high-content screens to in vivo assays. Epigenetics. 12 (12), 1065-1075 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados