A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تحضير عينة من وعاء واحد بمساعدة الناقل لتحليل البروتينات المستهدفة لأعداد صغيرة من الخلايا البشرية
In This Article
Summary
يعد تحضير عينة من وعاء واحد بمساعدة حامل البروتين إلى جانب الكروماتوغرافيا السائلة (LC) - مراقبة التفاعل المختار (SRM) المسمى cLC-SRM طريقة ملائمة لتحليل البروتينات المستهدفة متعددة الإرسال لأعداد صغيرة من الخلايا ، بما في ذلك الخلايا المفردة. إنه يستفيد من استخدام البروتين الخارجي المفرط كناقل وعالي الخصوصية LC-SRM للقياس الكمي المستهدف.
Abstract
يتم تحقيق تحليل البروتين لأعداد صغيرة من الخلايا البشرية بشكل أساسي عن طريق البروتينات المستهدفة باستخدام المقايسات المناعية القائمة على الأجسام المضادة ، والتي لها قيود متأصلة (على سبيل المثال ، تعدد الإرسال المنخفض وعدم توفر الأجسام المضادة للبروتينات الجديدة). ظهرت البروتينات المستهدفة القائمة على قياس الطيف الكتلي (MS) كبديل لأنها خالية من الأجسام المضادة ، وتعدد إرسال عالية ، ولها دقة عالية في التحديد والكمي. التطورات الحديثة في أجهزة MS تجعل البروتينات المستهدفة القائمة على MS ممكنة للقياس الكمي المتعدد للبروتينات الوفيرة للغاية في الخلايا المفردة. ومع ذلك ، هناك تحد تقني للمعالجة الفعالة للخلايا المفردة مع الحد الأدنى من فقدان العينة لتحليل مرض التصلب المتعدد. لمعالجة هذه المشكلة ، قمنا مؤخرا بتطوير مستحضر عينة مناسب بمساعدة حامل البروتين إلى جانب الكروماتوغرافيا السائلة (LC) - مراقبة التفاعل المختار (SRM) المسمى cLC-SRM لتحليل البروتينات المستهدفة لأعداد صغيرة من الخلايا البشرية. تستفيد هذه الطريقة من استخدام البروتين الخارجي المفرط المركب كناقل ومعالجة وعاء واحد منخفضة الحجم لتقليل خسائر امتصاص السطح بشكل كبير بالإضافة إلى LC-SRM عالي الجودة لمعالجة نطاق التركيز الديناميكي المتزايد بشكل فعال بسبب إضافة البروتين الناقل الخارجي. وقد أثبتت فائدته من خلال القياس الكمي الدقيق لمعظم البروتينات الوفيرة بشكل معتدل في أعداد صغيرة من الخلايا (على سبيل المثال ، 10-100 خلية) والبروتينات الوفيرة للغاية في الخلايا المفردة. تجعل الميزات سهلة التنفيذ وعدم الحاجة إلى أجهزة محددة هذه الطريقة في متناول معظم مختبرات البروتينات. لقد قدمنا هنا بروتوكولا مفصلا لتحليل cLC-SRM لأعداد صغيرة من الخلايا البشرية بما في ذلك فرز الخلايا ، وتحلل الخلايا وهضمها ، وتحليل LC-SRM ، وتحليل البيانات. هناك حاجة إلى مزيد من التحسينات في حساسية الكشف وإنتاجية العينة من أجل تحليل البروتينات أحادية الخلية المستهدفة. نتوقع أن يتم تطبيق cLC-SRM على نطاق واسع في البحوث الطبية الحيوية وبيولوجيا الأنظمة مع إمكانية تسهيل الطب الدقيق.
Introduction
تسمح التطورات التكنولوجية الحديثة في علم الجينوم (النسخ) بإجراء تحليل شامل ودقيق للجينوم (النسخ) في الخلايا المفردة1،2،3. ومع ذلك ، فإن تقنيات البروتينات أحادية الخلية متخلفة كثيرا ولكنها لا تقل أهمية عن تقنيات الجينوم (النسخ)4،5،6،7،8. علاوة على ذلك ، لا يمكن بالضرورة استنتاج وفرة البروتين من وفرة mRNA9 ، والبروتيوم أكثر تعقيدا وديناميكية من النسخ10. بالنظر إلى هذه التحديات ، يتم استخدام عدد كبير من المجموعات المختلطة من الخلايا (أي الخلايا السائبة) بشكل عام لتوليد بيانات بروتين شاملة11،12،13. ومع ذلك ، فإن مثل هذه القياسات السائبة تشير إلى متوسط الاختلافات العشوائية للخلايا الفردية ، وبالتالي تحجب التباين المهم من خلية إلى أخرى (أي عدم تجانس الخلية) 4،14. تصبح قيود مثل هذه القياسات المجمعة أكثر حدة عندما تمثل الخلايا ذات الأهمية جزءا صغيرا فقط من إجمالي مجموعات الخلايا (على سبيل المثال ، الخلايا الجذعية السرطانية داخل الأورام في مرحلة مبكرة من السرطان). لذلك ، هناك فجوة معرفية كبيرة بين البروتينات أحادية الخلية وعلم الجينوم (النسخ).
تستخدم المقايسات المناعية القائمة على الأجسام المضادة (على سبيل المثال ، التدفق أو قياس الكتلة الخلوية) في الغالب للتحليل البروتيني المستهدف للخلايا المفردة6،7،15،16،17،18. ومع ذلك ، فإنهم يعانون من تعدد الإرسال المنخفض ، والخصوصية المحدودة ، وعدم توفر الأجسام المضادة للبروتينات الجديدة ذات الأهمية. برزت البروتينات المستهدفة القائمة على قياس الطيف الكتلي (MS) كبديل لقياس البروتين بدقة نظرا لكونها خالية من الأجسام المضادة ، وتعدد الإرسال العالي (≥150 بروتينا في تحليلواحد 19) ، ودقة القياس الكمي العالية (الكميات أو التركيزات المطلقة) ، والخصوصية العالية وقابلية التكاثر (≤10٪ CV) 20،21،22،23. جعل التقدم الكبير الأخير في تحضير العينات البروتينات أحادية الخلية القائمة على التصلب العصبي المتعدد ممكنة للتحليل الكمي للبروتينات الوفيرة للغاية من الخلايا البشرية المفردة. ومع ذلك ، لا تزال البروتينات أحادية الخلية القائمة على التصلب العصبي المتعدد في مرحلة الطفولة المبكرة. على سبيل المثال ، يمكن لمنصة MS الأكثر تقدما إلى جانب معدلات تدفق RPLC ذات التدفق المنخفض للغاية أن تسمح فقط باكتشاف MS الخالي من الملصقات وتقدير كمية ~ 670-870 بروتين من إجمالي ≥12,000 بروتين في خلايا HeLa المفردة24,25.
حاليا ، هناك ستة مناهج للبروتينات أحادية الخلية قائمة على MS متاحة لتحليل خلايا الثدييات المفردة ، منها أربعة للبروتينات العالمية (nanoPOTS: تحضير قائم على nanowell في وعاء واحد لعينات التتبع26 ؛ iPAD-1: جهاز تحليل البروتين المتكامل لتحليل الخليةالمفردة 27 ؛ OAD (قطرة الزيت والهواء والقطرات) التحليل البروتيني أحادي الخلية القائمعلى الرقاقة 28 ؛ SCoPE-MS: البروتينات أحادية الخلية عن طريق قياس الطيف الكتلي29) والاثنان الآخران للبروتينات المستهدفة (cLC-SRM: كروماتوغرافيا السائل بمساعدة الموجة الحاملة (LC) - مراقبة التفاعل المختار (SRM) 30 ؛ cPRISM-SRM: ضغط عال بمساعدة الموجة الحاملة ، وفواصل عالية الدقة مع اختيار ذكي وتعدد إرسال مقترن ب SRM31). ومع ذلك ، فإن كل هذه الأساليب لها عيوب فنية. nanoPOTS و iPAD-1 و OAD حجم معالجة العينات المصغر إلى 2-200 nL وليست جاهزة للتطبيقات العريضة26،27،28. بالنسبة إلى SCoPE-MS ، تتم إضافة حامل TMT (علامة الكتلة الترادفية) بعد المعالجة أحادية الخلية ، لذلك لا يمكنه منع خسائر الامتزاز السطحي بشكل فعال أثناء معالجة العينة عند استخدام أنبوب واحد للمعالجة أحادية الخلية29 ، مما يؤدي إلى انخفاض قابلية التكرار مع معامل ارتباط ~ 0.2-0.4 فقط بين التكرارات32. بالنسبة إلى cLC-SRM و cPRISM-SRM ، يعد استخدام البروتينات الخارجية كناقل أكثر ملاءمة للبروتينات المستهدفة لأن الببتيدات من البروتينات الخارجية المفرطة يتم تسلسلها بشكل متكرر بواسطة MS / MS ، مما يقلل بشكل كبير من فرصة تسلسل الببتيدات الداخلية المنخفضة الوفرة30،31. على عكس البروتينات العالمية للقياس الكمي النسبي ، يمكن أن يوفر نهجان البروتينات المستهدفان تحليلا دقيقا أو مطلقا للبروتين لأعداد صغيرة من الخلايا ذات قابلية التكاثر العالية باستخدام معايير داخلية تحمل علامة النظائر الثقيلة بتركيزات معروفة. عند مقارنتها ب cPRISM-SRM التي تتطلب تجزئة PRISM سابقة عالية الدقة ، مما ينتج عنه العديد من عينات الكسور التي تحتاج إلى تحليل ، تتمتع cLC-SRM بميزة كبيرة في إنتاجية العينة دون تجزئة ويمكنها في نفس الوقت تحديد مئات البروتينات في تحليل واحد ولكن بحساسية كشف أقل نسبيا30. لذلك ، يمكن الوصول إلى cLC-SRM بشكل أكبر ويجب أن يكون له أدوات مساعدة أوسع لتحليل البروتين متعدد الإرسال الدقيق لأعداد صغيرة من الخلايا بالإضافة إلى العينات المحدودة الكتلة.
نصف هنا بروتوكولا مفصلا لإجراء cLC-SRM لتحليل البروتينات المستهدفة الملائمة لأعداد صغيرة من الخلايا البشرية ، بما في ذلك الخلايا المفردة. يتكون البروتوكول من الخطوات الرئيسية التالية: فرز الخلايا بواسطة FACS (فرز الخلايا المنشطة بالفلورة) ، وتحليل الخلايا والهضم المعالج في أنابيب تفاعل البوليميراز المتسلسل أحادي الحجم (PCR) منخفضة الحجم ، وجمع بيانات LC-SRM ، وتحليل بيانات SRM باستخدام برنامج Skyline المتاح للجمهور (الشكل 1). تم إثبات فائدته الواسعة جنبا إلى جنب مع فحوصات SRM الراسخة سابقا من خلال القياس الكمي المستهدف المطلق لبروتينات مسار EGFR / MAPK في خلايا 1-100 MCF7 أو MCF10A وتحديد نسخ بروتين المسار لكل خلية في نطاق ديناميكي واسع من التركيزات30. نتوقع أنه من خلال البروتوكول التفصيلي ، يمكن لمعظم الباحثين في علم البروتينات بسهولة تنفيذ cLC-SRM في مختبراتهم لتحليل البروتين الدقيق للعينات فائقة الحجم (على سبيل المثال ، الخلايا السرطانية النادرة) لتلبية احتياجات مشروعهم.
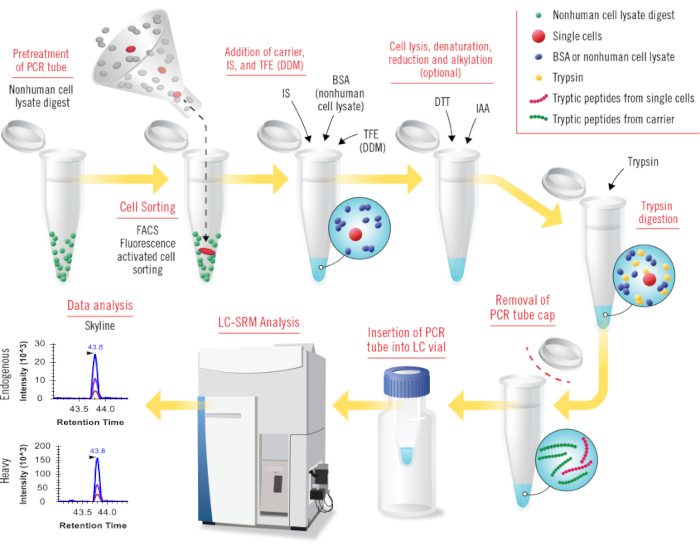
الشكل 1: نظرة عامة على جميع الخطوات في cLC-SRM (جإعداد عينة بمساعدة وعاء واحد إلى جانب liquid chromatography - sمختارة reaction monitoring). تستخدم هضم محللات الخلايا غير البشرية (على سبيل المثال ، Shewanella oneidensis) للمعالجة المسبقة لأنابيب تفاعل البوليميراز المتسلسل لطلاء سطح الأنبوب. يتم جمع أعداد صغيرة من الخلايا البشرية أو الخلايا المفردة التي تم فرزها بواسطة FACS في أنابيب PCR المعالجة مسبقا. يتم إضافة بروتين BSA (أو بروتين محلل الخلية غير البشرية) ، والمعيار الداخلي الثقيل (IS) ، و TFE (أو DDM) إلى أنابيب العينة بالتتابع لتسهيل تحلل الخلايا وتقليل خسائر امتصاص السطح. من الناحية المفاهيمية ، سيعمل حامل البروتين DDM والخلية غير البشرية بشكل جيد ل cLC-SRM. يتم إجراء تحلل الخلية عن طريق صوتنة ، ويتم تحقيق تمسخ البروتين عن طريق التسخين عند درجة حرارة عالية. تستخدم كواشف DTT و IAA للاختزال والألكلة ، على التوالي (هذه الخطوة اختيارية). يضاف التربسين للهضم بنسب أعلى بكثير من التربسين على كمية البروتين مقارنة بهضم التربسين القياسي. تتم إزالة غطاء أنبوب العينة ثم يتم إدخال أنبوب PCR في قارورة LC لتحليل LC-SRM المباشر. يتم تحليل بيانات SRM التي تم جمعها باستخدام برنامج Skyline المتاح للجمهور. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Access restricted. Please log in or start a trial to view this content.
Protocol
ملاحظة: يظهر تحليل cLC-SRM خطوة بخطوة في الشكل 1.
1. المعالجة المسبقة لأنابيب PCR
- أضف 100 ميكرولتر من هضم محللات الخلايا غير البشرية (على سبيل المثال ، Shewanella oneidensis) عند 0.2 ميكروغرام / ميكرولتر إلى أنابيب تفاعل البوليميراز المتسلسل (PCR). احتضن في درجة حرارة الغرفة طوال الليل لتغطية سطح أنبوب PCR.
- قم بإزالة هضم محللات الخلية عن طريق سحب العينة ، واشطف أنابيب PCR بماء HPLC 3 مرات ، ثم قم بتجفيف أنابيب PCR بالهواء في غطاء الدخان.
- قم بتخزين أنابيب PCR المطلية في فريزر 4 درجات مئوية حتى الاستخدام الآخر.
2. فرز FACS
- قم بمحاذاة فارز الخلايا المنشط بالفلورة (FACS) (انظر جدول المواد) في لوحة PCR سعة 96 بئرا باستخدام حبات الفلورسنت مع الأصباغ التي يمكن أن يثيرها مصدر الليزر لتوفير إشارات مرجعية موثوقة.
- تحقق من أن جهاز FACS موجه جيدا لفرز الخلايا في الجزء السفلي من لوحات PCR.
- اضبط زاوية تيار الفرز على الحد الأدنى الممكن لزيادة معدل النجاح الذي سيتم فرز الخلايا في قيعان الآبار بدلا من ضرب الجوانب.
- لتوجيه الجهاز ، استخدم دفق الاختبار "لفرز" قطرات المخزن المؤقت PBS (على سبيل المثال ، 100 قطرة) على بئر صفيحة فارغة للتأكد من أن القطرات المجمعة في الأسفل. إذا لم تهبط القطرات في الموضع الصحيح ، فأعد معايرة فارز الخلايا وكرر ذلك حتى يتم جمع القطرات في تيار الفرز بشكل صحيح.
- ضع أنابيب PCR المطلية في رف أنبوب PCR سعة 96 بئرا.
- قم بفرز الأعداد المرغوبة من خلايا الثدي البشرية (MCF7 أو MCF10A) في أنابيب PCR مطلية مسبقا.
- قم على الفور بالطرد المركزي لخلايا الثدي التي تم فرزها عند 100 × جم لمدة 10 دقائق عند 4 درجات مئوية لإبقائها في قاع الأنبوب لتجنب فقدان الخلايا المحتمل.
- قم بتخزين الخلايا التي تم فرزها في فريزر -80 درجة مئوية حتى مزيد من التحليل.
3. إضافة حامل البروتين ، والمعايير الداخلية الثقيلة ، و TFE
- أضف 4 ميكرولتر من 25 ملي مولار NH4HCO3 إلى أنابيب تفاعل البوليميراز المتسلسل التي تحتوي على خلايا مجمعة.
- أضف 1 ميكرولتر من 10 نانوغرام / ميكرولتر من ألبومين مصل الأبقار (BSA) في 25 ملي مولار NH4HCO3 في أنابيب العينة.
- أضف 0.3 ميكرولتر من معايير الببتيد الثقيلة الخام 100 fmol / μL (إجمالي 30 fmol) في أنابيب العينة.
ملاحظة: يتم تصنيع الحصص المستعارة من معايير الببتيد الثقيلة الخام لبروتينات مسار EGFR / MAPK بتركيز اسمي يبلغ 100 fmol / μL في 25 ملي مولار NH4HCO3. قم بتوزيع الحجم الصغير من معايير الببتيد الثقيلة الخام عند 100 fmol / μL في منتصف المحلول وتأكد من عدم وجود سائل متبقي في طرف الماصة. - أضف 9 ميكرولتر من 100٪ TFE إلى أنابيب العينة بتركيز نهائي ~ 60٪ TFE.
- جهاز طرد مركزي عند 1500 × جم لمدة 5 دقائق ثم دوامة برفق عند 100 × جم لمدة 3 دقائق.
4. تحلل الخلايا وتمسخ البروتين
- خلايا الثدي التي تم فرزها بواسطة SONICATE FACS على الجليد لمدة 1 دقيقة مع فواصل زمنية مدتها 1 دقيقة لما مجموعه 5 دورات بسعة 70٪ و 0.5 هرتز.
- احتضان العينات عند 90 درجة مئوية لمدة ساعة واحدة لتمسخ البروتين باستخدام جهاز تدوير حراري PCR مع خيار الغطاء المسخن.
ملاحظة: TFE هو مذيب متطاير بدرجة غليان تبلغ 74 درجة مئوية ، وبالتالي يتم تحديد خيار الغطاء الساخن لتجنب التجفيف الكامل مع فقدان العينة بشكل كبير. - قم بتبريد العينات إلى درجة حرارة الغرفة باستخدام الطرد المركزي عند 1500 × جم لمدة 3 دقائق.
5. الاختزال والألكلة (اختياري)
- أضف 0.6 ميكرولتر من 50 ملي مولار DTT للتركيز النهائي البالغ 2 ملليمتر.
ملاحظة: يجب تحضير حل DTT طازجا في كل مرة. تزن 77.5 مجم من DTT ثم تذوب في 1 مل من 25 ملي NH4HCO3 لصنع 500 ملي DTT. ثم يضاف 10 ميكرولتر من 500 ملي مولار DTT إلى 90 ميكرولتر من 25 ملي مولار NH4HCO3 لصنع 50 ملي مولار DTT. - جهاز طرد مركزي عند 1500 × جم لمدة 5 دقائق ثم اخلطه برفق عند 850 دورة في الدقيقة لمدة 3 دقائق.
- احتضان عند 56 درجة مئوية لمدة 1 ساعة مع رج لطيف عند 100 × جم.
- قم بتبريد العينات إلى درجة حرارة الغرفة وأجهزة الطرد المركزي عند 1500 × جم لمدة 3 دقائق.
- أضف 0.5 ميكرولتر من 60 ملي مولار IAA إلى أنبوب RT-PCR بتركيز نهائي ~ 2 ملليمتر.
ملاحظة: يجب إعداد حل IAA حديثا في كل مرة. تزن 74.3 مجم من IAA ثم تذوب في 1 مل من 25 ملي NH4HCO3 لصنع 400 ملي مولار IAA. يضاف 15 ميكرولتر من 400 ملي مولار IAA إلى 85 ميكرولتر من 25 ملي مولار NH4HCO3 لصنع 60 ملي مولار IAA. - احتضن في الظلام في درجة حرارة الغرفة لمدة 30 دقيقة مع رج لطيف عند 100 × جم.
ملاحظة: تكون خطوات الاختزال والألكلة اختيارية عندما لا تكون هناك ببتيدات تحتوي على السيستين مختارة للبروتينات المستهدفة.
6. هضم التربسين
- قلل حجم العينة إلى ~ 4 ميكرولتر باستخدام مكثف SpeedVac.
ملاحظة: TFE هو مذيب متطاير بدرجة غليان عند 74 درجة مئوية. نسبة عالية من TFE تمنع هضم التربسين. - أضف 9 ميكرولتر من 25 ملي مولار NH4HCO3 و 1-3 ميكرولتر من 15 نانوغرام / ميكرولتر من التربسين للهضم بتركيز التربسين النهائي 1-3 نانوغرام / ميكرولتر.
ملاحظة: بالنسبة ل 10 خلايا ثدي بشرية مصنفة بواسطة FACS (~ 1 نانوغرام) ، تكون نسبة إنزيم التربسين على البروتين ≥15: 1 ، أي أعلى بمقدار 750 ضعفا من تلك الموجودة في هضم التربسين القياسي (نسبة 1:50). - تخلط برفق عند 100 × جم لمدة 3 دقائق وتحتضن طوال الليل (~ 16 ساعة) عند 37 درجة مئوية.
- أضف 0.5 ميكرولتر من حمض الفورميك 5٪ لإيقاف التفاعل الأنزيمي.
- جهاز طرد مركزي لمدة 1 ساعة عند 1500 × جم.
- يحفظ في فريزر -80 درجة مئوية حتى مزيد من تحليل LC-SRM أو في فريزر 4 درجات مئوية للتحليل الفوري.
7. التحضير لتحليل LC-SRM المباشر
- قم بإزالة غطاء أنبوب تفاعل البوليميراز المتسلسل الذي تم استخدامه لجمع (الخطوة 2) ومعالجة (الخطوة 6) لخلايا الثدي التي تم فرزها بواسطة FACS.
- أدخل أنبوب PCR في قارورة LC.
- أغلق قارورة LC لتحليل LC-SRM.
ملاحظة: بالنسبة لأعداد صغيرة من الخلايا التي تمت معالجتها في لوحة PCR ذات 96 بئرا ، ستتم إضافة غطاء الحصيرة لإغلاق اللوحة لتحليل LC-SRM المباشر.
8. تحليل LC-SRM
- تحليل العينات باستخدام UPLC مقترنا بمطياف كتلة رباعي الأقطاب ثلاثي.
- قم بتوصيل الأعمدة الشعرية C18 (75 ميكرومتر هوية × 20 سم للتدرج القياسي و 100 ميكرومتر هوية × 10 سم للتدرج القصير) بباعث رش كهربائي من السيليكا محفور كيميائيا بحجم 20 ميكرومتر من السيليكا المنصهر عبر اتحاد معدني مقاوم للصدأ.
- استخدم حلقة عينة سعة 20 ميكرولتر لتحميل كل العينة مباشرة (~ 15 ميكرولتر في المجموع) في عمود LC لزيادة حساسية الكشف.
- استخدم حمض الفورميك 0.1٪ في الماء و 0.1٪ حمض الفورميك في 90٪ أسيتونيتريل كمرحلتين متنقلتين A و B لفصل RPLC الشعري.
- استخدم التدرج الثنائي LC بمعدلات تدفق تبلغ 300 نانولتر / دقيقة للحصول على تدرج قياسي عند قياس أعداد كبيرة من البروتينات المستهدفة (>10). التدرج القياسي: 5-20٪ B في 26 دقيقة ، 20-25٪ B في 10 دقائق ، 25-40٪ B في 8 دقائق ، 40-95٪ B في دقيقة واحدة و 95٪ B لمدة 7 دقائق لمدة 52 دقيقة والعمود التحليلي إعادة التوازن عند 99.5٪ A لمدة 8 دقائق.
- استخدم التدرج الثنائي LC بمعدلات تدفق تبلغ 400 نانولتر / دقيقة للحصول على تدرج قصير عند قياس أعداد صغيرة من البروتينات المستهدفة (≤10). تدرج قصير للفصل السريع: 5-95٪ B في 5 دقائق وإعادة توازن العمود التحليلي عند 99.5٪ A لمدة 5 دقائق مع إجمالي وقت تشغيل LC يبلغ 10 دقائق.
- قم بتشغيل مطياف الكتلة QQQ بجهد رش أيوني يبلغ 2,400 ± 100 فولت ، وجهد إزاحة شعري يبلغ 35 فولت ، وجهد إزاحة مقشدة يبلغ -5 فولت ودرجة حرارة مدخل شعري تبلغ 220 درجة مئوية.
- احصل على معلمات QQQ MS الأخرى من الضبط والمعايرة التلقائية دون مزيد من التحسين.
- قم برفع تركيزات مختلفة من معايير الببتيد الثقيلة الخام إلى 20-50 نانوغرام / ميكرولتر من هضم محللات الخلايا غير البشرية قبل تحليل LC-SRM للعينات لتحديد وقت الاحتفاظ بالببتيد LC (RT) لبناء طريقة SRM مجدولة وتحولات التداخل المحتملة من أجل القياس الكمي الدقيق.
- استخدم وضع SRM المجدول RT للقياس الكمي المتعدد مع نافذة المسح الضوئي التي تبلغ ≥6 دقائق.
ملاحظة: سيتم ضبط نافذة المسح وفقا لذلك اعتمادا على عدد الانتقالات في نافذة زمنية معينة. - قم بتعيين إجمالي وقت الدورة إلى 1 ثانية، ويتم ضبط وقت المكوث لكل انتقال تلقائيا بناء على عدد الانتقالات التي تم مسحها ضوئيا في فترات زمنية مختلفة للاستبقاء. هناك حاجة إلى وقت مكوث لا يقل عن 10 مللي ثانية لكل انتقال SRM. يمكن مراقبة جميع البروتينات المستهدفة في وقت واحد في تحليل LC-SRM واحد
9. تحليل البيانات
- قم باستيراد ملفات البيانات الأولية إلى برنامج Skyline33 المتاح للجمهور لتصور البيانات لكل ببتيد مستهدف لتحديد قابليتها للكشف.
- استخدم معيارين لتحديد اكتشاف ذروة SRM وتكاملها: 1) نفس وقت الاحتفاظ بLC ؛ 2) تقريبا نفس نسب كثافة ذروة SRM النسبية عبر انتقالات متعددة بين الببتيدات الداخلية (الخفيفة) ومعايير الببتيد الداخلية (الثقيلة) المقابلة لها (الثقيلة) المسماة بالنظائر
- افحص جميع بيانات SRM يدويا للتأكد من تخصيص ذروة SRM الصحيح وحدود ذروة SRM للقياس الكمي الموثوق به باستخدام المعيارين المذكورين أعلاه.
- استخدم أفضل انتقال مع إشارة SRM عالية ولكن بدون تدخل مصفوفة لقياس كمية البروتين ، واستخدم التحولات الأخرى كمرجع.
- استخدم تكرارات متعددة للحصول على الانحراف المعياري (SD) ومعامل التباين (CV).
- احسب نسبة الإشارة إلى الضوضاء (S/N) بواسطة شدة ذروة القمة على متوسط ضوضاء الخلفية ضمن منطقة زمنية للاحتفاظ بمقدار ±15 ثانية للتدرج القياسي و ±6 ث للتدرج القصير. يتم تعريف حد الكشف (LOD) وحد القياس الكمي (LOQ) على أنهما أدنى نقاط تركيز تكون عندها نسبة S / N 3 و 7 على الأقل ، على التوالي.
- قم بتطبيق معيار إضافي واحد لتحديد قيم LOQ بشكل متحفظ إلى جانب S / N ≥7: يجب أن تكون استجابة الببتيد البديل ضد أرقام الخلايا المختلفة ضمن النطاق الديناميكي الخطي.
- استخدم برنامج Excel لرسم جميع منحنيات المعايرة. قم بتحميل بيانات RAW من QQQ MS في برنامج Skyline المتاح للجمهور لعرض الرسوم البيانية للكروماتوجرامات الأيونية المستخرجة (XICs) للتحولات المتعددة للبروتينات المستهدفة التي تمت مراقبتها.
Access restricted. Please log in or start a trial to view this content.
النتائج
تم استخدام كميات صغيرة من محللات الخلايا MCF7 (0.5-20 نانوغرام أي ما يعادل 5-200 خلية) لأول مرة لتقييم أداء cLC-SRM عن طريق القياس الكمي المستهدف لبروتينات مسار EGFR / MAPK لأنها أكثر اتساقا مع اختلافات أقل عند مقارنتها بأعداد صغيرة من الخلايا التي تم فرزها بواسطة FACS. ك...
Access restricted. Please log in or start a trial to view this content.
Discussion
cLC-SRM هي طريقة بروتينات مستهدفة مريحة تتيح تحليلا دقيقا للبروتين متعدد الإرسال لأعداد صغيرة من الخلايا بما في ذلك الخلايا المفردة. تستفيد هذه الطريقة من تحضير عينة من وعاء واحد بمساعدة حامل البروتين ، حيث يتم تنفيذ جميع الخطوات بما في ذلك جمع الخلايا ، وتحلل الخلايا متعد?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
المؤلفون ليس لديهم ما يكشفون عنه.
Acknowledgements
تم دعم هذا العمل من قبل منح المعاهد الوطنية للصحة R21CA223715 (TS) و UG3CA256967 (TS و HL). تم تنفيذ العمل التجريبي الموصوف هنا في مختبر العلوم الجزيئية البيئية ، المختبر الوطني لشمال غرب المحيط الهادئ ، وهو مرفق علمي وطني للمستخدمين ترعاه وزارة الطاقة الأمريكية بموجب عقد DE-AC05-76RL0 1830.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 2 mL glass LC vial | Microsolv | 9502S-WCV | Vessel to hold PCR tube for autosample injection |
| BSA | Sigma-Aldrich | P0834-10×1mL | Carrier protein for greatly reducing surface adsorption losses |
| DTT | Thermo Scientific | A39255 | Reagent for reduction |
| Formic acid | Thermo Scientific | 28905 | Reagent for stopping enzyme reaction |
| IAA | Thermo Scientific | A39271 | Reagent for alkylation |
| Peptide internal standards | New England peptide | Targeted quantification of EGFR/MAPK pathway proteins | |
| RT-PCR tube | GeneMate Bioexpress | T-3035-1 | 0.2 mL PCR tube for one-pot sample preparation |
| Skyline software | University of Washington | Publicly available for SRM data analysis | |
| Sonicator | Hielscher Ultrasound Technology | UTR200 | Sonication on ice for cell lysis |
| Speed Vac concentrator | Thermo Scientific | Reduction of the percentage of TFE for effective trypsin digestion | |
| TFE | Sigma-Aldrich | 18370-10×1mL | 60% TFE for cell lysis |
| Thermocycler w/ heated lid | Peltier Thermal Cycler | PTC-200 | Heating for protein denaturation |
| Trypsin Gold | Promega | V528A | Enzyme for protein digestion |
| Waters BEH C18 column | Waters | C18 column for peptide separation |
References
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Miyamoto, D. T., et al. RNA-Seq of single prostate CTCs implicates noncanonical Wnt signaling in antiandrogen resistance. Science. 349 (6254), 1351-1356 (2015).
- Wang, Y., Navin, N. E. Advances and applications of single-cell sequencing technologies. Molecular Cell. 58 (4), 598-609 (2015).
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends in Genetics. 33 (2), 155-168 (2017).
- Gavasso, S., Gullaksen, S. E., Skavland, J., Gjertsen, B. T. Single-cell proteomics: potential implications for cancer diagnostics. Expert Review of Molecular Diagnostics. 16 (5), 579-589 (2016).
- Wu, M., Singh, A. K. Single-cell protein analysis. Current Opinion in Biotechnology. 23 (1), 83-88 (2012).
- Hughes, A. J., et al. Single-cell western blotting. Nature Methods. 11 (7), 749-755 (2014).
- Peterson, V. M., et al. Multiplexed quantification of proteins and transcripts in single cells. Nature Biotechnology. 35 (10), 936-939 (2017).
- Liu, Y., Beyer, A., Aebersold, R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. 165 (3), 535-550 (2016).
- Choudhary, C., Mann, M. Decoding signalling networks by mass spectrometry-based proteomics. Nature Reviews Molecular Cell Biology. 11 (6), 427-439 (2010).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Zhang, H., et al. Integrated Proteogenomic Characterization of Human High-Grade Serous Ovarian Cancer. Cell. 166 (3), 755-765 (2016).
- Zhang, B., et al. Proteogenomic characterization of human colon and rectal cancer. Nature. 513 (7518), 382-387 (2014).
- Gaudet, S., Miller-Jensen, K. Redefining Signaling Pathways with an Expanding Single-Cell Toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Di Palma, S., Bodenmiller, B. Unraveling cell populations in tumors by single-cell mass cytometry. Current Opinion in Biotechnology. 31, 122-129 (2015).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Current Protocols in Immunology. , Chapter 8 Unit 8 11-20 (2012).
- Willison, K. R., Klug, D. R. Quantitative single cell and single molecule proteomics for clinical studies. Current Opinion in Biotechnology. 24 (4), 745-751 (2013).
- Lee, J. Y., et al. Detection of Head and Neck Cancer Based on Longitudinal Changes in Serum Protein Abundance. Cancer Epidemiology, Biomarkers & Prevention. 29 (8), 1665-1672 (2020).
- Shi, T., et al. Advancing the sensitivity of selected reaction monitoring-based targeted quantitative proteomics. Proteomics. 12 (8), 1074-1092 (2012).
- Shi, T., et al. Advances in targeted proteomics and applications to biomedical research. Proteomics. 16 (15-16), 2160-2182 (2016).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Molecular Systems Biology. 4, 222(2008).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nature Methods. 9 (6), 555-566 (2012).
- Zhu, Y., et al. Proteomic Analysis of Single Mammalian Cells Enabled by Microfluidic Nanodroplet Sample Preparation and Ultrasensitive NanoLC-MS. Angewandte Chemie International Edition. 57 (38), 12370-12374 (2018).
- Cong, Y., et al. Improved Single-Cell Proteome Coverage Using Narrow-Bore Packed NanoLC Columns and Ultrasensitive Mass Spectrometry. Analytical Chemistry. 92 (3), 2665-2671 (2020).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9 (1), 882(2018).
- Shao, X., et al. Integrated Proteome Analysis Device for Fast Single-Cell Protein Profiling. Analytical Chemistry. 90 (23), 14003-14010 (2018).
- Li, Z. Y., et al. Nanoliter-Scale Oil-Air-Droplet Chip-Based Single Cell Proteomic Analysis. Analytical Chemistry. 90 (8), 5430-5438 (2018).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19 (1), 161(2018).
- Zhang, P., et al. Carrier-Assisted Single-Tube Processing Approach for Targeted Proteomics Analysis of Low Numbers of Mammalian Cells. Analytical Chemistry. 91 (2), 1441-1451 (2019).
- Shi, T., et al. Facile carrier-assisted targeted mass spectrometric approach for proteomic analysis of low numbers of mammalian cells. Communications Biology. 1, 103(2018).
- Vitrinel, B., Iannitelli, D. E., Mazzoni, E. O., Christiaen, L., Vogel, C. Simple Method to Quantify Protein Abundances from 1000 Cells. ACS Omega. 5 (25), 15537-15546 (2020).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26 (7), 966-968 (2010).
- Shi, T., et al. Conservation of protein abundance patterns reveals the regulatory architecture of the EGFR-MAPK pathway. Science Signaling. 9 (436), 6(2016).
- Addona, T. A., et al. Multi-site assessment of the precision and reproducibility of multiple reaction monitoring-based measurements of proteins in plasma. Nature Biotechnology. 27 (7), 633-641 (2009).
- He, J., et al. Analytical platform evaluation for quantification of ERG in prostate cancer using protein and mRNA detection methods. Journal of Translational Medicine. 13, 54(2015).
- Shi, T., et al. Antibody-free, targeted mass-spectrometric approach for quantification of proteins at low picogram per milliliter levels in human plasma/serum. Proceedings of the National Academy of Sciences. 109 (38), 15395-15400 (2012).
- Shi, T., et al. Targeted quantification of low ng/mL level proteins in human serum without immunoaffinity depletion. Journal of Proteome Research. 12 (7), 3353-3361 (2013).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved