JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
소량의 인간 세포에 대한 표적 단백질체학 분석을 위한 Carrier-assisted One-pot Sample Preparation
요약
cLC-SRM이라고 하는 액체 크로마토그래피(LC) - 선택 반응 모니터링(SRM)과 결합된 단백질 캐리어 지원 원팟 시료 전처리는 단일 세포를 포함한 소수의 세포에 대한 다중 표적 단백질체학 분석을 위한 편리한 방법입니다. 표적 정량화를 위해 과도한 외인성 단백질을 운반체로 사용하고 특이도가 높은 LC-SRM을 활용하는 활용합니다.
초록
소수의 인간 세포에 대한 단백질 분석은 주로 항체 기반 면역 분석을 통한 표적 단백질체학에 의해 이루어지며, 이는 내재적 한계(예: 낮은 멀티플렉스 및 새로운 단백질에 대한 항체 사용 불가)가 있습니다. 질량분석법(MS) 기반 표적 단백질체학은 항체가 없고 멀티플렉스가 높으며 특이성과 정량 정확도가 높기 때문에 대안으로 떠올랐습니다. 최근 MS 기기의 발전으로 MS 기반 표적 단백질체학(targeted proteomics)을 통해 단일 세포에서 매우 풍부한 단백질의 다중 정량화가 가능해졌습니다. 그러나 MS 분석을 위해 최소한의 시료 손실로 단일 세포를 효과적으로 처리하기 위해서는 기술적인 과제가 있습니다. 이 문제를 해결하기 위해 당사는 최근 소량의 인간 세포에 대한 표적 단백질체학 분석을 위해 cLC-SRM이라고 하는 액체 크로마토그래피(LC) - 선택 반응 모니터링(SRM)과 결합된 편리한 단백질 운반체 보조 원팟 시료 전처리를 개발했습니다. 이 방법은 결합된 과도한 외인성 단백질을 캐리어 및 저용량 원팟 공정으로 사용하여 표면 흡착 손실과 특이성이 높은 LC-SRM을 크게 줄여 외인성 캐리어 단백질의 추가로 인해 증가한 동적 농도 범위를 효과적으로 처리합니다. 그 유용성은 적은 수의 세포(예: 10-100개 세포)에서 가장 적당히 풍부한 단백질과 단일 세포에서 매우 풍부한 단백질의 정확한 정량화에 의해 입증되었습니다. 구현하기 쉬운 기능과 특정 장치가 필요하지 않기 때문에 대부분의 단백질체학 실험실에서 이 방법을 쉽게 사용할 수 있습니다. 본 연구에서는 세포 분류, 세포 용해 및 분해, LC-SRM 분석, 데이터 분석을 포함하여 소수의 인간 세포에 대한 cLC-SRM 분석을 위한 상세한 프로토콜을 제공했습니다. 표적 단일 세포 단백질체학 분석을 위해 검출 감도와 시료 처리량을 더욱 개선해야 합니다. 우리는 cLC-SRM이 정밀 의학을 촉진할 수 있는 잠재력을 가지고 생물 의학 연구 및 시스템 생물학에 광범위하게 적용될 것으로 기대합니다.
서문
최근 유전체학(transcriptomics)의 기술 발전으로 단일 세포의 게놈(transcriptome)을 포괄적이고 정밀하게 분석할 수 있게 되었습니다 1,2,3. 그러나 단세포 단백질체학 기술은 훨씬 뒤처져 있지만 유전체학(전사체학) 기술만큼 중요합니다 4,5,6,7,8. 더욱이, 단백질 풍부도는 반드시 mRNA풍부도9로부터 추론될 수 없으며, 단백질체는 전사체10보다 더 복잡하고 동적이다. 이러한 어려움을 감안할 때, 많은 수의 혼합 세포 집단(즉, 벌크 세포)이 일반적으로 포괄적인 단백질체 데이터를 생성하는 데 사용됩니다 11,12,13. 그러나 이러한 벌크 측정은 개별 세포의 확률적 변이를 평균화하여 중요한 세포 간 변동성(즉, 세포 이질성)을 모호하게 만듭니다 4,14. 이러한 벌크 측정의 한계는 관심 세포가 전체 세포 집단의 작은 부분만 차지할 때 훨씬 더 심각해집니다(예: 초기 단계의 암에서 종양 내 암 줄기세포). 따라서 단세포 단백질체학과 유전체학(transcriptomics) 사이에는 엄청난 지식 격차가 있습니다.
항체 기반 면역분석법(예: 유세포 또는 질량 세포 분석)은 주로 단일 세포 6,7,15,16,17,18의 표적 단백질체 분석에 사용됩니다. 그러나 이들은 낮은 멀티플렉스(multiplex), 제한된 특이성(specificity), 그리고 새로운 관심 단백질에 대한 항체의 가용성이 없다는 문제점을 가지고 있습니다. 질량분석법(MS) 기반 표적 단백질체학은 항체가 없고, 높은 멀티플렉스(단일 분석에서 ≥150개의 단백질19), 높은 정량 정확도(절대량 또는 농도), 높은 특이성 및 재현성(≤10% CV)20,21,22,23으로 인해 정확한 단백질 정량화를 위한 대안으로 부상했습니다. 최근 샘플 전처리의 상당한 발전으로 인해 단일 인간 세포에서 매우 풍부한 단백질의 정량 분석을 위해 MS 기반 단일 세포 단백질체학이 가능해졌습니다. 그러나 MS 기반 단세포 단백질체학은 아직 초기 단계에 있습니다. 예를 들어, 초저유량 RPLC 유속과 결합된 가장 진보된 MS 플랫폼은 단일 HeLa 세포24,25의 총 ≥12,000개 단백질 중 ~670-870개의 단백질에 대한 무표지 MS 검출 및 정량화만 허용할 수 있습니다.
현재 단일 포유류 세포 분석에 사용할 수 있는 6개의 MS 기반 단세포 단백질체학 접근법이 있으며, 그 중 4개는 글로벌 단백질체학을 위한 것입니다(nanoPOTS: 미량 시료를 위한 나노웰 기반 전처리26; iPAD-1: 단일 세포 분석을 위한 통합 단백질체 분석 장치27; OAD(oil-air-droplet) 칩 기반 단세포 단백질체 분석28; SCoPE-MS: 질량 분석법에 의한 단일 세포 단백질체학29) 및 다른 두 가지는 표적 단백질체학(cLC-SRM: 캐리어 보조 액체 크로마토그래피(LC) - 선택 반응 모니터링(SRM)30; cPRISM-SRM: SRM31과 결합된 지능형 선택 및 멀티플렉싱을 통한 캐리어 지원 고압, 고분해능 분리를 위한 것입니다.). 그러나 이러한 모든 접근 방식에는 기술적인 단점이 있습니다. nanoPOTS, iPAD-1 및 OAD는 시료 처리량을 2-200nL로 축소하고 광범위한 벤치탑 응용 분야에 적합하지 않습니다 26,27,28. SCoPE-MS의 경우, 단일 셀 처리 후 TMT(탠덤 질량 태그) 캐리어가 추가되므로 단일 셀 처리에 단일 튜브를 사용할 때 샘플 처리 중 표면 흡착 손실을 효과적으로 방지할 수 없습니다29 복제32 간 상관 계수 ~0.2-0.4에 불과한 낮은 재현성을 초래합니다. cLC-SRM 및 cPRISM-SRM의 경우, 과도한 외인성 단백질의 펩타이드가 MS/MS에 의해 자주 염기서열분석되기 때문에 외인성 단백질을 운반체로 사용하는 것이 표적 단백질체학에 더 적합하며, 이는 풍부한 내인성 펩타이드를 염기서열분석할 기회를 크게 감소시킵니다30,31. 상대적 정량화를 위한 글로벌 단백질체학과 달리, 두 가지 표적 단백질체학 접근법은 알려진 농도에서 중질 동위원소 표지 내부 표준물질을 사용하여 높은 재현성으로 소수의 세포에 대한 정확한 단백질 또는 절대 단백질 분석을 제공할 수 있습니다. 이전의 고분해능 PRISM 분획법이 필요하여 분석해야 할 분획 샘플이 많았던 cPRISM-SRM과 비교했을 때, cLC-SRM은 분획 없이 시료 처리량을 높일 수 있는 이점이 있으며 단일 분석에서 수백 개의 단백질을 동시에 정량화할 수 있지만 검출 감도는 상대적으로 낮습니다30. 따라서 cLC-SRM은 접근성이 더 높으며 질량이 제한된 샘플뿐만 아니라 적은 수의 세포에 대한 정확한 다중 단백질 분석을 위해 더 광범위한 유틸리티를 가져야 합니다.
본 명세서에서는 단일 세포를 포함한 소수의 인간 세포에 대한 편리한 표적 단백질체학 분석을 위해 cLC-SRM을 수행하기 위한 자세한 프로토콜에 대해 설명합니다. 프로토콜은 FACS(형광 활성화 세포 분류)에 의한 세포 분류, 저용량 단일 중합효소 연쇄 반응(PCR) 튜브에서 처리된 세포 용해 및 분해, LC-SRM 데이터 수집 및 공개적으로 사용 가능한 Skyline 소프트웨어를 사용한 SRM 데이터 분석의 주요 단계로 구성됩니다(그림 1). 1-100 MCF7 또는 MCF10A 세포에서 EGFR/MAPK 경로 단백질의 절대 표적 정량화와30의 광범위한 동적 농도에서 세포당 경로 단백질 사본 측정을 통해 이전에 잘 확립된 SRM 분석과 함께 광범위한 유용성이 입증되었습니다. 당사는 상세한 프로토콜을 통해 대부분의 단백질체학 연구자들이 프로젝트 요구 사항을 충족하기 위해 초소형 샘플(예: 희귀 종양 세포)의 정확한 단백질 분석을 위해 실험실에서 cLC-SRM을 즉시 구현할 수 있을 것으로 기대합니다.
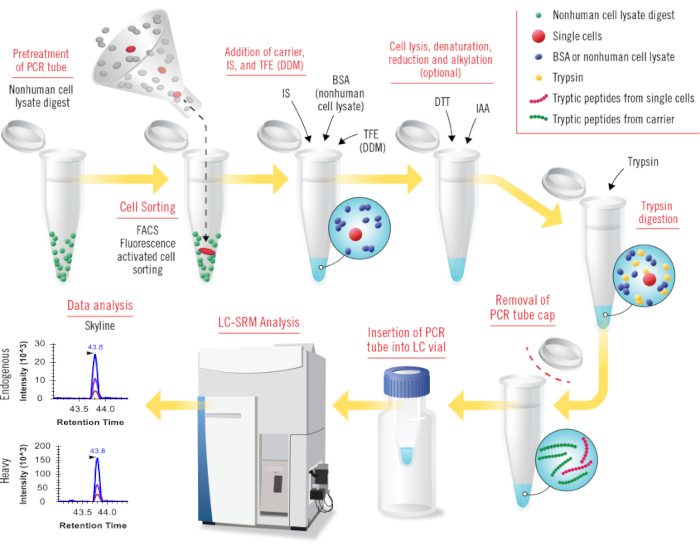
그림 1: cLC-SRM의 모든 단계 개요(liquid chromatography -selected reaction monitoring과 결합된 c arried-assisted one-pot 시료 준비).비인간 세포 용해물 분해물(예: Shewanella oneidensis)은 튜브 표면을 코팅하기 위해 PCR 튜브를 전처리하는 데 사용됩니다. FACS에 의해 분류된 소량의 인간 세포 또는 단일 세포를 전처리된 PCR 튜브에 수집합니다. BSA 단백질(또는 비인간 세포 용해물 단백질체) 운반체, 무거운 내부 표준물질(IS) 및 TFE(또는 DDM)를 세포 용해를 촉진하고 표면 흡착 손실을 줄이기 위해 샘플 튜브에 순차적으로 첨가합니다. 개념적으로 결합된 DDM과 비인간 세포 용해물 단백질체 운반체는 cLC-SRM에 잘 작동합니다. 세포 용해는 초음파 처리에 의해 수행되며 단백질 변성은 고온에서 가열하여 이루어집니다. DTT 및 IAA 시약은 각각 환원 및 알킬화에 사용됩니다(이 단계는 선택 사항임). 트립신은 소화를 위해 첨가되며, 표준 트립신 소화보다 단백질 양에 대한 트립신의 비율이 훨씬 높습니다. 시료 튜브의 캡을 제거한 다음 PCR 튜브를 LC 바이알에 삽입하여 직접 LC-SRM 분석을 수행합니다. 수집된 SRM 데이터는 공개적으로 사용 가능한 Skyline 소프트웨어를 사용하여 분석됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
참고: 단계별 cLC-SRM 분석은 그림 1에 나와 있습니다.
1. PCR 튜브의 전처리
- 0.2 μg/μL의 비인간(예: Shewanella oneidensis) 세포 용해물 분해물 100 μL를 PCR 튜브에 추가합니다. PCR 튜브 표면을 코팅하기 위해 실온에서 하룻밤 동안 배양합니다.
- 피펫팅으로 세포 용해물 분해물을 제거하고 PCR 튜브를 HPLC 등급의 물로 3회 헹구고 PCR 튜브를 흄 후드에서 공기 건조합니다.
- 코팅된 PCR 튜브는 나중에 사용할 때까지 4°C 냉동고에 보관하십시오.
2. FACS 정렬
- 신뢰할 수 있는 참조 신호를 제공하기 위해 레이저 소스에 의해 여기될 수 있는 염료가 있는 형광 비드를 사용하여 형광 활성화 세포 분류기(FACS)(재료 표 참조)를 96웰 PCR 플레이트에 정렬합니다.
- FACS 기계가 PCR 플레이트의 맨 아래로 세포를 분류하는 데 적합한지 확인합니다.
- 정렬 스트림의 각도를 가능한 최소로 설정하여 셀이 측면에 부딪히지 않고 웰 바닥으로 정렬되는 성공률을 최대화합니다.
- 기계를 조준하려면 테스트 스트림을 사용하여 PBS 버퍼의 방울(예: 100개 방울)을 빈 플레이트의 웰에 '정렬'하여 수집된 방울이 바닥에 있는지 확인합니다. 방울이 올바른 위치에 떨어지지 않으면 세포 분류기를 다시 보정하고 분류 스트림의 방울이 올바르게 수집될 때까지 반복합니다.
- 코팅된 PCR 튜브를 96웰 PCR 튜브 랙에 놓습니다.
- 원하는 수의 인간 유방 세포(MCF7 또는 MCF10A)를 사전 코팅된 PCR 튜브에 분류합니다.
- 분류된 유방 세포를 즉시 100 x g 에서 4°C에서 10분 동안 원심분리하여 튜브 바닥에 보관하여 잠재적인 세포 손실을 방지합니다.
- 추가 분석이 있을 때까지 분류된 셀을 -80°C 냉동고에 보관합니다.
3. 단백질 운반체, 무거운 내부 표준물질 및 TFE 추가
- 수집된 세포가 들어 있는 PCR 튜브에 4μL의 25mM NH4HCO3 를 추가합니다.
- 25mM NH4HCO3 에 10ng/μL 소 혈청 알부민(BSA) 1μL를 시료 튜브에 추가합니다.
- 0.3 μL의 100 fmol/μL 미정제 중펩타이드 표준물질(총 30 fmol)을 시료 튜브에 추가합니다.
참고: EGFR/MAPK 경로 단백질에 대한 미처리 중펩타이드 표준물질의 분취량은 25mM NH4HCO3 에서 100 fmol/μL의 공칭 농도로 만들어집니다. 100 fmol/μL의 소량의 미가공 펩타이드 표준물질을 용액 중간에 분배하고 피펫 팁에 액체가 남아 있지 않은지 확인합니다. - 최종 농도가 ~60% TFE인 시료 튜브에 9μL의 100% TFE를 추가합니다.
- 1500 x g에서 5분 동안 원심분리기를 사용한 다음 100 x g에서 3분 동안 부드럽게 와류로 회전시킵니다.
4. 세포 용해 및 단백질 변성
- FACS로 분류된 유방 세포를 얼음 위에서 1분 간격으로 1분 동안 70% 진폭 및 0.5Hz에서 총 5회 초음파 처리합니다.
- 단백질 변성을 위해 90°C에서 1시간 동안 가열식 뚜껑 옵션이 있는 PCR thermocycler를 사용하여 샘플을 배양합니다.
참고: TFE는 끓는점이 74°C인 휘발성 용매이므로 상당한 샘플 손실로 인한 완전 건조를 방지하기 위해 가열식 뚜껑 옵션을 선택합니다. - 1500 x g 에서 3분 동안 원심분리하여 샘플을 실온으로 냉각합니다.
5. (선택) 감소 그리고 알킬화
- 2mM의 최종 농도를 위해 0.6μL의 50mM DTT를 추가합니다.
참고: DTT 솔루션은 매번 새로 준비해야 합니다. 77.5mg의 DTT를 칭량한 다음 25mM NH4HCO3 1mL에 용해시켜 500mM DTT를 만듭니다. 그런 다음 10μL의 500mM DTT를 90μL의 25mM NH4HCO3에추가하여 50mM DTT를 만듭니다. - 1500 x g 에서 5분 동안 원심분리기를 사용한 다음 850rpm에서 3분 동안 부드럽게 혼합합니다.
- 56°C에서 1시간 동안 배양하고 100 x g에서 부드럽게 흔듭니다.
- 시료를 실온으로 냉각하고 1500 x g 에서 3분 동안 원심분리합니다.
- 최종 농도가 ~2mM인 RT-PCR 튜브에 0.5μL의 60mM IAA를 추가합니다.
참고: IAA 용액은 매번 새로 준비해야 합니다. IAA 74.3mg의 무게를 잰 다음 25mM NH4HCO3 1mL에 용해시켜 400mM IAA를 만듭니다. 15 μL의 400 mM IAA를 85 μL의 25 mM NH4HCO3 에 첨가하여 60 mM IAA를 만듭니다. - 실온의 어두운 곳에서 30분 동안 배양하고 100 x g에서 부드럽게 흔듭니다.
참고: 환원 및 알킬화 단계는 표적 단백질에 대해 선택된 시스테인 함유 펩타이드가 없는 경우 선택 사항입니다.
6. 트립신 소화
- SpeedVac 농축기를 사용하여 시료량을 ~4μL로 줄입니다.
참고 : TFE는 끓는점이 74 ° C인 휘발성 용매입니다. 높은 비율의 TFE는 트립신 소화를 억제합니다. - 분해를 위해 9 μL의 25 mM NH4HCO3 및 1-3 μL의 15 ng/μL 트립신을 첨가하고 최종 트립신 농도는 1-3 ng/μL입니다.
참고: 10개의 FACS로 분류된 인간 유방 세포(~1ng)의 경우 단백질에 대한 트립신 효소의 비율은 ≥15:1로 표준 트립신 분해(1:50의 비율)보다 750배 높습니다. - 100 x g 에서 3분 동안 부드럽게 혼합하고 37 °C에서 하룻밤(~16시간) 배양합니다.
- 0.5μL의 5% 포름산을 첨가하여 효소 반응을 중지합니다.
- 1500 x g에서 1시간 동안 원심분리기.
- 추가 LC-SRM 분석이 있을 때까지 -80°C 냉동고에 보관하거나 즉시 분석을 위해 4°C 냉동고에 보관하십시오.
7. 직접 LC-SRM 분석을 위한 준비
- FACS로 분류된 유방 세포의 수집(2단계) 및 처리(6단계)에 사용된 PCR 튜브의 캡을 제거합니다.
- PCR 튜브를 LC 바이알에 삽입합니다.
- LC-SRM 분석을 위해 LC 바이알을 닫습니다.
참고: 96-well PCR 플레이트에서 처리된 소량의 세포의 경우, 직접 LC-SRM 분석을 위해 플레이트를 밀봉하기 위해 매트 커버가 추가됩니다.
8. LC-SRM 분석
- 삼중 사중극자 질량 분석기와 결합된 UPLC를 사용하여 샘플을 분석합니다.
- 모세관 C18 컬럼(표준 그래디언트의 경우 75μm 내경 × 20cm, 짧은 그래디언트의 경우 100μm 내× 10cm)을 스테인리스 금속 유니온을 통해 화학적으로 에칭된 20μm 내경 용융 실리카 전기분무 이미터에 연결합니다.
- 20 μL 샘플 루프를 사용하여 모든 샘플(총 ~15 μL)을 LC 컬럼에 직접 로드하여 검출 감도를 극대화합니다.
- 모세관 RPLC 분리를 위한 이동상 A 및 B로 물에 0.1% 포름산을 사용하고 90% 아세토니트릴에 0.1% 포름산을 사용합니다.
- 많은 수의 표적 단백질(>10)을 측정할 때 표준 그래디언트를 위해 300nL/min의 유속에서 binary LC 그래디언트를 사용합니다. 표준 그래디언트: 26분에 5-20% B, 10분에 20-25% B, 8분에 25-40% B, 1분에 40-95% B, 7분 동안 95% B에서 총 52분 동안 그리고 분석 컬럼은 8분 동안 99.5% A에서 재평형화되었습니다.
- 적은 수의 표적 단백질(≤10)을 측정할 때 짧은 그래디언트를 위해 400nL/min의 유속에서 Binary LC 그래디언트를 사용합니다. 빠른 분리를 위한 짧은 그래디언트: 5분 내에 5-95% B, 분석 컬럼은 총 10분의 LC 실행 시간으로 5분 동안 99.5% A에서 재평형화되었습니다.
- 2,400 ± 100 V의 이온 스프레이 전압, 35 V의 모세관 오프셋 전압, -5 V의 스키머 오프셋 전압 및 220°C의 모세관 입구 온도로 QQQ 질량 분석기를 작동합니다.
- 추가 최적화 없이 자동 튜닝 및 캘리브레이션에서 다른 QQQ MS 파라미터를 얻을 수 있습니다.
- 시료의 LC-SRM 분석 전에 20-50 ng/μL의 비인간 세포 용해물 분해물에 다양한 농도의 미처리 펩타이드 표준물질을 스파이크하여 예정된 SRM 분석법 구축을 위한 펩타이드 LC 머무름 시간(RT) 및 정확한 정량화를 위한 잠재적 간섭 전이를 측정합니다.
- 스캔 창이 ≥6분인 다중화 정량화를 위해 RT 예약 SRM 모드를 사용합니다.
참고: 스캔 창은 특정 시간 창의 전환 수에 따라 적절하게 조정됩니다. - 총 주기 시간을 1초로 설정하면 각 전이의 유지 시간이 서로 다른 보존 시간 창에서 스캔된 전이 수에 따라 자동으로 조정됩니다. 각 SRM 전환에는 10ms의 최소 체류 시간이 필요합니다. 단일 LC-SRM 분석에서 모든 표적 단백질을 동시에 모니터링할 수 있습니다.
9. 데이터 분석
- 각 타겟 펩타이드의 데이터 시각화를 위해 공개적으로 사용 가능한 Skyline 소프트웨어33 으로 원시 데이터 파일을 가져와 검출 가능성을 결정할 수 있습니다.
- SRM 피크 검출 및 적분을 결정하기 위해 두 가지 기준을 사용하십시오: 1) 동일한 LC 머무름 시간; 2) 내인성(경량) 펩타이드와 이에 상응하는(무거운) 동위원소 표지 펩타이드 내부 표준물질 사이의 여러 전이에서 거의 동일한 상대적 SRM 피크 강도 비율.
- 위의 두 가지 기준을 사용하여 신뢰할 수 있는 정량화를 위해 올바른 SRM 피크 할당과 SRM 피크 경계를 보장하기 위해 모든 SRM 데이터를 수동으로 검사합니다.
- 단백질 정량화를 위해 매트릭스 간섭이 없는 높은 SRM 신호로 최상의 전이를 사용하고 다른 전이는 참조로 사용합니다.
- 다중 반복실험을 사용하여 표준 편차(SD)와 변동 계수(CV)를 구합니다.
- 표준 그래디언트의 경우 ±15초, 짧은 그래디언트의 경우 ±6초의 머무름 시간 영역 내에서 평균 배경 노이즈에 대한 피크 정점 강도로 신호 대 잡음비(S/N)를 계산합니다. 검출 한계(LOD)와 정량화 한계(LOQ)는 신호 대 잡음비가 각각 3과 7 이상인 가장 낮은 농도 지점으로 정의됩니다.
- LOQ 값을 보수적으로 결정하기 위해 S/N ≥7 외에 한 가지 추가 기준을 적용하십시오: 다른 세포 수에 대한 대리 펩타이드 반응은 선형 동적 범위 내에 있어야 합니다.
- Excel 소프트웨어를 사용하여 모든 검량 곡선을 플롯합니다. QQQ MS의 RAW 데이터를 공개적으로 사용 가능한 Skyline 소프트웨어로 로드하여 모니터링된 표적 단백질의 여러 전이에 대한 추출된 이온 크로마토그램(XIC)의 그래프를 표시합니다.
결과
소량의 MCF7 세포 용해물(5-200개 세포에 해당하는 0.5-20ng)은 FACS로 분류된 적은 수의 세포와 비교할 때 더 균일하고 변동이 적기 때문에 EGFR/MAPK 경로 단백질의 표적 정량화를 통해 cLC-SRM의 성능을 평가하기 위해 처음 사용되었습니다. 그림 2A에서 볼 수 있듯이, XIC는 MCF7 셀34당 ~220,000 카피에 존재하는 GRB2에서 ?...
토론
cLC-SRM은 단일 세포를 포함한 소수의 세포에 대한 정확한 다중 단백질 분석을 가능하게 하는 편리한 표적 단백질체학 방법입니다. 이 방법은 세포 채취, 다단계 세포 용해 및 분해, MS 분석을 위한 펩타이드 분해액을 모세관 LC 컬럼으로 옮기는 것을 포함한 모든 단계가 하나의 포트(예: 단일 튜브 또는 단일 웰)에서 수행되는 단백질 운반체 보조 원팟(one-pot) 시료 전처리를 ?...
공개
저자는 공개할 내용이 없습니다.
감사의 말
이 작업은 NIH Grant R21CA223715(TS) 및 UG3CA256967(TS 및 HL)의 지원을 받았습니다. 본 명세서에 기술된 실험 작업은 계약 DE-AC05-76RL0 1830에 따라 미국 에너지부(United States Department of Energy)가 후원하는 국립 과학 사용자 시설인 퍼시픽 노스웨스트 국립 연구소(Pacific Northwest National Laboratory)의 환경 분자 과학 실험실(Environmental Molecular Sciences Laboratory)에서 수행되었다.
자료
| Name | Company | Catalog Number | Comments |
| 2 mL glass LC vial | Microsolv | 9502S-WCV | Vessel to hold PCR tube for autosample injection |
| BSA | Sigma-Aldrich | P0834-10×1mL | Carrier protein for greatly reducing surface adsorption losses |
| DTT | Thermo Scientific | A39255 | Reagent for reduction |
| Formic acid | Thermo Scientific | 28905 | Reagent for stopping enzyme reaction |
| IAA | Thermo Scientific | A39271 | Reagent for alkylation |
| Peptide internal standards | New England peptide | Targeted quantification of EGFR/MAPK pathway proteins | |
| RT-PCR tube | GeneMate Bioexpress | T-3035-1 | 0.2 mL PCR tube for one-pot sample preparation |
| Skyline software | University of Washington | Publicly available for SRM data analysis | |
| Sonicator | Hielscher Ultrasound Technology | UTR200 | Sonication on ice for cell lysis |
| Speed Vac concentrator | Thermo Scientific | Reduction of the percentage of TFE for effective trypsin digestion | |
| TFE | Sigma-Aldrich | 18370-10×1mL | 60% TFE for cell lysis |
| Thermocycler w/ heated lid | Peltier Thermal Cycler | PTC-200 | Heating for protein denaturation |
| Trypsin Gold | Promega | V528A | Enzyme for protein digestion |
| Waters BEH C18 column | Waters | C18 column for peptide separation |
참고문헌
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Miyamoto, D. T., et al. RNA-Seq of single prostate CTCs implicates noncanonical Wnt signaling in antiandrogen resistance. Science. 349 (6254), 1351-1356 (2015).
- Wang, Y., Navin, N. E. Advances and applications of single-cell sequencing technologies. Molecular Cell. 58 (4), 598-609 (2015).
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends in Genetics. 33 (2), 155-168 (2017).
- Gavasso, S., Gullaksen, S. E., Skavland, J., Gjertsen, B. T. Single-cell proteomics: potential implications for cancer diagnostics. Expert Review of Molecular Diagnostics. 16 (5), 579-589 (2016).
- Wu, M., Singh, A. K. Single-cell protein analysis. Current Opinion in Biotechnology. 23 (1), 83-88 (2012).
- Hughes, A. J., et al. Single-cell western blotting. Nature Methods. 11 (7), 749-755 (2014).
- Peterson, V. M., et al. Multiplexed quantification of proteins and transcripts in single cells. Nature Biotechnology. 35 (10), 936-939 (2017).
- Liu, Y., Beyer, A., Aebersold, R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. 165 (3), 535-550 (2016).
- Choudhary, C., Mann, M. Decoding signalling networks by mass spectrometry-based proteomics. Nature Reviews Molecular Cell Biology. 11 (6), 427-439 (2010).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Zhang, H., et al. Integrated Proteogenomic Characterization of Human High-Grade Serous Ovarian Cancer. Cell. 166 (3), 755-765 (2016).
- Zhang, B., et al. Proteogenomic characterization of human colon and rectal cancer. Nature. 513 (7518), 382-387 (2014).
- Gaudet, S., Miller-Jensen, K. Redefining Signaling Pathways with an Expanding Single-Cell Toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Di Palma, S., Bodenmiller, B. Unraveling cell populations in tumors by single-cell mass cytometry. Current Opinion in Biotechnology. 31, 122-129 (2015).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Current Protocols in Immunology. , 11-20 (2012).
- Willison, K. R., Klug, D. R. Quantitative single cell and single molecule proteomics for clinical studies. Current Opinion in Biotechnology. 24 (4), 745-751 (2013).
- Lee, J. Y., et al. Detection of Head and Neck Cancer Based on Longitudinal Changes in Serum Protein Abundance. Cancer Epidemiology, Biomarkers & Prevention. 29 (8), 1665-1672 (2020).
- Shi, T., et al. Advancing the sensitivity of selected reaction monitoring-based targeted quantitative proteomics. Proteomics. 12 (8), 1074-1092 (2012).
- Shi, T., et al. Advances in targeted proteomics and applications to biomedical research. Proteomics. 16 (15-16), 2160-2182 (2016).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Molecular Systems Biology. 4, 222 (2008).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nature Methods. 9 (6), 555-566 (2012).
- Zhu, Y., et al. Proteomic Analysis of Single Mammalian Cells Enabled by Microfluidic Nanodroplet Sample Preparation and Ultrasensitive NanoLC-MS. Angewandte Chemie International Edition. 57 (38), 12370-12374 (2018).
- Cong, Y., et al. Improved Single-Cell Proteome Coverage Using Narrow-Bore Packed NanoLC Columns and Ultrasensitive Mass Spectrometry. Analytical Chemistry. 92 (3), 2665-2671 (2020).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9 (1), 882 (2018).
- Shao, X., et al. Integrated Proteome Analysis Device for Fast Single-Cell Protein Profiling. Analytical Chemistry. 90 (23), 14003-14010 (2018).
- Li, Z. Y., et al. Nanoliter-Scale Oil-Air-Droplet Chip-Based Single Cell Proteomic Analysis. Analytical Chemistry. 90 (8), 5430-5438 (2018).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19 (1), 161 (2018).
- Zhang, P., et al. Carrier-Assisted Single-Tube Processing Approach for Targeted Proteomics Analysis of Low Numbers of Mammalian Cells. Analytical Chemistry. 91 (2), 1441-1451 (2019).
- Shi, T., et al. Facile carrier-assisted targeted mass spectrometric approach for proteomic analysis of low numbers of mammalian cells. Communications Biology. 1, 103 (2018).
- Vitrinel, B., Iannitelli, D. E., Mazzoni, E. O., Christiaen, L., Vogel, C. Simple Method to Quantify Protein Abundances from 1000 Cells. ACS Omega. 5 (25), 15537-15546 (2020).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26 (7), 966-968 (2010).
- Shi, T., et al. Conservation of protein abundance patterns reveals the regulatory architecture of the EGFR-MAPK pathway. Science Signaling. 9 (436), 6 (2016).
- Addona, T. A., et al. Multi-site assessment of the precision and reproducibility of multiple reaction monitoring-based measurements of proteins in plasma. Nature Biotechnology. 27 (7), 633-641 (2009).
- He, J., et al. Analytical platform evaluation for quantification of ERG in prostate cancer using protein and mRNA detection methods. Journal of Translational Medicine. 13, 54 (2015).
- Shi, T., et al. Antibody-free, targeted mass-spectrometric approach for quantification of proteins at low picogram per milliliter levels in human plasma/serum. Proceedings of the National Academy of Sciences. 109 (38), 15395-15400 (2012).
- Shi, T., et al. Targeted quantification of low ng/mL level proteins in human serum without immunoaffinity depletion. Journal of Proteome Research. 12 (7), 3353-3361 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유