Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Az Sayıda İnsan Hücresinin Hedefli Proteomik Analizi için Taşıyıcı Destekli Tek Kap Numune Hazırlama
Bu Makalede
Özet
cLC-SRM olarak adlandırılan sıvı kromatografisi (LC) - seçilmiş reaksiyon izleme (SRM) ile birleştirilmiş bir protein taşıyıcı destekli tek kap numune hazırlama, tek hücreler de dahil olmak üzere az sayıda hücrenin çoğullanmış hedefli proteomik analizi için uygun bir yöntemdir. Hedeflenen miktar tayini için taşıyıcı olarak aşırı eksojen protein ve yüksek özgüllüklü LC-SRM kullanılmasından yararlanır.
Özet
Az sayıda insan hücresinin protein analizi, öncelikle, doğal sınırlamaları olan (örneğin, düşük multipleks ve yeni proteinler için antikorların bulunmaması) antikor bazlı immünolojik testlerle hedeflenen proteomiklerle elde edilir. Kütle spektrometresi (MS) tabanlı hedefe yönelik proteomikler, antikor içermeyen, yüksek multipleks ve yüksek özgüllük ve kantitasyon doğruluğuna sahip olması nedeniyle bir alternatif olarak ortaya çıkmıştır. MS enstrümantasyonundaki son gelişmeler, tek hücrelerde yüksek miktarda bulunan proteinlerin çoğullanmış miktar tayini için MS tabanlı hedefli proteomikleri mümkün kılmaktadır. Bununla birlikte, MS analizi için minimum numune kaybıyla tek hücrelerin etkili bir şekilde işlenmesi için teknik bir zorluk vardır. Bu sorunu çözmek için, yakın zamanda, az sayıda insan hücresinin hedefli proteomik analizi için cLC-SRM olarak adlandırılan sıvı kromatografisi (LC) - seçilmiş reaksiyon izleme (SRM) ile birleştirilmiş uygun bir protein taşıyıcı destekli tek kap numune hazırlama geliştirdik. Bu yöntem, yüzey adsorpsiyon kayıplarını büyük ölçüde azaltmak için bir taşıyıcı olarak kombine aşırı eksojen proteinin ve düşük hacimli tek kap işlemenin yanı sıra, ekzojen taşıyıcı proteinin eklenmesi nedeniyle artan dinamik konsantrasyon aralığını etkili bir şekilde ele almak için yüksek özgüllüklü LC-SRM'nin kullanılmasından yararlanır. Faydası, az sayıda hücrede (örneğin, 10-100 hücre) orta derecede bol proteinlerin ve tek hücrelerde yüksek miktarda bulunan proteinlerin doğru bir şekilde ölçülmesiyle gösterilmiştir. Uygulaması kolay özellikler ve belirli cihazlara ihtiyaç duyulmaması, bu yöntemi çoğu proteomik laboratuvarı için kolayca erişilebilir hale getirir. Burada, hücre sıralama, hücre lizisi ve sindirimi, LC-SRM analizi ve veri analizi dahil olmak üzere az sayıda insan hücresinin cLC-SRM analizi için ayrıntılı bir protokol sağladık. Hedeflenen tek hücreli proteomik analize yönelik olarak algılama hassasiyeti ve numune veriminde daha fazla iyileştirmeye ihtiyaç vardır. cLC-SRM'nin, hassas tıbbı kolaylaştırma potansiyeli ile biyomedikal araştırmalara ve sistem biyolojisine geniş çapta uygulanacağını tahmin ediyoruz.
Giriş
Genomikteki son teknolojik gelişmeler (transkriptomik), tek hücrelerdegenomun (transkriptom) kapsamlı ve kesin analizine izin verir 1,2,3. Bununla birlikte, tek hücreli proteomik teknolojiler çok geride kalmaktadır, ancak genomik (transkriptomik) teknolojiler kadar önemlidir 4,5,6,7,8. Ayrıca, protein bolluğu mutlaka mRNA bolluğu9'dan çıkarılamaz ve proteom, transkriptom10'dan daha karmaşık ve dinamiktir. Bu zorluklar göz önüne alındığında, kapsamlı proteom verileri oluşturmak için genellikle çok sayıda karışık hücre popülasyonu (yani toplu hücreler) kullanılır 11,12,13. Bununla birlikte, bu tür toplu ölçümler, tek tek hücrelerin stokastik varyasyonlarının ortalamasını alır, bu nedenle önemli hücreden hücreye değişkenliği (yani hücre heterojenliğini) gizler4,14. Bu tür toplu ölçümlerin sınırlamaları, ilgilenilen hücreler toplam hücre popülasyonlarının yalnızca küçük bir bölümünü oluşturduğunda (örneğin, erken evre bir kanserdeki tümörler içindeki kanser kök hücreleri) daha da şiddetli hale gelir. Bu nedenle, tek hücreli proteomik ve genomik (transkriptomik) arasında büyük bir bilgi boşluğu vardır.
Antikor bazlı immünolojik testler (ör., akış veya kütle sitometrisi) ağırlıklı olarak tek hücrelerinhedefli proteomik analizi için kullanılır 6,7,15,16,17,18. Bununla birlikte, düşük multipleks, sınırlı özgüllük ve ilgilenilen yeni proteinler için antikorların bulunmamasından muzdariptirler. Kütle spektrometresi (MS) tabanlı hedeflenmiş proteomik, antikor içermemesi, yüksek multipleks (tek bir analizde ≥150 protein19), yüksek kantitasyon doğruluğu (mutlak miktarlar veya konsantrasyonlar) ve yüksek özgüllük ve tekrarlanabilirlik (%≤10 CV) nedeniyle doğru protein miktar tayini için bir alternatif olarak ortaya çıkmıştır20,21,22,23. Numune hazırlamadaki son önemli ilerlemeler, tek insan hücrelerinden yüksek miktarda bulunan proteinlerin kantitatif analizi için MS bazlı tek hücreli proteomikleri mümkün kılmıştır. Bununla birlikte, MS tabanlı tek hücreli proteomik hala erken bebeklik aşamasındadır. Örneğin, ultra düşük akışlı RPLC akış hızlarıyla birleştirilmiş en gelişmiş MS platformu, tek HeLa hücrelerindeki toplam 670 proteinden yalnızca ~870-≥12,000 proteininetiketsiz MS tespitine ve miktar tayinine izin verebilir 24,25.
Şu anda, dördü küresel proteomikler için olmak üzere, tek memeli hücrelerinin analizi için altı MS tabanlı tek hücreli proteomik yaklaşım bulunmaktadır (nanoPOTS: İz Numuneler için Tek Kapta nanowell tabanlı Hazırlama26; iPAD-1: tek hücre analizi için entegre proteom analiz cihazı27; OAD (yağ-hava damlası) çip tabanlı tek hücreli proteomik analiz28; SCoPE-MS: kütle spektrometresi ile tek hücreli proteomikler29) ve diğer ikisi hedeflenen proteomikler içindir (cLC-SRM: taşıyıcı destekli sıvı kromatografisi (LC) - seçilmiş reaksiyon izleme (SRM)30; cPRISM-SRM: SRM31'e bağlı akıllı seçim ve çoğullama ile taşıyıcı destekli yüksek basınç, yüksek çözünürlüklü ayırmalar). Ancak, tüm bu yaklaşımların teknik dezavantajları vardır. nanoPOTS, iPAD-1 ve OAD, numune işleme hacmini 2-200 nL'ye düşürür ve geniş tezgah üstü uygulamalar için hazır değildir 26,27,28. SCoPE-MS için, tek hücreli işlemeden sonra bir TMT (tandem kütle etiketi) taşıyıcısı eklenir, bu nedenle, tek hücreli işleme29 için tek bir tüp kullanıldığında numune işleme sırasında yüzey adsorpsiyon kayıplarını etkili bir şekilde önleyemez, bu da replikatlar arasında yalnızca ~ 0.2-0.4'lük bir korelasyon katsayısı ile düşük tekrarlanabilirlik ile sonuçlanır32. cLC-SRM ve cPRISM-SRM için, taşıyıcı olarak eksojen proteinlerin kullanılması, hedeflenen proteomikler için daha uygundur, çünkü aşırı eksojen proteinlerden elde edilen peptitler sıklıkla MS/MS tarafından dizilenir, bu da düşük bol miktarda endojen peptitlerin dizilenmesi şansını büyük ölçüde azaltır30,31. Bağıl kantitasyon için küresel proteomikten farklı olarak, hedeflenen iki proteomik yaklaşım, bilinen konsantrasyonlarda ağır izotop etiketli iç standartları kullanarak yüksek tekrarlanabilirliğe sahip az sayıda hücrenin doğru veya mutlak protein analizini sağlayabilir. Önceden yüksek çözünürlüklü PRISM fraksiyonlaması gerektiren ve analiz edilmesi gereken birçok fraksiyon numunesi ile sonuçlanan cPRISM-SRM ile karşılaştırıldığında, cLC-SRM, fraksiyonlama olmadan numune veriminde önemli bir avantaja sahiptir ve tek bir analizde yüzlerce proteini aynı anda ölçebilir, ancak nispeten daha düşük algılama hassasiyeti ile30. Bu nedenle, cLC-SRM daha erişilebilirdir ve az sayıda hücrenin yanı sıra kütle sınırlı numunelerin doğru çoğullanmış protein analizi için daha geniş yardımcı programlara sahip olmalıdır.
Burada, tek hücreler de dahil olmak üzere az sayıda insan hücresinin uygun hedefli proteomik analizi için cLC-SRM'yi gerçekleştirmek için ayrıntılı bir protokol açıklıyoruz. Protokol aşağıdaki ana adımlardan oluşur: FACS (floresan ile aktive edilmiş hücre sıralaması) ile hücre sıralaması, düşük hacimli tek polimeraz zincir reaksiyonu (PCR) tüplerinde işlenen hücre lizisi ve sindirimi, LC-SRM veri toplama ve halka açık Skyline yazılımı kullanılarak SRM veri analizi (Şekil 1). Geniş faydası, 1-100 MCF7 veya MCF10A hücrelerinde EGFR / MAPK yol proteinlerinin mutlak hedefli miktar tayini ve geniş bir dinamik konsantrasyon aralığında hücre başına yol protein kopyalarının belirlenmesi ile daha önce iyi bilinen SRM testlerimizle birlikte gösterilmiştir30. Ayrıntılı protokol ile çoğu proteomik araştırmacısının, proje ihtiyaçlarını karşılamak için ultra küçük numunelerin (örneğin, nadir tümör hücreleri) doğru protein analizi için laboratuvarlarında cLC-SRM'yi kolayca uygulayabileceğini tahmin ediyoruz.
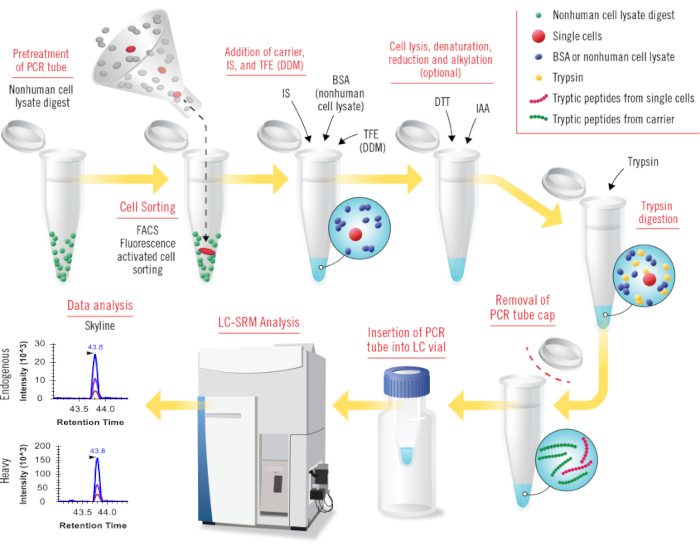
Şekil 1: cLC-SRM'deki tüm adımlara genel bakış (c iquid chromatografi -sseçilmiş reaction monitoring ile birleştirilmiş c arried destekli tek kap numune hazırlama).İnsan dışı hücre lizat sindirimleri (örneğin, Shewanella oneidensis), tüp yüzeyini kaplamak için PCR tüplerini ön işleme tabi tutmak için kullanılır. FACS ile sıralanan az sayıda insan hücresi veya tek hücre, ön işleme tabi tutulmuş PCR tüplerine toplanır. BSA proteini (veya insan olmayan hücre lizatı proteomu) taşıyıcısı, ağır dahili standart (IS) ve TFE (veya DDM), hücre lizizini kolaylaştırmak ve yüzey adsorpsiyon kayıplarını azaltmak için numune tüplerine sırayla eklenir. Kavramsal olarak, birleşik DDM ve insan olmayan hücre lizat proteom taşıyıcısı, cLC-SRM için iyi çalışacaktır. Hücre lizisi sonikasyon ile gerçekleştirilir ve protein denatürasyonu yüksek sıcaklıkta ısıtılarak elde edilir. DTT ve IAA reaktifleri sırasıyla redüksiyon ve alkilasyon için kullanılır (bu adım isteğe bağlıdır). Tripsin, standart tripsin sindirimi için olandan çok daha yüksek protein miktarı üzerinde tripsin oranları ile sindirim için eklenir. Numune tüpünün kapağı çıkarılır ve daha sonra PCR tüpü doğrudan LC-SRM analizi için LC şişesine yerleştirilir. Toplanan SRM verileri, halka açık Skyline yazılımı kullanılarak analiz edilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protokol
NOT: Adım adım cLC-SRM analizi Şekil 1'de gösterilmiştir.
1. PCR tüplerinin ön tedavisi
- PCR tüplerine 0,2 μg/μL'de 100 μL insan dışı (örneğin, Shewanella oneidensis) hücre lizat sindirimi ekleyin. PCR tüpü yüzeyini kaplamak için gece boyunca oda sıcaklığında inkübe edin.
- Pipetleme yoluyla hücre lizat çürütücülerini çıkarın, PCR tüplerini HPLC dereceli suyla 3 kez durulayın ve ardından PCR tüplerini bir davlumbazda havayla kurutun.
- Kaplanmış PCR tüplerini daha fazla kullanana kadar 4 °C'lik bir dondurucuda saklayın.
2. FACS sıralama
- Güvenilir referans sinyalleri sağlamak için lazer kaynağı tarafından uyarılabilen boyalara sahip floresan boncuklar kullanarak floresanla aktive edilen bir hücre sıralayıcıyı (FACS) (Malzeme Tablosuna bakın) 96 oyuklu bir PCR plakasına hizalayın.
- FACS makinesinin, hücreleri PCR plakalarının altına yerleştirmek için iyi bir şekilde hedeflendiğini doğrulayın.
- Hücrelerin kenarlara çarpmak yerine kuyucukların dibine sıralanacağı başarı oranını en üst düzeye çıkarmak için sıralama akışının açısını mümkün olan en düşük seviyeye ayarlayın.
- Makineyi hedeflemek için, toplanan damlacıkların dipte olduğundan emin olmak için PBS tampon damlacıklarını (örn. 100 damlacık) boş bir plakanın kuyusuna 'sıralamak' için test akışını kullanın. Damlacıklar doğru konuma inmezse, hücre sıralayıcıyı yeniden kalibre edin ve sıralama akışındaki damlacıklar doğru şekilde toplanana kadar tekrarlayın.
- Kaplanmış PCR tüplerini 96 oyuklu bir PCR tüp rafına yerleştirin.
- İstenilen sayıda insan meme hücresini (MCF7 veya MCF10A) önceden kaplanmış PCR tüplerine sıralayın.
- Sıralanan göğüs hücrelerini, potansiyel hücre kaybını önlemek için tüpün dibinde tutmak için 100 x g'da 4 ° C'de 10 dakika boyunca hemen santrifüjleyin.
- Sıralanan hücreleri daha fazla analize kadar -80 ° C'lik bir dondurucuda saklayın.
3. Protein taşıyıcısı, ağır iç standartlar ve TFE'nin eklenmesi
- Toplanan hücreleri içeren PCR tüplerine 4 μL 25 mMNH4HCO3 ekleyin.
- Numune tüplerine 25 mM NH4HCO3'te 1 μL 10 ng / μL sığır serum albümini (BSA) ekleyin.
- Numune tüplerine 0,3 μL 100 fmol/μL ham ağır peptit standardı (toplam 30 fmol) ekleyin.
NOT: EGFR / MAPK yolağı proteinleri için ham ağır peptit standartlarının alikotları, 25 mMNH4HCO3'te 100 fmol / μL nominal konsantrasyonda yapılır. 100 fmol / μL'de küçük hacimli ham ağır peptit standartlarını çözeltinin ortasına dağıtın ve pipet ucunda artık sıvı olmadığından emin olun. - Nihai konsantrasyonu ~%60 TFE olan numune tüplerine 9 μL %100 TFE ekleyin.
- 1500 x g'da 5 dakika santrifüjleyin ve ardından 100 x g'da 3 dakika boyunca hafifçe girdap yapın.
4. Hücre lizisi ve protein denatürasyonu
- % 70 genlik ve 0.5 Hz'de toplam 5 döngü için 1 dakikalık aralıklarla buz üzerinde FACS ile sıralanmış meme hücrelerini sonikleştirin.
- Isıtmalı kapak seçeneğine sahip bir PCR termodöngüleyici kullanarak protein denatürasyonu için numuneleri 90 °C'de 1 saat inkübe edin.
NOT: TFE, kaynama noktası 74 °C olan uçucu bir çözücüdür ve bu nedenle, önemli numune kaybıyla tam kurumayı önlemek için ısıtmalı kapak seçeneği seçilmiştir. - Numuneleri 3 dakika boyunca 1500 x g'da santrifüjleme ile oda sıcaklığına soğutun.
5. İndirgeme ve alkilasyon (isteğe bağlı)
- 2 mM'lik bir nihai konsantrasyon için 0,6 μL 50 mM DTT ekleyin.
NOT: DTT çözeltisinin her seferinde taze olarak hazırlanması gerekir. 77.5 mg DTT tartın ve daha sonra 500 mM DTT yapmak için 1 mL 25 mM NH4HCO3'te çözün. Daha sonra 50 mM DTT yapmak için 90 μL 25 mMNH4HCO3'e 10 μL 500 mM DTT eklenir. - 1500 x g'da 5 dakika santrifüjleyin ve ardından 850 rpm'de 3 dakika yavaşça karıştırın.
- 56°C'de 1 saat boyunca 100 x g'da hafifçe çalkalayarak inkübe edin.
- Numuneleri oda sıcaklığına soğutun ve 1500 x g'da 3 dakika santrifüjleyin.
- Son konsantrasyonu ~2 mM olan RT-PCR tüpüne 0,5 μL 60 mM IAA ekleyin.
NOT: IAA çözeltisinin her seferinde taze olarak hazırlanması gerekir. 74.3 mg IAA tartın ve daha sonra 400 mM IAA yapmak için 1 mL 25 mMNH4HCO3'e çözün. 60 mM IAA yapmak için 85 μL 25 mMNH4HCO3'e 15 μL 400 mM IAA eklenir. - Karanlıkta oda sıcaklığında 30 dakika boyunca 100 x g'da hafifçe çalkalayarak inkübe edin.
NOT: İndirgeme ve alkilasyon adımları, hedef proteinler için seçilmiş sistein içeren peptitler olmadığında isteğe bağlıdır.
6. Tripsin sindirimi
- Bir SpeedVac yoğunlaştırıcı kullanarak örnek hacmini ~4 μL'ye düşürün.
NOT: TFE, kaynama noktası 74°C'de olan uçucu bir çözücüdür. Yüksek yüzde TFE, tripsin sindirimini inhibe eder. - Sindirim için 9 μL 25 mM,NH4HCO3 ve 1-3 μL 15 ng/μL tripsin ekleyin, son tripsin konsantrasyonu 1-3 ng/μL'dir.
NOT: FACS'a göre sıralanmış 10 insan meme hücresi (~ 1 ng) için, tripsin enziminin protein üzerindeki oranı ≥15: 1, standart tripsin sindirimindekinden (1: 50 oranı) 750 kat daha yüksektir. - 100 x g'da 3 dakika boyunca nazikçe karıştırın ve gece boyunca (~ 16 saat) 37 ° C'de inkübe edin.
- Enzimatik reaksiyonu durdurmak için 0,5 μL %5 formik asit ekleyin.
- 1500 x g'da 1 saat santrifüjleyin.
- Daha fazla LC-SRM analizine kadar -80 ° C dondurucuda veya anında analiz için 4 ° C dondurucuda saklayın.
7. Doğrudan LC-SRM analizi için hazırlık
- FACS ile sıralanmış meme hücrelerinin toplanması (Adım 2) ve işlenmesi (Adım 6) için kullanılan PCR tüpünün kapağını çıkarın.
- PCR tüpünü LC şişesine yerleştirin.
- LC-SRM analizi için LC şişesini kapatın.
NOT: 96 oyuklu PCR plakasında işlenen az sayıda hücre için, doğrudan LC-SRM analizi için plakayı kapatmak için paspas kapağı eklenecektir.
8. LC-SRM analizi
- Üçlü dört kutuplu kütle spektrometresine bağlı UPLC kullanarak numuneleri analiz edin.
- Kılcal C18 kolonlarını (standart gradyan için 75 μm iç × 20 cm ve kısa gradyan için 100 μm iç × 10 cm) paslanmaz metal bir rakor aracılığıyla kimyasal olarak kazınmış 20 μm id erimiş silika elektrosprey yayıcıya bağlayın.
- Algılama hassasiyetini en üst düzeye çıkarmak için tüm numuneyi (toplamda ~15 μL) doğrudan LC kolonuna yüklemek için 20 μL'lik bir numune döngüsü kullanın.
- Kılcal RPLC ayrımı için mobil faz A ve B olarak suda %0.1 formik asit ve %90 asetonitrilde %0.1 formik asit kullanın.
- Çok sayıda hedef protein (>10) ölçüldüğünde standart bir gradyan için 300 nL/dk akış hızlarında ikili LC gradyanını kullanın. Standart gradyan: 26 dakikada% 5-20 B, 10 dakikada% 20-25 B, 8 dakikada% 25-40 B, 1 dakikada% 40-95 B ve% 95 B'de 7 dakika boyunca toplam 52 dakika ve analitik sütun 8 dakika boyunca% 99,5 A'da yeniden dengelendi.
- Az sayıda hedef protein (≤10) ölçüldüğünde kısa bir gradyan için 400 nL/dk akış hızlarında ikili LC gradyanını kullanın. Hızlı ayırma için kısa gradyan: 5 dakikada %5-95 B ve analitik kolon, toplam 10 dakikalık LC çalışma süresi ile 5 dakika boyunca %99,5 A'da yeniden dengelenir.
- QQQ kütle spektrometresini 2,400 ± 100 V iyon püskürtme voltajları, 35 V kılcal ofset voltajı, -5 V sıyırıcı ofset voltajı ve 220 ° C kılcal giriş sıcaklığı ile çalıştırın.
- Daha fazla optimizasyon olmadan otomatik ayarlama ve kalibrasyondan diğer QQQ MS parametrelerini elde edin.
- Planlanmış bir SRM yöntemi oluşturmak için peptit LC tutma süresini (RT) ve doğru miktar tayini için potansiyel girişim geçişlerini belirlemek için numunelerin LC-SRM analizinden önce farklı konsantrasyonlarda ham ağır peptit standartlarını 20-50 ng/μL insan dışı hücre lizat sindirimlerine dökün.
- ≥6 dakikalık tarama penceresi ile çoğullanmış niceleme için RT programlanmış SRM modunu kullanın.
NOT: Tarama penceresi, belirli bir zaman penceresindeki geçiş sayısına bağlı olarak buna göre ayarlanacaktır. - Toplam döngü süresini 1 sn olarak ayarlayın ve her geçiş için bekleme süresi, farklı bekletme süresi pencerelerinde taranan geçiş sayısına bağlı olarak otomatik olarak ayarlanır. Her SRM geçişi için minimum 10 ms'lik bir bekleme süresi gereklidir. Tüm hedef proteinler tek bir LC-SRM analizinde eş zamanlı olarak izlenebilir
9. Veri Analizi
- Ham veri dosyalarını, tespit edilebilirliklerini belirlemek için her bir hedef peptidin veri görselleştirmesi için halka açık Skyline yazılımına33 aktarın.
- SRM tepe tespiti ve entegrasyonunu belirlemek için iki kriter kullanın: 1) aynı LC tutma süresi; 2) endojen (hafif) peptitler ve bunlara karşılık gelen (ağır) izotop etiketli peptit iç standartları arasındaki çoklu geçişler boyunca yaklaşık olarak aynı nispi SRM tepe yoğunluğu oranları.
- Yukarıdaki iki kriteri kullanarak güvenilir niceleme için doğru SRM tepe atamasını ve SRM tepe sınırını sağlamak için tüm SRM verilerini manuel olarak inceleyin.
- Protein miktar tayini için yüksek SRM sinyaline sahip ancak matris girişimi olmayan en iyi geçişi kullanın ve diğer geçişleri referans olarak kullanın.
- Standart sapma (SD) ve varyasyon katsayısını (CV) elde etmek için birden fazla kopya kullanın.
- Standart gradyan için ±15 sn ve kısa gradyan için ±6 s'lik bir tutma süresi bölgesi içinde ortalama arka plan gürültüsü üzerindeki tepe tepe yoğunluğuna göre sinyal-gürültü oranını (S/N) hesaplayın. Tespit limiti (LOD) ve miktar tayini limiti (LOQ), S/N oranının sırasıyla en az 3 ve 7 olduğu en düşük konsantrasyon noktaları olarak tanımlanır.
- LOQ değerlerini konservatif olarak belirlemek için S/N ≥7'nin yanı sıra bir ek kriter uygulayın: farklı hücre sayılarına karşı vekil peptit yanıtı doğrusal dinamik aralık içinde olmalıdır.
- Tüm kalibrasyon eğrilerini çizmek için Excel yazılımını kullanın. İzlenen hedef proteinlerin çoklu geçişlerinin ekstrakte edilmiş iyon kromatogramlarının (XIC'ler) grafiklerini görüntülemek için RAW verilerini QQQ MS'den halka açık Skyline yazılımına yükleyin.
Sonuçlar
Küçük miktarlarda MCF7 hücre lizatları (5-200 hücreye eşdeğer 0.5-20 ng) ilk olarak EGFR / MAPK yol proteinlerinin hedeflenen miktar tayini ile cLC-SRM'nin performansını değerlendirmek için kullanıldı, çünkü bunlar daha az varyasyonla daha homojendir ve FACS'a göre sıralanan az sayıda hücreye kıyasla. Şekil 2A'da gösterildiği gibi, XIC'ler, MCF7 hücresi34 başına ~220.000 kop...
Tartışmalar
cLC-SRM, tek hücreler de dahil olmak üzere az sayıda hücrenin doğru çoğullanmış protein analizini sağlayan uygun bir hedefli proteomik yöntemdir. Bu yöntem, hücre toplama, çok aşamalı hücre lizisi ve sindirimi ve peptit sindirimlerinin MS analizi için kılcal LC kolonuna aktarılması dahil olmak üzere tüm adımların tek bir kapta (örneğin, tek tüp veya tek kuyucuk) gerçekleştirildiği protein taşıyıcı destekli tek kap numune hazırlamadan yararlanır (
Açıklamalar
Yazarların ifşa edecek hiçbir şeyi yok.
Teşekkürler
Bu çalışma NIH Grant R21CA223715 (TS) ve UG3CA256967 (TS ve HL) tarafından desteklenmiştir. Burada açıklanan deneysel çalışma, DE-AC05-76RL0 1830 Sözleşmesi kapsamında Amerika Birleşik Devletleri Enerji Bakanlığı tarafından desteklenen ulusal bir bilimsel kullanıcı tesisi olan Pasifik Kuzeybatı Ulusal Laboratuvarı, Çevresel Moleküler Bilimler Laboratuvarı'nda gerçekleştirilmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 2 mL glass LC vial | Microsolv | 9502S-WCV | Vessel to hold PCR tube for autosample injection |
| BSA | Sigma-Aldrich | P0834-10×1mL | Carrier protein for greatly reducing surface adsorption losses |
| DTT | Thermo Scientific | A39255 | Reagent for reduction |
| Formic acid | Thermo Scientific | 28905 | Reagent for stopping enzyme reaction |
| IAA | Thermo Scientific | A39271 | Reagent for alkylation |
| Peptide internal standards | New England peptide | Targeted quantification of EGFR/MAPK pathway proteins | |
| RT-PCR tube | GeneMate Bioexpress | T-3035-1 | 0.2 mL PCR tube for one-pot sample preparation |
| Skyline software | University of Washington | Publicly available for SRM data analysis | |
| Sonicator | Hielscher Ultrasound Technology | UTR200 | Sonication on ice for cell lysis |
| Speed Vac concentrator | Thermo Scientific | Reduction of the percentage of TFE for effective trypsin digestion | |
| TFE | Sigma-Aldrich | 18370-10×1mL | 60% TFE for cell lysis |
| Thermocycler w/ heated lid | Peltier Thermal Cycler | PTC-200 | Heating for protein denaturation |
| Trypsin Gold | Promega | V528A | Enzyme for protein digestion |
| Waters BEH C18 column | Waters | C18 column for peptide separation |
Referanslar
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Miyamoto, D. T., et al. RNA-Seq of single prostate CTCs implicates noncanonical Wnt signaling in antiandrogen resistance. Science. 349 (6254), 1351-1356 (2015).
- Wang, Y., Navin, N. E. Advances and applications of single-cell sequencing technologies. Molecular Cell. 58 (4), 598-609 (2015).
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends in Genetics. 33 (2), 155-168 (2017).
- Gavasso, S., Gullaksen, S. E., Skavland, J., Gjertsen, B. T. Single-cell proteomics: potential implications for cancer diagnostics. Expert Review of Molecular Diagnostics. 16 (5), 579-589 (2016).
- Wu, M., Singh, A. K. Single-cell protein analysis. Current Opinion in Biotechnology. 23 (1), 83-88 (2012).
- Hughes, A. J., et al. Single-cell western blotting. Nature Methods. 11 (7), 749-755 (2014).
- Peterson, V. M., et al. Multiplexed quantification of proteins and transcripts in single cells. Nature Biotechnology. 35 (10), 936-939 (2017).
- Liu, Y., Beyer, A., Aebersold, R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. 165 (3), 535-550 (2016).
- Choudhary, C., Mann, M. Decoding signalling networks by mass spectrometry-based proteomics. Nature Reviews Molecular Cell Biology. 11 (6), 427-439 (2010).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Zhang, H., et al. Integrated Proteogenomic Characterization of Human High-Grade Serous Ovarian Cancer. Cell. 166 (3), 755-765 (2016).
- Zhang, B., et al. Proteogenomic characterization of human colon and rectal cancer. Nature. 513 (7518), 382-387 (2014).
- Gaudet, S., Miller-Jensen, K. Redefining Signaling Pathways with an Expanding Single-Cell Toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Di Palma, S., Bodenmiller, B. Unraveling cell populations in tumors by single-cell mass cytometry. Current Opinion in Biotechnology. 31, 122-129 (2015).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Current Protocols in Immunology. , 11-20 (2012).
- Willison, K. R., Klug, D. R. Quantitative single cell and single molecule proteomics for clinical studies. Current Opinion in Biotechnology. 24 (4), 745-751 (2013).
- Lee, J. Y., et al. Detection of Head and Neck Cancer Based on Longitudinal Changes in Serum Protein Abundance. Cancer Epidemiology, Biomarkers & Prevention. 29 (8), 1665-1672 (2020).
- Shi, T., et al. Advancing the sensitivity of selected reaction monitoring-based targeted quantitative proteomics. Proteomics. 12 (8), 1074-1092 (2012).
- Shi, T., et al. Advances in targeted proteomics and applications to biomedical research. Proteomics. 16 (15-16), 2160-2182 (2016).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Molecular Systems Biology. 4, 222 (2008).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nature Methods. 9 (6), 555-566 (2012).
- Zhu, Y., et al. Proteomic Analysis of Single Mammalian Cells Enabled by Microfluidic Nanodroplet Sample Preparation and Ultrasensitive NanoLC-MS. Angewandte Chemie International Edition. 57 (38), 12370-12374 (2018).
- Cong, Y., et al. Improved Single-Cell Proteome Coverage Using Narrow-Bore Packed NanoLC Columns and Ultrasensitive Mass Spectrometry. Analytical Chemistry. 92 (3), 2665-2671 (2020).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9 (1), 882 (2018).
- Shao, X., et al. Integrated Proteome Analysis Device for Fast Single-Cell Protein Profiling. Analytical Chemistry. 90 (23), 14003-14010 (2018).
- Li, Z. Y., et al. Nanoliter-Scale Oil-Air-Droplet Chip-Based Single Cell Proteomic Analysis. Analytical Chemistry. 90 (8), 5430-5438 (2018).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19 (1), 161 (2018).
- Zhang, P., et al. Carrier-Assisted Single-Tube Processing Approach for Targeted Proteomics Analysis of Low Numbers of Mammalian Cells. Analytical Chemistry. 91 (2), 1441-1451 (2019).
- Shi, T., et al. Facile carrier-assisted targeted mass spectrometric approach for proteomic analysis of low numbers of mammalian cells. Communications Biology. 1, 103 (2018).
- Vitrinel, B., Iannitelli, D. E., Mazzoni, E. O., Christiaen, L., Vogel, C. Simple Method to Quantify Protein Abundances from 1000 Cells. ACS Omega. 5 (25), 15537-15546 (2020).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26 (7), 966-968 (2010).
- Shi, T., et al. Conservation of protein abundance patterns reveals the regulatory architecture of the EGFR-MAPK pathway. Science Signaling. 9 (436), 6 (2016).
- Addona, T. A., et al. Multi-site assessment of the precision and reproducibility of multiple reaction monitoring-based measurements of proteins in plasma. Nature Biotechnology. 27 (7), 633-641 (2009).
- He, J., et al. Analytical platform evaluation for quantification of ERG in prostate cancer using protein and mRNA detection methods. Journal of Translational Medicine. 13, 54 (2015).
- Shi, T., et al. Antibody-free, targeted mass-spectrometric approach for quantification of proteins at low picogram per milliliter levels in human plasma/serum. Proceedings of the National Academy of Sciences. 109 (38), 15395-15400 (2012).
- Shi, T., et al. Targeted quantification of low ng/mL level proteins in human serum without immunoaffinity depletion. Journal of Proteome Research. 12 (7), 3353-3361 (2013).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır