Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Однореакторная пробоподготовка с помощью носителей для таргетного протеомного анализа небольшого числа клеток человека
В этой статье
Резюме
Однореакторная пробоподготовка с помощью носителей белка в сочетании с жидкостной хроматографией (LC) - мониторингом выбранных реакций (SRM), называемой cLC-SRM, является удобным методом мультиплексного таргетного протеомного анализа небольшого числа клеток, включая одиночные клетки. Он извлекает выгоду из использования избыточного экзогенного белка в качестве носителя и высокоспецифичного LC-SRM для целевого количественного определения.
Аннотация
Анализ белков небольшого числа клеток человека в основном достигается с помощью таргетированной протеомики с иммунологическими анализами на основе антител, которые имеют присущие им ограничения (например, низкая мультиплексность и недоступность антител к новым белкам). Таргетная протеомика на основе масс-спектрометрии (МС) появилась в качестве альтернативы, поскольку она не содержит антител, обладает высокой мультиплексностью и высокой специфичностью и точностью количественного анализа. Последние достижения в области аппаратуры для РС делают возможной таргетную протеомику на основе РС для мультиплексного количественного определения высокораспространенных белков в отдельных клетках. Тем не менее, существует техническая проблема эффективной обработки одиночных клеток с минимальными потерями образца для анализа МС. Чтобы решить эту проблему, мы недавно разработали удобную однокамерную пробоподготовку с помощью носителей белка в сочетании с жидкостной хроматографией (LC) - выбранным мониторингом реакций (SRM), называемой cLC-SRM, для целевого протеомного анализа небольшого количества клеток человека. Этот метод основан на использовании комбинированного избыточного экзогенного белка в качестве носителя и малообъемной однореакторной обработке для значительного снижения поверхностных потерь на адсорбции, а также на высокоспецифичном LC-SRM для эффективного решения проблемы повышенного динамического диапазона концентраций из-за добавления экзогенного белка-носителя. Его полезность была продемонстрирована точным количественным определением наиболее умеренно распространенных белков в небольшом количестве клеток (например, 10-100 клеток) и высокораспространенных белков в отдельных клетках. Простота реализации функций и отсутствие необходимости в специальных устройствах делают этот метод легко доступным для большинства лабораторий протеомики. В данной работе мы предоставили подробный протокол для анализа cLC-SRM небольшого числа клеток человека, включая сортировку клеток, лизис и переваривание клеток, анализ LC-SRM и анализ данных. Необходимо дальнейшее повышение чувствительности обнаружения и пропускной способности образца для целевого протеомного анализа одиночных клеток. Мы ожидаем, что cLC-SRM будет широко применяться в биомедицинских исследованиях и системной биологии с потенциалом для содействия точной медицине.
Введение
Последние технологические достижения в области геномики (транскриптомики) позволяют проводить всесторонний и точный анализ генома (транскриптома) в отдельных клетках 1,2,3. Тем не менее, технологии протеомики одиночных клеток сильно отстают, но не менее важны, чем технологии геномики (транскриптомики) 4,5,6,7,8. Кроме того, распространенность белка не обязательно может быть выведена из обилия мРНК9, и протеом является более сложным и динамичным, чем транскриптом10. Учитывая эти проблемы, большое количество смешанных популяций клеток (т.е. объемных клеток) обычно используется для получения всеобъемлющих данных протеома 11,12,13. Тем не менее, такие объемные измерения усредняют стохастические вариации отдельных клеток, тем самым скрывая важную межклеточную изменчивость (т.е. гетерогенность клеток)4,14. Ограничения таких объемных измерений становятся еще более серьезными, когда на представляющие интерес клетки приходится лишь небольшая часть от общего числа популяций клеток (например, раковые стволовые клетки в опухолях на ранней стадии рака). Таким образом, существует огромный разрыв в знаниях между протеомикой одиночных клеток и геномикой (транскриптомикой).
Иммунологические анализы на основе антител (например, проточная или массовая цитометрия) преимущественно используются для таргетного протеомного анализа одиночных клеток 6,7,15,16,17,18. Тем не менее, они страдают от низкой мультиплексности, ограниченной специфичности и недоступности антител к новым белкам, представляющим интерес. Таргетная протеомика на основе масс-спектрометрии (МС) стала альтернативой точному количественному определению белка благодаря отсутствию антител, высокой мультиплексности (≥150 белков в одноманализе19), высокой точности количественного определения (абсолютные количества или концентрации), а также высокой специфичности и воспроизводимости (≤10% CV)20,21,22,23. Недавний значительный прогресс в подготовке образцов сделал возможным протеомику одиночных клеток на основе РС для количественного анализа широко распространенных белков из отдельных клеток человека. Тем не менее, протеомика одиночных клеток на основе рассеянного склероза все еще находится на ранней стадии младенчества. Например, самая передовая платформа МС в сочетании со сверхнизкими скоростями потока RPLC может обеспечить обнаружение и количественное определение МС без меток только ~670-870 белков из общего числа ≥12 000 белков в одиночных клетках HeLa24,25.
В настоящее время для анализа одиночных клеток млекопитающих доступно шесть подходов к протеомике одиночных клеток на основе МС, четыре из которых предназначены для глобальной протеомики (nanoPOTS: nano-well based Preparation in One pot for Trace Samples26; iPAD-1: интегрированное устройство для анализа протеома для анализа одиночных клеток27; OAD (oil-air-droplet) протеомный анализ одиночных клеток на основе чипов28; SCoPE-MS: протеомика одиночных клеток методом масс-спектрометрии29), а два других предназначены для целевой протеомики (cLC-SRM: жидкостная хроматография с использованием носителей (LC) - selected reaction monitoring (SRM)30; cPRISM-SRM: разделение высокого давления с высоким разрешением и интеллектуальным отбором и мультиплексированием в сочетании с SRM31). Однако все эти подходы имеют технические недостатки. nanoPOTS, iPAD-1 и OAD уменьшают объем обработки образцов до 2-200 нл и не готовы к широкому настольному применению 26,27,28. Для SCoPE-MS после обработки одиночных клеток добавляется носитель TMT (тандемная метка массы), поэтому он не может эффективно предотвращать поверхностные адсорбционные потери во время обработки образца, когда для обработки одиночных клеток используется одна пробирка29, что приводит к низкой воспроизводимости с коэффициентом корреляции всего ~0,2-0,4 между репликами32. Для cLC-SRM и cPRISM-SRM использование экзогенных белков в качестве носителя больше подходит для таргетной протеомики, поскольку пептиды из избыточного количества экзогенных белков часто секвенируются MS/MS, что значительно снижает вероятность секвенирования низкораспространенных эндогенных пептидов30,31. В отличие от глобальной протеомики для относительного количественного определения, два целевых подхода к протеомике могут обеспечить точный или абсолютный анализ белка небольшого числа клеток с высокой воспроизводимостью с использованием тяжелых изотопных меченных внутренних стандартов в известных концентрациях. По сравнению с cPRISM-SRM, который требует предварительного фракционирования PRISM с высоким разрешением, в результате чего необходимо анализировать множество образцов фракций, cLC-SRM имеет значительное преимущество в пропускной способности образцов без фракционирования и может одновременно количественно определять сотни белков в одном анализе, но с относительно более низкой чувствительностью детектирования.. Таким образом, cLC-SRM более доступен и должен иметь более широкие возможности для точного мультиплексного анализа белков небольшого числа клеток, а также образцов с ограниченной массой.
В данной статье мы подробно описываем протокол выполнения cLC-SRM для удобного целевого протеомного анализа небольшого количества клеток человека, включая одиночные клетки. Протокол состоит из следующих основных этапов: сортировка клеток с помощью FACS (флуоресцентно активированная сортировка клеток), лизис и расщепление клеток, обработанные в пробирках для однополимеразной цепной реакции (ПЦР) в малом объеме, сбор данных LC-SRM и анализ данных SRM с использованием общедоступного программного обеспечения Skyline (рис. 1). Его широкая полезность была продемонстрирована наряду с нашими ранее хорошо зарекомендовавшими себя анализами SRM путем абсолютного целевого количественного определения белков пути EGFR/MAPK в клетках 1-100 MCF7 или MCF10A и определения копий белков пути на клетку в широком динамическом диапазоне концентраций30. Мы ожидаем, что с подробным протоколом большинство исследователей протеомики смогут легко внедрить cLC-SRM в своих лабораториях для точного анализа белков ультрамалых образцов (например, редких опухолевых клеток) в соответствии с потребностями своего проекта.
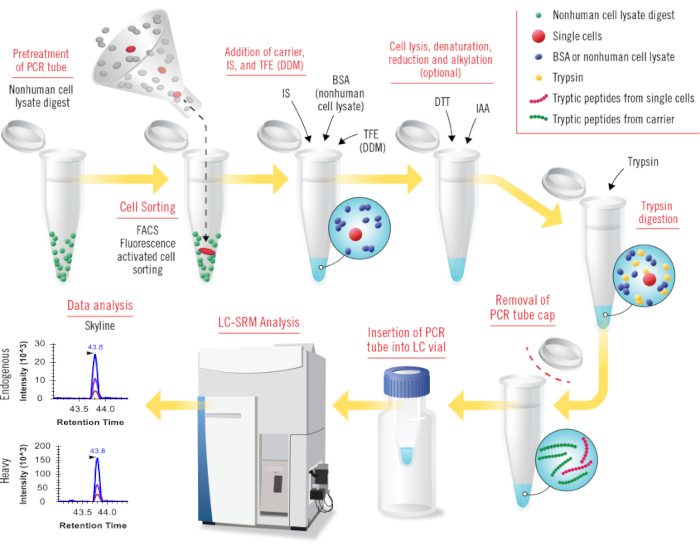
Рисунок 1: Обзор всех этапов cLC-SRM (подготовкаобразцов с помощью одного горшка в сочетании с liquid chromatography -sselected reaction monitoring). Лизаты нечеловеческих клеток (например, Shewanella oneidensis) используются для предварительной обработки ПЦР-пробирок для покрытия поверхности пробирок. Небольшое количество человеческих клеток или отдельных клеток, отсортированных с помощью FACS, собираются в предварительно обработанные ПЦР-пробирки. Носитель белка BSA (или протеома лизата клеток), тяжелый внутренний стандарт (IS) и TFE (или DDM) добавляются в пробирки с образцами последовательно для облегчения лизиса клеток и снижения потерь на поверхностную адсорбцию. Концептуально, комбинированный DDM и переносчик протеома нечеловеческого клеточного лизата будут хорошо работать для cLC-SRM. Лизис клеток проводится путем ультразвуковой обработки, а денатурация белков достигается путем нагревания при высокой температуре. Реагенты DTT и IAA используются для восстановления и алкилирования соответственно (этот этап необязателен). Трипсин добавляется для переваривания с гораздо более высоким соотношением трипсина к количеству белка, чем при стандартном переваривании трипсина. Крышка пробирки с образцом снимается, а затем пробирка для ПЦР вставляется в флакон с ЛК для прямого анализа ЖХ-СРМ. Собранные данные SRM анализируются с помощью общедоступного программного обеспечения Skyline. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
протокол
ПРИМЕЧАНИЕ: Пошаговый анализ cLC-SRM показан на рисунке 1.
1. Предварительная обработка ПЦР-пробирок
- Добавьте в ПЦР-пробирки 100 мкл нечеловеческого (например, Shewanella oneidensis) клеточного лизата в концентрации 0,2 мкг/мкл. Инкубируйте при комнатной температуре в течение ночи, чтобы покрыть поверхность ПЦР-пробирки.
- Удалите расщепленные клеточные лизаты с помощью пипетирования, промойте ПЦР-пробирки водой класса ВЭЖХ 3 раза, а затем высушите ПЦР-пробирки на воздухе в вытяжном шкафу.
- Храните ПЦР-пробирки с покрытием в морозильной камере при температуре 4 °C до дальнейшего использования.
2. Сортировка FACS
- Совместите флуоресцентно-активируемый сортировщик клеток (FACS) (см. Таблицу материалов) в 96-луночный ПЦР-планшет с использованием флуоресцентных шариков с красителями, которые могут возбуждаться лазерным источником для получения надежных эталонных сигналов.
- Убедитесь, что машина FACS хорошо предназначена для сортировки ячеек в нижней части ПЦР-планшетов.
- Установите угол потока сортировки на минимально возможный, чтобы максимизировать вероятность того, что ячейки будут сортироваться на дне лунок, а не по бокам.
- Чтобы навести машину, используйте тестовый поток для «сортировки» капель PBS-буфера (например, 100 капель) на лунку пустой пластины, чтобы убедиться, что собранные капли находятся на дне. Если капли не попадают в нужное положение, повторно откалибруйте сортировщик клеток и повторяйте эти действия, пока капли в потоке сортировки не будут собраны правильно.
- Поместите ПЦР-пробирки с покрытием в штатив для ПЦР-пробирок на 96 лунок.
- Отсортируйте желаемое количество клеток молочной железы человека (MCF7 или MCF10A) в предварительно покрытые пробирки для ПЦР.
- Немедленно центрифугируйте отсортированные клетки молочной железы при давлении 100 x g в течение 10 минут при 4 °C, чтобы они оставались на дне пробирки во избежание потенциальной потери клеток.
- Храните отсортированные ячейки в морозильной камере при температуре -80 °C до дальнейшего анализа.
3. Добавление белкового носителя, тяжелых внутренних стандартов и TFE
- Добавьте 4 мкл 25 мМ NH4HCO3 в ПЦР-пробирки, содержащие собранные клетки.
- Добавьте 1 мкл 10 нг/мкл бычьего сывороточного альбумина (БСА) в 25 мМ NH4HCO3 в пробирки для проб.
- Добавьте 0,3 мкл 100 фмоль/мкл стандартов сырых тяжелых пептидов (всего 30 фмоль) в пробирки для образцов.
ПРИМЕЧАНИЕ: Аликвоты стандартов сырых тяжелых пептидов для белков пути EGFR/MAPK получают в номинальной концентрации 100 фмоль/мкл в 25 мМ NH4HCO3. Диспонируйте небольшой объем сырого стандартного тяжелого пептида в концентрации 100 фмоль/мкл в середину раствора и убедитесь, что в наконечнике пипетки нет остатков жидкости. - Добавьте 9 мкл 100% TFE в пробирки для образцов с конечной концентрацией ~60% TFE.
- Центрифугируйте при 1500 x g в течение 5 минут, а затем осторожно переваривайте при 100 x g в течение 3 минут.
4. Лизис клеток и денатурация белков
- Отсортированные с помощью FACS ультразвуком клетки молочной железы на льду в течение 1 минуты с интервалом 1 минута, всего 5 циклов с амплитудой 70% и частотой 0,5 Гц.
- Инкубируйте образцы при температуре 90 °C в течение 1 ч для денатурации белка с помощью термоамплификатора ПЦР с опцией нагрева крышки.
ПРИМЕЧАНИЕ: TFE является летучим растворителем с температурой кипения 74 °C, поэтому вариант с нагреваемой крышкой выбран таким образом, чтобы избежать полного высыхания со значительными потерями образца. - Охладите образцы до комнатной температуры с помощью центрифугирования при 1500 x g в течение 3 минут.
5. Восстановление и алкилирование (по желанию)
- Добавьте 0,6 мкл 50 мМ DTT для получения конечной концентрации 2 мМ.
ПРИМЕЧАНИЕ: Решение DTT каждый раз должно быть свежеприготовленным. Взвесьте 77,5 мг DTT, а затем растворите в 1 мл 25 мл NH4HCO3 для получения 500 мл DTT. Затем 10 мкл 500 мМ DTT добавляют к 90 мкл 25 мМ NH4HCO3 для получения 50 мМ DTT. - Центрифугируйте при 1500 x g в течение 5 минут, а затем осторожно перемешивайте при 850 об/мин в течение 3 минут.
- Выдерживать при температуре 56°C в течение 1 ч, осторожно встряхивая при 100 x g.
- Охладите образцы до комнатной температуры и центрифугируйте при 1500 x g в течение 3 минут.
- Добавьте 0,5 мкл 60 мМ IAA в пробирку ОТ-ПЦР с конечной концентрацией ~2 мМ.
ПРИМЕЧАНИЕ: Решение IAA каждый раз должно быть свежеприготовленным. Взвесьте 74,3 мг IAA, а затем растворите в 1 мл 25 мл NH4HCO3 для получения 400 мМ IAA. 15 мкл 400 мМ IAA добавляют к 85 мкл 25 мМ NH4HCO3 для получения 60 мМ IAA. - Выдерживать в темноте при комнатной температуре в течение 30 минут, осторожно встряхивая при 100 х г.
Примечание: Этапы восстановления и алкилирования являются необязательными, если для белков-мишеней не выбраны цистеинсодержащие пептиды.
6. Усвоение трипсина
- Уменьшите объем образца до ~4 мкл с помощью концентратора SpeedVac.
ПРИМЕЧАНИЕ: TFE является летучим растворителем с температурой кипения при 74°C. Высокий процент TFE подавляет переваривание трипсина. - Добавьте 9 мкл 25 мМ NH4HCO3 и 1-3 мкл 15 нг/мкл трипсина для переваривания с конечной концентрацией трипсина 1-3 нг/мкл.
ПРИМЕЧАНИЕ: Для 10 отсортированных с помощью FACS клеток молочной железы человека (~1 нг) соотношение фермента трипсина и белка составляет ≥15:1, что в 750 раз выше, чем при стандартном расщеплении трипсина (соотношение 1:50). - Осторожно перемешайте при 100 x g в течение 3 минут и выдерживайте в течение ночи (~16 часов) при температуре 37 °C.
- Добавьте 0,5 мкл 5% муравьиной кислоты, чтобы остановить ферментативную реакцию.
- Центрифуга в течение 1 ч при 1500 x g.
- Хранить в морозильной камере при температуре -80 °C до дальнейшего анализа LC-SRM или в морозильной камере при температуре 4 °C для немедленного анализа.
7. Подготовка к прямому анализу LC-SRM
- Снимите колпачок с ПЦР-пробирки, которая использовалась для сбора (Шаг 2) и обработки (Шаг 6) клеток молочной железы, отсортированных с помощью FACS.
- Вставьте ПЦР-пробирку во флакон LC.
- Закройте флакон LC для анализа LC-SRM.
ПРИМЕЧАНИЕ: Для небольшого количества клеток, обработанных в 96-луночном ПЦР-планшете, будет добавлена крышка коврика, чтобы герметизировать планшет для прямого анализа LC-SRM.
8. Анализ LC-SRM
- Анализируйте образцы с помощью UPLC, соединенного с тройным квадрупольным масс-спектрометром.
- Соедините капиллярные колонки C18 (внутренний диаметр 75 μм × 20 см для стандартного градиента и внутренний диаметр 100 μм × 10 см для короткого градиента) к химически вытравленному электронапыляемому излучателю с внутренним диаметром 20 μм плавленого кремнезема с помощью соединения из нержавеющей стали.
- Используйте контур выборки объемом 20 мкл для прямой загрузки всего образца (всего ~15 мкл) в колонку LC для максимальной чувствительности обнаружения.
- Используйте 0,1% муравьиной кислоты в воде и 0,1% муравьиной кислоты в 90% ацетонитриле в качестве подвижных фаз А и В для разделения капиллярного RPLC.
- Используйте бинарный градиент LC при скорости потока 300 нл/мин для стандартного градиента при измерении большого количества целевых белков (>10). Стандартный градиент: 5-20% В за 26 мин, 20-25% В за 10 мин, 25-40% В за 8 мин, 40-95% В за 1 мин и при 95% В за 7 мин всего 52 мин и аналитическая колонка повторно уравновешивается при 99,5% А в течение 8 мин.
- Используйте бинарный градиент LC при скорости потока 400 нл/мин для короткого градиента, когда измеряется небольшое количество белков-мишеней (≤10). Короткий градиент для быстрой сепарации: 5-95% B за 5 мин и повторное выравнивание аналитической колонны при 99,5% А в течение 5 мин с общим временем работы LC 10 мин.
- Масс-спектрометр QQQ работает с напряжением ионного распыления 2 400 ± 100 В, напряжением капиллярного смещения 35 В, напряжением смещения скиммера -5 В и температурой капиллярного входа 220° C.
- Получение других параметров QQQ MS в результате автоматической настройки и калибровки без дополнительной оптимизации.
- Увеличьте различные концентрации стандартных тяжелых пептидов сырой нефти в 20-50 нг/мкл перевариваемых лизатов клеток, не являющихся человеческими, перед анализом образцов LC-SRM для определения времени удержания пептида LC (RT) для построения запланированного метода SRM и потенциальных интерференционных переходов для точного количественного определения.
- Используйте запланированный режим SRM RT для количественного определения мультиплексирования с окном сканирования ≥6 мин.
ПРИМЕЧАНИЕ: Окно сканирования будет настроено соответствующим образом в зависимости от количества переходов в определенном временном окне. - Установите общее время цикла равным 1 с, и время задержки для каждого перехода автоматически корректируется в зависимости от количества переходов, отсканированных в разных временных окнах. Минимальное время задержки в 10 мс требуется для каждого перехода SRM. Все целевые белки можно одновременно контролировать в одном анализе LC-SRM
9. Анализ данных
- Импортируйте файлы необработанных данных в общедоступное программное обеспечение Skyline33 для визуализации данных каждого целевого пептида с целью определения их обнаруживаемости.
- Используйте два критерия для определения обнаружения и интеграции пиковых значений SRM: 1) одинаковое время удержания LC; 2) примерно одинаковые относительные отношения пиковой интенсивности SRM при множественных переходах между эндогенными (легкими) пептидами и их соответствующими (тяжелыми) внутренними стандартами меченых изотопами пептидов.
- Проверьте все данные SRM вручную, чтобы убедиться в правильном назначении пика SRM и границе пика SRM для надежной количественной оценки с использованием двух вышеуказанных критериев.
- Используйте наилучший переход с высоким сигналом SRM, но без матричной интерференции для количественного определения белка, и используйте другие переходы в качестве эталона.
- Используйте несколько репликаций для получения стандартного отклонения (SD) и коэффициента вариации (CV).
- Рассчитайте отношение сигнал/шум (S/N) по пиковой интенсивности пика над средним фоновым шумом в области времени удержания ±15 с для стандартного градиента и ±6 с для короткого градиента. Предел обнаружения (LOD) и предел количественной оценки (LOQ) определяются как самые низкие точки концентрации, при которых отношение S/N составляет не менее 3 и 7 соответственно.
- Примените еще один критерий для консервативного определения значений LOQ, помимо S/N ≥7: суррогатный пептидный ответ на различные номера клеток должен находиться в линейном динамическом диапазоне.
- Используйте программное обеспечение Excel для построения всех калибровочных кривых. Загрузите необработанные данные из QQQ MS в общедоступное программное обеспечение Skyline для отображения графиков хроматограмм экстрагированных ионов (XIC) нескольких переходов целевых белков.
Результаты
Небольшие количества клеточных лизатов MCF7 (0,5-20 нг, что эквивалентно 5-200 клеткам) были впервые использованы для оценки эффективности cLC-SRM путем целенаправленного количественного определения белков пути EGFR/MAPK, поскольку они более однородны с меньшими...
Обсуждение
cLC-SRM — это удобный метод таргетной протеомики, который позволяет проводить точный мультиплексный анализ белков небольшого числа клеток, включая одиночные клетки. Этот метод основан на однореакторной пробоподготовке с помощью носителей белка, в которой все этапы, вк?...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа была поддержана грантами NIH R21CA223715 (TS) и UG3CA256967 (TS и HL). Описанная здесь экспериментальная работа была выполнена в Лаборатории молекулярных наук об окружающей среде Тихоокеанской северо-западной национальной лаборатории, национальном научном пользовательском объекте, спонсируемом Министерством энергетики Соединенных Штатов Америки в соответствии с контрактом DE-AC05-76RL0 1830.
Материалы
| Name | Company | Catalog Number | Comments |
| 2 mL glass LC vial | Microsolv | 9502S-WCV | Vessel to hold PCR tube for autosample injection |
| BSA | Sigma-Aldrich | P0834-10×1mL | Carrier protein for greatly reducing surface adsorption losses |
| DTT | Thermo Scientific | A39255 | Reagent for reduction |
| Formic acid | Thermo Scientific | 28905 | Reagent for stopping enzyme reaction |
| IAA | Thermo Scientific | A39271 | Reagent for alkylation |
| Peptide internal standards | New England peptide | Targeted quantification of EGFR/MAPK pathway proteins | |
| RT-PCR tube | GeneMate Bioexpress | T-3035-1 | 0.2 mL PCR tube for one-pot sample preparation |
| Skyline software | University of Washington | Publicly available for SRM data analysis | |
| Sonicator | Hielscher Ultrasound Technology | UTR200 | Sonication on ice for cell lysis |
| Speed Vac concentrator | Thermo Scientific | Reduction of the percentage of TFE for effective trypsin digestion | |
| TFE | Sigma-Aldrich | 18370-10×1mL | 60% TFE for cell lysis |
| Thermocycler w/ heated lid | Peltier Thermal Cycler | PTC-200 | Heating for protein denaturation |
| Trypsin Gold | Promega | V528A | Enzyme for protein digestion |
| Waters BEH C18 column | Waters | C18 column for peptide separation |
Ссылки
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Miyamoto, D. T., et al. RNA-Seq of single prostate CTCs implicates noncanonical Wnt signaling in antiandrogen resistance. Science. 349 (6254), 1351-1356 (2015).
- Wang, Y., Navin, N. E. Advances and applications of single-cell sequencing technologies. Molecular Cell. 58 (4), 598-609 (2015).
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends in Genetics. 33 (2), 155-168 (2017).
- Gavasso, S., Gullaksen, S. E., Skavland, J., Gjertsen, B. T. Single-cell proteomics: potential implications for cancer diagnostics. Expert Review of Molecular Diagnostics. 16 (5), 579-589 (2016).
- Wu, M., Singh, A. K. Single-cell protein analysis. Current Opinion in Biotechnology. 23 (1), 83-88 (2012).
- Hughes, A. J., et al. Single-cell western blotting. Nature Methods. 11 (7), 749-755 (2014).
- Peterson, V. M., et al. Multiplexed quantification of proteins and transcripts in single cells. Nature Biotechnology. 35 (10), 936-939 (2017).
- Liu, Y., Beyer, A., Aebersold, R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. 165 (3), 535-550 (2016).
- Choudhary, C., Mann, M. Decoding signalling networks by mass spectrometry-based proteomics. Nature Reviews Molecular Cell Biology. 11 (6), 427-439 (2010).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Zhang, H., et al. Integrated Proteogenomic Characterization of Human High-Grade Serous Ovarian Cancer. Cell. 166 (3), 755-765 (2016).
- Zhang, B., et al. Proteogenomic characterization of human colon and rectal cancer. Nature. 513 (7518), 382-387 (2014).
- Gaudet, S., Miller-Jensen, K. Redefining Signaling Pathways with an Expanding Single-Cell Toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Di Palma, S., Bodenmiller, B. Unraveling cell populations in tumors by single-cell mass cytometry. Current Opinion in Biotechnology. 31, 122-129 (2015).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Current Protocols in Immunology. , 11-20 (2012).
- Willison, K. R., Klug, D. R. Quantitative single cell and single molecule proteomics for clinical studies. Current Opinion in Biotechnology. 24 (4), 745-751 (2013).
- Lee, J. Y., et al. Detection of Head and Neck Cancer Based on Longitudinal Changes in Serum Protein Abundance. Cancer Epidemiology, Biomarkers & Prevention. 29 (8), 1665-1672 (2020).
- Shi, T., et al. Advancing the sensitivity of selected reaction monitoring-based targeted quantitative proteomics. Proteomics. 12 (8), 1074-1092 (2012).
- Shi, T., et al. Advances in targeted proteomics and applications to biomedical research. Proteomics. 16 (15-16), 2160-2182 (2016).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Molecular Systems Biology. 4, 222 (2008).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nature Methods. 9 (6), 555-566 (2012).
- Zhu, Y., et al. Proteomic Analysis of Single Mammalian Cells Enabled by Microfluidic Nanodroplet Sample Preparation and Ultrasensitive NanoLC-MS. Angewandte Chemie International Edition. 57 (38), 12370-12374 (2018).
- Cong, Y., et al. Improved Single-Cell Proteome Coverage Using Narrow-Bore Packed NanoLC Columns and Ultrasensitive Mass Spectrometry. Analytical Chemistry. 92 (3), 2665-2671 (2020).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9 (1), 882 (2018).
- Shao, X., et al. Integrated Proteome Analysis Device for Fast Single-Cell Protein Profiling. Analytical Chemistry. 90 (23), 14003-14010 (2018).
- Li, Z. Y., et al. Nanoliter-Scale Oil-Air-Droplet Chip-Based Single Cell Proteomic Analysis. Analytical Chemistry. 90 (8), 5430-5438 (2018).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19 (1), 161 (2018).
- Zhang, P., et al. Carrier-Assisted Single-Tube Processing Approach for Targeted Proteomics Analysis of Low Numbers of Mammalian Cells. Analytical Chemistry. 91 (2), 1441-1451 (2019).
- Shi, T., et al. Facile carrier-assisted targeted mass spectrometric approach for proteomic analysis of low numbers of mammalian cells. Communications Biology. 1, 103 (2018).
- Vitrinel, B., Iannitelli, D. E., Mazzoni, E. O., Christiaen, L., Vogel, C. Simple Method to Quantify Protein Abundances from 1000 Cells. ACS Omega. 5 (25), 15537-15546 (2020).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26 (7), 966-968 (2010).
- Shi, T., et al. Conservation of protein abundance patterns reveals the regulatory architecture of the EGFR-MAPK pathway. Science Signaling. 9 (436), 6 (2016).
- Addona, T. A., et al. Multi-site assessment of the precision and reproducibility of multiple reaction monitoring-based measurements of proteins in plasma. Nature Biotechnology. 27 (7), 633-641 (2009).
- He, J., et al. Analytical platform evaluation for quantification of ERG in prostate cancer using protein and mRNA detection methods. Journal of Translational Medicine. 13, 54 (2015).
- Shi, T., et al. Antibody-free, targeted mass-spectrometric approach for quantification of proteins at low picogram per milliliter levels in human plasma/serum. Proceedings of the National Academy of Sciences. 109 (38), 15395-15400 (2012).
- Shi, T., et al. Targeted quantification of low ng/mL level proteins in human serum without immunoaffinity depletion. Journal of Proteome Research. 12 (7), 3353-3361 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены