Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Preparación de muestras en un recipiente asistida por portadores para el análisis proteómico dirigido de pequeñas cantidades de células humanas
En este artículo
Resumen
Una preparación de muestras asistida por transportadores de proteínas en un solo recipiente junto con cromatografía líquida (LC) - monitoreo de reacción seleccionada (SRM) denominada cLC-SRM es un método conveniente para el análisis proteómico dirigido multiplexado de pequeñas cantidades de células, incluidas las células individuales. Aprovecha el uso excesivo de proteínas exógenas como portadoras y LC-SRM de alta especificidad para la cuantificación dirigida.
Resumen
El análisis de proteínas de un pequeño número de células humanas se logra principalmente mediante proteómica dirigida con inmunoensayos basados en anticuerpos, que tienen limitaciones inherentes (por ejemplo, baja multiplexación y falta de disponibilidad de anticuerpos para nuevas proteínas). La proteómica dirigida basada en espectrometría de masas (MS) ha surgido como una alternativa porque está libre de anticuerpos, es de alta multiplexación y tiene una alta especificidad y precisión de cuantificación. Los avances recientes en la instrumentación de MS hacen posible la proteómica dirigida basada en MS para la cuantificación multiplexada de proteínas altamente abundantes en células individuales. Sin embargo, existe un reto técnico para el procesamiento eficaz de células individuales con una pérdida mínima de muestras para el análisis de MS. Para abordar este problema, hemos desarrollado recientemente una conveniente preparación de muestras asistida por transportadores de proteínas en un solo recipiente junto con cromatografía líquida (LC) - monitoreo de reacción seleccionada (SRM) denominado cLC-SRM para el análisis proteómico dirigido de pequeñas cantidades de células humanas. Este método aprovecha el uso de la proteína exógena excesiva combinada como portadora y el procesamiento de bajo volumen en un solo recipiente para reducir en gran medida las pérdidas de adsorción en la superficie, así como la LC-SRM de alta especificidad para abordar de manera efectiva el aumento del rango de concentración dinámica debido a la adición de proteína transportadora exógena. Su utilidad se ha demostrado mediante la cuantificación precisa de las proteínas más moderadamente abundantes en un pequeño número de células (por ejemplo, 10-100 células) y proteínas altamente abundantes en células individuales. Las características fáciles de implementar y la ausencia de necesidad de dispositivos específicos hacen que este método sea fácilmente accesible para la mayoría de los laboratorios de proteómica. Aquí hemos proporcionado un protocolo detallado para el análisis cLC-SRM de pequeñas cantidades de células humanas, incluida la clasificación de células, la lisis y digestión celular, el análisis LC-SRM y el análisis de datos. Se necesitan más mejoras en la sensibilidad de detección y el rendimiento de las muestras hacia el análisis proteómico unicelular dirigido. Anticipamos que cLC-SRM se aplicará ampliamente a la investigación biomédica y la biología de sistemas, con el potencial de facilitar la medicina de precisión.
Introducción
Los recientes avances tecnológicos en genómica (transcriptómica) permiten un análisis exhaustivo y preciso del genoma (transcriptoma) en células individuales 1,2,3. Sin embargo, las tecnologías de proteómica unicelular están muy rezagadas, pero son tan importantes como las tecnologías genómicas (transcriptómicas) 4,5,6,7,8. Además, la abundancia de proteínas no se puede inferir necesariamente de la abundancia de ARNm9, y el proteoma es más complejo y dinámico que el transcriptoma10. Dados estos desafíos, generalmente se utiliza un gran número de poblaciones mixtas de células (es decir, células a granel) para generar datos completos del proteoma 11,12,13. Sin embargo, tales mediciones masivas promedian las variaciones estocásticas de las células individuales, lo que oscurece una importante variabilidad de célula a célula (es decir, la heterogeneidad celular)4,14. Las limitaciones de tales mediciones masivas se vuelven aún más severas cuando las células de interés solo representan una pequeña porción de las poblaciones totales de células (por ejemplo, células madre cancerosas dentro de tumores en una etapa temprana de cáncer). Por lo tanto, existe una gran brecha de conocimiento entre la proteómica unicelular y la genómica (transcriptómica).
Los inmunoensayos basados en anticuerpos (p. ej., citometría de flujo o de masas) se utilizan principalmente para el análisis proteómico dirigido de células individuales 6,7,15,16,17,18. Sin embargo, sufren de baja multiplexación, especificidad limitada y falta de disponibilidad de anticuerpos para nuevas proteínas de interés. La proteómica dirigida basada en espectrometría de masas (MS) ha surgido como una alternativa para la cuantificación precisa de proteínas debido a que está libre de anticuerpos, es de alta multiplexación (≥150 proteínas en un solo análisis19), tiene una alta precisión de cuantificación (cantidades o concentraciones absolutas) y es de alta especificidad y reproducibilidad (≤10% CV)20,21,22,23. Los recientes avances significativos en la preparación de muestras han hecho posible la proteómica unicelular basada en MS para el análisis cuantitativo de proteínas muy abundantes a partir de células humanas individuales. Sin embargo, la proteómica unicelular basada en la EM aún se encuentra en una etapa temprana de infancia. Por ejemplo, la plataforma de MS más avanzada, junto con caudales de RPLC de flujo ultra bajo, solo puede permitir la detección y cuantificación de MS sin etiquetas de ~ 670-870 proteínas del total de ≥ 12,000 proteínas en células HeLa individuales24,25.
En la actualidad, hay seis enfoques proteómicos unicelulares basados en MS disponibles para el análisis de células individuales de mamíferos, de los cuales cuatro son para la proteómica global (nanoPOTS: nanowell based Preparation in One pot for Trace Samples26; iPAD-1: dispositivo integrado de análisis de proteoma para el análisis de células individuales27; análisis proteómico de una sola célula basado en chips OAD (gotas de aceite)28; SCoPE-MS: proteómica unicelular por espectrometría de masas29) y las otras dos son para proteómica dirigida (cLC-SRM: cromatografía líquida asistida por portadores (LC) - monitorización de reacciones seleccionadas (SRM)30; cPRISM-SRM: separaciones de alta presión y alta resolución asistidas por portadores con selección inteligente y multiplexación acopladas a SRM31). Sin embargo, todos estos enfoques tienen inconvenientes técnicos. nanoPOTS, iPAD-1 y OAD reducen el volumen de procesamiento de muestras a 2-200 nL y no están preparados para aplicaciones de sobremesa amplias 26,27,28. Para SCoPE-MS, se agrega un portador TMT (etiqueta de masa en tándem) después del procesamiento de una sola celda, por lo que no puede prevenir eficazmente las pérdidas de adsorción superficial durante el procesamiento de muestras cuando se usa un solo tubo para el procesamiento de una sola celda29, lo que resulta en una baja reproducibilidad con un coeficiente de correlación de solo ~ 0.2-0.4 entre las réplicas32. En el caso de cLC-SRM y cPRISM-SRM, el uso de proteínas exógenas como portador es más adecuado para la proteómica dirigida, ya que los péptidos de proteínas exógenas excesivas son frecuentemente secuenciados por MS/MS, lo que reduce en gran medida la posibilidad de secuenciación de péptidos endógenos de baja abundancia30,31. A diferencia de la proteómica global para la cuantificación relativa, los dos enfoques de proteómica dirigida pueden proporcionar un análisis de proteínas preciso o absoluto de un pequeño número de células con alta reproducibilidad utilizando estándares internos marcados con isótopos pesados a concentraciones conocidas. En comparación con cPRISM-SRM que requiere un fraccionamiento previo de PRISM de alta resolución, lo que da como resultado muchas muestras de fracción que necesitan ser analizadas, cLC-SRM tiene una ventaja significativa en el rendimiento de muestras sin fraccionamiento y puede cuantificar simultáneamente cientos de proteínas en un solo análisis, pero con una sensibilidad de detección relativamente menor30. Por lo tanto, cLC-SRM es más accesible y debería tener utilidades más amplias para el análisis preciso de proteínas multiplexadas de pequeñas cantidades de células, así como de muestras de masa limitada.
En este trabajo describimos un protocolo detallado para realizar cLC-SRM para un análisis proteómico dirigido conveniente de pequeñas cantidades de células humanas, incluidas las células individuales. El protocolo consta de los siguientes pasos principales: clasificación celular por FACS (clasificación celular activada por fluorescencia), lisis celular y digestión procesada en tubos de reacción en cadena de polimerasa (PCR) de bajo volumen, recopilación de datos LC-SRM y análisis de datos SRM utilizando el software Skyline disponible públicamente (Figura 1). Su amplia utilidad se demostró junto con nuestros ensayos SRM previamente bien establecidos mediante la cuantificación absoluta dirigida de las proteínas de la vía EGFR/MAPK en células 1-100 MCF7 o MCF10A y la determinación de copias de proteínas de la vía por célula en un amplio rango dinámico de concentraciones30. Anticipamos que con el protocolo detallado, la mayoría de los investigadores de proteómica pueden implementar fácilmente cLC-SRM en sus laboratorios para el análisis preciso de proteínas de muestras ultrapequeñas (por ejemplo, células tumorales raras) para satisfacer las necesidades de su proyecto.
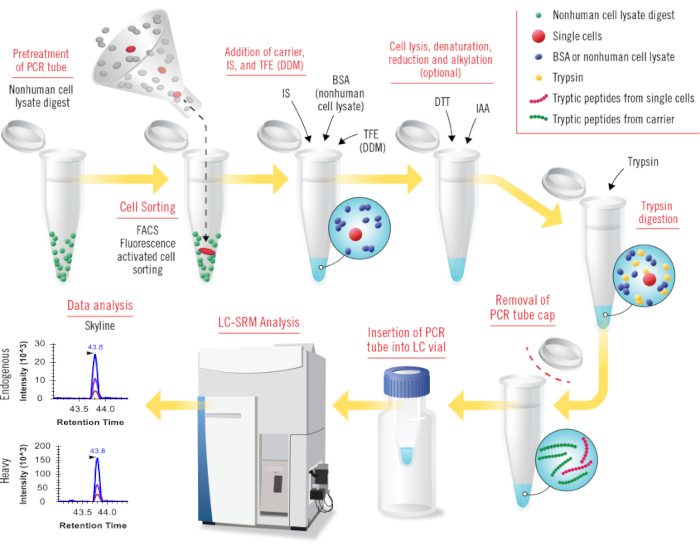
Figura 1: Resumen de todos los pasos de la cLC-SRM (preparación de muestras en un solo recipiente asistida por c arado junto con lidídica hromatografía -s monitora de reacción elegida).Los digestores de lisado de células no humanas (por ejemplo, Shewanella oneidensis) se utilizan para pretratar los tubos de PCR para recubrir la superficie del tubo. Pequeñas cantidades de células humanas o células individuales clasificadas por FACS se recogen en tubos de PCR pretratados. El portador de proteína BSA (o proteoma de lisado de células no humanas), el estándar interno pesado (IS) y el TFE (o DDM) se añaden a los tubos de muestra de forma secuencial para facilitar la lisis celular y reducir las pérdidas de adsorción superficial. Conceptualmente, el portador combinado del proteoma de DDM y lisado de células no humanas funcionará bien para cLC-SRM. La lisis celular se lleva a cabo mediante sonicación, y la desnaturalización de las proteínas se logra mediante calentamiento a alta temperatura. Los reactivos DTT e IAA se utilizan para la reducción y la alquilación, respectivamente (este paso es opcional). La tripsina se agrega para la digestión con proporciones mucho más altas de tripsina sobre la cantidad de proteínas que para la digestión estándar de tripsina. Se retira la tapa del tubo de muestra y luego se inserta el tubo de PCR en el vial de LC para el análisis directo de LC-SRM. Los datos SRM recopilados se analizan mediante el uso del software Skyline disponible públicamente. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
NOTA: El análisis cLC-SRM paso a paso se muestra en la Figura 1.
1. Pretratamiento de tubos de PCR
- Añadir 100 μL de digestión de lisado de células no humanas (p. ej., Shewanella oneidensis) a 0,2 μg/μL a los tubos de PCR. Incubar a temperatura ambiente durante toda la noche para cubrir la superficie del tubo de PCR.
- Retire los digestores de lisado celular mediante pipeteo, enjuague los tubos de PCR con agua de grado HPLC 3 veces y luego seque los tubos de PCR al aire en una campana extractora.
- Guarde los tubos de PCR recubiertos en un congelador a 4 °C hasta su uso posterior.
2. Clasificación FACS
- Alinee un clasificador de células activadas por fluorescencia (FACS) (consulte la Tabla de materiales) en una placa de PCR de 96 pocillos utilizando perlas fluorescentes con tintes que pueden ser excitados por la fuente láser para proporcionar señales de referencia confiables.
- Verifique que la máquina FACS esté bien orientada para clasificar las células en la parte inferior de las placas de PCR.
- Establezca el ángulo del flujo de clasificación al mínimo posible para maximizar la tasa de éxito de que las celdas se ordenen en el fondo de los pozos en lugar de golpear los lados.
- Para apuntar la máquina, use el flujo de prueba para "clasificar" las gotas de tampón PBS (por ejemplo, 100 gotas) en el pozo de una placa vacía para asegurarse de que las gotas recolectadas estén en el fondo. Si las gotas no caen en la posición correcta, vuelva a calibrar el clasificador de celdas y repita hasta que las gotas en el flujo de clasificación se recojan correctamente.
- Coloque los tubos de PCR recubiertos en una gradilla de tubos de PCR de 96 pocillos.
- Clasifique la cantidad deseada de células mamarias humanas (MCF7 o MCF10A) en tubos de PCR prerrecubiertos.
- Centrifugar inmediatamente las células mamarias clasificadas a 100 x g durante 10 minutos a 4 °C para mantenerlas en el fondo del tubo y evitar una posible pérdida de células.
- Guarde las celdas clasificadas en un congelador a -80 °C hasta su posterior análisis.
3. Adición de portador de proteínas, patrones internos pesados y TFE
- Añadir 4 μL de 25 mM de NH4HCO3 en los tubos de PCR que contienen las células recogidas.
- Añadir 1 μL de 10 ng/μL de albúmina sérica bovina (BSA) en 25 mM de NH4HCO3 en los tubos de muestra.
- Agregue 0.3 μL de 100 fmol/μL de patrones de péptidos pesados crudos (30 fmol en total) en los tubos de muestra.
NOTA: Las alícuotas de los patrones de péptidos pesados crudos para las proteínas de la vía EGFR/MAPK se elaboran a una concentración nominal de 100 fmol/μL en 25 mM de NH4HCO3. Dispense el pequeño volumen de patrones de péptidos pesados crudos a 100 fmol/μL en el centro de la solución y asegúrese de que no queden restos de líquido en la punta de la pipeta. - Agregue 9 μL de 100% TFE en los tubos de muestra con la concentración final de ~60% TFE.
- Centrifugar a 1500 x g durante 5 min y luego vórtice suavemente a 100 x g durante 3 min.
4. Lisis celular y desnaturalización de proteínas
- Sonicar células mamarias clasificadas por FACS en hielo durante 1 minuto con intervalos de 1 minuto durante un total de 5 ciclos con una amplitud del 70% y 0,5 Hz.
- Incubar muestras a 90 °C durante 1 h para la desnaturalización de proteínas utilizando un termociclador de PCR con la opción de tapa calentada.
NOTA: TFE es un disolvente volátil con un punto de ebullición de 74 °C, por lo que se selecciona la opción de tapa calentada para evitar el secado completo con una pérdida sustancial de la muestra. - Enfriar las muestras a temperatura ambiente con centrifugación a 1500 x g durante 3 min.
5. Reducción y alquilación (opcional)
- Añadir 0,6 μL de 50 mM de DTT para una concentración final de 2 mM.
NOTA: La solución TDT debe estar recién preparada cada vez. Pesar 77,5 mg de DTT y luego disolver en 1 mL de 25 mM de NH4HCO3 para hacer 500 mM de DTT. A continuación, se añaden 10 μL de TDT de 500 mM a 90 μL de NH4HCO3 de 25 mM para hacer TDT de 50 mM. - Centrifugar a 1500 x g durante 5 min y luego mezclar suavemente a 850 rpm durante 3 min.
- Incubar a 56°C durante 1 h con una suave agitación a 100 x g.
- Enfríe las muestras a temperatura ambiente y centrifuga a 1500 x g durante 3 min.
- Añadir 0,5 μL de 60 mM de IAA al tubo de RT-PCR con una concentración final de ~2 mM.
NOTA: La solución IAA debe estar recién preparada cada vez. Pesar 74,3 mg de AAI y luego disolver en 1 mL de 25 mM de NH4HCO3 para producir 400 mM de AAI. Se añaden 15 μL de 400 mM de IAA a 85 μL de 25 mM de NH4HCO3 para producir 60 mM de IAA. - Incubar en la oscuridad a temperatura ambiente durante 30 min con una suave agitación a 100 x g.
NOTA: Los pasos de reducción y alquilación son opcionales cuando no hay péptidos que contengan cisteína seleccionados para las proteínas objetivo.
6. Digestión de tripsina
- Reduzca el volumen de la muestra a ~4 μL utilizando un concentrador SpeedVac.
NOTA: TFE es un disolvente volátil con un punto de ebullición a 74 °C. El alto porcentaje de TFE inhibe la digestión de la tripsina. - Añadir 9 μL de 25 mM de NH4HCO3 y 1-3 μL de 15 ng/μL de tripsina para la digestión con la concentración final de tripsina de 1-3 ng/μL.
NOTA: Para 10 células mamarias humanas clasificadas por FACS (~ 1 ng), la proporción de enzima tripsina sobre la proteína es ≥15:1, 750 veces mayor que la de la digestión estándar de tripsina (la proporción de 1:50). - Mezclar suavemente a 100 x g durante 3 min e incubar toda la noche (~16 horas) a 37 °C.
- Añadir 0,5 μL de ácido fórmico al 5% para detener la reacción enzimática.
- Centrífuga durante 1 h a 1500 x g.
- Almacene en un congelador de -80 °C hasta un análisis posterior de LC-SRM o en un congelador de 4 °C para un análisis inmediato.
7. Preparación para el análisis LC-SRM directo
- Retire la tapa del tubo de PCR que se utilizó para la recolección (Paso 2) y el procesamiento (Paso 6) de las células mamarias clasificadas por FACS.
- Inserte el tubo de PCR en el vial de LC.
- Cierre el vial de LC para el análisis de LC-SRM.
NOTA: Para un pequeño número de células procesadas en una placa de PCR de 96 pocillos, se agregará la cubierta del tapete para sellar la placa para el análisis directo de LC-SRM.
8. Análisis LC-SRM
- Analice muestras utilizando UPLC acoplado a un espectrómetro de masas de triple cuadrupolo.
- Conecte las columnas capilares C18 (75 μm de diámetro interno × 20 cm para gradiente estándar y 100 μm de diámetro interno × 10 cm para gradiente corto) a un emisor de electrospray de sílice fundida de 20 μm de diámetro interno grabado químicamente a través de una unión de metal inoxidable.
- Utilice un bucle de muestra de 20 μL para cargar directamente toda la muestra (~15 μL en total) en la columna LC para maximizar la sensibilidad de detección.
- Utilice ácido fórmico al 0,1 % en agua y ácido fórmico al 0,1 % en acetonitrilo al 90 % como fases móviles A y B para la separación por RPLC capilar.
- Utilice el gradiente binario de LC a caudales de 300 nL/min para obtener un gradiente estándar cuando se midan grandes cantidades de proteínas diana (>10). Gradiente estándar: 5-20% B en 26 min, 20-25% B en 10 min, 25-40% B en 8 min, 40-95% B en 1 min y al 95% B durante 7 min durante un total de 52 min y la columna analítica reequilibrada al 99,5% A durante 8 min.
- Utilice el gradiente LC binario a caudales de 400 nL/min para un gradiente corto cuando se miden pequeñas cantidades de proteínas objetivo (≤10). Gradiente corto para una separación rápida: 5-95% B en 5 min y la columna analítica reequilibrada al 99,5% A durante 5 min con un tiempo de ejecución total de LC de 10 min.
- Opere el espectrómetro de masas QQQ con voltajes de pulverización de iones de 2.400 ± 100 V, un voltaje de compensación capilar de 35 V, un voltaje de compensación del skimmer de -5 V y una temperatura de entrada capilar de 220 ° C.
- Obtenga los demás parámetros de QQQ MS mediante ajuste y calibración automáticos sin necesidad de optimización adicional.
- Aumente diferentes concentraciones de patrones de péptidos pesados crudos en 20-50 ng/μL de digestión de lisado de células no humanas antes del análisis LC-SRM de muestras para determinar el tiempo de retención (RT) de LC del péptido para construir un método SRM programado y las posibles transiciones de interferencia para una cuantificación precisa.
- Utilice el modo SRM programado RT para la cuantificación multiplexada con una ventana de exploración de ≥6 min.
NOTA: La ventana de escaneo se ajustará en consecuencia dependiendo del número de transiciones en una determinada ventana de tiempo. - Establezca el tiempo de ciclo total en 1 s, y el tiempo de permanencia para cada transición se ajusta automáticamente en función del número de transiciones escaneadas en diferentes ventanas de tiempo de retención. Se necesita un tiempo de permanencia mínimo de 10 ms para cada transición de SRM. Todas las proteínas diana pueden monitorizarse simultáneamente en un único análisis LC-SRM
9. Análisis de datos
- Importe archivos de datos sin procesar en el software Skyline33 disponible públicamente para la visualización de datos de cada péptido objetivo para determinar su detectabilidad.
- Utilice dos criterios para determinar la detección e integración de picos de SRM: 1) el mismo tiempo de retención de LC; 2) aproximadamente las mismas relaciones relativas de intensidad máxima de SRM a través de múltiples transiciones entre péptidos endógenos (ligeros) y sus correspondientes estándares internos de péptidos marcados con isótopos (pesados).
- Inspeccione todos los datos de SRM manualmente para asegurarse de que la asignación de picos de SRM y el límite de picos de SRM sean correctos para una cuantificación confiable utilizando los dos criterios anteriores.
- Utilice la mejor transición con una señal SRM alta pero sin interferencia de la matriz para la cuantificación de proteínas, y utilice las otras transiciones como referencia.
- Utilice varias réplicas para obtener la desviación estándar (DE) y el coeficiente de variación (CV).
- Calcule la relación señal-ruido (S/N) por la intensidad máxima del ápice sobre el ruido de fondo medio dentro de una región de tiempo de retención de ±15 s para gradiente estándar y ±6 s para gradiente corto. El límite de detección (LOD) y el límite de cuantificación (LOQ) se definen como los puntos de concentración más bajos en los que la relación S/N es de al menos 3 y 7, respectivamente.
- Aplique un criterio adicional para determinar de manera conservadora los valores de LOQ además de S/N ≥7: la respuesta del péptido sustituto contra diferentes números de celdas debe estar dentro del rango dinámico lineal.
- Utilice el software Excel para trazar todas las curvas de calibración. Cargue los datos RAW de QQQ MS en el software Skyline disponible públicamente para mostrar gráficos de cromatogramas iónicos extraídos (XIC) de múltiples transiciones de proteínas objetivo monitoreadas.
Resultados
Se utilizaron por primera vez pequeñas cantidades de lisados de células MCF7 (0,5-20 ng, equivalentes a 5-200 células) para evaluar el rendimiento de cLC-SRM mediante la cuantificación dirigida de las proteínas de la vía EGFR/MAPK, ya que son más uniformes con menos variaciones en comparación con un pequeño número de células clasificadas por FACS. Como se muestra en la Figura 2A, XICs muestra claramente la detecci?...
Discusión
cLC-SRM es un conveniente método de proteómica dirigida que permite el análisis preciso de proteínas multiplexadas de pequeñas cantidades de células, incluidas células individuales. Este método aprovecha la preparación de muestras asistida por transportadores de proteínas en un solo recipiente, en la que todos los pasos, incluida la recolección de células, la lisis y digestión celular de varios pasos y la transferencia de digestión de péptidos a la columna capilar de LC pa...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo contó con el apoyo de los NIH Grant R21CA223715 (TS) y UG3CA256967 (TS y HL). El trabajo experimental descrito en este documento se llevó a cabo en el Laboratorio de Ciencias Moleculares Ambientales del Laboratorio Nacional del Noroeste del Pacífico, una instalación nacional de usuarios científicos patrocinada por el Departamento de Energía de los Estados Unidos de América bajo el contrato DE-AC05-76RL0 1830.
Materiales
| Name | Company | Catalog Number | Comments |
| 2 mL glass LC vial | Microsolv | 9502S-WCV | Vessel to hold PCR tube for autosample injection |
| BSA | Sigma-Aldrich | P0834-10×1mL | Carrier protein for greatly reducing surface adsorption losses |
| DTT | Thermo Scientific | A39255 | Reagent for reduction |
| Formic acid | Thermo Scientific | 28905 | Reagent for stopping enzyme reaction |
| IAA | Thermo Scientific | A39271 | Reagent for alkylation |
| Peptide internal standards | New England peptide | Targeted quantification of EGFR/MAPK pathway proteins | |
| RT-PCR tube | GeneMate Bioexpress | T-3035-1 | 0.2 mL PCR tube for one-pot sample preparation |
| Skyline software | University of Washington | Publicly available for SRM data analysis | |
| Sonicator | Hielscher Ultrasound Technology | UTR200 | Sonication on ice for cell lysis |
| Speed Vac concentrator | Thermo Scientific | Reduction of the percentage of TFE for effective trypsin digestion | |
| TFE | Sigma-Aldrich | 18370-10×1mL | 60% TFE for cell lysis |
| Thermocycler w/ heated lid | Peltier Thermal Cycler | PTC-200 | Heating for protein denaturation |
| Trypsin Gold | Promega | V528A | Enzyme for protein digestion |
| Waters BEH C18 column | Waters | C18 column for peptide separation |
Referencias
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Miyamoto, D. T., et al. RNA-Seq of single prostate CTCs implicates noncanonical Wnt signaling in antiandrogen resistance. Science. 349 (6254), 1351-1356 (2015).
- Wang, Y., Navin, N. E. Advances and applications of single-cell sequencing technologies. Molecular Cell. 58 (4), 598-609 (2015).
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends in Genetics. 33 (2), 155-168 (2017).
- Gavasso, S., Gullaksen, S. E., Skavland, J., Gjertsen, B. T. Single-cell proteomics: potential implications for cancer diagnostics. Expert Review of Molecular Diagnostics. 16 (5), 579-589 (2016).
- Wu, M., Singh, A. K. Single-cell protein analysis. Current Opinion in Biotechnology. 23 (1), 83-88 (2012).
- Hughes, A. J., et al. Single-cell western blotting. Nature Methods. 11 (7), 749-755 (2014).
- Peterson, V. M., et al. Multiplexed quantification of proteins and transcripts in single cells. Nature Biotechnology. 35 (10), 936-939 (2017).
- Liu, Y., Beyer, A., Aebersold, R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. 165 (3), 535-550 (2016).
- Choudhary, C., Mann, M. Decoding signalling networks by mass spectrometry-based proteomics. Nature Reviews Molecular Cell Biology. 11 (6), 427-439 (2010).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Zhang, H., et al. Integrated Proteogenomic Characterization of Human High-Grade Serous Ovarian Cancer. Cell. 166 (3), 755-765 (2016).
- Zhang, B., et al. Proteogenomic characterization of human colon and rectal cancer. Nature. 513 (7518), 382-387 (2014).
- Gaudet, S., Miller-Jensen, K. Redefining Signaling Pathways with an Expanding Single-Cell Toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Di Palma, S., Bodenmiller, B. Unraveling cell populations in tumors by single-cell mass cytometry. Current Opinion in Biotechnology. 31, 122-129 (2015).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Current Protocols in Immunology. , 11-20 (2012).
- Willison, K. R., Klug, D. R. Quantitative single cell and single molecule proteomics for clinical studies. Current Opinion in Biotechnology. 24 (4), 745-751 (2013).
- Lee, J. Y., et al. Detection of Head and Neck Cancer Based on Longitudinal Changes in Serum Protein Abundance. Cancer Epidemiology, Biomarkers & Prevention. 29 (8), 1665-1672 (2020).
- Shi, T., et al. Advancing the sensitivity of selected reaction monitoring-based targeted quantitative proteomics. Proteomics. 12 (8), 1074-1092 (2012).
- Shi, T., et al. Advances in targeted proteomics and applications to biomedical research. Proteomics. 16 (15-16), 2160-2182 (2016).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Molecular Systems Biology. 4, 222 (2008).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nature Methods. 9 (6), 555-566 (2012).
- Zhu, Y., et al. Proteomic Analysis of Single Mammalian Cells Enabled by Microfluidic Nanodroplet Sample Preparation and Ultrasensitive NanoLC-MS. Angewandte Chemie International Edition. 57 (38), 12370-12374 (2018).
- Cong, Y., et al. Improved Single-Cell Proteome Coverage Using Narrow-Bore Packed NanoLC Columns and Ultrasensitive Mass Spectrometry. Analytical Chemistry. 92 (3), 2665-2671 (2020).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9 (1), 882 (2018).
- Shao, X., et al. Integrated Proteome Analysis Device for Fast Single-Cell Protein Profiling. Analytical Chemistry. 90 (23), 14003-14010 (2018).
- Li, Z. Y., et al. Nanoliter-Scale Oil-Air-Droplet Chip-Based Single Cell Proteomic Analysis. Analytical Chemistry. 90 (8), 5430-5438 (2018).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19 (1), 161 (2018).
- Zhang, P., et al. Carrier-Assisted Single-Tube Processing Approach for Targeted Proteomics Analysis of Low Numbers of Mammalian Cells. Analytical Chemistry. 91 (2), 1441-1451 (2019).
- Shi, T., et al. Facile carrier-assisted targeted mass spectrometric approach for proteomic analysis of low numbers of mammalian cells. Communications Biology. 1, 103 (2018).
- Vitrinel, B., Iannitelli, D. E., Mazzoni, E. O., Christiaen, L., Vogel, C. Simple Method to Quantify Protein Abundances from 1000 Cells. ACS Omega. 5 (25), 15537-15546 (2020).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26 (7), 966-968 (2010).
- Shi, T., et al. Conservation of protein abundance patterns reveals the regulatory architecture of the EGFR-MAPK pathway. Science Signaling. 9 (436), 6 (2016).
- Addona, T. A., et al. Multi-site assessment of the precision and reproducibility of multiple reaction monitoring-based measurements of proteins in plasma. Nature Biotechnology. 27 (7), 633-641 (2009).
- He, J., et al. Analytical platform evaluation for quantification of ERG in prostate cancer using protein and mRNA detection methods. Journal of Translational Medicine. 13, 54 (2015).
- Shi, T., et al. Antibody-free, targeted mass-spectrometric approach for quantification of proteins at low picogram per milliliter levels in human plasma/serum. Proceedings of the National Academy of Sciences. 109 (38), 15395-15400 (2012).
- Shi, T., et al. Targeted quantification of low ng/mL level proteins in human serum without immunoaffinity depletion. Journal of Proteome Research. 12 (7), 3353-3361 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados