È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Preparazione di campioni one-pot assistita da carrier per l'analisi proteomica mirata di un piccolo numero di cellule umane
In questo articolo
Riepilogo
Una preparazione di campioni one-pot assistita da carrier proteico abbinata alla cromatografia liquida (LC) - monitoraggio della reazione selezionata (SRM) denominata cLC-SRM è un metodo conveniente per l'analisi proteomica mirata multiplexata di un piccolo numero di cellule, comprese le singole cellule. Sfrutta l'utilizzo di un'eccessiva proteina esogena come vettore e LC-SRM ad alta specificità per la quantificazione mirata.
Abstract
L'analisi delle proteine di un piccolo numero di cellule umane si ottiene principalmente mediante proteomica mirata con saggi immunologici basati su anticorpi, che hanno limitazioni intrinseche (ad esempio, basso multiplex e indisponibilità di anticorpi per nuove proteine). La proteomica mirata basata sulla spettrometria di massa (MS) è emersa come alternativa perché è priva di anticorpi, altamente multiplex e ha un'elevata specificità e accuratezza di quantificazione. I recenti progressi nella strumentazione per la SM rendono possibile la proteomica mirata basata sulla SM per la quantificazione multiplexata di proteine altamente abbondanti in singole cellule. Tuttavia, esiste una sfida tecnica per l'elaborazione efficace di singole cellule con una perdita minima di campione per l'analisi MS. Per risolvere questo problema, abbiamo recentemente sviluppato una comoda preparazione di campioni one-pot assistita da trasportatori proteici abbinata alla cromatografia liquida (LC) - monitoraggio delle reazioni selezionate (SRM) denominata cLC-SRM per l'analisi proteomica mirata di un piccolo numero di cellule umane. Questo metodo sfrutta l'utilizzo dell'eccesso combinato di proteina esogena come vettore e l'elaborazione one-pot a basso volume per ridurre notevolmente le perdite di adsorbimento superficiale, nonché LC-SRM ad alta specificità per affrontare efficacemente l'aumento dell'intervallo di concentrazione dinamica dovuto all'aggiunta di proteina trasportatrice esogena. La sua utilità è stata dimostrata dalla quantificazione accurata delle proteine più moderatamente abbondanti in un piccolo numero di cellule (ad esempio, 10-100 cellule) e delle proteine altamente abbondanti in singole cellule. Le caratteristiche di facile implementazione e l'assenza di dispositivi specifici rendono questo metodo facilmente accessibile alla maggior parte dei laboratori di proteomica. Qui abbiamo fornito un protocollo dettagliato per l'analisi cLC-SRM di un piccolo numero di cellule umane, tra cui la selezione cellulare, la lisi e la digestione cellulare, l'analisi LC-SRM e l'analisi dei dati. Sono necessari ulteriori miglioramenti nella sensibilità di rilevamento e nella produttività dei campioni per un'analisi proteomica mirata su singola cellula. Prevediamo che cLC-SRM sarà ampiamente applicato alla ricerca biomedica e alla biologia dei sistemi con il potenziale di facilitare la medicina di precisione.
Introduzione
I recenti progressi tecnologici nel campo della genomica (trascrittomica) consentono un'analisi completa e precisa del genoma (trascrittoma) in singole cellule 1,2,3. Tuttavia, le tecnologie di proteomica a singola cellula sono molto indietro, ma sono altrettanto importanti delle tecnologie genomiche (trascrittomica) 4,5,6,7,8. Inoltre, l'abbondanza proteica non può necessariamente essere dedotta dall'abbondanza dell'mRNA9 e il proteoma è più complesso e dinamico del trascrittoma10. Date queste sfide, un gran numero di popolazioni miste di cellule (cioè cellule di massa) viene generalmente utilizzato per generare dati completi sul proteoma 11,12,13. Tuttavia, tali misurazioni di massa mediano le variazioni stocastiche delle singole cellule, oscurando così un'importante variabilità da cellula a cellula (cioè l'eterogeneità cellulare)4,14. Le limitazioni di tali misurazioni di massa diventano ancora più gravi quando le cellule di interesse rappresentano solo una piccola parte delle popolazioni totali di cellule (ad esempio, cellule staminali tumorali all'interno di tumori in stadio iniziale). Pertanto, esiste un enorme divario di conoscenze tra la proteomica a singola cellula e la genomica (trascrittomica).
I saggi immunologici basati su anticorpi (ad es. citometria a flusso o di massa) sono utilizzati prevalentemente per l'analisi proteomica mirata di singole cellule 6,7,15,16,17,18. Tuttavia, soffrono di un basso multiplex, di una specificità limitata e dell'indisponibilità di anticorpi per nuove proteine di interesse. La proteomica mirata basata sulla spettrometria di massa (MS) è emersa come alternativa per la quantificazione accurata delle proteine grazie all'assenza di anticorpi, all'elevato multiplex (≥150 proteine in una singola analisi19), all'elevata accuratezza di quantificazione (quantità o concentrazioni assolute) e all'elevata specificità e riproducibilità (≤10% CV)20,21,22,23. I recenti progressi significativi nella preparazione dei campioni hanno reso possibile la proteomica a singola cellula basata sulla MS per l'analisi quantitativa di proteine altamente abbondanti provenienti da singole cellule umane. Tuttavia, la proteomica a singola cellula basata sulla SM è ancora nella fase iniziale dell'infanzia. Ad esempio, la piattaforma MS più avanzata abbinata a velocità di flusso RPLC a flusso ultrabasso può consentire solo il rilevamento e la quantificazione della MS senza marcatura di ~670-870 proteine su un totale di ≥12.000 proteine in singole cellule HeLa 24,25.
Attualmente, sono disponibili sei approcci di proteomica a singola cellula basati sulla MS per l'analisi di singole cellule di mammifero, di cui quattro per la proteomica globale (nanoPOTS: nanowell-based Preparation in One pot for Trace Samples26; iPAD-1: dispositivo integrato per l'analisi del proteoma per l'analisi di singole cellule27; Analisi proteomica a singola cellula basata su chip OAD (oil-air-droplet)28; SCoPE-MS: proteomica a singola cellula mediante spettrometria di massa29) e gli altri due sono per la proteomica mirata (cLC-SRM: cromatografia liquida assistita da carrier (LC) - monitoraggio di reazioni selezionate (SRM)30; cPRISM-SRM: separazioni ad alta pressione e ad alta risoluzione assistite da carrier con selezione intelligente e multiplexing accoppiati a SRM31). Tuttavia, tutti questi approcci presentano svantaggi tecnici. nanoPOTS, iPAD-1 e OAD riducono il volume di elaborazione del campione a 2-200 nL e non sono pronti per ampie applicazioni da banco 26,27,28. Per SCoPE-MS, un vettore TMT (tandem mass tag) viene aggiunto dopo l'elaborazione di singole cellule, in modo che non possa prevenire efficacemente le perdite di adsorbimento superficiale durante l'elaborazione del campione quando una singola provetta viene utilizzata per l'elaborazione di singole cellule29, con conseguente bassa riproducibilità con un coefficiente di correlazione di soli ~0,2-0,4 tra le repliche32. Per cLC-SRM e cPRISM-SRM, l'utilizzo di proteine esogene come vettore è più adatto per la proteomica mirata perché i peptidi provenienti da proteine esogene eccessive sono frequentemente sequenziati da MS/MS, il che riduce notevolmente la possibilità di sequenziamento di peptidi endogeni a bassa abbondanza30,31. A differenza della proteomica globale per la quantificazione relativa, i due approcci di proteomica mirata possono fornire analisi proteiche accurate o assolute di un piccolo numero di cellule con un'elevata riproducibilità utilizzando standard interni marcati con isotopi pesanti a concentrazioni note. Rispetto a cPRISM-SRM, che richiede un precedente frazionamento PRISM ad alta risoluzione, con conseguente necessità di analizzare molti campioni frazionati, cLC-SRM presenta un vantaggio significativo in termini di produttività del campione senza frazionamento e può quantificare simultaneamente centinaia di proteine in una singola analisi, ma con una sensibilità di rilevamento relativamente inferiore30. Pertanto, cLC-SRM è più accessibile e dovrebbe avere utilità più ampie per un'accurata analisi multiplex delle proteine di un piccolo numero di cellule e di campioni a massa limitata.
Qui descriviamo un protocollo dettagliato per eseguire cLC-SRM per un'analisi proteomica mirata di un piccolo numero di cellule umane, comprese le singole cellule. Il protocollo consiste nelle seguenti fasi principali: smistamento cellulare mediante FACS (smistamento cellulare attivato da fluorescenza), lisi e digestione cellulare trattate in provette a reazione a catena della polimerasi (PCR) a basso volume, raccolta di dati LC-SRM e analisi dei dati SRM utilizzando il software Skyline disponibile al pubblico (Figura 1). La sua ampia utilità è stata dimostrata insieme ai nostri saggi SRM precedentemente consolidati mediante quantificazione mirata assoluta delle proteine della via EGFR/MAPK in cellule 1-100 MCF7 o MCF10A e determinazione delle copie delle proteine della via per cellula a un ampio intervallo dinamico di concentrazioni30. Prevediamo che con il protocollo dettagliato la maggior parte dei ricercatori di proteomica possa facilmente implementare cLC-SRM nei loro laboratori per un'analisi accurata delle proteine di campioni ultrapiccoli (ad esempio, cellule tumorali rare) per soddisfare le esigenze del loro progetto.
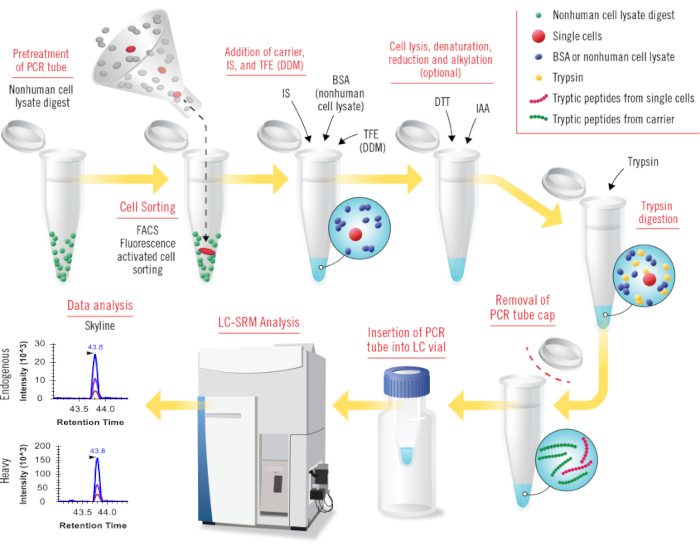
Figura 1: Panoramica di tutte le fasi della cLC-SRM (preparazione del campione in un vaso assistita da c arried accoppiata con cromatografia a base di liquido liquido).I digesti di lisato di cellule non umane (ad esempio, Shewanella oneidensis) vengono utilizzati per pretrattare le provette PCR per il rivestimento della superficie delle provette. Un piccolo numero di cellule umane o singole cellule selezionate mediante FACS vengono raccolte in provette PCR pretrattate. Il vettore della proteina BSA (o proteoma del lisato di cellule non umane), lo standard interno pesante (IS) e il TFE (o DDM) vengono aggiunti in sequenza alle provette del campione per facilitare la lisi cellulare e ridurre le perdite di adsorbimento superficiale. Concettualmente, il DDM combinato e il trasportatore di proteoma del lisato cellulare non umano funzioneranno bene per cLC-SRM. La lisi cellulare viene condotta mediante sonicazione e la denaturazione delle proteine si ottiene mediante riscaldamento ad alta temperatura. I reagenti DTT e IAA vengono utilizzati rispettivamente per la riduzione e l'alchilazione (questo passaggio è facoltativo). La tripsina viene aggiunta per la digestione con rapporti molto più elevati di tripsina rispetto alla quantità di proteine rispetto a quella per la digestione standard della tripsina. Il tappo della provetta del campione viene rimosso e quindi la provetta PCR viene inserita nella fiala LC per l'analisi LC-SRM diretta. I dati SRM raccolti vengono analizzati utilizzando il software Skyline disponibile pubblicamente. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
NOTA: L'analisi cLC-SRM passo dopo passo è mostrata nella Figura 1.
1. Pretrattamento delle provette per PCR
- Aggiungere 100 μL di lisato cellulare non umano (ad es. Shewanella oneidensis) digerito a 0,2 μg/μL alle provette PCR. Incubare a temperatura ambiente per una notte per rivestire la superficie della provetta PCR.
- Rimuovere il lisato cellulare digerito mediante pipettaggio, sciacquare le provette PCR con acqua HPLC 3 volte e quindi asciugare le provette PCR all'aria in una cappa aspirante.
- Conservare le provette PCR rivestite in un congelatore a 4 °C fino a nuovo utilizzo.
2. Smistamento FACS
- Allineare un selezionatore cellulare attivato a fluorescenza (FACS) (vedere la Tabella dei materiali) in una piastra PCR a 96 pozzetti utilizzando perle fluorescenti con coloranti che possono essere eccitati dalla sorgente laser per fornire segnali di riferimento affidabili.
- Verificare che la macchina FACS sia ben mirata per lo smistamento delle celle sul fondo delle piastre PCR.
- Impostare l'angolo del flusso di ordinamento al minimo possibile per massimizzare la percentuale di successo in cui le celle verranno ordinate sul fondo dei pozzetti anziché colpire i lati.
- Per puntare la macchina, utilizzare il flusso di prova per "ordinare" le goccioline di tampone PBS (ad esempio, 100 goccioline) sul pozzetto di una piastra vuota per assicurarsi che le goccioline raccolte siano sul fondo. Se le goccioline non atterrano nella posizione corretta, ricalibrare il selezionatore di celle e ripetere l'operazione fino a quando le goccioline nel flusso di smistamento non vengono raccolte correttamente.
- Posizionare le provette PCR rivestite in un rack per provette PCR a 96 pozzetti.
- Ordina il numero desiderato di cellule mammarie umane (MCF7 o MCF10A) in provette PCR prerivestite.
- Centrifugare immediatamente le cellule mammarie selezionate a 100 x g per 10 minuti a 4 °C per mantenerle sul fondo della provetta ed evitare potenziali perdite cellulari.
- Conservare le cellule selezionate in un congelatore a -80 °C fino a nuova analisi.
3. Aggiunta di vettore proteico, standard interni pesanti e TFE
- Aggiungere 4 μl di 25 mM NH4HCO3 in provette PCR contenenti le cellule raccolte.
- Aggiungere 1 μL di 10 ng/μL di albumina sierica bovina (BSA) in 25 mM NH4HCO3 in provette per campioni.
- Aggiungere 0,3 μl di standard di peptidi pesanti grezzi da 100 fmol/μl (totale 30 fmol) nelle provette del campione.
NOTA: Aliquote di standard di peptidi pesanti grezzi per le proteine della via EGFR/MAPK sono prodotte a una concentrazione nominale di 100 fmol/μL in 25 mM NH4HCO3. Erogare il piccolo volume di standard di peptidi pesanti grezzi a 100 fmol/μL al centro della soluzione e assicurarsi che non vi siano residui di liquido nel puntale della pipetta. - Aggiungere 9 μl di TFE al 100% nelle provette del campione con la concentrazione finale di ~60% TFE.
- Centrifugare a 1500 x g per 5 min e poi agitare delicatamente a 100 x g per 3 min.
4. Lisi cellulare e denaturazione delle proteine
- Sonicare le cellule mammarie selezionate FACS su ghiaccio per 1 minuto con intervalli di 1 minuto per un totale di 5 cicli al 70% di ampiezza e 0,5 Hz.
- Incubare i campioni a 90 °C per 1 ora per la denaturazione delle proteine utilizzando un termociclatore PCR con l'opzione del coperchio riscaldato.
NOTA: Il TFE è un solvente volatile con un punto di ebollizione di 74 °C, quindi l'opzione con coperchio riscaldato è selezionata per evitare l'essiccazione completa con una sostanziale perdita di campione. - Raffreddare i campioni a temperatura ambiente con centrifugazione a 1500 x g per 3 minuti.
5. Riduzione e alchilazione (opzionale)
- Aggiungere 0,6 μl di 50 mM DTT per una concentrazione finale di 2 mM.
NOTA: La soluzione DTT deve essere preparata ogni volta di fresco. Pesare 77,5 mg di DTT e poi sciogliere in 1 mL di 25 mM NH4HCO3 per ottenere 500 mM di DTT. 10 μL di 500 mM DTT vengono quindi aggiunti a 90 μL di 25 mM NH4HCO3 per ottenere 50 mM DTT. - Centrifugare a 1500 x g per 5 min e poi mescolare delicatamente a 850 rpm per 3 min.
- Incubare a 56°C per 1 ora agitando delicatamente a 100 x g.
- Raffreddare i campioni a temperatura ambiente e centrifugare a 1500 x g per 3 minuti.
- Aggiungere 0,5 μl di 60 mM IAA alla provetta RT-PCR con la concentrazione finale di ~2 mM.
NOTA: La soluzione IAA deve essere preparata ogni volta di fresco. Pesare 74,3 mg di IAA e poi sciogliere in 1 mL di 25 mM NH4HCO3 per ottenere 400 mM di IAA. 15 μL di 400 mM IAA vengono aggiunti a 85 μL di 25 mM NH4HCO3 per ottenere 60 mM IAA. - Incubare al buio a temperatura ambiente per 30 minuti agitando delicatamente a 100 x g.
NOTA: Le fasi di riduzione e alchilazione sono facoltative quando non sono selezionati peptidi contenenti cisteina per le proteine bersaglio.
6. Digestione della tripsina
- Ridurre il volume del campione a ~4 μl utilizzando un concentratore SpeedVac.
NOTA: Il TFE è un solvente volatile con punto di ebollizione a 74°C. L'alta percentuale di TFE inibisce la digestione della tripsina. - Aggiungere 9 μL di 25 mM NH4HCO3 e 1-3 μL di 15 ng/μL di tripsina per la digestione con la concentrazione finale di tripsina di 1-3 ng/μL.
NOTA: Per 10 cellule mammarie umane selezionate con FACS (~1 ng), il rapporto tra l'enzima tripsina e le proteine è ≥15:1, 750 volte superiore a quello della digestione standard della tripsina (il rapporto di 1:50). - Miscelare delicatamente a 100 x g per 3 minuti e incubare per una notte (~16 ore) a 37 °C.
- Aggiungere 0,5 μl di acido formico al 5% per fermare la reazione enzimatica.
- Centrifugare per 1 ora a 1500 x g.
- Conservare in congelatore a -80 °C fino a ulteriori analisi LC-SRM o in congelatore a 4 °C per un'analisi immediata.
7. Preparazione per l'analisi diretta LC-SRM
- Rimuovere il tappo della provetta per PCR utilizzata per la raccolta (Fase 2) e l'elaborazione (Fase 6) delle cellule mammarie selezionate con FACS.
- Inserire la provetta per PCR nella fiala LC.
- Chiudere la fiala LC per l'analisi LC-SRM.
NOTA: Per un numero ridotto di cellule processate in una piastra PCR a 96 pozzetti, verrà aggiunto il coperchio del tappetino per sigillare la piastra per l'analisi LC-SRM diretta.
8. Analisi LC-SRM
- Analizza i campioni utilizzando UPLC accoppiato a uno spettrometro di massa a triplo quadrupolo.
- Collegare le colonne capillari C18 (75 μm i.d. × 20 cm per gradiente standard e 100 μm i.d. × 10 cm per gradiente corto) a un emettitore elettrospray di silice fusa da 20 μm inciso chimicamente tramite un raccordo in metallo inossidabile.
- Utilizzare un loop di campionamento da 20 μl per caricare direttamente tutto il campione (~15 μl in totale) nella colonna LC per massimizzare la sensibilità di rilevamento.
- Utilizzare lo 0,1% di acido formico in acqua e lo 0,1% di acido formico in acetonitrile al 90% come fasi mobili A e B per la separazione capillare dell'RPLC.
- Utilizzare il gradiente LC binario a velocità di flusso di 300 nL/min per un gradiente standard quando viene misurato un gran numero di proteine bersaglio (>10). Gradiente standard: 5-20% B in 26 min, 20-25% B in 10 min, 25-40% B in 8 min, 40-95% B in 1 min e al 95% B per 7 min per un totale di 52 min e colonna analitica riequilibrata al 99,5% A per 8 min.
- Utilizzare il gradiente LC binario a velocità di flusso di 400 nL/min per un gradiente breve quando vengono misurati piccoli numeri di proteine bersaglio (≤10). Gradiente corto per una separazione rapida: 5-95% B in 5 min e colonna analitica riequilibrata al 99,5% A per 5 min con un tempo di esecuzione LC totale di 10 min.
- Aziona lo spettrometro di massa QQQ con tensioni di spruzzo ionico di 2.400 ± 100 V, una tensione di offset capillare di 35 V, una tensione di offset dello skimmer di -5 V e una temperatura di ingresso capillare di 220 °C.
- Ottieni gli altri parametri QQQ MS dalla sintonizzazione e calibrazione automatiche senza ulteriori ottimizzazioni.
- Aumentare diverse concentrazioni di standard di peptidi pesanti grezzi in 20-50 ng/μL di lisato cellulare non umano digerito prima dell'analisi LC-SRM dei campioni per determinare il tempo di ritenzione LC (RT) del peptide per la creazione di un metodo SRM programmato e le potenziali transizioni di interferenza per una quantificazione accurata.
- Utilizzare la modalità SRM programmata RT per la quantificazione multiplexata con una finestra di scansione di ≥6 min.
NOTA: La finestra di scansione verrà regolata di conseguenza in base al numero di transizioni in una determinata finestra temporale. - Imposta il tempo di ciclo totale su 1 s e il tempo di permanenza per ogni transizione viene regolato automaticamente in base al numero di transizioni scansionate in diverse finestre di tempo di conservazione. Per ogni transizione SRM è necessario un tempo di permanenza minimo di 10 ms. Tutte le proteine bersaglio possono essere monitorate simultaneamente in un'unica analisi LC-SRM
9. Analisi dei dati
- Importa i file di dati grezzi nel software Skyline33 disponibile al pubblico per la visualizzazione dei dati di ciascun peptide target per determinarne la rilevabilità.
- Utilizzare due criteri per determinare il rilevamento e l'integrazione dei picchi SRM: 1) stesso tempo di ritenzione LC; 2) approssimativamente gli stessi rapporti di intensità di picco SRM relativi attraverso transizioni multiple tra peptidi endogeni (leggeri) e i loro corrispondenti standard interni di peptidi marcati con isotopi (pesanti).
- Ispezionare manualmente tutti i dati SRM per garantire la corretta assegnazione dei picchi SRM e il limite dei picchi SRM per una quantificazione affidabile utilizzando i due criteri precedenti.
- Utilizzare la transizione migliore con un segnale SRM elevato ma senza interferenza della matrice per la quantificazione delle proteine e utilizzare le altre transizioni come riferimento.
- Utilizzare più repliche per ottenere la deviazione standard (SD) e il coefficiente di variazione (CV).
- Calcola il rapporto segnale/rumore (S/N) in base all'intensità apicale di picco rispetto al rumore di fondo medio entro un intervallo di tempo di ritenzione di ±15 s per il gradiente standard e di ±6 s per il gradiente breve. Il limite di rilevabilità (LOD) e il limite di quantificazione (LOQ) sono definiti come i punti di concentrazione più bassi in cui il rapporto S/N è rispettivamente di almeno 3 e 7.
- Applicare un criterio aggiuntivo per determinare in modo conservativo i valori LOQ oltre a S/N ≥7: la risposta del peptide surrogato contro diversi numeri di cellule deve essere all'interno dell'intervallo dinamico lineare.
- Utilizzare il software Excel per tracciare tutte le curve di calibrazione. Carica i dati RAW da QQQ MS nel software Skyline disponibile al pubblico per visualizzare i grafici dei cromatogrammi ionici estratti (XIC) di più transizioni di proteine bersaglio monitorate.
Risultati
Piccole quantità di lisati cellulari MCF7 (0,5-20 ng equivalenti a 5-200 cellule) sono state utilizzate per la prima volta per valutare le prestazioni di cLC-SRM mediante quantificazione mirata delle proteine della via EGFR/MAPK perché sono più uniformi con meno variazioni rispetto a un piccolo numero di cellule selezionate da FACS. Come mostrato nella Figura 2A, gli XIC mostrano chiaramente il rilevamento di transizioni S...
Discussione
cLC-SRM è un comodo metodo di proteomica mirata che consente un'analisi accurata delle proteine multiplex di un piccolo numero di cellule, comprese le singole cellule. Questo metodo sfrutta la preparazione di campioni one-pot assistita da trasportatori proteici, in cui tutte le fasi, tra cui la raccolta cellulare, la lisi cellulare e la digestione in più fasi e il trasferimento dei peptidi digestati alla colonna LC capillare per l'analisi MS, vengono eseguite in un unico vaso (ad esemp...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da NIH Grant R21CA223715 (TS) e UG3CA256967 (TS e HL). Il lavoro sperimentale qui descritto è stato eseguito presso l'Environmental Molecular Sciences Laboratory, Pacific Northwest National Laboratory, una struttura scientifica nazionale sponsorizzata dal Dipartimento dell'Energia degli Stati Uniti d'America nell'ambito del contratto DE-AC05-76RL0 1830.
Materiali
| Name | Company | Catalog Number | Comments |
| 2 mL glass LC vial | Microsolv | 9502S-WCV | Vessel to hold PCR tube for autosample injection |
| BSA | Sigma-Aldrich | P0834-10×1mL | Carrier protein for greatly reducing surface adsorption losses |
| DTT | Thermo Scientific | A39255 | Reagent for reduction |
| Formic acid | Thermo Scientific | 28905 | Reagent for stopping enzyme reaction |
| IAA | Thermo Scientific | A39271 | Reagent for alkylation |
| Peptide internal standards | New England peptide | Targeted quantification of EGFR/MAPK pathway proteins | |
| RT-PCR tube | GeneMate Bioexpress | T-3035-1 | 0.2 mL PCR tube for one-pot sample preparation |
| Skyline software | University of Washington | Publicly available for SRM data analysis | |
| Sonicator | Hielscher Ultrasound Technology | UTR200 | Sonication on ice for cell lysis |
| Speed Vac concentrator | Thermo Scientific | Reduction of the percentage of TFE for effective trypsin digestion | |
| TFE | Sigma-Aldrich | 18370-10×1mL | 60% TFE for cell lysis |
| Thermocycler w/ heated lid | Peltier Thermal Cycler | PTC-200 | Heating for protein denaturation |
| Trypsin Gold | Promega | V528A | Enzyme for protein digestion |
| Waters BEH C18 column | Waters | C18 column for peptide separation |
Riferimenti
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Miyamoto, D. T., et al. RNA-Seq of single prostate CTCs implicates noncanonical Wnt signaling in antiandrogen resistance. Science. 349 (6254), 1351-1356 (2015).
- Wang, Y., Navin, N. E. Advances and applications of single-cell sequencing technologies. Molecular Cell. 58 (4), 598-609 (2015).
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends in Genetics. 33 (2), 155-168 (2017).
- Gavasso, S., Gullaksen, S. E., Skavland, J., Gjertsen, B. T. Single-cell proteomics: potential implications for cancer diagnostics. Expert Review of Molecular Diagnostics. 16 (5), 579-589 (2016).
- Wu, M., Singh, A. K. Single-cell protein analysis. Current Opinion in Biotechnology. 23 (1), 83-88 (2012).
- Hughes, A. J., et al. Single-cell western blotting. Nature Methods. 11 (7), 749-755 (2014).
- Peterson, V. M., et al. Multiplexed quantification of proteins and transcripts in single cells. Nature Biotechnology. 35 (10), 936-939 (2017).
- Liu, Y., Beyer, A., Aebersold, R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. 165 (3), 535-550 (2016).
- Choudhary, C., Mann, M. Decoding signalling networks by mass spectrometry-based proteomics. Nature Reviews Molecular Cell Biology. 11 (6), 427-439 (2010).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Zhang, H., et al. Integrated Proteogenomic Characterization of Human High-Grade Serous Ovarian Cancer. Cell. 166 (3), 755-765 (2016).
- Zhang, B., et al. Proteogenomic characterization of human colon and rectal cancer. Nature. 513 (7518), 382-387 (2014).
- Gaudet, S., Miller-Jensen, K. Redefining Signaling Pathways with an Expanding Single-Cell Toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Di Palma, S., Bodenmiller, B. Unraveling cell populations in tumors by single-cell mass cytometry. Current Opinion in Biotechnology. 31, 122-129 (2015).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Current Protocols in Immunology. , 11-20 (2012).
- Willison, K. R., Klug, D. R. Quantitative single cell and single molecule proteomics for clinical studies. Current Opinion in Biotechnology. 24 (4), 745-751 (2013).
- Lee, J. Y., et al. Detection of Head and Neck Cancer Based on Longitudinal Changes in Serum Protein Abundance. Cancer Epidemiology, Biomarkers & Prevention. 29 (8), 1665-1672 (2020).
- Shi, T., et al. Advancing the sensitivity of selected reaction monitoring-based targeted quantitative proteomics. Proteomics. 12 (8), 1074-1092 (2012).
- Shi, T., et al. Advances in targeted proteomics and applications to biomedical research. Proteomics. 16 (15-16), 2160-2182 (2016).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Molecular Systems Biology. 4, 222 (2008).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nature Methods. 9 (6), 555-566 (2012).
- Zhu, Y., et al. Proteomic Analysis of Single Mammalian Cells Enabled by Microfluidic Nanodroplet Sample Preparation and Ultrasensitive NanoLC-MS. Angewandte Chemie International Edition. 57 (38), 12370-12374 (2018).
- Cong, Y., et al. Improved Single-Cell Proteome Coverage Using Narrow-Bore Packed NanoLC Columns and Ultrasensitive Mass Spectrometry. Analytical Chemistry. 92 (3), 2665-2671 (2020).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9 (1), 882 (2018).
- Shao, X., et al. Integrated Proteome Analysis Device for Fast Single-Cell Protein Profiling. Analytical Chemistry. 90 (23), 14003-14010 (2018).
- Li, Z. Y., et al. Nanoliter-Scale Oil-Air-Droplet Chip-Based Single Cell Proteomic Analysis. Analytical Chemistry. 90 (8), 5430-5438 (2018).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19 (1), 161 (2018).
- Zhang, P., et al. Carrier-Assisted Single-Tube Processing Approach for Targeted Proteomics Analysis of Low Numbers of Mammalian Cells. Analytical Chemistry. 91 (2), 1441-1451 (2019).
- Shi, T., et al. Facile carrier-assisted targeted mass spectrometric approach for proteomic analysis of low numbers of mammalian cells. Communications Biology. 1, 103 (2018).
- Vitrinel, B., Iannitelli, D. E., Mazzoni, E. O., Christiaen, L., Vogel, C. Simple Method to Quantify Protein Abundances from 1000 Cells. ACS Omega. 5 (25), 15537-15546 (2020).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26 (7), 966-968 (2010).
- Shi, T., et al. Conservation of protein abundance patterns reveals the regulatory architecture of the EGFR-MAPK pathway. Science Signaling. 9 (436), 6 (2016).
- Addona, T. A., et al. Multi-site assessment of the precision and reproducibility of multiple reaction monitoring-based measurements of proteins in plasma. Nature Biotechnology. 27 (7), 633-641 (2009).
- He, J., et al. Analytical platform evaluation for quantification of ERG in prostate cancer using protein and mRNA detection methods. Journal of Translational Medicine. 13, 54 (2015).
- Shi, T., et al. Antibody-free, targeted mass-spectrometric approach for quantification of proteins at low picogram per milliliter levels in human plasma/serum. Proceedings of the National Academy of Sciences. 109 (38), 15395-15400 (2012).
- Shi, T., et al. Targeted quantification of low ng/mL level proteins in human serum without immunoaffinity depletion. Journal of Proteome Research. 12 (7), 3353-3361 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon