このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
少数のヒト細胞の標的プロテオミクス分析のためのキャリア支援ワンポットサンプル調製
要約
cLC-SRMと呼ばれる液体クロマトグラフィー(LC)選択反応モニタリング(SRM)と組み合わせたタンパク質キャリア支援ワンポットサンプル調製は、単一細胞を含む少数の細胞のマルチプレックス標的プロテオミクス分析に便利な方法です。過剰な外因性タンパク質をキャリアとして使用し、標的定量のための高特異性LC-SRMを活用します。
要約
少数のヒト細胞のタンパク質解析は、主に抗体ベースのイムノアッセイを用いた標的プロテオミクスによって達成されますが、これには固有の制限があります(例:マルチプレックスが低い、新しいタンパク質に対する抗体が利用できない)。質量分析(MS)ベースのターゲットプロテオミクスは、抗体を含まず、マルチプレックスが高く、特異性と定量精度が高いため、代替手段として浮上しています。近年のMS装置の進歩により、MSベースのターゲットプロテオミクスにより、単一細胞内の存在量の多いタンパク質のマルチプレックス定量が可能になりました。しかし、MS分析のためにサンプル損失を最小限に抑えてシングルセルを効果的に処理するには、技術的な課題があります。この問題に対処するために、私たちは最近、少数のヒト細胞の標的プロテオミクス分析のために、液体クロマトグラフィー(LC)選択反応モニタリング(SRM)と組み合わせた便利なタンパク質キャリア支援ワンポットサンプル調製を開発しました。この方法は、過剰な外因性タンパク質をキャリアとして使用し、少量のワンポット処理で表面吸着損失を大幅に低減するとともに、高特異性LC-SRMを利用して、外来性キャリアタンパク質の添加によるダイナミック濃度範囲の増加に効果的に対処します。その有用性は、少数の細胞(10〜100個の細胞など)で最も適度に存在量の多いタンパク質と、単一細胞内の非常に豊富なタンパク質の正確な定量によって実証されています。実装が簡単で、特定のデバイスを必要としないため、この分析法はほとんどのプロテオミクスラボで容易に利用できます。ここでは、細胞ソーティング、細胞溶解および消化、LC-SRM分析、データ解析など、少数のヒト細胞のcLC-SRM解析のための詳細なプロトコールを提供しています。ターゲットシングルセルプロテオミクス分析には、検出感度とサンプルスループットのさらなる改善が必要です。cLC-SRMは、生物医学研究やシステムバイオロジーに広く適用され、プレシジョンメディシンを促進する可能性を秘めていると期待しています。
概要
近年のゲノミクス(トランスクリプトミクス)の技術的進歩により、単一細胞1,2,3のゲノム(トランスクリプトーム)を包括的かつ精密に解析することが可能になりました。しかし、シングルセルプロテオミクス技術は大きく遅れをとっていますが、ゲノミクス(トランスクリプトミクス)技術と同じくらい重要です4,5,6,7,8。さらに、タンパク質の存在量は必ずしもmRNAの存在量9から推測できるわけではなく、プロテオームはトランスクリプトーム10よりも複雑で動的である。これらの課題を考慮すると、多数の混合細胞集団(すなわち、バルク細胞)が一般に、包括的なプロテオームデータを生成するために使用される11,12,13。しかし、このようなバルク測定は、個々の細胞の確率的変動を平均化するため、重要な細胞間の変動性(すなわち、細胞の不均一性)を不明瞭にする4,14。このようなバルク測定の限界は、目的の細胞が細胞の全集団のごく一部しか占めていない場合(例えば、早期がんの腫瘍内のがん幹細胞)ではさらに厳しくなります。したがって、シングルセルプロテオミクスとゲノミクス(トランスクリプトミクス)の間には大きな知識のギャップがあります。
抗体ベースのイムノアッセイ(例えば、フローサイトメトリーまたはマスサイトメトリー)は、主に単一細胞の標的プロテオミクス分析に使用されます6、7、15、16、17、18。しかし、マルチプレックスが低く、特異性が限られており、目的の新しいタンパク質に対する抗体が利用できないという問題があります。質量分析(MS)ベースの標的プロテオミクスは、抗体を含まず、高いマルチプレックス(1回の分析で≥150のタンパク質19)、高い定量精度(絶対量または濃度)、および高い特異性と再現性(≤10%CV)20,21,22,23により、正確なタンパク質定量の代替手段として浮上しています20,21,22,23。.近年のサンプル調製の大幅な進歩により、MSベースのシングルセルプロテオミクスは、ヒト単一細胞から存在量の多いタンパク質を定量的に分析することが可能になりました。しかし、MSベースのシングルセルプロテオミクスは、まだ初期段階にあります。例えば、最先端のMSプラットホームと超低流量RPLC流量の組み合わせでは、単一HeLa細胞24,25に含まれる合計≥12,000のタンパク質のうち、~670-870のタンパク質のラベルフリーMS検出と定量しかできません。
現在、単一哺乳類細胞の解析には6つのMSベースのシングルセルプロテオミクスアプローチが利用可能であり、そのうち4つはグローバルプロテオミクス用です(nanoPOTS:Nanowell-based Preparation in One Pot for Trace Samples26; iPAD-1:シングルセル解析用の統合プロテオーム解析デバイス27;OAD(油-空気滴)チップベースのシングルセルプロテオミクス解析28;SCoPE-MS:質量分析によるシングルセルプロテオミクス29)と他の2つはターゲットプロテオミクス用です(cLC-SRM:キャリア支援液体クロマトグラフィー(LC)-選択反応モニタリング(SRM)30;cPRISM-SRM:SRM31に結合されたインテリジェント選択とマルチプレックスによるキャリア支援高圧、高分解能分離).ただし、これらのアプローチにはすべて技術的な欠点があります。nanoPOTS、iPAD-1、およびOADは、サンプル処理量を2〜200nLにダウンスケールし、広範なベンチトップアプリケーションには準備ができていません26,27,28。SCoPE-MSでは、シングルセルプロセシング後にTMT(タンデムマスタグ)キャリアを付加するため、シングルセルプロセッシングにシングルチューブを用いると、サンプルプロセッシング中の表面吸着損失を効果的に防ぐことができず29、リピート間の相関係数が~0.2-0.4程度と再現性が低くなる32。cLC-SRMおよびcPRISM-SRMの場合、過剰な外因性タンパク質からのペプチドはMS/MSによって頻繁に配列決定されるため、外因性タンパク質をキャリアとして使用することは、標的プロテオミクスにより適しており、これにより、存在量が少ない内在性ペプチドの配列決定の機会が大幅に減少します30,31。相対定量のためのグローバルプロテオミクスとは異なり、2つのターゲットプロテオミクスアプローチは、既知の濃度で重同位体標識内部標準を使用して、少数の細胞の高精度または絶対的なタンパク質分析を高い再現性で提供できます。cPRISM-SRMは、事前に高分解能PRISM分画が必要で、その結果、多くの分画サンプルを分析する必要があるのに対し、cLC-SRMは分画なしのサンプルスループットに大きな利点があり、1回の分析で数百のタンパク質を同時に定量できますが、検出感度は比較的低くなっています30。.したがって、cLC-SRMはよりアクセスしやすく、少数の細胞や質量が限られているサンプルの正確なマルチプレックスタンパク質分析のための幅広い有用性を備えている必要があります。
ここでは、単一細胞を含む少数のヒト細胞の便利な標的プロテオミクス解析のためのcLC-SRMを実施するための詳細なプロトコールについて説明します。このプロトコールは、FACS(蛍光活性化細胞ソーティング)による細胞ソーティング、少量の単一ポリメラーゼ連鎖反応(PCR)チューブで処理された細胞溶解および消化、LC-SRMデータ収集、および公開されているSkylineソフトウェアを使用したSRMデータ解析(図1)の主要なステップで構成されています。その広範な有用性は、1-100 MCF7またはMCF10A細胞におけるEGFR/MAPK経路タンパク質の絶対標的定量、および広いダイナミックレンジの濃度での細胞当たりの経路タンパク質コピーの測定により、以前に確立されたSRMアッセイとともに実証されました30。詳細なプロテオミクス研究者は、詳細なプロトコルにより、プロジェクトのニーズを満たすために、超微小サンプル(希少腫瘍細胞など)の正確なタンパク質分析のために、cLC-SRMを研究室に容易に導入できると予想しています。
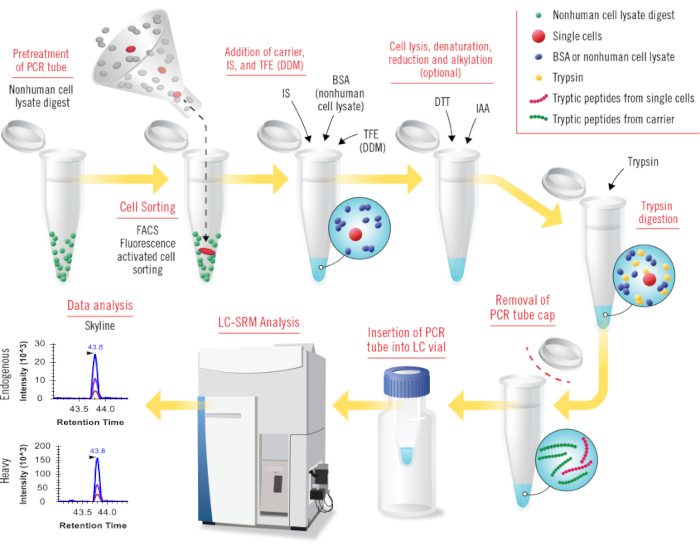
図1:cLC-SRMのすべてのステップの概要(carried-assisted one-pot sample preparation coupled with liquid chromatography -selected reaction monitoring)。 非ヒト細胞ライセート消化物( 例:Shewanella oneidensis)は、チューブ表面をコーティングするためのPCRチューブの前処理に使用されます。FACSによって選別された少数のヒト細胞または単一細胞を、前処理されたPCRチューブに収集します。BSAタンパク質(または非ヒト細胞溶解物プロテオーム)キャリア、重質内部標準(IS)、およびTFE(またはDDM)をサンプルチューブに順次添加することで、細胞溶解を促進し、表面吸着損失を低減します。概念的には、DDMと非ヒト細胞ライセートプロテオームキャリアの組み合わせは、cLC-SRMで良好に機能します。細胞溶解は超音波処理によって行われ、タンパク質の変性は高温で加熱することによって達成されます。DTT試薬とIAA試薬は、それぞれ還元とアルキル化に使用されます(このステップはオプションです)。トリプシンは標準的なトリプシンの消化のためのそれより蛋白質の量上のトリプシンの大いに高い比率の消化のために加えられます。サンプルチューブのキャップを取り外し、PCRチューブをLCバイアルに挿入して、直接LC-SRM分析を行います。収集された SRM データは、公開されている Skyline ソフトウェアを使用して分析されます。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
注:ステップバイステップのcLC-SRM分析を 図1に示します。
1. PCRチューブの前処理
- 0.2 μg/μLの非ヒト細胞( Shewanella oneidensisなど)細胞ライセート消化物100 μLをPCRチューブに加えます。室温で一晩インキュベートし、PCRチューブ表面をコーティングします。
- ピペッティングで細胞ライセート消化物を除去し、PCRチューブをHPLCグレードの水で3回すすぎ、次にPCRチューブをドラフト内で空気乾燥させます。
- コーティングされたPCRチューブは、さらに使用するまで4°Cの冷凍庫で保管してください。
2. FACSの選別
- 蛍光活性化セルソーター(FACS)(材料表を参照)を96ウェルPCRプレートに整列させ、レーザー光源によって励起できる色素を含む蛍光ビーズを使用して、信頼性の高い参照シグナルを提供します。
- FACSマシンがPCRプレートの底部に細胞を選別することを目的としていることを確認します。
- ソートストリームの角度を可能な限り最小に設定して、セルが側面にヒットするのではなくウェルの底にソートされる成功率を最大化します。
- マシンを照準するには、テストストリームを使用してPBSバッファーの液滴(100個の液滴など)を空のプレートのウェルに「選別」し、収集された液滴が底にあることを確認します。液滴が正しい位置に着弾しない場合は、セルソーターを再キャリブレーションし、ソーティングストリーム内の液滴が正しく収集されるまで繰り返します。
- コーティングされたPCRチューブを96ウェルPCRチューブラックに入れます。
- 必要な数のヒト乳房細胞(MCF7またはMCF10A)をプレコートPCRチューブにソーティングします。
- 選別した乳房細胞を直ちに100 x g で4°Cで10分間遠心分離し、細胞の損失を防ぐためにチューブの底に保持します。
- 選別した細胞は、さらなる分析が行われるまで-80°Cの冷凍庫で保存します。
3. タンパク質担体、重質内部標準物質、TFEの添加
- 4 μL の 25 mM NH4HCO3 を、採取した細胞を含む PCR チューブに加えます。
- 1 μL の 10 ng/μL ウシ血清アルブミン (BSA) を 25 mM NH4HCO3 に 1 μL 加えます。
- 0.3 μL の 100 fmol/μL 粗重ペプチド標準試料 (合計 30 fmol) をサンプルチューブに加えます。
注:EGFR/MAPK経路タンパク質用の粗重ペプチドスタンダードのアリコートは、25 mM NH4HCO3中の公称濃度100 fmol/μLで調製されます。少量の粗重ペプチドスタンダードを100 fmol/μLで溶液の中央に分注し、ピペットチップに液体が残っていないことを確認します。 - 9 μL の 100% TFE をサンプルチューブに加え、最終濃度が ~60% TFE になるようにします。
- 1500 x gで5分間遠心分離し、次に100 x gで3分間穏やかにボルテックスします。
4. 細胞溶解とタンパク質変性
- FACSで選別した乳房細胞を氷上で1分間、1分間隔で、70%の振幅と0.5Hzで合計5サイクル超音波処理します。
- サンプルを90°Cで1時間インキュベートし、PCRサーモサイクラーと加熱蓋オプションを使用してタンパク質変性を行います。
注:TFEは沸点が74°Cの揮発性溶媒であるため、サンプルの損失が大幅に発生する完全な乾燥を避けるために、加熱蓋オプションが選択されています。 - サンプルを室温まで冷却し、1500 x g で3分間遠心分離します。
5.還元とアルキル化(オプション)
- 0.6 μL の 50 mM DTT を添加して、最終濃度を 2 mM にします。
注:DTT溶液は毎回新たに調製する必要があります。77.5 mg の DTT を秤量し、1 mL の 25 mM NH4HCO3 に溶解して 500 mM DTT を作ります。次に、10 μL の 500 mM DTT を 90 μL の 25 mM NH4HCO3 に加えて、50 mM DTT を作成します。 - 1500 x g で5分間遠心分離し、850 rpmで3分間穏やかに混合します。
- 56°Cで1時間インキュベートし、100 x gで穏やかに振とうします。
- サンプルを室温まで冷却し、1500 x g で3分間遠心分離します。
- 0.5 μL の 60 mM IAA を RT-PCR チューブに 0.5 μL を加え、最終濃度を ~2 mM にします。
注:IAA溶液は、毎回新たに調製する必要があります。74.3 mg の IAA を秤量し、次に 1 mL の 25 mM NH4HCO3 に溶解して 400 mM IAA を作ります。15 μL の 400 mM IAA を 85 μL の 25 mM NH4HCO3 に添加して、60 mM IAA を調製します。 - 暗所で室温で30分間インキュベートし、100 x gで穏やかに振とうします。
注:標的タンパク質に選択されたシステイン含有ペプチドがない場合、還元およびアルキル化ステップはオプションです。
6.トリプシン消化
- SpeedVac濃縮器を使用して、サンプル量を~4 μLに減らします。
注:TFEは、沸点が74°Cの揮発性溶媒です。 TFEの割合が高いと、トリプシンの消化が阻害されます。 - 9 μLの25 mM NH4HCO3 と1-3 μLの15 ng/μLトリプシンを添加して、最終トリプシン濃度が1-3 ng/μLになるように分解します。
注:10個のFACS選別ヒト乳房細胞(~1 ng)の場合、タンパク質に対するトリプシン酵素の比率は≥15:1で、標準的なトリプシン消化(1:50の比率)よりも750倍高くなります。 - 100 x g で3分間穏やかに混合し、37°Cで一晩(~16時間)インキュベートします。
- 0.5 μLの5%ギ酸を添加して酵素反応を停止します。
- 1500 x gで1時間遠心分離します。
- LC-SRM分析が進むまで-80°Cのフリーザーで保存するか、すぐに分析するために4°Cのフリーザーで保存してください。
7. 直接LC-SRM分析の準備
- FACSで選別した乳房細胞の採取(ステップ2)と処理(ステップ6)に使用したPCRチューブのキャップを外します。
- PCRチューブをLCバイアルに挿入します。
- LC-SRM分析のためにLCバイアルを閉じます。
注:96ウェルPCRプレートで処理された少数の細胞の場合、直接LC-SRM分析のためにプレートをシールするためにマットカバーが追加されます。
8. LC-SRM分析
- UPLCとトリプル四重極質量分析計を組み合わせてサンプルを分析します。
- キャピラリー C18 カラム(標準グラジエントでは内径 75 μm × 20 cm、内径 100 μm ×短グラジエントでは 10 cm)を、化学エッチングされた内径 20 μm のフューズドシリカエレクトロスプレーエミッターにステンレス製の金属ユニオンを介して接続します。
- 20 μLのサンプルループを使用して、すべてのサンプル(合計~15 μL)をLCカラムに直接ロードし、検出感度を最大化します。
- 0.1% ギ酸水溶液、0.1% ギ酸 90% アセトニトリル溶液を移動相 A および B として使用して、キャピラリー RPLC 分離を行います。
- 300 nL/minの流速でバイナリLCグラジエントを、多数の標的タンパク質(>10)を測定する標準的なグラジエントに使用します。標準グラジエント:26分で5-20%B、10分で20-25%B、8分で25-40%B、1分で40-95%B、95%Bで7分間合計52分間、分析カラムを99.5%Aで8分間再平衡化。
- 400 nL/minの流速でバイナリLCグラジエントを使用し、少数の標的タンパク質(≤10)を測定する短いグラジエントに使用します。迅速な分離のための短いグラジエント:5 分で 5-95% B を測定し、分析カラムを 99.5% A で 5 分間再平衡化し、合計 10 分間の LC 分析時間で分析します。
- QQQ質量分析計は、イオン噴霧電圧2,400±100V、キャピラリーオフセット電圧35V、スキマーオフセット電圧-5V、キャピラリーインレット温度220°Cで操作します。
- 他の QQQ MS パラメーターは、自動チューニングおよびキャリブレーションから取得でき、さらに最適化する必要はありません。
- サンプルの LC-SRM 分析の前に、異なる濃度の粗重ペプチド標準試料を 20 〜 50 ng/μL の非ヒト細胞ライセート消化物にスパイクして、スケジュールされた SRM メソッドを構築するためのペプチド LC 保持時間(RT)と、正確な定量のための潜在的な干渉トランジションを決定します。
- RTスケジュールSRMモードを使用して、スキャンウィンドウ≥6分でマルチプレックス定量を行います。
注意: スキャンウィンドウは、特定の時間枠内の遷移の数に応じてそれに応じて調整されます。 - 合計サイクル時間を 1 秒に設定すると、各トランジションの滞留時間は、異なる保持時間ウィンドウでスキャンされたトランジションの数に応じて自動的に調整されます。各 SRM 遷移には、最小 10 ミリ秒の滞留時間が必要です。すべての標的タンパク質を1回のLC-SRM分析で同時にモニター可能
9. データ分析
- 生データファイルを一般に入手可能なSkylineソフトウェア33 にインポートして、各標的ペプチドのデータを視覚化し、それらの検出可能性を決定する。
- SRMピークの検出と波形解析を決定するには、2つの基準を使用します:1)同じLC保持時間。2)内因性(軽)ペプチドとそれに対応する(重)同位体標識ペプチド内部標準との間の複数のトランジションにわたる相対SRMピーク強度比がほぼ同じである。
- 上記の 2 つの基準を使用して信頼性の高い定量を行うには、すべての SRM データを手動で検査し、正しい SRM ピークの割り当てと SRM ピーク境界を確認します。
- タンパク質の定量には、高いSRMシグナルを持ちながらマトリックス干渉のない最適なトランジションを使用し、他のトランジションをリファレンスとして使用します。
- 複数の反復を使用して、標準偏差(SD)と変動係数(CV)を求めます。
- S/N比(S/N)は、標準グラジエントで±15秒、ショートグラジエントで±6秒の保持時間領域内の平均バックグラウンドノイズに対するピーク頂点強度で計算します。検出限界(LOD)と定量限界(LOQ)は、S/N比がそれぞれ3と7以上になる最低濃度ポイントとして定義されます。
- S/N ≥7以外のLOQ値を控えめに決定するためのもう1つの基準を適用します:異なる細胞数に対する代理ペプチド応答は、線形ダイナミックレンジ内にある必要があります。
- Excelソフトウェアを使用して、すべての検量線をプロットします。QQQ MS の RAW データを一般に公開されている Skyline ソフトウェアにロードして、モニター対象の標的タンパク質の複数のトランジションの抽出イオンクロマトグラム(XIC)のグラフを表示します。
結果
EGFR/MAPK経路タンパク質の標的定量によるcLC-SRMの性能評価には、FACSで選別した少数の細胞と比較して、より均一で変動が少ないため、少量のMCF7細胞ライセート(0.5-20 ng、5-200細胞相当)が最初に使用されました。図2Aに示されているように、XICは、MCF7セル34あたり~220,000コピーに存在するGRB2から導出された...
ディスカッション
cLC-SRMは、単一細胞を含む少数の細胞の正確なマルチプレックスタンパク質解析を可能にする便利なターゲットプロテオミクス法です。この分析法は、タンパク質キャリア支援によるワンポットサンプル調製を利用しており、細胞収集、マルチステップの細胞溶解および消化、MS分析のためのキャピラリーLCカラムへのペプチド消化物の移し替えを含むすべてのステッ?...
開示事項
著者は何も開示していません。
謝辞
この研究は、NIH Grant R21CA223715 (TS) と UG3CA256967 (TS および HL) の支援を受けました。本明細書に記載の実験作業は、米国エネルギー省が後援する全米科学ユーザー施設である太平洋岸北西部国立研究所の環境分子科学研究所で、契約DE-AC05-76RL0 1830の下で行われた。
資料
| Name | Company | Catalog Number | Comments |
| 2 mL glass LC vial | Microsolv | 9502S-WCV | Vessel to hold PCR tube for autosample injection |
| BSA | Sigma-Aldrich | P0834-10×1mL | Carrier protein for greatly reducing surface adsorption losses |
| DTT | Thermo Scientific | A39255 | Reagent for reduction |
| Formic acid | Thermo Scientific | 28905 | Reagent for stopping enzyme reaction |
| IAA | Thermo Scientific | A39271 | Reagent for alkylation |
| Peptide internal standards | New England peptide | Targeted quantification of EGFR/MAPK pathway proteins | |
| RT-PCR tube | GeneMate Bioexpress | T-3035-1 | 0.2 mL PCR tube for one-pot sample preparation |
| Skyline software | University of Washington | Publicly available for SRM data analysis | |
| Sonicator | Hielscher Ultrasound Technology | UTR200 | Sonication on ice for cell lysis |
| Speed Vac concentrator | Thermo Scientific | Reduction of the percentage of TFE for effective trypsin digestion | |
| TFE | Sigma-Aldrich | 18370-10×1mL | 60% TFE for cell lysis |
| Thermocycler w/ heated lid | Peltier Thermal Cycler | PTC-200 | Heating for protein denaturation |
| Trypsin Gold | Promega | V528A | Enzyme for protein digestion |
| Waters BEH C18 column | Waters | C18 column for peptide separation |
参考文献
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Miyamoto, D. T., et al. RNA-Seq of single prostate CTCs implicates noncanonical Wnt signaling in antiandrogen resistance. Science. 349 (6254), 1351-1356 (2015).
- Wang, Y., Navin, N. E. Advances and applications of single-cell sequencing technologies. Molecular Cell. 58 (4), 598-609 (2015).
- Macaulay, I. C., Ponting, C. P., Voet, T. Single-Cell Multiomics: Multiple Measurements from Single Cells. Trends in Genetics. 33 (2), 155-168 (2017).
- Gavasso, S., Gullaksen, S. E., Skavland, J., Gjertsen, B. T. Single-cell proteomics: potential implications for cancer diagnostics. Expert Review of Molecular Diagnostics. 16 (5), 579-589 (2016).
- Wu, M., Singh, A. K. Single-cell protein analysis. Current Opinion in Biotechnology. 23 (1), 83-88 (2012).
- Hughes, A. J., et al. Single-cell western blotting. Nature Methods. 11 (7), 749-755 (2014).
- Peterson, V. M., et al. Multiplexed quantification of proteins and transcripts in single cells. Nature Biotechnology. 35 (10), 936-939 (2017).
- Liu, Y., Beyer, A., Aebersold, R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. 165 (3), 535-550 (2016).
- Choudhary, C., Mann, M. Decoding signalling networks by mass spectrometry-based proteomics. Nature Reviews Molecular Cell Biology. 11 (6), 427-439 (2010).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Zhang, H., et al. Integrated Proteogenomic Characterization of Human High-Grade Serous Ovarian Cancer. Cell. 166 (3), 755-765 (2016).
- Zhang, B., et al. Proteogenomic characterization of human colon and rectal cancer. Nature. 513 (7518), 382-387 (2014).
- Gaudet, S., Miller-Jensen, K. Redefining Signaling Pathways with an Expanding Single-Cell Toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Di Palma, S., Bodenmiller, B. Unraveling cell populations in tumors by single-cell mass cytometry. Current Opinion in Biotechnology. 31, 122-129 (2015).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Current Protocols in Immunology. , 11-20 (2012).
- Willison, K. R., Klug, D. R. Quantitative single cell and single molecule proteomics for clinical studies. Current Opinion in Biotechnology. 24 (4), 745-751 (2013).
- Lee, J. Y., et al. Detection of Head and Neck Cancer Based on Longitudinal Changes in Serum Protein Abundance. Cancer Epidemiology, Biomarkers & Prevention. 29 (8), 1665-1672 (2020).
- Shi, T., et al. Advancing the sensitivity of selected reaction monitoring-based targeted quantitative proteomics. Proteomics. 12 (8), 1074-1092 (2012).
- Shi, T., et al. Advances in targeted proteomics and applications to biomedical research. Proteomics. 16 (15-16), 2160-2182 (2016).
- Lange, V., Picotti, P., Domon, B., Aebersold, R. Selected reaction monitoring for quantitative proteomics: a tutorial. Molecular Systems Biology. 4, 222 (2008).
- Picotti, P., Aebersold, R. Selected reaction monitoring-based proteomics: workflows, potential, pitfalls and future directions. Nature Methods. 9 (6), 555-566 (2012).
- Zhu, Y., et al. Proteomic Analysis of Single Mammalian Cells Enabled by Microfluidic Nanodroplet Sample Preparation and Ultrasensitive NanoLC-MS. Angewandte Chemie International Edition. 57 (38), 12370-12374 (2018).
- Cong, Y., et al. Improved Single-Cell Proteome Coverage Using Narrow-Bore Packed NanoLC Columns and Ultrasensitive Mass Spectrometry. Analytical Chemistry. 92 (3), 2665-2671 (2020).
- Zhu, Y., et al. Nanodroplet processing platform for deep and quantitative proteome profiling of 10-100 mammalian cells. Nature Communications. 9 (1), 882 (2018).
- Shao, X., et al. Integrated Proteome Analysis Device for Fast Single-Cell Protein Profiling. Analytical Chemistry. 90 (23), 14003-14010 (2018).
- Li, Z. Y., et al. Nanoliter-Scale Oil-Air-Droplet Chip-Based Single Cell Proteomic Analysis. Analytical Chemistry. 90 (8), 5430-5438 (2018).
- Budnik, B., Levy, E., Harmange, G., Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome Biology. 19 (1), 161 (2018).
- Zhang, P., et al. Carrier-Assisted Single-Tube Processing Approach for Targeted Proteomics Analysis of Low Numbers of Mammalian Cells. Analytical Chemistry. 91 (2), 1441-1451 (2019).
- Shi, T., et al. Facile carrier-assisted targeted mass spectrometric approach for proteomic analysis of low numbers of mammalian cells. Communications Biology. 1, 103 (2018).
- Vitrinel, B., Iannitelli, D. E., Mazzoni, E. O., Christiaen, L., Vogel, C. Simple Method to Quantify Protein Abundances from 1000 Cells. ACS Omega. 5 (25), 15537-15546 (2020).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26 (7), 966-968 (2010).
- Shi, T., et al. Conservation of protein abundance patterns reveals the regulatory architecture of the EGFR-MAPK pathway. Science Signaling. 9 (436), 6 (2016).
- Addona, T. A., et al. Multi-site assessment of the precision and reproducibility of multiple reaction monitoring-based measurements of proteins in plasma. Nature Biotechnology. 27 (7), 633-641 (2009).
- He, J., et al. Analytical platform evaluation for quantification of ERG in prostate cancer using protein and mRNA detection methods. Journal of Translational Medicine. 13, 54 (2015).
- Shi, T., et al. Antibody-free, targeted mass-spectrometric approach for quantification of proteins at low picogram per milliliter levels in human plasma/serum. Proceedings of the National Academy of Sciences. 109 (38), 15395-15400 (2012).
- Shi, T., et al. Targeted quantification of low ng/mL level proteins in human serum without immunoaffinity depletion. Journal of Proteome Research. 12 (7), 3353-3361 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved