A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
التعرف التلقائي على الفروع التشجرية وتوجهها
In This Article
Summary
المقدمة هي أداة حسابية تسمح قياس تلقائي بسيط ومباشر للتوجهات الفروع التشجر العصبية من الصور الفلورية 2D.
Abstract
بنية الأشجار التشجر العصبي يلعب دورا رئيسيا في دمج المدخلات متشابك في الخلايا العصبية. لذلك ، فإن توصيف مورفولوجيا التشعبات أمر ضروري لفهم أفضل لوظيفة الخلايا العصبية. ومع ذلك ، فإن تعقيد الأشجار التشجرية ، سواء عندما تكون معزولة وخاصة عندما تقع داخل شبكات الخلايا العصبية ، لم يكن مفهوما تماما. طورنا أداة حسابية جديدة ، SOA (تحليل التقسيم والتوجيه) ، والتي تسمح للقياس التلقائي لتوجيه الفروع التشجرية من الصور الفلورية للثقافات العصبية ثنائية الأبعاد. SOA، مكتوبة في بيثون، ويستخدم تجزئة لتمييز الفروع dendritic من خلفية الصورة ويتراكم قاعدة بيانات عن الاتجاه المكاني لكل فرع. ثم يتم استخدام قاعدة البيانات لحساب المعلمات المورفولوجية مثل التوزيع الاتجاهي للفروع التشجرية في الشبكة وانتشار نمو الفرع المتوازي. ويمكن استخدام البيانات التي تم الحصول عليها للكشف عن التغيرات الهيكلية في التشعبات استجابة لنشاط الخلايا العصبية والمحفزات البيولوجية والصيدلانية.
Introduction
تكوين المورفوجين Dendritic هو موضوع مركزي في علم الأعصاب ، حيث يؤثر هيكل الشجرة التغصنية على الخصائص الحسابية للتكامل المتشابك في الخلايا العصبية1،2،3. وعلاوة على ذلك، فإن التشوهات المورفولوجية والتعديلات في الفروع التغصنية متورطة في اضطرابات تنكسية وعصبية النمو4,5,6. في الثقافات العصبية حيث يمكن تصور تشعبات التشجر بسهولة أكبر ، تنظم التفاعلات بين الفروع غير الشقيقة مواقع ومدى التجمع المتشابك على طول الفروع ، وهو سلوك قد يؤثر على التفاعل المتشابك واللدونة7،8،9. لذلك، توصيف المعلمات المورفولوجية للشجرة التغصنية باستخدام الثقافات العصبية ثنائية الأبعاد (2D) مفيد لفهم مورفوجينيسيس التغصني ووظائف واحدة وشبكات الخلايا العصبية. ومع ذلك ، هذه مهمة صعبة لأن الفروع التشجرية تشكل شبكة معقدة حتى في الثقافات العصبية ثنائية الأبعاد "المبسطة".
وقد تم تطوير العديد من الأدوات لتتبع وتحليل الهياكل التغصنية تلقائيا10,11,12,13. ومع ذلك، تم تصميم معظم هذه الأدوات لشبكات الخلايا العصبية ثلاثية الأبعاد وهي بطبيعة الحال معقدة للغاية لاستخدامها مع الشبكات ثنائية الأبعاد. وعلى النقيض من ذلك، تنطوي أدوات التحليل المورفولوجي الأقل تقدما عادة على مكون هام من العمل اليدوي بمساعدة الحاسوب، وهو يستغرق وقتا طويلا جدا وعرضة لتحيز المشغل14. الأدوات شبه التلقائية الحالية ، مثل "ImageJ'15 (حزمة معالجة الصور مفتوحة المصدر من NIH مع مجموعة واسعة من أدوات تحليل الصور البيولوجية المطورة من المجتمع) ، تقلل إلى حد كبير من العمل اليدوي للمستخدم. ومع ذلك، لا تزال هناك حاجة إلى بعض التدخلات اليدوية أثناء معالجة الصور، ويمكن أن تكون جودة التقسيم أقل من المرغوب فيه.
تقدم هذه الورقة SOA ، وهي أداة آلية بسيطة تسمح بتقسيم مباشر وتحليل اتجاه الفروع التغصنية داخل شبكات الخلايا العصبية ثنائية الأبعاد. يمكن للSA الكشف عن مختلف الأجسام الشبيهة بالخط في الصور 2D وتوصيف خصائصها المورفولوجية. هنا ، استخدمنا SOA لتقسيم الفروع التشجرية في صور مضانة 2D من الشبكات التشجرية في الثقافة. ويحدد البرنامج الفروع التشجرية وينجح في إجراء قياسات للمعلمات المورفولوجية مثل التوازي والتوزيع المكاني. ويمكن تكييف ال SOA بسهولة لتحليل العمليات الخلوية لأنواع الخلايا الأخرى ولدراسة الشبكات غير البيولوجية.
Protocol
ملاحظة: وافقت وزارة الصحة الإسرائيلية على استخدام الفئران بموجب البروتوكول IL-218-01-21 للاستخدام الأخلاقي للحيوانات التجريبية. SOA متوافق فقط مع ويندوز 10 وبيثون 3.9. وهي متاحة كشفرة مفتوحة المصدر: https://github.com/inbar2748/DendriteProject. في هذا الارتباط، هناك أيضا README. DM يحتوي على توجيهات لتحميل البرنامج، وصلة إلى موقع البرنامج، وملف متطلبات تحتوي على معلومات عن الإصدارات المطلوبة من جميع الحزم. وقدمت أمثلة إضافية للتحليل الذي تم إجراؤه باستخدام البرنامج هناك أيضا.
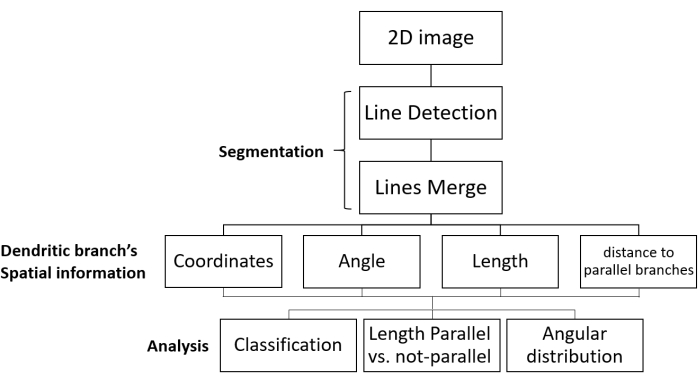
الشكل 1: سير عمل ال SOA لتحليل التقسيم واتجاه النمو. تظهر خطوات معالجة الصور الفلورية للشبكات المتشعبة وتحليل البيانات. يتم تحميل الصورة ثنائية الأبعاد، وتجزئة (في خطوتين: يتم الكشف عن الفروع المتشعبة كخطوط، ومن ثم يتم دمج الخطوط ذات الصلة)، ويتم الحصول على المعلومات المكانية لكل فرع دندريتيك. يتم جمع البيانات لجميع الفروع dendritic في الصورة. وأخيرا، يتم تحليل البيانات لإعطاء المعلمات المورفولوجية المطلوبة. اختصار: SOA = تحليل التقسيم والتوجه. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
1. افتح تطبيق SOA.
- فتح عنوان URL: https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA، والعثور على المجلد SOA.zip مضغوط، وتحميل ملف ZIP بالنقر المزدوج.
- فك ضغط المجلد بالنقر بزر الماوس الأيمن على SOA.zip واختيار استخراج الملفات. مراقبة مسار الاستخراج والخيارات الإطار الذي يفتح مربع النص عنوان الوجهة الذي يعرض المسار للملفات المستخرجة. لاستخراج إلى موقع مختلف، انقر على أحد المجلدات في لوحة الإطار الأيمن لجعله المجلد الوجهة. انقر فوق موافق لاستخراج الملفات إلى هذا المجلد.
- افتح ملف SOA المستخرج وانقر نقرا مزدوجا فوق SOA.exe. انتظر حتى يتم فتح إطار أسود، وبعد ذلك سيظهر التطبيق.

الشكل 2: مثال لسير العمل باستخدام واجهة المستخدم الرسومية الخاصة ب SOA. العمود الأيسر: مقاطع GUI لسير العمل. العمود الأوسط: صورة لشبكة dendritic، تتم معالجتها أثناء سير العمل (شريط المقياس: 20 ميكرومتر). العمود الأيمن: تكبير المنطقة التي تم تمييزها ب مستطيل أحمر في صور العمود الأوسط (شريط المقياس: 4 ميكرومتر). الخطوة 1: تحديد صورة وتحميلها. الخطوة 2: المرحلة الأولى من التقسيم هي الكشف عن الخطوط التي تمثل الفروع التشجرية المحددة. الخطوة 3: المرحلة الثانية من التقسيم هي الاندماج القائم على القرب من بطانة القطاع في الفروع التشجرية الفردية. يمكن تعديل إعدادات كافة الخطوات. المختصرات: SOA = تحليل التقسيم والتوجيه؛ GUI = واجهة المستخدم الرسومية. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
2. افتح صورة للتحليل.
- في شريط القائمة تحميل عارض SOA | حدد اختيار | الملف اختيار صورة من ملفات الكمبيوتر | انقر عليه (.png .jpg .tif .bmp الملفات فقط) | فتح | مراقبة مسار الملف | مقبل.
3. تحسين التقسيم
ملاحظة: في شريط القائمة خصائص عارض SOA تغيير قيم المعلمات المحددة لضبط إعدادات عملية التقسيم. ويرد وصف مفصل للمعلمات، مثل العتبة، في المواد التكميلية.
- في الحواف، اضبط عتبة العرض عن طريق تحديد العتبة وإدخال رقم.
ملاحظة: كلما انخفض عدد العتبة، كلما تم الكشف عن المزيد من الأسطر. العتبة هي رقم يتراوح من 0 إلى 255. تم تعيين القيمة الافتراضية إلى 0. - في خطوط دمج:
- اضبط الحد الأدنى للمسافة لدمجها للعرض عن طريق تحديد مسافة الحد الأدنى لدمجها وإدخال رقم.
ملاحظة: المسافة الحد الأدنى لدمج يتراوح من 0 إلى 30 بكسل. يتم تعيين القيمة الافتراضية إلى 20. - ضبط الحد الأدنى من زاوية لدمج للعرض عن طريق تحديد زاوية دقيقة لدمج وإدخال رقم.
ملاحظة: زاوية Min لدمج يتراوح من 0 إلى 30 درجة. يتم تعيين القيمة الافتراضية إلى 10.
- اضبط الحد الأدنى للمسافة لدمجها للعرض عن طريق تحديد مسافة الحد الأدنى لدمجها وإدخال رقم.
- انقر على إنشاء معاينة تجزئة الصورة.
ملاحظة: سيتم عرض صورة معاينة لنتائج التقسيم وفقا للقيم المحدثة. بالإضافة إلى ذلك، سيتم عرض عدد الأسطر قبل الدمج وعدد الأسطر بعد الدمج. - تغيير المعلمات لتحقيق أقصى تعريف للشرائح. إذا كانت هناك حاجة لتغيير خصائص، انقر على إغلاق النافذة زر واتبع الخطوات 3.1-3.4.
4. إنشاء ملفات الإخراج.
- اضغط موافق لتصور الصور تجزئة والرسوم البيانية تحليل. مراقبة الإطار الذي يظهر لتحديد موقع حيث سيتم حفظ الملف .xlsx.
- إدراج اسم ملف | اختر حفظ | انتظر حتى يتم إنشاء ملف .xlsx مع البيانات وحفظها.
ملاحظة: بالإضافة إلى الملف .xlsx، سيتم عرض الملفات التالية تلقائيا: ملف يعرض الصورة الأصلية، صورة التعرف على الأسطر، الصورة النهائية للتجزئة، وثلاثة رسوم بيانية للتحليل.
5. شريط أدوات التنقل
ملاحظة: يتم تضمين شريط أدوات التنقل في كافة إطارات الشكل ويمكن استخدامه للتنقل خلال مجموعة البيانات. يتم وصف كل زر من الأزرار الموجودة أسفل شريط الأدوات أدناه.
- للتنقل ذهابا وإيابا بين طرق العرض المعرفة مسبقا، استخدم الزرين للأمام والخلف.
ملاحظة: أزرار الصفحة الرئيسية وإلى الأمام والظهر مشابهة لعناصر التحكم الرئيسية وإلى الأمام والظهر على مستعرض ويب. يعود المنزل إلى الشاشة الافتراضية، الصورة الأصلية. - استخدم زر التكبير/التصغير لتكبير/تصغير. لتنشيط التحريك والتكبير، اضغط على زر التكبير/التصغير ، ثم حرك الماوس إلى موقع مرغوب فيه في الصورة.
- التحريك الشكل اضغط باستمرار على زر الماوس الأيسر أثناء سحبه إلى موضع جديد. حرر زر الماوس، وستظهر النقطة المحددة في الصورة في الموضع الجديد. أثناء تحريك المفاتيح، اضغط باستمرار على مفاتيح x أو y لتقييد الحركة إلى محاور x أو y ، على التوالي.
- للتكبير، اضغط باستمرار على زر الماوس الأيمن واسحبه إلى موقع جديد. تحرك لليمين لتكبير المحور x وتحرك يسارا للتصغير على المحور x. افعل الشيء نفسه بالنسبة لمحور ص وحركات لأعلى/لأسفل. عند التكبير، لاحظ أن النقطة الموجودة تحت الماوس تبقى ثابتة، مما يسمح بالتكبير أو التصغير حول تلك النقطة. استخدم مفاتيح التعديل x أو y أو CONTROL للحد من التكبير/التصغير إلى الحفاظ على نسبة العرض إلى الارتفاع x أو y أو العرض إلى الارتفاع على التوالي.
- لتنشيط وضع التكبير/التصغير إلى المستطيل، انقر فوق الزر تكبير/تصغير إلى مستطيل . ضع المؤشر فوق الصورة واضغط على زر الماوس الأيسر. اسحب الماوس إلى موقع جديد أثناء الضغط على الزر لتحديد منطقة مستطيلة.
ملاحظة: سيتم تكبير حدود عرض المحاور إلى المنطقة المعرفة عند الضغط على زر الماوس الأيسر. سيتم تكبير حدود عرض المحاور عند الضغط على زر الماوس الأيمن، ووضع المحاور الأصلية في المنطقة المحددة. - استخدم أداة تكوين Subplot لتكوين مظهر المؤامرة الفرعية.
ملاحظة: يمكن أن تمتد أو ضغط الجوانب اليسرى، اليمنى، العلوية والسفلية من المؤامرة الفرعية، وكذلك المسافة بين الصفوف والأعمدة. - لفتح مربع حوار حفظ ملف، انقر فوق الزر حفظ وحفظ الملف بالتنسيقات التالية: .png أو .ps أو .eps أو .svg أو .pdf.
النتائج
10- وقد أجريت دراسة تمثيلية لصور شبكات التشجر في الثقافة. تم استخراج الخلايا كما وصفها بارانس وآخرون. 16,17. لفترة وجيزة، تم استخراج خلايا فرس النهر من أدمغة الفئران بعد الولادة وزرعها على أغطية الزجاج 2D لمدة 1-2 أسابيع. ثم تم إصلاح الثقافات وملطخة من خلال ال...
Discussion
وهناك حاجة ماسة إلى استراتيجيات فعالة لاستخراج المعلومات المورفولوجية من الصور ثلاثية الأبعاد لمواكبة بيانات التصوير البيولوجي. على الرغم من أنه يمكن توليد بيانات التصوير في ساعات ، إلا أن التحليل المتعمق للصور يستغرق وقتا طويلا. ونتيجة لذلك، أصبح من الواضح أن معالجة الصور تشكل عقبة رئي...
Disclosures
ويعلن أصحاب البلاغ أنه ليس لديهم مصالح مالية متنافسة.
Acknowledgements
ويود المؤلفون أن يشكروا الدكتور أورلي فايس على إعداد الصور الثقافية.
Materials
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
References
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083 (2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782 (2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206 (2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ Available from: https://imagej.nih.gov/ij (2021)
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved