Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Dendritik Dalların Otomatik Tanımlanması ve Oryantasyonları
Bu Makalede
Özet
Sunulan, 2D floresan görüntülerden nöronal dendritik dalların yönelimlerinin basit ve doğrudan otomatik olarak ölçülmasını sağlayan bir hesaplama aracıdır.
Özet
Nöronal dendritik ağaçların yapısı, sinaptik girdilerin nöronlara entegrasyonunda kilit rol oynar. Bu nedenle, nöronal fonksiyonun daha iyi anlaşılması için dendritlerin morfolojisinin karakterizasyonu esastır. Bununla birlikte, hem izole edildiğinde hem de özellikle nöronal ağlar içinde bulunduğunda, dendritik ağaçların karmaşıklığı tam olarak anlaşılamamıştır. 2B nöronal kültürlerin floresan görüntülerinden dendritik dalların yönünün otomatik olarak ölçülmesine izin veren yeni bir hesaplama aracı olan SOA 'yı (Segmentasyon ve Oryantasyon Analizi) geliştirdik. Python ile yazılan SOA, dendritik dalları görüntü arka planından ayırmak için segmentasyon kullanır ve her dalın uzamsal yönü üzerinde bir veritabanı biriktirir. Veritabanı daha sonra bir ağdaki dendritik dalların yönlü dağılımı ve paralel dendritik dal büyümesinin yaygınlığı gibi morfolojik parametreleri hesaplamak için kullanılır. Elde edilen veriler, nöronal aktiviteye yanıt olarak dendritlerdeki yapısal değişiklikleri tespit etmek ve biyolojik ve farmakolojik uyaranlara karşı kullanılabilir.
Giriş
Dendritik morfogenez, nöronlarda sinaptik entegrasyonun hesaplama özelliklerini etkilediği için, dendritik ağacın yapısı nörobilimde merkezi bir konudur1,2,3. Ayrıca dendritik branşlarda morfolojik anormallikler ve modifikasyonlar dejeneratif ve nöro-gelişimsel bozukluklara bulaşmıştır4,5,6. Dendritik sonuçların daha kolay görselleştirilebildiği nöronal kültürlerde, kardeş olmayan dendritik dallar arasındaki etkileşimler, kollar boyunca sinaptik kümelenmenin alanlarını ve kapsamını düzenler, sinaptik koaktiviteyi ve plastisiteyi etkileyebilecek bir davranış7,8,9. Bu nedenle, dendritik ağacın morfolojik parametrelerinin iki boyutlu (2D) nöronal kültürler kullanılarak karakterizasyonu, dendritik morfogenez ve tek ve nöron ağlarının işlevselliğini anlamak için avantajlıdır. Yine de, bu zorlu bir iştir, çünkü dendritik dallar "basitleştirilmiş" 2D nöronal kültürlerde bile karmaşık bir ağ oluşturur.
Dendritik yapıları otomatik olarak izlemek ve analiz etmek için çeşitli araçlar geliştirilmiştir10,11,12,13. Bununla birlikte, bu araçların çoğu 3D nöronal ağlar için tasarlanmıştır ve doğal olarak 2D ağlarla kullanılamayacak kadar karmaşıktır. Buna karşılık, daha az gelişmiş morfolojik analiz araçları tipik olarak bilgisayar destekli el emeğinin önemli bir bileşenini içerir, bu da çok zaman alıcı ve operatör önyargısına karşı hassastır14. 'ImageJ'15 (topluluk tarafından geliştirilen biyolojik görüntü analiz araçlarının geniş bir koleksiyonuna sahip bir NIH açık kaynaklı görüntü işleme paketi) gibi mevcut yarı otomatik araçlar, kullanıcı el emeğini büyük ölçüde azaltır. Bununla birlikte, görüntü işleme sırasında bazı manuel müdahalelere hala ihtiyaç vardır ve segmentasyonun kalitesi arzu edilenden daha az olabilir.
Bu makale, 2B nöronal ağlardaki dendritik dalların doğrudan segmentasyon ve oryantasyon analizine izin veren basit bir otomatik araç olan SOA'yı sunun. SOA, 2D görüntülerdeki çeşitli çizgi benzeri nesneleri algılayabilir ve morfolojik özelliklerini karakterize edebilir. Burada, kültürdeki dendritik ağların 2D floresan görüntülerinde dendritik dalları segmentlere ayırırken SOA'yı kullandık. Yazılım dendritik dalları tanımlar ve paralellik ve mekansal dağılım gibi morfolojik parametrelerin ölçümlerini başarıyla gerçekleştirir. SOA, diğer hücre tiplerinin hücresel süreçlerinin analizi ve biyolojik olmayan ağların incelenmesi için kolayca uyarlanabilir.
Protokol
NOT: İsrail Sağlık Bakanlığı, deney hayvanlarının etik kullanımı için IL-218-01-21 protokolü kapsamında fare kullanımını onayladı. SOA yalnızca Windows 10 ve Python 3.9 ile uyumludur. Açık kaynak kod olarak mevcuttur: https://github.com/inbar2748/DendriteProject. Bu bağlantıda, bir README de vardır. Yazılımı indirmek için yol tarifi, yazılımın web sitesine bir bağlantı ve tüm paketlerin gerekli sürümleri hakkında bilgi içeren bir gereksinim dosyası içeren DM dosyası. Yazılım kullanılarak gerçekleştirilen ek analiz örnekleri de burada sağlanmıştır.
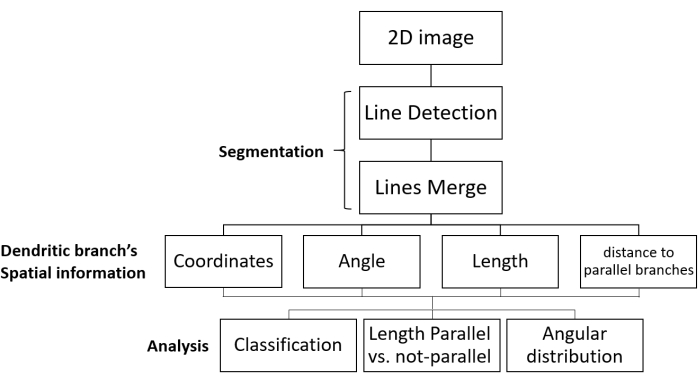
Şekil 1: Segmentasyon ve büyüme yönü analizi için SOA iş akışı. Dendritik ağların floresan görüntülerinin ve veri analizinin işleme adımları gösterilmiştir. 2B görüntü yüklenir, bölümlere ayırılır (iki adımda: dendritik dallar çizgi olarak algılanır ve daha sonra ilgili satırlar birleştirilir) ve her dendritik dalın uzamsal bilgileri elde edilir. Veriler görüntüdeki tüm dendritik dallar için toplanır. Son olarak, veriler istenen morfolojik parametreleri vermek için analiz edilir. Kısaltma: SOA = segmentasyon ve oryantasyon analizi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
1. SOA uygulamasını açın.
- URL adresini açın: https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA, SOA.zip sıkıştırılmış klasörünü bulun ve zip dosyasını çift tıklatarak indirin.
- SOA'ya sağ tıklayarak klasörün ferş .zip ve Dosyaları Ayıkla'yı seçin. Açılan Ayıklama Yolu ve Seçenekleri penceresini ve ayıklanan dosyaların yolunu görüntüleyen Hedef Adres metin kutusunu gözlemleyin. Farklı bir konuma ayıklamak için, pencerenin sağ panelinde bulunan klasörlerden birini tıklatarak hedef klasör haline getirin. Dosyaları bu klasöre ayıklamak için Tamam'ı tıklatın.
- Ayıklanan SOA dosyasını açın ve SOA.exe'e çift tıklayın. Siyah bir pencerenin açılmasını bekleyin, ardından uygulama görünecektir.

Şekil 2: SOA'nın GUI'sini kullanan iş akışı örneği. Sol sütun: İş akışının GUI bölümleri. Orta sütun: iş akışı sırasında işlenen bir dendritik ağın görüntüsü (Ölçek çubuğu: 20 μm). Sağ sütun: orta sütunun görüntülerinde kırmızı bir dikdörtgenle işaretlenmiş alanın büyütülmesi (Ölçek çubuğu: 4 μm). Adım 1: Bir görüntünün seçimi ve yüklenmesi. Adım 2: Segmentasyonun ilk aşaması, tanımlanan dendritik dalları temsil eden çizgilerin algılanmasıdır. Adım 3: Segmentasyonun ikinci aşaması, segment astarının bireysel dendritik dallarda yakınlık tabanlı birleşmesidir. Tüm adımların ayarları değiştirilebilir. Kısaltmalar: SOA = segmentasyon ve oryantasyon analizi; GUI = grafik kullanıcı arayüzü. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
2. Analiz etmek için bir resim açın.
- SOA Görüntüleyici Karşıya Yükleme menü çubuğunda | Dosya Seç |'nı seçin bilgisayar dosyalarından bir görüntü seçin| üzerine tıklayın (yalnızca .png .jpg .tif .bmp dosyalar) | | Aç dosyanın yolunu gözlemleyin | Önümüzdeki.
3. Segmentasyon optimizasyonu
NOT: SOA Görüntüleyici Özellikleri menü çubuğunda, segmentasyon işlemi ayarlarını ayarlamak için seçili parametrelerin değerlerini değiştirin. Ek Malzeme'de Eşik gibi parametrelerin ayrıntılı bir açıklaması verilmiştir.
- Kenarlar'da, Eşiği seçip bir sayı girerek ekranın eşiğini ayarlayın.
NOT: Eşik için sayı ne kadar düşük olursa, o kadar çok satır algılanır. Eşik, 0 ile 255 arasında değişen bir sayıdır. Varsayılan değer 0 olarak ayarlandı. - Satırları Birleştir'de:
- Birleştirilecek Min mesafesini seçip bir sayı girerek ekran için birleştirilecek minimum mesafeyi ayarlayın.
NOT: Birleştirme için Min mesafesi 0 ile 30 piksel arasında değişir. Varsayılan değer 20 olarak ayarlanır. - Birleştirilecek Min açısını seçip bir sayı girerek ekran için birleştirilecek minimum açıyı ayarlayın.
NOT: Birleştirilecek Min açısı 0 ile 30° arasında değişir. Varsayılan değer 10 olarak ayarlanır.
- Birleştirilecek Min mesafesini seçip bir sayı girerek ekran için birleştirilecek minimum mesafeyi ayarlayın.
- Önizleme Segmentasyon Görüntüsü Oluştur'a tıklayın.
NOT: Segmentasyon sonuçlarının önizleme görüntüsü güncelleştirilmiş değerlere göre görüntülenir. Ayrıca, birleştirmeden önceki satır sayısı ve birleştirmeden sonraki satır sayısı görüntülenir. - Segmentlerin maksimum tanımlamasını elde etmek için parametreleri değiştirin. Özellikleri değiştirme ihtiyacı varsa, Pencereyi kapat düğmesine tıklayın ve 3.1-3.4 adımlarını izleyin.
4. Çıktı dosyalarını oluşturun.
- Segmentasyon görüntülerini ve çözümleme grafiklerini görselleştirmek için Tamam'a basın. .xlsx dosyasının kaydedileceği konumu seçmek için görüntülenen pencereyi gözlemleyin.
- dosya adı ekleme | Kaydet |' i seçin verilerin oluşturulup kaydedilmesini içeren .xlsx dosyasını bekleyin.
NOT: .xlsx dosyasına ek olarak, aşağıdaki dosyalar otomatik olarak görüntülenir: orijinal görüntüyü, çizgi tanıma görüntüsünü, segmentasyonun son görüntüsünü ve üç analiz grafiğini sunan bir dosya.
5. Gezinti araç çubuğu
NOT: Gezinti araç çubuğu tüm şekil pencerelerinde bulunur ve veri kümesinde gezinmek için kullanılabilir. Araç çubuğunun altındaki düğmelerin her biri aşağıda açıklanmıştır.
- Önceden tanımlanmış görünümler arasında ileri geri gezinmek için İleri ve Geri düğmelerini kullanın.
NOT: Giriş, İleri ve Geri düğmeleri, web tarayıcısındaki Giriş, İleri ve Geri denetimlerine benzer. Giriş varsayılan ekrana, orijinal görüntüye döner. - Kaydırmak ve yakınlaştırmak için Yakınlaştır düğmesini kullanın. Kaydırma ve yakınlaştırmayı etkinleştirmek için Yakınlaştır düğmesine basın, ardından fareyi görüntüde istediğiniz konuma getirin.
- Şekli kaydırmak için, sol fare düğmesini yeni bir konuma sürüklerken basılı tutun. Fare düğmesini bırakın ve görüntüdeki seçili nokta yeni konumda görünecektir. Kaydırma sırasında, hareketi sırasıyla x veya y eksenleriyle sınırlamak için x veya y tuşlarını basılı tutun.
- Yakınlaştırmak için sağ fare düğmesini basılı tutun ve yeni bir konuma sürükleyin. X eksenini yakınlaştırmak için sağa, x ekseninde uzaklaştırmak için sola hareket edin. Aynı şeyi y ekseni ve yukarı/aşağı hareketler için de yapın. Yakınlaştırırken, farenin altındaki noktanın sabit kaldığını ve bu noktanın yakınlaştırmasına veya uzaklaştırmasına izin verdiğini unutmayın. Yakınlaştırmayı sırasıyla x, y veya en boy oranı korumasıyla sınırlamak için x, y veya CONTROL değiştirici tuşlarını kullanın.
- Dikdörtgene yakınlaştırma modunu etkinleştirmek için Dikdörtgene yakınlaştır düğmesini tıklatın. İmleci görüntünün üzerine yerleştirin ve sol fare düğmesine basın. Dikdörtgen bir bölge tanımlamak için düğmeyi basılı tutarken fareyi yeni bir konuma sürükleyin.
NOT: Sol fare düğmesine basıldığında eksen görünümü sınırları tanımlanan bölgeye yakınlaştırılacaktır. Sağ fare düğmesine basıldığında eksen görünümü sınırları uzaklaştırılacak ve orijinal eksenler tanımlanan bölgeye yerleştirilecektir. - Alt pilotun görünümünü yapılandırmak için Alt Pilot yapılandırma aracını kullanın.
NOT: Alt pilotun sol, sağ, üst ve alt kenarlarının yanı sıra satırlar ve sütunlar arasındaki boşluk uzatılabilir veya sıkıştırılabilir. - Dosya kaydetme iletişim kutusunu açmak için Kaydet düğmesini tıklatın ve dosyayı aşağıdaki biçimlerde kaydedin: .png, .ps, .eps, .svg veya .pdf.
Sonuçlar
Kültürdeki dendritik ağların görüntüleri üzerinde temsili bir analiz yapıldı. Hücreler Baranes ve ark. 16,17. Kısaca, hipokampal hücreler doğum sonrası sıçanların beyinlerinden çıkarıldı ve 1-2 hafta boyunca 2D cam kapaklar üzerinde yetiştirildi. Kültürler daha sonra dendritik protein belirteci, mikrotübül ilişkili protein 2'ye (MAP2) karşı bir antikor kullanılarak dolaylı immünofluoresans yoluyla sabitlendi ve lekelendi...
Tartışmalar
Biyolojik görüntüleme verilerine ayak uydurmak için 2D görüntülerden morfolojik bilgi çıkarmak için etkili stratejiler gereklidir. Görüntüleme verileri saatler içinde oluşturulabilse de, görüntülerin derinlemesine analizi uzun zaman alır. Sonuç olarak, görüntü işleme açıkça birçok alanda büyük bir engel haline gelmiştir. Bu kısmen, özellikle biyolojik örneklerle uğraşırken verilerin yüksek karmaşıklığından kaynaklanmaktadır. Ayrıca, birçok kullanıcı özel programlama ve gö...
Açıklamalar
Yazarlar rakip finansal çıkarları olmadığını beyan ederler.
Teşekkürler
Yazarlar, kültür görüntülerinin hazırlanması için Dr. Orly Weiss'a teşekkür eder.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
Referanslar
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083 (2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782 (2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206 (2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ Available from: https://imagej.nih.gov/ij (2021)
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005 (2019).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır