Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Identification automatique des branches dendritiques et de leur orientation
Dans cet article
Résumé
Présenté est un outil de calcul qui permet une mesure automatique simple et directe des orientations des branches dendritiques neuronales à partir d’images de fluorescence 2D.
Résumé
La structure des arbres dendritiques neuronaux joue un rôle clé dans l’intégration des entrées synaptiques dans les neurones. Par conséquent, la caractérisation de la morphologie des dendrites est essentielle pour une meilleure compréhension de la fonction neuronale. Cependant, la complexité des arbres dendritiques, à la fois lorsqu’ils sont isolés et surtout lorsqu’ils sont situés dans des réseaux neuronaux, n’a pas été complètement comprise. Nous avons développé un nouvel outil de calcul, SOA (Segmentation and Orientation Analysis), qui permet de mesurer automatiquement l’orientation des branches dendritiques à partir d’images de fluorescence de cultures neuronales 2D. SOA, écrit en Python, utilise la segmentation pour distinguer les branches dendritiques de l’arrière-plan de l’image et accumule une base de données sur la direction spatiale de chaque branche. La base de données est ensuite utilisée pour calculer des paramètres morphologiques tels que la distribution directionnelle des branches dendritiques dans un réseau et la prévalence de la croissance des branches dendritiques parallèles. Les données obtenues peuvent être utilisées pour détecter des changements structurels dans les dendrites en réponse à l’activité neuronale et aux stimuli biologiques et pharmacologiques.
Introduction
La morphogenèse dendritique est un sujet central en neurosciences, car la structure de l’arbre dendritique affecte les propriétés computationnelles de l’intégration synaptique dans les neurones1,2,3. De plus, des anomalies morphologiques et des modifications dans les branches dendritiques sont impliquées dans les troubles dégénératifs et neuro-développementaux4,5,6. Dans les cultures neuronales où la ramification dendritique peut être plus facilement visualisée, les interactions entre les branches dendritiques non sœurs régulent les sites et l’étendue du regroupement synaptique le long des branches, un comportement qui peut affecter la coactivité synaptique et la plasticité7,8,9. Par conséquent, la caractérisation des paramètres morphologiques de l’arbre dendritique à l’aide de cultures neuronales bidimensionnelles (2D) est avantageuse pour comprendre la morphogenèse dendritique et la fonctionnalité des neurones uniques et des réseaux de neurones. Pourtant, c’est une tâche difficile car les branches dendritiques forment un maillage complexe, même dans les cultures neuronales 2D « simplifiées ».
Plusieurs outils ont été développés pour tracer et analyser automatiquement les structures dendritiques10,11,12,13. Cependant, la plupart de ces outils sont conçus pour les réseaux neuronaux 3D et sont naturellement trop complexes pour être utilisés avec des réseaux 2D. En revanche, les outils d’analyse morphologique moins avancés impliquent généralement une composante importante du travail manuel assisté par ordinateur, qui prend beaucoup de temps et est sensible au biais de l’opérateur14. Les outils semi-automatiques existants, tels que « ImageJ»15 (un package de traitement d’image open source des NIH avec une vaste collection d’outils d’analyse d’images biologiques développés par la communauté), réduisent considérablement le travail manuel de l’utilisateur. Cependant, certaines interventions manuelles sont encore nécessaires pendant le traitement de l’image, et la qualité de la segmentation peut être moins que souhaitable.
Cet article présente le SOA, un outil automatisé simple qui permet la segmentation directe et l’analyse d’orientation des branches dendritiques au sein des réseaux neuronaux 2D. La SOA peut détecter divers objets ressemblant à des lignes dans des images 2D et caractériser leurs propriétés morphologiques. Ici, nous avons utilisé la SOA pour segmenter les branches dendritiques dans des images de fluorescence 2D de réseaux dendritiques en culture. Le logiciel identifie les branches dendritiques et effectue avec succès des mesures de paramètres morphologiques tels que le parallélisme et la distribution spatiale. La SOA peut être facilement adaptée pour l’analyse des processus cellulaires d’autres types de cellules et pour l’étude des réseaux non biologiques.
Access restricted. Please log in or start a trial to view this content.
Protocole
NOTE: Le ministère israélien de la Santé a approuvé l’utilisation de souris en vertu du protocole IL-218-01-21 pour l’utilisation éthique des animaux de laboratoire. SOA n’est compatible qu’avec Windows 10 et Python 3.9. Il est disponible en tant que code open-source : https://github.com/inbar2748/DendriteProject. Sur ce lien, il y a aussi un fichier README. DM contient des instructions pour télécharger le logiciel, un lien vers le site Web du logiciel et un fichier de configuration requise contenant des informations sur les versions requises de tous les packages. D’autres exemples d’analyses effectuées à l’aide du logiciel y ont également été fournis.
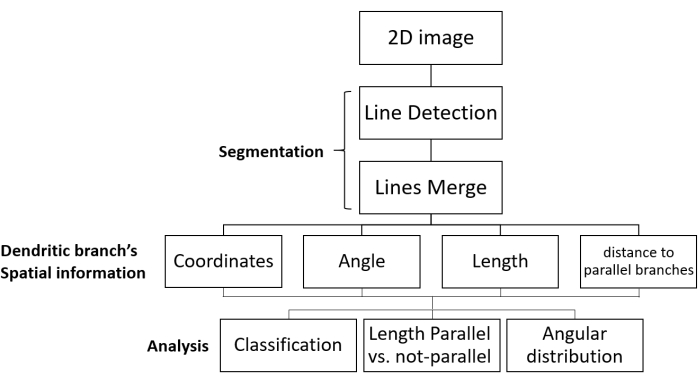
Figure 1 : Flux de travail SOA pour l’analyse de la segmentation et de la direction de croissance. Les étapes de traitement des images fluorescentes des réseaux dendritiques et l’analyse des données sont présentées. L’image 2D est téléchargée, segmentée (en deux étapes: les branches dendritiques sont détectées sous forme de lignes, puis les lignes pertinentes sont fusionnées), et les informations spatiales de chaque branche dendritique sont obtenues. Les données sont collectées pour toutes les branches dendritiques de l’image. Enfin, les données sont analysées pour donner les paramètres morphologiques souhaités. Abréviation : SOA = analyse de segmentation et d’orientation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Ouvrez l’application SOA.
- Ouvrez l’adresse URL : https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA, recherchez le dossier compressé SOA.zip et téléchargez le fichier ZIP en double-cliquant dessus.
- Décompressez le dossier en cliquant avec le bouton droit sur SOA.zip et choisissez Extraire les fichiers. Observez la fenêtre Chemin d’extraction et options qui s’ouvre et la zone de texte Adresse de destination qui affiche le chemin d’accès des fichiers extraits. Pour extraire vers un autre emplacement, cliquez sur l’un des dossiers dans le panneau de droite de la fenêtre pour en faire le dossier de destination. Cliquez sur OK pour extraire les fichiers dans ce dossier.
- Ouvrez le fichier SOA extrait et double-cliquez sur SOA.exe. Attendez qu’une fenêtre noire s’ouvre, après quoi l’application apparaîtra.

Figure 2 : Exemple de workflow utilisant l’interface graphique de la SOA. Colonne de gauche : sections GUI du workflow. Colonne du milieu : image d’un réseau dendritique, traitée pendant le workflow (Barre d’échelle : 20 μm). Colonne de droite : grossissement de la zone marquée par un rectangle rouge dans les images de la colonne du milieu (Barre d’échelle : 4 μm). Étape 1 : Sélection et téléchargement d’une image. Étape 2 : La première étape de la segmentation est la détection des lignes qui représentent les branches dendritiques identifiées. Étape 3: La deuxième étape de la segmentation est la fusion basée sur la proximité du revêtement de segment dans les branches dendritiques individuelles. Les paramètres de toutes les étapes peuvent être modifiés. Abréviations : SOA = analyse de segmentation et d’orientation; GUI = interface utilisateur graphique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Ouvrez une image à analyser.
- Dans la barre de menus Téléchargement de LA VISIONNEUSE SOA , | sélectionnez Choisir un fichier | choisissez une image dans les fichiers de l’ordinateur | cliquez dessus (fichiers .png .jpg .tif .bmp uniquement) | Ouvrir | observer le chemin d’accès du fichier | Prochain.
3. Optimisation de la segmentation
Remarque : Dans la barre de menus Propriétés de la visionneuse SOA , modifiez les valeurs des paramètres sélectionnés pour ajuster les paramètres du processus de segmentation. Une description détaillée des paramètres, tels que le seuil, est donnée dans le document supplémentaire.
- Dans Arêtes, ajustez le seuil d’affichage en sélectionnant le Seuil et en entrant un nombre.
REMARQUE: Plus le nombre de seuils est bas, plus le nombre de lignes détectées est élevé. Le seuil est un nombre compris entre 0 et 255. La valeur par défaut a été définie sur 0. - Dans Fusionner les lignes :
- Ajustez la distance minimale à fusionner pour l’affichage en sélectionnant la distance minimale à fusionner et en entrant un nombre.
REMARQUE: La distance minimale pour fusionner varie de 0 à 30 pixels. La valeur par défaut est définie sur 20. - Ajustez l’angle minimal à fusionner pour l’affichage en sélectionnant l’angle min à fusionner et en entrant un nombre.
REMARQUE: L’angle min pour fusionner varie de 0 à 30 °. La valeur par défaut est définie sur 10.
- Ajustez la distance minimale à fusionner pour l’affichage en sélectionnant la distance minimale à fusionner et en entrant un nombre.
- Cliquez sur Créer une image de segmentation d’aperçu.
REMARQUE: Une image d’aperçu des résultats de la segmentation sera affichée en fonction des valeurs mises à jour. De plus, le nombre de lignes avant la fusion et le nombre de lignes après la fusion seront affichés. - Modifiez les paramètres pour obtenir une identification maximale des segments. S’il est nécessaire de modifier les propriétés, cliquez sur le bouton Fermer la fenêtre et suivez les étapes 3.1 à 3.4.
4. Créez les fichiers de sortie.
- Appuyez sur OK pour visualiser les images de segmentation et les graphiques d’analyse. Observez la fenêtre qui s’affiche pour sélectionner un emplacement où le fichier .xlsx sera enregistré.
- insérer un nom de fichier | Choisissez Enregistrer | attendez que le fichier .xlsx contenant des données soit créé et enregistré.
REMARQUE: En plus du fichier .xlsx, les fichiers suivants seront automatiquement affichés: un fichier qui présente l’image d’origine, l’image de reconnaissance de ligne, l’image finale de la segmentation et trois graphiques d’analyse.
5. Barre d’outils de navigation
REMARQUE : Une barre d’outils de navigation est incluse dans toutes les fenêtres de figure et peut être utilisée pour naviguer dans l’ensemble de données. Chacun des boutons en bas de la barre d’outils est décrit ci-dessous.
- Pour naviguer entre les vues précédemment définies, utilisez les boutons Suivant et Précédent .
REMARQUE : Les boutons Accueil, Suivant et Précédent sont similaires aux contrôles Accueil, Suivant et Précédent d’un navigateur Web. Accueil revient à l’écran par défaut, l’image d’origine. - Utilisez le bouton Zoom pour effectuer un panoramique et un zoom. Pour activer le panoramique et le zoom, appuyez sur le bouton Zoom , puis déplacez la souris à l’emplacement souhaité dans l’image.
- Pour effectuer un panoramique de la figure, maintenez enfoncé le bouton gauche de la souris tout en le faisant glisser vers une nouvelle position. Relâchez le bouton de la souris et le point sélectionné dans l’image apparaîtra dans la nouvelle position. Pendant le panoramique, maintenez les touches x ou y enfoncées pour limiter le mouvement aux axes x ou y , respectivement.
- Pour zoomer, maintenez le bouton droit de la souris enfoncé et faites-le glisser vers un nouvel emplacement. Déplacez-vous vers la droite pour effectuer un zoom avant sur l’axe des x et vers la gauche pour effectuer un zoom arrière sur l’axe des x. Faites de même pour l’axe des y et les mouvements haut/bas. Lors du zoom, notez que le point sous la souris reste immobile, ce qui permet d’effectuer un zoom avant ou arrière autour de ce point. Utilisez les touches de modification x, y ou CTRL pour limiter le zoom à la préservation des rapports x, y ou des rapports L/H, respectivement.
- Pour activer le mode Zoom sur rectangle, cliquez sur le bouton Zoom sur rectangle . Placez le curseur sur l’image et appuyez sur le bouton gauche de la souris. Faites glisser la souris vers un nouvel emplacement tout en maintenant le bouton enfoncé pour définir une région rectangulaire.
REMARQUE: Les limites d’affichage des axes seront zoomées sur la région définie lorsque vous appuyez sur le bouton gauche de la souris. Les limites d’affichage des axes sont dézoomées lorsque vous appuyez sur le bouton droit de la souris, plaçant les axes d’origine dans la région définie. - Utilisez l’outil de configuration de sous-tracé pour configurer l’apparence du sous-tracé.
REMARQUE : Les côtés gauche, droit, supérieur et inférieur du sous-tracé, ainsi que l’espace entre les lignes et les colonnes, peuvent être étirés ou compressés. - Pour ouvrir une boîte de dialogue d’enregistrement de fichier, cliquez sur le bouton Enregistrer et enregistrez le fichier aux formats suivants : .png, .ps, .eps, .svg ou .pdf.
Access restricted. Please log in or start a trial to view this content.
Résultats
Une analyse représentative a été réalisée sur des images de réseaux dendritiques en culture. Les cellules ont été extraites comme décrit par Baranes et al. 16,17. En bref, les cellules de l’hippocampe ont été extraites du cerveau de rats postnatals et cultivées sur des couvertures en verre 2D pendant 1 à 2 semaines. Les cultures ont ensuite été fixées et colorées par immunofluorescence indirecte à l’aide d’un anticorps contre le m...
Access restricted. Please log in or start a trial to view this content.
Discussion
Des stratégies efficaces pour extraire des informations morphologiques à partir d’images 2D sont nécessaires de toute urgence pour suivre le rythme des données d’imagerie biologique. Bien que les données d’imagerie puissent être générées en quelques heures, l’analyse approfondie des images prend beaucoup de temps. En conséquence, le traitement de l’image est clairement devenu un obstacle majeur dans de nombreux domaines. Cela est dû en partie à la grande complexité des données, en particulier lors...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents.
Remerciements
Les auteurs tiennent à remercier le Dr Orly Weiss pour la préparation des images de culture.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
Références
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083(2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782(2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206(2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ. , Available from: https://imagej.nih.gov/ij (2021).
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005(2019).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon