Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Автоматическая идентификация дендритных ветвей и их ориентация
В этой статье
Резюме
Представлен вычислительный инструмент, позволяющий просто и напрямую автоматически измерять ориентации нейронных дендритных ветвей по 2D флуоресцентным изображениям.
Аннотация
Структура нейронных дендритных деревьев играет ключевую роль в интеграции синаптических входов в нейроны. Поэтому характеристика морфологии дендритов необходима для лучшего понимания функции нейронов. Однако сложность дендритных деревьев, как при изоляции, так и особенно при расположении в нейронных сетях, не была полностью понята. Мы разработали новый вычислительный инструмент SOA (Segmentation and Orientation Analysis), который позволяет автоматически измерять ориентацию дендритных ветвей по флуоресцентным изображениям 2D нейронных культур. SOA, написанная на Python, использует сегментацию для отличия дендритных ветвей от фона изображения и аккумулирует базу данных о пространственном направлении каждой ветви. Затем база данных используется для расчета морфологических параметров, таких как направленное распределение дендритных ветвей в сети и распространенность параллельного роста дендритных ветвей. Полученные данные могут быть использованы для выявления структурных изменений дендритов в ответ на активность нейронов и на биологические и фармакологические раздражители.
Введение
Дендритный морфогенез является центральным предметом в нейробиологии, так как структура дендритного дерева влияет на вычислительные свойства синаптической интеграции в нейронах1,2,3. Кроме того, морфологические аномалии и модификации в дендритных ветвях связаны с дегенеративными и нервно-психическими расстройствами развития4,5,6. В нейронных культурах, где дендритное разветвление может быть более легко визуализировано, взаимодействия между неродственными дендритными ветвями регулируют участки и степень синаптической кластеризации вдоль ветвей, поведение, которое может влиять на синаптическую коактивность и пластичность7,8,9. Поэтому характеристика морфологических параметров дендритного дерева с использованием двумерных (2D) нейрональных культур выгодна для понимания дендритного морфогенеза и функциональности одиночных и сетей нейронов. Тем не менее, это сложная задача, потому что дендритные ветви образуют сложную сетку даже в «упрощенных» 2D-нейронных культурах.
Было разработано несколько инструментов для автоматического отслеживания и анализа дендритных структур10,11,12,13. Однако большинство из этих инструментов предназначены для 3D-нейронных сетей и, естественно, слишком сложны для использования с 2D-сетями. Напротив, менее продвинутые инструменты морфологического анализа обычно включают значительный компонент ручного труда с помощью компьютера, который очень трудоемкий и восприимчивый к смещению оператора14. Существующие полуавтоматические инструменты, такие как ImageJ'15 (пакет обработки изображений с открытым исходным кодом NIH с обширной коллекцией разработанных сообществом инструментов анализа биологических изображений), в значительной степени сокращают ручной труд пользователя. Тем не менее, некоторые ручные вмешательства все еще необходимы во время обработки изображения, и качество сегментации может быть менее желательным.
В этой статье представлен SOA, простой автоматизированный инструмент, который позволяет напрямую сегментировать и анализировать ориентацию дендритных ветвей в 2D-нейронных сетях. SOA может обнаруживать различные линейно-подобные объекты на 2D-изображениях и характеризовать их морфологические свойства. Здесь мы использовали SOA для сегментации дендритных ветвей в 2D-флуоресцентных изображениях дендритных сетей в культуре. Программное обеспечение идентифицирует дендритные ветви и успешно выполняет измерения морфологических параметров, таких как параллелизм и пространственное распределение. SOA может быть легко адаптирована для анализа клеточных процессов других типов клеток и для изучения небиологических сетей.
протокол
ПРИМЕЧАНИЕ: Министерство здравоохранения Израиля одобрило использование мышей по протоколу IL-218-01-21 для этического использования подопытными животными. SOA совместима только с Windows 10 и Python 3.9. Он доступен в виде открытого исходного кода: https://github.com/inbar2748/DendriteProject. По этой ссылке также есть файл README. DM-файл, содержащий инструкции по загрузке программного обеспечения, ссылку на веб-сайт программного обеспечения и файл требований, содержащий информацию о требуемых версиях всех пакетов. Там же были приведены дополнительные примеры анализа, выполненного с использованием программного обеспечения.
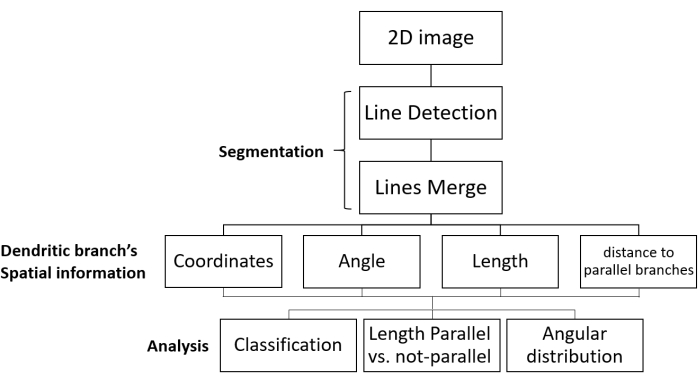
Рисунок 1: Рабочий процесс SOA для сегментации и анализа направлений роста. Показаны этапы обработки флуоресцентных изображений дендритных сетей и анализа данных. 2D-изображение загружается, сегментируется (в два этапа: дендритные ветви обнаруживаются как линии, а затем соответствующие линии объединяются), и получается пространственная информация каждой дендритной ветви. Данные собираются для всех дендритных ветвей на изображении. Наконец, данные анализируются для получения желаемых морфологических параметров. Аббревиатура: SOA = сегментация и ориентационный анализ. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
1. Откройте SOA-приложение.
- Откройте URL-адрес: https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA, найдите сжатую папку SOA.zip и загрузите ZIP-файл двойным щелчком мыши.
- Распакуйте папку, щелкнув правой кнопкой мыши по SOA.zip и выберите «Извлечь файлы». Обратите внимание на открывающееся окно «Путь и параметры извлечения» и текстовое поле «Адрес назначения », в котором отображается путь к извлеченным файлам. Чтобы выполнить извлечение в другое место, нажмите на одну из папок на правой панели окна, чтобы сделать ее папкой назначения. Нажмите кнопку ОК, чтобы извлечь файлы в эту папку.
- Откройте извлеченный SOA-файл и дважды щелкните на SOA.exe. Дождитесь открытия черного окна, после чего появится приложение.

Рисунок 2: Пример рабочего процесса, использующего графический интерфейс SOA. Левый столбец: разделы рабочего процесса с графическим интерфейсом. Средняя колонка: изображение дендритной сети, обработанной во время рабочего процесса (шкала: 20 мкм). Правый столбец: увеличение области, отмеченной красным прямоугольником на изображениях среднего столбца (шкала: 4 мкм). Шаг 1: Выбор и загрузка изображения. Шаг 2: Первым этапом сегментации является обнаружение линий, которые представляют идентифицированные дендритные ветви. Шаг 3: Вторым этапом сегментации является слияние сегментной футеровки в отдельных дендритных ветвях на основе близости. Настройки всех шагов могут быть изменены. Сокращения: SOA = сегментация и ориентационный анализ; GUI = графический интерфейс пользователя. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
2. Откройте изображение для анализа.
- В строке меню ЗАГРУЗКА SOA Viewer | выберите Выбрать файл | Выберите изображение из файлов компьютера, | нажмите на него (только .png .jpg .tif .bmp файлы) | Открыть | обратите внимание на путь к файлу | Следующий.
3. Оптимизация сегментации
ПРИМЕЧАНИЕ: В строке меню Свойства SOA Viewer измените значения выбранных параметров, чтобы настроить параметры процесса сегментации. Подробное описание параметров, таких как Пороговое значение, приведено в Дополнительном материале.
- В окне Edges настройте пороговое значение для дисплея, выбрав Пороговое значение и введя число.
ПРИМЕЧАНИЕ: Чем меньше число порогового значения, тем больше строк обнаруживается. Пороговое значение — это число, которое находится в диапазоне от 0 до 255. Значение по умолчанию установлено равным 0. - В разделе Слияние строк:
- Отрегулируйте минимальное расстояние для слияния для отображения, выбрав Минимальное расстояние для слияния и введя число.
ПРИМЕЧАНИЕ: Минимальное расстояние для слияния колеблется от 0 до 30 пикселей. Значение по умолчанию равно 20. - Отрегулируйте минимальный угол слияния для отображения, выбрав Минимальный угол для слияния и введя число.
ПРИМЕЧАНИЕ: Минимальный угол слияния колеблется от 0 до 30°. Значение по умолчанию равно 10.
- Отрегулируйте минимальное расстояние для слияния для отображения, выбрав Минимальное расстояние для слияния и введя число.
- Нажмите «Создать изображение сегментации предварительного просмотра».
ПРИМЕЧАНИЕ: Предварительное изображение результатов сегментации будет отображаться в соответствии с обновленными значениями. Кроме того, будет отображаться количество строк перед слиянием и количество строк после слияния. - Измените параметры для достижения максимальной идентификации сегментов. Если есть необходимость изменить Свойства, нажмите на кнопку Закрыть окно и выполните шаги 3.1-3.4.
4. Создайте выходные файлы.
- Нажмите OK , чтобы визуализировать изображения сегментации и анализирующие графики. Обратите внимание на появившееся окно для выбора места, в котором будет сохранен .xlsx файл.
- Вставьте имя файла | Выберите Сохранить | дождитесь создания и сохранения файла .xlsx с данными.
ПРИМЕЧАНИЕ: В дополнение к файлу .xlsx автоматически будут отображаться следующие файлы: файл, в котором представлено исходное изображение, изображение распознавания строк, окончательное изображение сегментации и три графика анализа.
5. Навигационная панель инструментов
ПРИМЕЧАНИЕ: Навигационная панель инструментов включена во все окна рисунков и может использоваться для навигации по набору данных. Каждая из кнопок в нижней части панели инструментов описана ниже.
- Для перемещения вперед и назад между ранее определенными представлениями используйте кнопки Вперед и Назад .
ПРИМЕЧАНИЕ: Кнопки «Домой», «Вперед» и «Назад» аналогичны элементам управления «Домой», «Вперед» и «Назад» в веб-браузере. Главная возвращается к экрану по умолчанию, исходному изображению. - Используйте кнопку «Масштаб» для панорамирования и масштабирования. Чтобы активировать сдвиг и масштабирование, нажмите кнопку «Масштаб», затем переместите мышь в нужное место на изображении.
- Чтобы сдвинуть фигуру, нажмите и удерживайте левую кнопку мыши, перетаскивая ее в новое положение. Отпустите кнопку мыши, и выбранная точка на изображении появится в новом положении. Во время сдвига удерживайте клавиши x или y , чтобы ограничить движение осями x или y соответственно.
- Чтобы увеличить масштаб, удерживайте правую кнопку мыши и перетащите ее в новое место. Переместите вправо, чтобы увеличить масштаб по оси X, и влево, чтобы уменьшить масштаб по оси X. Проделайте то же самое для движений по оси Y и вверх/вниз. При масштабировании обратите внимание, что точка под мышью остается неподвижной, что позволяет увеличивать или уменьшать масштаб вокруг этой точки. Используйте клавиши-модификаторы x, y или CONTROL , чтобы ограничить масштаб сохранением x, y или пропорций соответственно.
- Чтобы активировать режим масштабирования в прямоугольник, нажмите кнопку Масштабировать в прямоугольник . Наведите курсор на изображение и нажмите левую кнопку мыши. Перетащите мышь в новое место, удерживая кнопку, чтобы определить прямоугольную область.
ПРИМЕЧАНИЕ: Ограничения представления осей будут увеличены до определенной области при нажатии левой кнопки мыши. Ограничения представления осей будут уменьшены при нажатии правой кнопки мыши, помещая исходные оси в определенную область. - Используйте средство настройки подсюжета для настройки внешнего вида подсюжета.
ПРИМЕЧАНИЕ: Левая, правая, верхняя и нижняя стороны подсюжета, а также пространство между строками и столбцами могут быть растянуты или сжаты. - Чтобы открыть диалоговое окно сохранения файла, нажмите кнопку Сохранить и сохраните файл в следующих форматах: .png, PS, .eps, .svg или .pdf.
Результаты
Был проведен репрезентативный анализ изображений дендритных сетей в культуре. Клетки были извлечены, как описано Baranes et al. 16,17. Вкратце, клетки гиппокампа были извлечены из мозга постнатальных крыс и культивированы на 2D стеклянных крышках в течение 1-...
Обсуждение
Эффективные стратегии извлечения морфологической информации из 2D-изображений срочно необходимы, чтобы идти в ногу с данными биологической визуализации. Хотя данные визуализации могут быть сгенерированы за несколько часов, углубленный анализ изображений занимает много времени. В рез...
Раскрытие информации
Авторы заявляют, что у них нет конкурирующих финансовых интересов.
Благодарности
Авторы хотели бы поблагодарить доктора Орли Вайса за подготовку изображений культуры.
Материалы
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
Ссылки
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083 (2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782 (2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206 (2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ Available from: https://imagej.nih.gov/ij (2021)
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005 (2019).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены