É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Identificação Automática de Ramos Dendráticos e sua Orientação
Neste Artigo
Resumo
Apresentado é uma ferramenta computacional que permite a medição automática simples e direta das orientações de ramos dendríticas neuronais a partir de imagens de fluorescência 2D.
Resumo
A estrutura das árvores dendríticas neuronais desempenha um papel fundamental na integração de insumos sinápticos nos neurônios. Portanto, a caracterização da morfologia dos dendritos é essencial para uma melhor compreensão da função neuronal. No entanto, a complexidade das árvores dendríticas, tanto quando isoladas quanto especialmente quando localizadas dentro das redes neuronais, não foi completamente compreendida. Desenvolvemos uma nova ferramenta computacional, SOA (Segmentação e Análise de Orientação), que permite a medição automática da orientação de ramos dendríticos a partir de imagens de fluorescência de culturas neuronais 2D. SOA, escrito em Python, usa segmentação para distinguir ramos dendríticos do fundo da imagem e acumula um banco de dados na direção espacial de cada ramo. O banco de dados é então utilizado para calcular parâmetros morfológicos, como a distribuição direcional de ramos dendráticos em uma rede e a prevalência de crescimento dendrático paralelo do ramo. Os dados obtidos podem ser usados para detectar alterações estruturais nos dendritos em resposta à atividade neuronal e a estímulos biológicos e farmacológicos.
Introdução
A morfogênese dendrítica é um assunto central na neurociência, pois a estrutura da árvore dendrítica afeta as propriedades computacionais da integração sináptica nos neurônios1,2,3. Além disso, anormalidades morfológicas e modificações em ramos dendráticos estão implicadas em distúrbios degenerativos e neurodesenvolvimento4,5,6. Nas culturas neuronais onde a ramificação dendrítica pode ser mais facilmente visualizada, as interações entre ramos dendráticos não-irmãos regulam os locais e a extensão do agrupamento sináptico ao longo dos ramos, comportamento que pode afetar a coatividade sináptica e a plasticidade7,8,9. Portanto, a caracterização dos parâmetros morfológicos da árvore dendrítica utilizando culturas neuronais bidimensionais (2D) é vantajosa para a compreensão da morfogênese dendrítica e funcionalidade de redes únicas e de neurônios. No entanto, esta é uma tarefa desafiadora porque os ramos dendráticos formam uma malha complexa mesmo em culturas neuronais 2D "simplificadas".
Várias ferramentas foram desenvolvidas para rastrear e analisar automaticamente estruturas dendríticas10,11,12,13. No entanto, a maioria dessas ferramentas são projetadas para redes neuronais 3D e são naturalmente muito complexas para usar com redes 2D. Em contraste, ferramentas de análise morfológica menos avançadas normalmente envolvem um componente significativo do trabalho manual assistido por computador, que é muito demorado e suscetível ao viés do operador14. Ferramentas semiautomáticas existentes, como o 'ImageJ'15 (um pacote de processamento de imagem de código aberto do NIH com uma vasta coleção de ferramentas de análise biológica desenvolvidas pela comunidade), reduzem em grande parte o trabalho manual do usuário. No entanto, algumas intervenções manuais ainda são necessárias durante o processamento da imagem, e a qualidade da segmentação pode ser menor do que desejável.
Este artigo apresenta o SOA, uma ferramenta automatizada simples que permite a segmentação direta e a análise de orientação de ramos dendríticos dentro de redes neuronais 2D. O SOA pode detectar vários objetos semelhantes a linhas em imagens 2D e caracterizar suas propriedades morfológicas. Aqui, utilizamos o SOA para segmentar ramos dendríticos em imagens de fluorescência 2D de redes dendríticas na cultura. O software identifica os ramos dendráticos e realiza com sucesso medições de parâmetros morfológicos, como paralelismo e distribuição espacial. O SOA pode ser facilmente adaptado para a análise de processos celulares de outros tipos celulares e para o estudo de redes não biológicas.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: O Ministério da Saúde israelense aprovou o uso de camundongos sob o protocolo IL-218-01-21 para o uso ético de animais experimentais. O SOA só é compatível com o Windows 10 e o Python 3.9. Está disponível como um código de código aberto: https://github.com/inbar2748/DendriteProject. Neste link, há também uma README. Arquivo DM que tem instruções para baixar o software, um link para o site do software e um arquivo de requisitos contendo informações sobre as versões necessárias de todos os pacotes. Outros exemplos de análises realizadas utilizando o software também foram fornecidos lá.
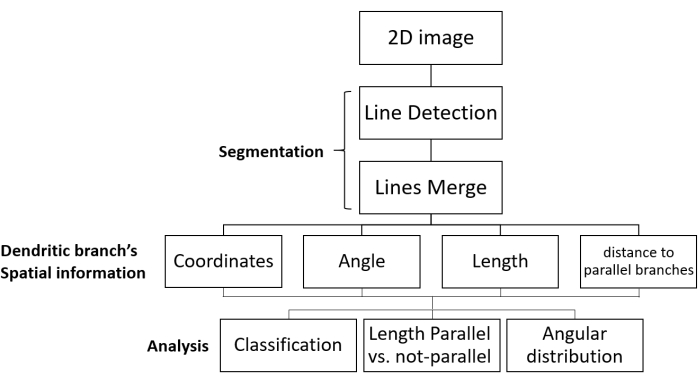
Figura 1: Fluxo de trabalho SOA para segmentação e análise de direção de crescimento. São mostradas as etapas de processamento de imagens fluorescentes de redes dendríticas e análise de dados. A imagem 2D é carregada, segmentada (em duas etapas: ramos dendráticos são detectados como linhas e, em seguida, as linhas relevantes são mescladas), e as informações espaciais de cada ramo dendrático são obtidas. Os dados são coletados para todos os ramos dendráticos da imagem. Por fim, os dados são analisados para dar os parâmetros morfológicos desejados. Abreviação: SOA = segmentação e análise de orientação. Clique aqui para ver uma versão maior desta figura.
1. Abra o aplicativo SOA.
- Abra o endereço de URL: https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA, encontre a pasta com zíper .ZIP SOA e baixe o arquivo ZIP clicando duas vezes.
- Descompacte a pasta clicando com o botão direito do mouse no SOA.zip e escolha Extrair arquivos. Observe a janela Caminho de Extração e Opções que abre e a caixa de texto Endereço de Destino que exibe o caminho para os arquivos extraídos. Para extrair para um local diferente, clique em uma das pastas no painel direito da janela para torná-la a pasta de destino. Clique em OK para extrair os arquivos para essa pasta.
- Abra o arquivo SOA extraído e clique duas vezes no SOA.exe. Aguarde a abertura de uma janela preta, após a qual o aplicativo será exibido.

Figura 2: Exemplo de fluxo de trabalho usando a GUI do SOA. Coluna esquerda: seções gui do fluxo de trabalho. Coluna média: imagem de uma rede dendrítica, processada durante o fluxo de trabalho (Barra de escala: 20 μm). Coluna direita: ampliação da área marcada por um retângulo vermelho nas imagens da coluna do meio (Barra de escala: 4 μm). Passo 1: Seleção e upload de uma imagem. Passo 2: A primeira etapa da segmentação é a detecção de linhas que representam os ramos dendráticos identificados. Passo 3: A segunda etapa da segmentação é a fusão baseada em proximidade de forro de segmento em filiais dendríticas individuais. As configurações de todas as etapas podem ser modificadas. Abreviaturas: SOA = segmentação e análise de orientação; GUI = interface gráfica do usuário. Clique aqui para ver uma versão maior desta figura.
2. Abra uma imagem para analisar.
- Na barra de menus SOA Viewer Upload | selecionar Escolher arquivo | escolha uma imagem dos arquivos do computador | clique nele (somente .png .jpg .tif .bmp arquivos) | | observar o caminho do arquivo | Próximo.
3. Otimização de segmentação
NOTA: Na barra de menuS SOA Viewer Properties , altere os valores dos parâmetros selecionados para ajustar as configurações do processo de segmentação. Uma descrição detalhada dos parâmetros, como o Limiar, é dada no Material Suplementar.
- Em Bordas, ajuste o limiar para o display selecionando o Limiar e digitando um número.
NOTA: Quanto menor o número do limiar, mais linhas são detectadas. Limiar é um número que varia de 0 a 255. O valor padrão foi definido para 0. - Em Linhas de Mesclagem:
- Ajuste a distância mínima para mesclar para o display selecionando a distância Min para mesclar e digitar um número.
NOTA: A distância min para mesclar varia de 0 a 30 pixels. O valor padrão está definido para 20. - Ajuste o ângulo mínimo para mesclar para o display selecionando o ângulo Min para mesclar e digitar um número.
NOTA: O ângulo Min para mesclar varia de 0 a 30°. O valor padrão está definido para 10.
- Ajuste a distância mínima para mesclar para o display selecionando a distância Min para mesclar e digitar um número.
- Clique em Criar imagem de segmentação de visualização.
NOTA: Uma imagem de visualização dos resultados de segmentação será exibida de acordo com os valores atualizados. Além disso, o número de linhas antes da fusão e o número de linhas após a fusão serão exibidos. - Alterar os parâmetros para alcançar a máxima identificação dos segmentos. Se houver necessidade de alterar as Propriedades, clique no botão Fechar a janela e siga os passos 3.1-3.4.
4. Crie os arquivos de saída.
- Pressione OK para visualizar as imagens de segmentação e os gráficos de análise. Observe a janela que aparece para selecionar um local onde o arquivo .xlsx será salvo.
- inserir um nome de arquivo | Escolha salvar | esperar que o arquivo .xlsx com dados sejam criados e salvos.
NOTA: Além do arquivo .xlsx, os seguintes arquivos serão exibidos automaticamente: um arquivo que apresenta a imagem original, a imagem de reconhecimento de linha, a imagem final da segmentação e três gráficos de análise.
5. Barra de ferramentas de navegação
NOTA: Uma barra de ferramentas de navegação está incluída em todas as janelas de figuras e pode ser usada para navegar através do conjunto de dados. Cada um dos botões na parte inferior da barra de ferramentas está descrito abaixo.
- Para navegar para frente e para trás entre as visualizações previamente definidas, use os botões Para frente e para trás .
NOTA: Os botões Home, Forward e Back são semelhantes aos controles Home, Forward e Back em um navegador da Web. Home retorna à tela padrão, à imagem original. - Use o botão Zoom para panela e zoom. Para ativar o garimpo e o zoom, pressione o botão Zoom e mova o mouse para um local desejado na imagem.
- Para a panela da figura, pressione e segure o botão esquerdo do mouse enquanto arrasta-o para uma nova posição. Solte o botão do mouse e o ponto selecionado na imagem aparecerá na nova posição. Enquanto garimpo, segure as teclas x ou y para restringir o movimento aos eixos x ou y , respectivamente.
- Para ampliar, segure o botão direito do mouse e arraste-o para um novo local. Mova-se para a direita para ampliar o eixo x e mova-se para a esquerda para diminuir o zoom no eixo x. Faça o mesmo para os movimentos y-axis e para cima/para baixo. Ao dar zoom, observe que o ponto sob o mouse permanece parado, permitindo ampliar ou sair em torno desse ponto. Use as teclas modificador x, y ou CONTROL para limitar o zoom à reserva de x, y ou proporção, respectivamente.
- Para ativar o modo Zoom-to-retângulo, clique no botão Zoom-to-retângulo . Coloque o cursor sobre a imagem e pressione o botão esquerdo do mouse. Arraste o mouse para um novo local enquanto segura o botão para definir uma região retangular.
NOTA: Os limites de visualização dos eixos serão ampliados para a região definida quando o botão esquerdo do mouse for pressionado. Os limites de visualização dos eixos serão ampliados quando o botão direito do mouse for pressionado, colocando os eixos originais na região definida. - Use a ferramenta de configuração de subtrama para configurar a aparência da subtrama.
NOTA: Os lados esquerdo, direito, superior e inferior da subtrama, bem como o espaço entre linhas e colunas, podem ser esticados ou comprimidos. - Para abrir uma caixa de diálogo de salvamento de arquivos, clique no botão Salvar e salve o arquivo nos seguintes formatos: .png, .ps, .eps, .svg ou .pdf.
Access restricted. Please log in or start a trial to view this content.
Resultados
Uma análise representativa foi realizada em imagens de redes dendríticas na cultura. As células foram extraídas como descrito por Baranes et al. 16,17. Resumidamente, as células hipocampais foram extraídas do cérebro de ratos pós-natais e cultivadas em tampas de vidro 2D por 1-2 semanas. As culturas foram então fixadas e manchadas através da imunofluorescência indireta usando um anticorpo contra o marcador de proteína dendrítica, proteína a...
Access restricted. Please log in or start a trial to view this content.
Discussão
Estratégias eficazes para extrair informações morfológicas de imagens 2D são urgentemente necessárias para acompanhar os dados biológicos de imagem. Embora os dados de imagem possam ser gerados em horas, a análise aprofundada das imagens leva muito tempo. Como resultado, o processamento de imagens tornou-se claramente um grande obstáculo em muitos campos. Isso se deve, em parte, à alta complexidade dos dados, especialmente quando se trata de amostras biológicas. Além disso, como muitos usuários não possuem ...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Os autores gostariam de agradecer ao Dr. Orly Weiss pela preparação das imagens culturais.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
Referências
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083(2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782(2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206(2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ. , Available from: https://imagej.nih.gov/ij (2021).
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005(2019).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados