Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Automatische Identifizierung von dendritischen Ästen und deren Orientierung
In diesem Artikel
Zusammenfassung
Vorgestellt wird ein Computerwerkzeug, das eine einfache und direkte automatische Messung der Orientierungen neuronaler dendritischer Verzweigungen aus 2D-Fluoreszenzbildern ermöglicht.
Zusammenfassung
Die Struktur neuronaler dendritischer Bäume spielt eine Schlüsselrolle bei der Integration synaptischer Inputs in Neuronen. Daher ist die Charakterisierung der Morphologie von Dendriten für ein besseres Verständnis der neuronalen Funktion unerlässlich. Die Komplexität dendritischer Bäume, sowohl isoliert als auch insbesondere in neuronalen Netzwerken, ist jedoch noch nicht vollständig verstanden. Wir haben ein neues Berechnungswerkzeug entwickelt, SOA (Segmentation and Orientation Analysis), das die automatische Messung der Orientierung dendritischer Zweige aus Fluoreszenzbildern neuronaler 2D-Kulturen ermöglicht. SOA, geschrieben in Python, verwendet Segmentierung, um dendritische Verzweigungen vom Bildhintergrund zu unterscheiden und sammelt eine Datenbank in der räumlichen Richtung jedes Zweiges an. Die Datenbank wird dann verwendet, um morphologische Parameter wie die richtungsweisende Verteilung dendritischer Zweige in einem Netzwerk und die Prävalenz des parallelen dendritischen Zweigwachstums zu berechnen. Die gewonnenen Daten können verwendet werden, um strukturelle Veränderungen in Dendriten als Reaktion auf neuronale Aktivität und auf biologische und pharmakologische Reize nachzuweisen.
Einleitung
Die dendritische Morphogenese ist ein zentrales Thema in den Neurowissenschaften, da die Struktur des dendritischen Baumes die Recheneigenschaften der synaptischen Integration in Neuronen beeinflusst1,2,3. Darüber hinaus sind morphologische Anomalien und Modifikationen in dendritischen Ästen mit degenerativen und neurologischen Entwicklungsstörungen assoziiert4,5,6. In neuronalen Kulturen, in denen die dendritische Verzweigung leichter sichtbar gemacht werden kann, regulieren die Wechselwirkungen zwischen nicht-schwesterlichen dendritischen Ästen die Stellen und das Ausmaß der synaptischen Clusterbildung entlang der Zweige, ein Verhalten, das die synaptische Koaktivität und Plastizität beeinflussen kann7,8,9. Daher ist die Charakterisierung der morphologischen Parameter des dendritischen Baumes unter Verwendung zweidimensionaler (2D) neuronaler Kulturen vorteilhaft, um die dendritische Morphogenese und Funktionalität einzelner und Netzwerke von Neuronen zu verstehen. Dies ist jedoch eine herausfordernde Aufgabe, da dendritische Zweige selbst in "vereinfachten" neuronalen 2D-Kulturen ein komplexes Netz bilden.
Mehrere Werkzeuge wurden entwickelt, um dendritische Strukturen automatisch zu verfolgen und zu analysieren10,11,12,13. Die meisten dieser Werkzeuge sind jedoch für neuronale 3D-Netzwerke konzipiert und natürlich zu komplex, um sie mit 2D-Netzwerken zu verwenden. Im Gegensatz dazu beinhalten weniger fortgeschrittene morphologische Analysewerkzeuge typischerweise eine bedeutende Komponente computergestützter manueller Arbeit, die sehr zeitaufwendig und anfällig für Bedienerverzerrungen ist14. Bestehende halbautomatische Tools wie "ImageJ'15" (ein Open-Source-Bildverarbeitungspaket der NIH mit einer umfangreichen Sammlung von von der Community entwickelten biologischen Bildanalysewerkzeugen) reduzieren die manuelle Arbeit des Benutzers weitgehend. Während der Bildverarbeitung sind jedoch noch einige manuelle Eingriffe erforderlich, und die Qualität der Segmentierung kann weniger als wünschenswert sein.
Dieser Artikel stellt die SOA vor, ein einfaches automatisiertes Werkzeug, das eine direkte Segmentierung und Orientierungsanalyse von dendritischen Verzweigungen innerhalb neuronaler 2D-Netzwerke ermöglicht. Die SOA kann verschiedene linienartige Objekte in 2D-Bildern erkennen und deren morphologische Eigenschaften charakterisieren. Hier verwendeten wir die SOA zur Segmentierung dendritischer Zweige in 2D-Fluoreszenzbildern von dendritischen Netzwerken in Kultur. Die Software identifiziert die dendritischen Verzweigungen und führt erfolgreich Messungen morphologischer Parameter wie Parallelität und räumliche Verteilung durch. Die SOA kann leicht für die Analyse zellulärer Prozesse anderer Zelltypen und für die Untersuchung nicht-biologischer Netzwerke angepasst werden.
Protokoll
HINWEIS: Das israelische Gesundheitsministerium genehmigte die Verwendung von Mäusen gemäß Protokoll IL-218-01-21 für die ethische Verwendung von Versuchstieren. SOA ist nur mit Windows 10 und Python 3.9 kompatibel. Es ist als Open-Source-Code verfügbar: https://github.com/inbar2748/DendriteProject. Unter diesem Link gibt es auch eine README. DM-Datei mit Anweisungen zum Herunterladen der Software, einem Link zur Website der Software und einer Anforderungsdatei mit Informationen zu den erforderlichen Versionen aller Pakete. Weitere Beispiele für Analysen, die mit der Software durchgeführt wurden, wurden dort ebenfalls bereitgestellt.
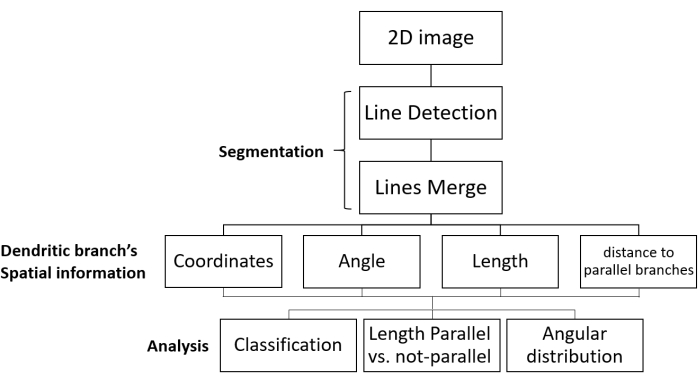
Abbildung 1: SOA-Workflow für die Segmentierung und Wachstumsrichtungsanalyse. Gezeigt werden die Verarbeitungsschritte von Fluoreszenzbildern dendritischer Netzwerke und die Datenanalyse. Das 2D-Bild wird hochgeladen, segmentiert (in zwei Schritten: dendritische Verzweigungen werden als Linien erkannt, und dann werden die relevanten Linien zusammengeführt), und die räumlichen Informationen jedes dendritischen Zweiges werden erhalten. Die Daten werden für alle dendritischen Zweige im Bild gesammelt. Schließlich werden die Daten analysiert, um die gewünschten morphologischen Parameter zu erhalten. Abkürzung: SOA = Segmentierungs- und Orientierungsanalyse. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Öffnen Sie die SOA-Anwendung.
- Öffnen Sie die URL-Adresse: https://mega.nz/folder/bKZhmY4I#4WAaec4biiGt4_1lJlL4WA, suchen Sie die SOA.zip gezippten Ordner und laden Sie die ZIP-Datei per Doppelklick herunter.
- Entpacken Sie den Ordner, indem Sie mit der rechten Maustaste auf SOA klicken.zip und wählen Sie Dateien extrahieren. Beachten Sie das daraufhin geöffnete Fenster Extraktionspfad und Optionen und das Textfeld Zieladresse , in dem der Pfad für die extrahierten Dateien angezeigt wird. Um an einen anderen Speicherort zu extrahieren, klicken Sie auf einen der Ordner im rechten Fensterbereich, um ihn zum Zielordner zu machen. Klicken Sie auf OK, um die Dateien in diesen Ordner zu extrahieren.
- Öffnen Sie die extrahierte SOA-Datei und doppelklicken Sie auf SOA.exe. Warten Sie, bis sich ein schwarzes Fenster öffnet, nach dem die Anwendung angezeigt wird.

Abbildung 2: Beispiel für einen Workflow mit der grafischen Benutzeroberfläche der SOA. Linke Spalte: GUI-Abschnitte des Workflows. Mittlere Spalte: Bild eines dendritischen Netzwerks, das während des Workflows verarbeitet wird (Maßstabsleiste: 20 μm). Rechte Spalte: Vergrößerung des durch ein rotes Rechteck markierten Bereichs in den Bildern der mittleren Spalte (Maßstabsleiste: 4 μm). Schritt 1: Auswahl und Hochladen eines Bildes. Schritt 2: Die erste Stufe der Segmentierung ist die Erkennung von Linien, die die identifizierten dendritischen Zweige darstellen. Schritt 3: Die zweite Stufe der Segmentierung ist die näherungsbasierte Verschmelzung der Segmentauskleidung in einzelnen dendritischen Ästen. Die Einstellungen aller Schritte können geändert werden. Abkürzungen: SOA = Segmentierungs- und Orientierungsanalyse; GUI = grafische Benutzeroberfläche. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Öffnen Sie ein Bild zur Analyse.
- In der SOA Viewer-Menüleiste Hochladen | Wählen Sie Datei auswählen | Wählen Sie ein Bild aus den Computerdateien | Klicken Sie darauf (nur .png .jpg .tif .bmp Dateien) | | Beachten Sie den Pfad der Datei | Nächster.
3. Segmentierungsoptimierung
HINWEIS: Ändern Sie in der SoA Viewer-Menüleiste Eigenschaften die Werte der ausgewählten Parameter, um die Einstellungen für den Segmentierungsprozess anzupassen. Eine detaillierte Beschreibung der Parameter, wie z. B . der Schwellenwert, finden Sie im Ergänzungsmaterial.
- Passen Sie unter Kanten den Schwellenwert für die Anzeige an, indem Sie den Schwellenwert auswählen und eine Zahl eingeben.
HINWEIS: Je niedriger die Zahl für den Schwellenwert ist, desto mehr Zeilen werden erkannt. Schwellenwert ist eine Zahl, die zwischen 0 und 255 liegt. Der Standardwert wurde auf 0 festgelegt. - In Seriendruckzeilen:
- Passen Sie den Mindestabstand für die Zusammenführung für die Anzeige an, indem Sie den Mindestabstand zum Zusammenführen auswählen und eine Zahl eingeben.
HINWEIS: Der Mindestabstand zum Zusammenführen reicht von 0 bis 30 Pixeln. Der Standardwert ist auf 20 festgelegt. - Passen Sie den minimalen Winkel zum Zusammenführen für die Anzeige an, indem Sie den zu verschmelzenden Mindestwinkel auswählen und eine Zahl eingeben.
HINWEIS: Der Min-Winkel zum Zusammenführen reicht von 0 bis 30°. Der Standardwert ist auf 10 festgelegt.
- Passen Sie den Mindestabstand für die Zusammenführung für die Anzeige an, indem Sie den Mindestabstand zum Zusammenführen auswählen und eine Zahl eingeben.
- Klicken Sie auf Vorschau-Segmentierungsbild erstellen.
HINWEIS: Ein Vorschaubild der Segmentierungsergebnisse wird entsprechend den aktualisierten Werten angezeigt. Zusätzlich wird die Anzahl der Zeilen vor dem Zusammenführen und die Anzahl der Zeilen nach dem Zusammenführen angezeigt. - Ändern Sie die Parameter, um eine maximale Identifizierung der Segmente zu erreichen. Wenn Sie die Eigenschaften ändern müssen, klicken Sie auf die Schaltfläche Fenster schließen und folgen Sie den Schritten 3.1-3.4.
4. Erstellen Sie die Ausgabedateien.
- Drücken Sie OK , um die Segmentierungsbilder und die Analysediagramme zu visualisieren. Beobachten Sie das angezeigte Fenster, in dem Sie einen Speicherort auswählen, an dem die .xlsx Datei gespeichert werden soll.
- Fügen Sie einen Dateinamen | Wählen Sie Save | Warten Sie, bis die .xlsx Datei mit Daten erstellt und gespeichert wurde.
HINWEIS: Zusätzlich zur .xlsx Datei werden automatisch die folgenden Dateien angezeigt: eine Datei, die das Originalbild, das Linienerkennungsbild, das endgültige Bild der Segmentierung und drei Analysediagramme darstellt.
5. Navigationssymbolleiste
HINWEIS: Eine Navigationssymbolleiste ist in allen Abbildungsfenstern enthalten und kann zum Navigieren durch den Datensatz verwendet werden. Jede der Schaltflächen am unteren Rand der Symbolleiste wird im Folgenden beschrieben.
- Um zwischen zuvor definierten Ansichten hin und her zu navigieren, verwenden Sie die Schaltflächen Vorwärts und Zurück .
HINWEIS: Die Schaltflächen Start, Vorwärts und Zurück ähneln den Steuerelementen Home, Vorwärts und Zurück in einem Webbrowser. Home kehrt zum Standardbildschirm, dem Originalbild, zurück. - Verwenden Sie die Schaltfläche Zoom zum Schwenken und Zoomen. Um das Schwenken und Zoomen zu aktivieren, drücken Sie die Zoom-Taste und bewegen Sie dann die Maus an die gewünschte Stelle im Bild.
- Um die Figur zu schwenken, halten Sie die linke Maustaste gedrückt, während Sie sie an eine neue Position ziehen. Lassen Sie die Maustaste los, und der ausgewählte Punkt im Bild wird an der neuen Position angezeigt. Halten Sie während des Schwenkens die Tasten x oder y gedrückt, um die Bewegung auf die x - bzw. y-Achse zu beschränken.
- Halten Sie zum Vergrößern die rechte Maustaste gedrückt und ziehen Sie sie an eine neue Position. Bewegen Sie sich nach rechts, um auf der x-Achse zu vergrößern, und nach links, um auf der x-Achse zu verkleinern. Machen Sie dasselbe für die y-Achse und die Aufwärts-/Abwärtsbewegungen. Beachten Sie beim Zoomen, dass der Punkt unter der Maus stationär bleibt, sodass Sie um diesen Punkt herum vergrößern oder verkleinern können. Verwenden Sie die Modifikatortasten x, y oder CTRL , um den Zoom auf die Beibehaltung von x, y bzw. Seitenverhältnis zu beschränken.
- Um den Zoom-zu-Rechteck-Modus zu aktivieren, klicken Sie auf die Schaltfläche Zoom-zu-Rechteck . Bewegen Sie den Mauszeiger über das Bild und drücken Sie die linke Maustaste. Ziehen Sie die Maus an eine neue Position, während Sie die Schaltfläche gedrückt halten, um einen rechteckigen Bereich zu definieren.
HINWEIS: Die Achsenansichtsgrenzen werden auf den definierten Bereich gezoomt, wenn die linke Maustaste gedrückt wird. Die Achsenansichtsgrenzen werden verkleinert, wenn die rechte Maustaste gedrückt wird, wobei die ursprünglichen Achsen im definierten Bereich platziert werden. - Verwenden Sie das Subplot-Konfigurationstool , um das Erscheinungsbild der Subplot zu konfigurieren.
HINWEIS: Die linke, rechte, obere und untere Seite der Nebenhandlung sowie der Abstand zwischen Zeilen und Spalten können gestreckt oder komprimiert werden. - Um ein Dialogfeld zum Speichern einer Datei zu öffnen, klicken Sie auf die Schaltfläche Speichern und speichern Sie die Datei in den folgenden Formaten: .png, .ps, .eps, .svg oder .pdf.
Ergebnisse
Eine repräsentative Analyse wurde an Bildern von dendritischen Netzwerken in Kultur durchgeführt. Zellen wurden extrahiert, wie von Baranes et al. beschrieben. 16,17. Kurz gesagt, Hippocampuszellen wurden aus den Gehirnen postnataler Ratten extrahiert und auf 2D-Glasdeckgläsern für 1-2 Wochen kultiviert. Die Kulturen wurden dann fixiert und durch indirekte Immunfluoreszenz unter Verwendung eines Antikörpers gegen den dendritischen Proteinmarker, das...
Diskussion
Effektive Strategien zur Extraktion morphologischer Informationen aus 2D-Bildern sind dringend erforderlich, um mit den biologischen Bildgebungsdaten Schritt zu halten. Obwohl Bilddaten in Stunden generiert werden können, dauert eine eingehende Analyse der Bilder sehr lange. Infolgedessen ist die Bildverarbeitung in vielen Bereichen eindeutig zu einem großen Hindernis geworden. Das liegt unter anderem an der hohen Komplexität der Daten, insbesondere beim Umgang mit biologischen Proben. Da viele Benutzer keine speziell...
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
Die Autoren danken Dr. Orly Weiss für die Aufbereitung der Kulturbilder.
Materialien
| Name | Company | Catalog Number | Comments |
| Matplotlib | 2002 - 2012 John Hunter, Darren Dale, Eric Firing, Michael Droettboom and the Matplotlib development team; 2012 - 2021 The Matplotlib development team. | 3.4.2 | a Python 2D plotting library |
| matplotlib-scalebar | Philippe Pinard | 0.7.2 | artist for matplotlib to display a scale bar |
| NumPy | The NumPy community. | 1.20.3 | fundamental package for scientific computing library |
| OpenCV | OpenCV team | 4.5.2.54 | Open Source Computer Vision Library |
| PyCharm | JetBrains | 2020.3.1 (Community Edition) version | Build #PC-203.6682.86, built on December 18, 2020. Runtime version: 11.0.9.1+11-b1145.37 amd64. VM: OpenJDK 64-Bit Server VM by JetBrains s.r.o. Windows 10 10.0. Memory: 978M, Cores: 4 |
| PyQt5 | Riverbank Computing | 5.15.4 | manage the GUI |
| python | Python Software Foundation License | 3.9 version | |
| Qt Designer | The QT Company Ltd. | 5.11.1 version | |
| scipy | Community library project | 1.6.3 | Python-based ecosystem of open-source software for mathematics, science, and engineering |
| Seaborn | Michael Waskom. | 0.11.1 | Python's Statistical Data Visualization Library. |
| Windows 10 | Microsoft | ||
| Xlsxwriter | John McNamara | 1.4.3 | Python module for creating Excel XLSX files |
Referenzen
- Ferrante, M., Migliore, M., Ascoli, G. Functional impact of dendritic branch-point morphology. Journal of Neuroscience. 33 (5), 2156-2165 (2013).
- Spruston, N. Pyramidal neurons: dendritic structure and synaptic integration. Nature Reviews Neuroscience. 9 (3), 206-221 (2008).
- Chklovskii, D. Synaptic Connectivity and Neuronal Morphology: Two Sides of the Same Coin. Neuron. 43 (5), 609-617 (2004).
- Chapleau, C., Larimore, J., Theibert, A., Pozzo-Miller, L. Modulation of dendritic spine development and plasticity by BDNF and vesicular trafficking: fundamental roles in neurodevelopmental disorders associated with mental retardation and autism. Journal of Neurodevelopmental Disorders. 1 (3), 185-196 (2009).
- Irwin, S. Dendritic Spine Structural Anomalies in Fragile-X Mental Retardation Syndrome. Cerebral Cortex. 10 (10), 1038-1044 (2000).
- Kaufmann, W. Dendritic anomalies in disorders associated with mental retardation. Cerebral Cortex. 10 (10), 981-991 (2000).
- Pinchas, M., Baranes, D. Dendritic branch intersections are structurally regulated targets for efficient axonal wiring and synaptic clustering. PLoS ONE. 8 (12), 82083 (2013).
- Cove, J., Blinder, P., Baranes, D. Contacts among non-sister dendritic branches at bifurcations shape neighboring dendritic branches and pattern their synaptic inputs. Brain Research. 1251, 30-41 (2009).
- Blinder, P., Cove, J., Foox, M., Baranes, D. Convergence among non-sister dendritic branches: An activity-controlled mean to strengthen network connectivity. PLoS ONE. 3 (11), 3782 (2008).
- Glaser, J., Glaser, E. Neuron imaging with neurolucida - PC-based system for image combining microscopy. Computerized Medical Imaging and Graphics. 14 (5), 307-317 (1990).
- Scorcioni, R., Polavaram, S., Ascoli, G. L-Measure: a web-accessible tool for the analysis, comparison and search of digital reconstructions of neuronal morphologies. Nature Protocols. 3 (5), 866-876 (2008).
- Torben-Nielsen, B. An efficient and extendable python library to analyze neuronal morphologies. Neuroinformatics. 12 (4), 619-622 (2014).
- Parekh, R., Ascoli, G. Neuronal morphology goes digital: A research hub for cellular and system neuroscience. Neuron. 78 (1), 206 (2013).
- heng, J., Zhou, X., Sabatini, B. L., Wong, S. T. C. NeuronIQ: A novel computational approach for automatic dendrite SPINES detection and analysis. 2007 IEEE/NIH Life Science Systems and Applications Workshop. , 168-171 (2007).
- Image processing and analysis in Java. NIH. ImageJ Available from: https://imagej.nih.gov/ij (2021)
- Peretz, H., Talpalar, A. E., Vago, R., Baranes, D. Superior survival and durability of neurons and astrocytes on 3-dimensional aragonite biomatrices. Tissue Engineering. 13, 461-472 (2007).
- Morad, T. I., Hendler, R. M., Weiss, O. E., Canji, E. A., Merfeld, I., Dubinsky, Z., Minnes, R., Francis, Y. I., Baranes, D. Gliosis of astrocytes cultivated on coral skeleton is regulated by the matrix surface topography. Biomedical Materials. 14 (4), 045005 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten