Method Article
تسلسل الجينوم الكامل للتوصيف السريع لفيروس داء باستخدام تقنية النانو
In This Article
Summary
هنا ، نقدم سير عمل سريع وفعال من حيث التكلفة لتوصيف جينومات فيروس داء (RABV) باستخدام تقنية المسام النانوية. يهدف سير العمل إلى دعم المراقبة الواعية بالجينوم على المستوى المحلي ، وتوفير معلومات حول سلالات RABV المتداولة ووضعها داخل السلالات الإقليمية لتوجيه تدابير مكافحة داء.
Abstract
يمكن استخدام البيانات الجينومية لتتبع انتقال الأمراض المعدية وانتشارها الجغرافي. ومع ذلك، لا تزال القدرة على تحديد التسلسل الجيني اللازمة للترصد الجينومي محدودة في العديد من البلدان المنخفضة والمتوسطة الدخل، حيث يشكل داء و/أو داء الذي تنقله البرية مثل الخفافيش مصاصة الدماء شواغل صحية واقتصادية كبيرة. نقدم هنا سير عمل سريع وبأسعار معقولة من العينة إلى التسلسل إلى التفسير باستخدام تقنية المسام النانوية. يتم وصف بروتوكولات جمع العينات وتشخيص داء بإيجاز ، متبوعة بتفاصيل سير عمل تسلسل الجينوم الكامل الأمثل ، بما في ذلك تصميم التمهيدي والتحسين لتفاعل البوليميراز المتسلسل المتعدد (PCR) ، وإعداد مكتبة تسلسل معدلة ومنخفضة التكلفة ، والتسلسل مع الاتصال الأساسي المباشر وغير المتصل بالإنترنت ، وتعيين النسب الجيني ، وتحليل النشوء والتطور. يتم عرض تنفيذ سير العمل ، ويتم تسليط الضوء على الخطوات الحاسمة للنشر المحلي ، مثل التحقق من صحة خط الأنابيب ، وتحسين التمهيدي ، وإدراج الضوابط السلبية ، واستخدام البيانات المتاحة للجمهور والأدوات الجينومية (GLUE ، MADDOG) للتصنيف والتنسيب داخل السلالات الإقليمية والعالمية. الوقت المستغرق لسير العمل هو 2-3 أيام ، وتتراوح التكلفة من 25 دولارا لكل عينة لتشغيل عينة 96 إلى 80 دولارا لكل عينة لتشغيل 12 عينة. نستنتج أن إنشاء المراقبة الجينومية لفيروس داء في البلدان المنخفضة والمتوسطة الدخل أمر ممكن ويمكن أن يدعم التقدم نحو الهدف العالمي المتمثل في القضاء على وفيات داء البشري بوساطة بحلول عام 2030 ، فضلا عن تعزيز رصد انتشار داء في الحياة البرية. علاوة على ذلك ، يمكن تكييف المنصة مع مسببات الأمراض الأخرى ، مما يساعد على بناء قدرة جينومية متعددة الاستخدامات تساهم في التأهب للأوبئة والجوائح.
Introduction
فيروس داء (RABV) هو فيروس ليسا في عائلة Rhabdoviridae يسبب مرضا عصبيا قاتلا في الثدييات1. على الرغم من أن داء يمكن الوقاية منه بنسبة 100٪ عن طريق التطعيم ، إلا أنه لا يزال مصدر قلق كبير للصحة العامة والاقتصادية في البلدان الموبوءة. من بين 60000 حالة وفاة بشرية بسبب داء يقدر حدوثها كل عام ، يوجد أكثر من 95٪ في إفريقيا وآسيا حيث هي المستودع الأساسي2. في المقابل ، أدى تطعيم إلى القضاء على داء بوساطة في جميع أنحاء أوروبا الغربية وأمريكا الشمالية وجزء كبير من أمريكا اللاتينية. في هذه المناطق ، تقتصر خزانات داء الآن على الحياة البرية ، مثل الخفافيش والراكون والظربان البرية3. في جميع أنحاء أمريكا اللاتينية ، يعد الخفاش مصاص الدماء الشائع مصدرا إشكاليا لداء بسبب الانتقال المنتظم غير المباشر من الخفافيش إلى كل من البشر والماشية أثناء تغذية الدم ليلا4. ويقدر الأثر الاقتصادي العالمي السنوي لداء بنحو 8.6 مليار دولار، وتمثل خسائر الثروة الحيوانية 6٪5.
يمكن أن توفر بيانات التسلسل من مسببات الأمراض الفيروسية جنبا إلى جنب مع البيانات الوصفية حول توقيت ومصدر العدوى رؤى وبائية قوية6. بالنسبة ل RABV ، تم استخدام التسلسل للتحقيق في أصل الفاشيات 7,8 ، وتحديد الارتباطات المضيفة مع الحياة البرية أو الأليفة 8،9،10،11،12 ، وتتبع مصادر الحالات البشرية13،14. وقد أشارت استقصاءات الفاشية باستخدام التحليل الوراثي إلى أن داء ظهر في مقاطعة بالي التي كانت خالية من داء سابقا في إندونيسيا، من خلال إدخال واحد من المناطق الموبوءة القريبة في كاليمانتان أو سولاويزي15. وفي الوقت نفسه ، في الفلبين ، ثبت أن تفشي المرض في جزيرة تابلاس بمقاطعة رومبلون قد تم إدخاله من جزيرة لوزون16 الرئيسية. كما تم استخدام البيانات الجينومية الفيروسية لفهم ديناميكيات انتقال مسببات الأمراض المطلوبة لاستهداف تدابير المكافحة جغرافيا بشكل أفضل. على سبيل المثال ، يوضح التوصيف الجينومي ل RABV التجميع الجغرافي للمجموعات 17،18،19 ، والدوران المشترك للسلالات 20،21،22 ، والحركة الفيروسية بوساطة الإنسان 17،23،24 ، وديناميكيات metapopulation 25،26.
تعد مراقبة الأمراض إحدى الوظائف المهمة للمراقبة الجينومية التي تم تعزيزها مع الزيادة العالمية في القدرة على التسلسل استجابة لوباء SARS-CoV-2. دعمت المراقبة الجينومية التتبع في الوقت الفعلي لمتغيرات SARS-COV-2 المثيرة للقلق27,28 والتدابير المضادة المرتبطةبها 6. أدى التقدم في تكنولوجيا التسلسل التي يمكن الوصول إليها ، مثل تقنية المسام النانوية ، إلى بروتوكولات محسنة وبأسعار معقولة للتسلسل السريع لكل من مسببات الأمراضالبشرية 29،30،31،32 والحيوانية33،34،35. ومع ذلك ، في العديد من البلدان الموبوءة بداء ، لا تزال هناك حواجز أمام تفعيل المراقبة الجينومية لمسببات الأمراض ، كما يتضح من التفاوتات العالمية في قدرة تسلسل SARS-CoV-236. إن القيود المفروضة على البنية التحتية للمختبرات وسلاسل التوريد والمعرفة التقنية تجعل إنشاء المراقبة الجينومية وروتينها أمرا صعبا. في هذه الورقة ، نوضح كيف يمكن نشر سير عمل تسلسل الجينوم الكامل الأمثل والسريع وبأسعار معقولة لمراقبة RABV في إعدادات محدودة الموارد.
Protocol
وتمت الموافقة على الدراسة من قبل لجنة تنسيق البحوث الطبية التابعة للمعهد الوطني للبحوث الطبية (NIMR/HQ/R.8a/vol. IX/2788)، ووزارة الإدارة الإقليمية والحكم المحلي (AB.81/288/01)، ومجلس المراجعة المؤسسية لمعهد إيفاكارا الصحي (IHI/IRB/No:22-2014) في تنزانيا؛ معهد جامعة نيروبي للأمراض المدارية والمعدية (P947/11/2019) ومعهد كينيا للبحوث الطبية (KEMRI-SERU ؛ البروتوكول رقم 3268) في كينيا ؛ ومعهد بحوث طب المناطق الحارة (RITM) ، وزارة الصحة (2019-023) في الفلبين. وأجري تسلسل العينات القادمة من نيجيريا على مواد التشخيص المحفوظة التي جمعت كجزء من الترصد الوطني.
ملاحظة: القسم 1-4 هي متطلبات أساسية. يصف القسم 5-16 سير عمل العينة إلى التسلسل إلى التفسير لتسلسل المسام النانوية RABV (الشكل 1). بالنسبة للخطوات اللاحقة في البروتوكول التي تحتاج إلى الطرد المركزي النبضي ، أجهزة الطرد المركزي عند 10-15000 × جم لمدة 5-15 ثانية.
1. إعداد البيئة الحسابية للتسلسل وتحليل البيانات
- افتح موقع Oxford Nanopore Technology (ONT)37 وأنشئ حسابا للوصول إلى الموارد الخاصة بالمسام النانوية.
- قم بتسجيل الدخول وتثبيت تسلسل ONT وبرنامج الاتصال الأساسي38.
- افتح GitHub39 وأنشئ حسابا.
- انتقل إلى مستودعات artic-rabv40 و MADDOG41 واتبع تعليمات التثبيت.
2. تصميم أو تحديث مخطط التمهيدي المتعدد
ملاحظة: مخططات RABV الحالية متوفرة في مستودع artic-rabv40. عند استهداف منطقة جغرافية جديدة ، يجب تصميم مخطط جديد ، أو تعديل مخطط قائم لدمج تنوع إضافي.
- اختر مجموعة مرجعية للجينوم لتمثيل التنوع في منطقة الدراسة ؛ عادة ما تكون هذه مجموعة من التسلسلات المتاحة للجمهور (على سبيل المثال ، من NCBI GenBank) أو البيانات الداخلية الأولية. اتبع الخطوة 2.1.1 لاستخدام RABV-GLUE42 ، وهو مورد بيانات تسلسل RABV ، لتصفية وتنزيل تسلسلات NCBI والبيانات الوصفية المرتبطة بها.
ملاحظة: اختر تسلسلات مرجعية ذات جينومات كاملة (أي بدون فجوات وقواعد مقنعة). يوصى باختيار ما يصل إلى 10 تسلسلات كمجموعة مرجعية لتصميم التمهيدي. إذا كانت بيانات التسلسل المتاحة غير كاملة أو غير ممثلة لمنطقة الدراسة ، فراجع المشورة43،44،45 في الملف التكميلي 1.- انتقل إلى NCBI RABV تسلسلات بواسطة Clade صفحة من القائمة المنسدلة بيانات التسلسل في RABV-GLUE. انقر فوق رابط فيروس داء (RABV) للوصول إلى جميع البيانات المتاحة أو تحديد مجموعة معينة من الاهتمام. استخدم خيار التصفية لإضافة عوامل تصفية تناسب المعايير المطلوبة (على سبيل المثال، بلد المنشأ وطول التسلسل). تنزيل التسلسلات والبيانات الوصفية.
- قم بإنشاء مخطط تمهيدي لتفاعل البوليميراز المتسلسل المتعدد (PCR) باتباع التعليمات المقدمة من المخطط البدائي46. يوصى باستخدام مخطط 400 نقطة أساس مع تداخل 50 نقطة أساس لتسلسل عينات منخفضة الجودة. قم بتنزيل وحفظ جميع المخرجات (لا تقم بتحرير أسماء الملفات أو التمهيدي).
ملاحظة: سيتم فهرسة المخطط إلى التسلسل الأول في فاستا الإدخال ، ويشار إليه من الآن فصاعدا باسم "مرجع الفهرس" (الشكل 2). راجع الملف التكميلي 1 للحصول على خيارات لتحسين أداء التمهيدي.
3. إعداد خطوط أنابيب RAMPART والمعلوماتية الحيوية ARTIC
- راجع الملف التكميلي 2 لإعداد بنية دليل لإدارة ملفات الإدخال / الإخراج ل RAMPART وخط أنابيب المعلوماتية الحيوية ARTIC.
4. السلامة البيولوجية وإعداد المختبرات
- التعامل مع العينات التي يحتمل أن تكون إيجابية لداء في ظروف مستوى السلامة البيولوجية (BSL) 2 أو 3.
- التأكد من أن موظفي المختبر قد أتموا التطعيم ضد داء قبل التعرض ويخضعون لرصد المناعة وفقا لتوصيات منظمة الصحة العالمية(WHO) 3.
- ضمان وجود إجراءات تشغيل موحدة مخصصة وتقييمات للمخاطر، باتباع المبادئ التوجيهية الوطنية أو الدولية، للمختبر.
- إعداد المختبر المطلوب: تقليل التلوث عن طريق الحفاظ على الفصل المادي بين مناطق ما قبل وبعد تفاعل البوليميراز المتسلسل. في المختبرات ذات المساحة المحدودة أو إعدادات المختبر الميداني ، استخدم صناديق القفازات المحمولة أو محطات المختبر المؤقتة لتقليل التلوث.
- في هذا البروتوكول ، تأكد من تعيين مناطق منفصلة ل:
- استخراج العينة: قم بإعداد خزانة / صندوق قفازات BSL2 / 3 للتعامل مع المواد البيولوجية وإجراء التعطيل واستخراج الحمض النووي الريبي.
- منطقة القالب: قم بإعداد خزانة / صندوق قفازات BSL1 لإضافة قالب (RNA / cDNA) إلى المزيج الرئيسي للتفاعل المعد مسبقا.
- منطقة الخلط الرئيسية: قم بإعداد منطقة نظيفة مخصصة (خزانة BSL1 / صندوق قفازات) لإعداد الخلطات الرئيسية للكاشف. يجب ألا يكون هناك قالب في هذا المجال.
- منطقة ما بعد تفاعل البوليميراز المتسلسل: قم بإعداد منطقة منفصلة للعمل على amplicons وإعداد مكتبة التسلسل.
ملاحظة: يجب تنظيف جميع المناطق باستخدام مطهر سطحي وتعقيم الأشعة فوق البنفسجية (UV) قبل الاستخدام وبعده.
5. جمع العينات الميدانية والتشخيص
ملاحظة: يجب جمع العينات من قبل موظفين مدربين ومحصنين يرتدون معدات الحماية الشخصية ويتبعون الإجراءات القياسية المشار إليها47،48،49.
- اجمع العينة عبر الثقبة العظمى (أي الطريق القذالي) ، كما هو موضح بالتفصيل في Mauti et al.50.
- تشخيص داء في الميدان من خلال الاختبارات التشخيصية السريعة والتأكيد في المختبر باستخدام الإجراءات الموصى بها47 ، مثل اختبار الأجسام المضادة الفلورية المباشرة (DFA) ، أو الاختبار الكيميائي المناعي السريع المباشر (DRIT) 51,52 ، أو النسخ العكسي في الوقت الفعلي (RT) -PCR 53.
- استخدم عينات الدماغ الإيجابية المؤكدة لاستخراج الحمض النووي الريبي أو تخزينها في الثلاجة عند -20 درجة مئوية لمدة 2-3 أشهر أو -80 درجة مئوية لفترات أطول. الحفاظ على الحمض النووي الريبي للتخزين والنقل باستخدام وسيط تثبيت الحمض النووي / الحمض النووي الريبي المناسب.
6. تحضير العينة واستخراج الحمض النووي الريبي (3 ساعات)
ملاحظة: استخدم مجموعة استخراج الحمض النووي الريبي الفيروسي القائمة على عمود الدوران المناسبة لنوع العينة.
- قم بإعداد أنبوبين من الخرز الخزفي عن طريق ملء أنبوب PCR سعة 2 مل بأنبوب 200 ميكرولتر تقريبا مليء بخرز السيراميك مقاس 1.4 مم وقم بتسمية الأنبوب.
- أضف الحجم الموصى به من المخزن المؤقت للتحلل الموجود في مجموعة استخراج الحمض النووي الريبي إلى أنبوب PCR المسمى.
- احصل على مكعب 3 مم تقريبا من عينة الدماغ المؤكدة بعدوى داء باستخدام قضيب خشبي وضعه في أنبوب ملصق مع معرف العينة و 100 ميكرولتر من الماء الخالي من النيوكلياز في الأنبوب المسمى بالتحكم السلبي.
ملاحظة: استخدم التجانس القائم على حبة الأنبوب المغلق للحد من التعرض للعينة. إذا لم يكن ذلك ممكنا ، فاستخدم عوامل تعطيل ميكانيكية مناسبة أخرى (على سبيل المثال ، قائمة على الدوار) أو مدقة دقيقة يدوية. ومع ذلك ، قد تكون هذه أقل فعالية من ضرب الخرزة على سطح صلب لتعطيل الأنسجة (قد تتصلب عينات الأنسجة في بعض وسائط التخزين). - قم بتعطيل أنسجة المخ يدويا باستخدام عصا قضيب خشبية ثم دوامة بأقصى سرعة حتى يتم تحقيق التجانس الكامل للأنسجة.
- أجهزة الطرد المركزي المحللة وفقا لتعليمات الشركة المصنعة واستخدام ماصة لنقل المادة الطافية إلى أنبوب طرد مركزي دقيق جديد مسمى. استخدم هذا الطافي فقط في الخطوات اللاحقة.
- اتبع تعليمات عمود الدوران لمجموعة استخراج الحمض النووي الريبي للحصول على الحمض النووي الريبي المنقى.
- قم بتضمين عنصر تحكم في الاستخراج السلبي (NEC) هنا وانتقل إلى مرحلة التسلسل.
7. تحضير cDNA (20 دقيقة)
- في منطقة المزيج الرئيسي ، قم بإعداد مزيج رئيسي لتخليق cDNA الأول وفقا لعدد العينات والضوابط التي سيتم معالجتها (مع حجم زائد بنسبة 10٪ لضمان الكاشف الكافي ؛ الجدول 1). يجب تضمين عنصر تحكم بدون قالب (NTC) في هذه المرحلة.
- قم بتسمية أنابيب شريط PCR سعة 0.2 مل و aliquot 5 ميكرولتر من المزيج الرئيسي في الأنابيب.
- خذ الأنابيب المعدة إلى منطقة القالب. أضف 5 ميكرولتر من الحمض النووي الريبي في كل أنبوب مسمى ، بما في ذلك NEC. أضف 5 ميكرولتر من الماء الخالي من النيوكلياز (NFW) إلى NTC.
- احتضان في دورة حرارية وفقا للشروط المذكورة في الجدول 1.
ملاحظة: نقطة توقف اختيارية: يمكن تخزين cDNA في -20 درجة مئوية لمدة تصل إلى 1 شهر إذا لزم الأمر ، ولكن يفضل الانتقال إلى تفاعل البوليميراز المتسلسل.
8. إعداد مخزون بركة التمهيدي (1 ساعة)
ملاحظة: هذه الخطوة ضرورية فقط في حالة إنشاء مخزون جديد من البادئات الفردية ، وبعد ذلك يمكن استخدام حلول المخزون المعدة مسبقا.
- قم بإعداد مجموعة أولية من مخزون 100 ميكرومتر في منطقة المزيج الرئيسي.
- أعد تعليق البادئات المجففة بالتجميد في 1x tris-EDTA (TE) عازلة أو NFW بتركيز 100 ميكرومتر لكل منهما. دوامة بدقة وتدور لأسفل.
ملاحظة: في الخطوات التالية، يتم فصل البادئات الفردية إلى اثنين من تجمعات التمهيدي - مرقمة فردية (تسمى تجمع A) وزوجية مرقمة (تسمى تجمع B) - لتجنب التفاعلات بين البادئات المحيطة بتداخلات amplicon. تولد هذه المجموعات من البادئات مضخمات متداخلة تبلغ 400 bp تمتد عبر الجينوم المستهدف. - رتب جميع البرايمرات ذات الأرقام الفردية في رف أنبوبي. قم بإنشاء مخزون تجمع تمهيدي عن طريق إضافة 5 ميكرولتر من كل جهاز تمهيدي إلى أنبوب طرد مركزي دقيق سعة 1.5 مل يسمى "اسم مخطط التمهيدي - المسبح A (100 ميكرومتر)".
- كرر العملية لجميع البادئات ذات الأرقام الزوجية والتسمية ك "اسم مخطط التمهيدي - المسبح B (100 ميكرومتر)".
- قم بتخفيف كل حوض تمهيدي 1:10 في ماء من الدرجة الجزيئية لتوليد مخزون تمهيدي 10 ميكرومتر.
ملاحظة: قم بعمل حصص متعددة من التخفيفات التمهيدية 10 ميكرومتر وقم بتجميدها في حالة التدهور أو التلوث.
9. PCR متعدد الإرسال (5 ساعات)
- قم بإعداد مزيجين رئيسيين PCR ، واحد لكل تجمع تمهيدي في منطقة المزيج الرئيسي.
- استخدم تركيزا نهائيا قدره 0.015 ميكرومتر لكل برايمر. احسب حجم تجمع التمهيدي المطلوب لتفاعل PCR (الجدول 2) باستخدام الصيغة التالية:
حجم تجمع التمهيدي = عدد البادئات × حجم التفاعل × 0.015 / تركيز (ميكرومتر) من مخزون التمهيدي
- استخدم تركيزا نهائيا قدره 0.015 ميكرومتر لكل برايمر. احسب حجم تجمع التمهيدي المطلوب لتفاعل PCR (الجدول 2) باستخدام الصيغة التالية:
- Aliquot 10 ميكرولتر لكل من المزيج الرئيسي للمسبح A والمزيج الرئيسي للمسبح B لأنابيب شريط PCR المسماة في منطقة القالب. لكل عينة ، أضف 2.5 ميكرولتر من cDNA (من الخطوة 3) إلى كل من تفاعلات المجموعة A و B الأولية المناظرة. يمكن تخزين cDNA الزائد عند -20 درجة مئوية.
- تخلط عن طريق نفض الغبار بلطف ونبض الطرد المركزي.
- احتضان العينات بالشروط المذكورة في الجدول 2 على جهاز PCR.
ملاحظة: لا يتضمن البرنامج خطوة تمديد محددة بسبب وقت التلدين الطويل البالغ 5 دقائق (مطلوب بسبب العدد الكبير من البادئات) وقصر طول الأمبليكونات (400 نقطة أساس) وهو ما يكفي للتمديد.
10. تنظيف PCR والقياس الكمي (3.5 ساعة)
- قم بتنفيذ جميع الأعمال من هذه النقطة فصاعدا في منطقة ما بعد تفاعل البوليميراز المتسلسل.
- حبات الشلل العكسي للمرحلة الصلبة (SPRI) في أنابيب الطرد المركزي الدقيقة من الزجاجة الرئيسية. يحفظ في درجة حرارة 4 درجة مئوية.
- قم بتسخين حصصة حبة SPRI إلى درجة حرارة الغرفة (RT ؛ ~ 20 °C) ودوامة تماما حتى يتم إعادة تعليق الخرزات بالكامل في المحلول.
- في أنابيب سعة 1.5 مل ، ادمج منتجات التمهيدي Pool A و Primer Pool B PCR لكل عينة. إذا لزم الأمر ، أضف الماء لرفع الحجم إلى 25 ميكرولتر.
- أضف 25 ميكرولتر من حبات SPRI إلى كل عينة (1: 1 حبة: نسبة العينة). امزج عن طريق السحب لأعلى ولأسفل أو النقر برفق على الأنبوب.
- احتضان في RT لمدة 10 دقائق ، وأحيانا قلب أو تحريك الأنابيب.
- ضعه على رف مغناطيسي حتى تنفصل الحبيبات والمحلول تماما. قم بإزالة المادة الطافية والتخلص منها ، مع الحرص على عدم إزعاج حبيبات الخرزة.
- يغسل مرتين مع 80 ٪ من الإيثانول (تحسنت إلى RT).
- أضف 200 ميكرولتر من الإيثانول إلى الحبيبات. انتظر لمدة 30 ثانية للتأكد من غسل الخرز بشكل صحيح.
- قم بإزالة المادة الطافية والتخلص منها بعناية ، محاولا عدم لمس حبيبات الخرزة.
- كرر الخطوات 10.8.1-10.8.2 لغسل الحبيبات مرة ثانية.
- قم بإزالة جميع آثار الإيثانول باستخدام طرف 10 ميكرولتر. يجف في الهواء حتى يتبخر الإيثانول النزرة (مع حبات صغيرة يحدث هذا بسرعة ، ~ 30 ثانية) ؛ عندما يحدث هذا ، يجب أن تنتقل الحبيبات من لامعة إلى غير لامعة. احرص على عدم الإفراط في الجفاف (إذا كانت الحبيبات تتشقق ، فهي جافة جدا) ، لأن ذلك سيؤثر على استعادة الحمض النووي.
- أعد تعليق الخرزات في 15 ميكرولتر من NFW واحتضانها في RT (إيقاف الرف المغناطيسي) لمدة 10 دقائق.
- ارجع إلى الرف المغناطيسي وانقل المادة الطافية (المنتج النظيف) إلى أنبوب جديد سعة 1.5 مل.
- قم بإعداد تخفيف 1:10 لكل عينة في أنبوب منفصل (2 ميكرولتر من المنتج + 18 ميكرولتر من NFW).
ملاحظة: كن حذرا جدا في هذه المرحلة لتجنب التلوث المتبادل. لديك أنبوب amplicon واحد فقط مفتوح في كل مرة. قسمة 18 ميكرولتر من الماء في الأنابيب أولا (في منطقة خلط رئيسية نظيفة). - قم بقياس تركيز الحمض النووي لكل عينة مخففة باستخدام مقياس فلوري شديد الحساسية ومحدد ، كما هو موضح في protocols.io54,55.
11. التطبيع (30 دقيقة)
- استخدم نموذج التطبيع (الملف التكميلي 3) وتركيز الحمض النووي (نانوغرام / ميكرولتر) لكل عينة لحساب حجم العينة المخففة (أو الأنيقة) المطلوبة ل 200 fmol من كل عينة في حجم إجمالي قدره 5 ميكرولتر.
- قم بتسمية أنابيب PCR الجديدة وإضافة أحجام محسوبة من NFW والعينة للحصول على الحمض النووي الطبيعي.
- استخدم الحجم المحسوب للعينات غير المخففة (الأنيقة) إذا كانت هناك حاجة إلى أكثر من 5 ميكرولتر من العينة المخففة للحصول على 200 fmol.
ملاحظة: نقطة توقف اختيارية: في هذه المرحلة ، يمكن تخزين منتج PCR الذي تم تنظيفه عند 4 °C لمدة تصل إلى 1 أسبوع أو وضعه في -20 °C للتخزين على المدى الطويل إذا لزم الأمر.
12. نهاية الإعداد والباركود (1.5 ساعة)
ملاحظة: تفترض الخطوات التالية استخدام كواشف محددة من مجموعات الترميز الشريطي وتسلسل الربط الخاصة بالمسام النانوية (انظر جدول المواد للحصول على التفاصيل). البروتوكول قابل للنقل عبر إصدارات كيميائية مختلفة ، ولكن يجب على المستخدمين الحرص على استخدام مجموعات متوافقة ، وفقا لتعليمات الشركة المصنعة.
- إصلاح النهاية و dA-tailing
- قم بإعداد تفاعل التحضير النهائي لكل عينة مذكورة في الجدول 3. قم بإعداد مزيج رئيسي وفقا لعدد العينات (بالإضافة إلى 10٪ فائض). توخ الحذر عند سحب الكواشف لأن الكواشف لزجة.
- أضف 5 ميكرولتر من المزيج الرئيسي في كل أنبوب من الحمض النووي الطبيعي (5 ميكرولتر). يجب أن يكون إجمالي مزيج التفاعل 10 ميكرولتر. قم بتغيير الأطراف في كل مرة وافتح أنبوبا واحدا فقط في كل مرة.
- احتضان في دورة حرارية في ظل الظروف المذكورة في الجدول 3.
- الترميز الشريطي
- قم باقتباس الرموز الشريطية من مجموعة الترميز الشريطي إلى أنابيب شريط PCR عند 1.25 ميكرولتر / أنبوب ، وسجل الرمز الشريطي المخصص لكل عينة.
- أضف 0.75 ميكرولتر من العينة المعدة نهائيا إلى حصص الباركود المخصصة لها.
- قم بإعداد مزيج رئيسي للربط وفقا لعدد العينات (بالإضافة إلى 10٪ فائض) (الجدول 4).
- أضف 8 ميكرولتر من المزيج الرئيسي للربط إلى العينة المعدة نهائيا + الرموز الشريطية ، مما يعطي تفاعلا إجماليا قدره 10 ميكرولتر.
- احتضان في دورة حرارية باستخدام الشروط المذكورة في الجدول 4.
- تنظيف حبة SPRI وقياس كمية الحمض النووي
- قم بإذابة المخزن المؤقت للشظايا القصيرة (SFB) في RT ، واخلطه عن طريق الدوامة ، وأجهزة الطرد المركزي النبضي ، وضعه على الجليد.
- قم بتجميع جميع العينات المشفرة معا في أنبوب طرد مركزي صغير لوبيند سعة 1.5 مل. حتى لا تجعل حجم التنظيف كبيرا جدا بحيث لا يمكن استخدامه ، قم بتجميع 12-24 عينة (10 ميكرولتر / عينة) ، أو ما يصل إلى 48 عينة (5 ميكرولتر / عينة) ، أو ما يصل إلى 96 عينة (2.5 ميكرولتر / عينة) من كل تفاعل تشفير شريطي أصلي.
- أضف حجم 0.4x من حبات SPRI إلى تجمع الباركود. تخلط برفق (نفض الغبار أو الماصة) وتحتضن في RT لمدة 5 دقائق.
- ضع العينات على مغناطيس حتى تتكبئ الحبيبات ويصبح العنصر الطافي واضحا تماما (~ 2 دقيقة). قم بإزالة وتجاهل المادة الطافية. احرص على عدم إزعاج الخرز.
- يغسل مرتين مع 250 ميكرولتر من SFB.
- قم بإزالة الأنبوب من المغناطيس وأعد تعليق الحبيبات بالكامل في 250 ميكرولتر من SFB. احتضان لمدة 30 ثانية ، نبض الطرد المركزي ، والعودة إلى المغناطيس.
- إزالة طاف وتجاهل.
- كرر الخطوة 12.3.5 لإجراء غسل SFB ثان.
- نبض الطرد المركزي وإزالة أي SFB المتبقية.
- أضف 200 ميكرولتر من 80٪ (RT) إيثانول لاستحمام الحبيبات. قم بإزالة الإيثانول والتخلص منه ، مع الحرص على عدم إزعاج حبيبات الخرزة. جففه في الهواء لمدة 30 ثانية أو حتى تفقد الحبيبات لمعانها.
- أعد التعليق في 22 ميكرولتر من NFW عند RT لمدة 10 دقائق.
- ضعه على المغناطيس ، واتركه ليستقر لمدة ~ 2 دقيقة ، ثم قم بإزالة المحلول بعناية وانقله إلى أنبوب طرد مركزي نظيف سعة 1.5 مل.
- استخدم 1 ميكرولتر للحصول على تركيز الحمض النووي ، كما هو موضح سابقا (الخطوة 10.13).
ملاحظة: نقطة توقف اختيارية: في هذه المرحلة ، يمكن تخزين المكتبة في 4 درجة مئوية لمدة تصل إلى 1 أسبوع أو -20 درجة مئوية للتخزين على المدى الطويل ، ولكن من الأفضل الاستمرار في ربط المحول والتسلسل.
13. التسلسل (48 ساعة كحد أقصى)
- قم بإعداد الكمبيوتر (راجع أيضا أقسام المتطلبات الأساسية 1-4).
- تحقق من وجود مساحة كافية لتخزين البيانات الجديدة (150 جيجابايت على الأقل) ، ويتم نسخ / نقل البيانات من عمليات التشغيل القديمة احتياطيا إلى خادم قبل الحذف ، ويتم تثبيت أحدث إصدار من MinKNOW.
- أخرج خلية التدفق المخزنة من الثلاجة واتركها تصل إلى RT.
- ربط المحول (1 ساعة)
- نبض الطرد المركزي محول مزيج وligase ومكان على الجليد.
- المخزن المؤقت لشطف الذوبان (EB) و SFB ومخزن الربط المؤقت عند RT. امزج عن طريق الدوامة وأجهزة الطرد المركزي النبضي وضعها على الجليد.
- قم بإعداد المزيج الرئيسي لربط المحول (الجدول 5) ، مع الجمع بين الكواشف بالترتيب المحدد في أنبوب ربط منخفض.
ملاحظة: يمكن استخدام بدائل كواشف المزيج الرئيسي لربط المهايئ (الجدول 5) حسب التوافر في المختبر. انظر الملف التكميلي 3 وجدول المواد للحصول على قائمة بالبدائل. استخدم الحساب في ورقة عمل الملف التكميلي 3 للحصول على حجم مكتبة الحمض النووي المكافئ ل 200 fmol. إذا تم حساب أقل من 20 ميكرولتر ، أضف NFW لتكوين ما يصل إلى 20 ميكرولتر. - تخلط عن طريق نفض الغبار لطيف والنبض الطرد المركزي. احتضان في RT لمدة 20 دقيقة.
ملاحظة: أثناء الحضانة ، ابدأ في تحضير خلية التدفق (القسم 13.5).
- نظف باستخدام حبات SPRI (لا تستخدم الإيثانول كما في عمليات التنظيف السابقة).
- أضف حجم 0.4x من حبات SPRI (RT) إلى العينات. احتضن في RT لمدة 10 دقائق ، ونفض برفق بشكل متقطع للمساعدة في الخلط.
- ضعه على المغناطيس حتى تنفصل الحبيبات والمحلول تماما (~ 5 دقائق). إزالة وتجاهل طاف ؛ احرص على عدم إزعاج حبة بيليه.
- يغسل مرتين مع 125 ميكرولتر من SFB.
- أعد تعليق الحبيبات تماما باستخدام 125 ميكرولتر من SFB عن طريق الخلط مع ماصة. اتركيه لاحتضانه لمدة 30 ثانية.
- نبض الطرد المركزي لجمع السائل في قاعدة الأنبوب ووضعه على المغناطيس. إزالة طاف وتجاهل.
- كرر الخطوات من 13.4.4 إلى 13.4.5 لغسل الحبيبات مرة ثانية.
- نبض الطرد المركزي وإزالة SFB الزائدة.
- يعاد التعليق في 15 ميكرولتر من EB واحتضانه لمدة 10 دقائق في RT.
- ارجع إلى المغناطيس لمدة ~ 2 دقيقة ثم انقل المحلول بعناية إلى أنبوب طرد مركزي صغير نظيف سعة 1.5 مل.
- حدد كمية 1 ميكرولتر من المكتبة المستحثة ، كما هو موضح سابقا في الخطوة 10.13
ملاحظة: للحصول على أفضل النتائج ، انتقل مباشرة إلى تسلسل MinION ؛ ومع ذلك ، يمكن تخزين المكتبة النهائية في EB عند 4 °C لمدة تصل إلى 1 أسبوع إذا لزم الأمر.
- قم بتشغيل فحص جودة خلية التدفق.
- قم بتوصيل جهاز التسلسل بجهاز كمبيوتر محمول وافتح برنامج التسلسل.
- حدد نوع خلية التدفق، ثم انقر فوق التحقق من خلية التدفق وبدء الاختبار.
- بمجرد الانتهاء ، سيتم عرض العدد الإجمالي للمسام النشطة (أي القابلة للحياة). يجب أن تحتوي خلية التدفق الجديدة على >800 مسام نشطة ؛ إذا لم يحدث ذلك ، فاتصل بالشركة المصنعة للحصول على بديل.
- تحضير وتحميل خلية التدفق (20 دقيقة)
- قم بإذابة الكواشف التالية في RT ثم ضع المخزن المؤقت للتسلسل ، وحبل التدفق ، ومخزن مؤقت للتدفق ، وخرز التحميل على الجليد.
- دوامة المخزن المؤقت للتسلسل والمخزن المؤقت للتدفق ، وأجهزة الطرد المركزي النبضية ، ووضعها على الجليد.
- نبض الطرد المركزي الحبل دافق وتخلط عن طريق ماصة ؛ ثم ضع على الجليد.
- قم بإعداد مزيج تحضير خلايا التدفق عن طريق إضافة 30 ميكرولتر من حبل التدفق مباشرة إلى أنبوب المخزن المؤقت للتدفق من مجموعة تحضير خلايا التدفق واخلطها عن طريق الماصة.
- امزج حبات التحميل عن طريق السحب مباشرة قبل الاستخدام لأنها تستقر بسرعة.
- في أنبوب جديد ، قم بإعداد التخفيف النهائي للمكتبة للتسلسل ، كما هو مذكور في الجدول 5.
ملاحظة: استخدم الحساب في ورقة عمل الملف التكميلي 3 للحصول على حجم مكتبة الحمض النووي المكافئ ل 50 fmol. إذا تم حساب أقل من 12 ميكرولتر ، أضف EB لتكوين ما يصل إلى 12 ميكرولتر. - اقلب غطاء جهاز التسلسل للخلف وحرك غطاء منفذ التحضير في اتجاه عقارب الساعة بحيث يكون منفذ التحضير مرئيا (الشكل 3)
- قم بإزالة فقاعات الهواء بعناية عن طريق ضبط ماصة P1000 على 200 ميكرولتر ، وأدخل الطرف في منفذ التحضير ، وأدر العجلة حتى يظهر حجم صغير يدخل طرف الماصة (الحد الأقصى للدوران إلى 230 ميكرولتر).
- قم بتحميل 800 ميكرولتر من مزيج تحضير خلايا التدفق في خلية التدفق عبر منفذ التحضير ، مع الحرص على تجنب الفقاعات.
- اتركيه لمدة 5 دقائق.
- ارفع غطاء منفذ العينة برفق وقم بتحميل 200 ميكرولتر من مزيج التحضير في خلية التدفق عبر منفذ التحضير باستخدام ماصة P1000.
- ماصة تمتزج المكتبة لأعلى ولأسفل قبل التحميل ، مما يضمن إعادة تعليق حبات التحميل في المزيج الرئيسي قبل التحميل.
- قم بتحميل 75 ميكرولتر من مزيج المكتبة إلى خلية التدفق عبر منفذ العينة بطريقة قطرة. تأكد من أن كل قطرة تتدفق إلى المنفذ قبل إضافة التالي.
- استبدل غطاء منفذ العينة برفق، مع التأكد من دخول البونغ إلى منفذ العينة.
- أغلق منفذ التحضير واستبدل غطاء جهاز التسلسل.
- تشغيل التسلسل (48 ساعة كحد أقصى)
- قم بتوصيل جهاز التسلسل بالكمبيوتر المحمول وافتح برنامج التسلسل.
- انقر فوق ابدأ ثم انقر فوق بدء التسلسل.
- انقر فوق تجربة جديدة واتبع سير عمل واجهة المستخدم الرسومية (GUI) لبرنامج التسلسل لإعداد معلمات التشغيل.
- اكتب اسم التجربة ونموذج المعرف (على سبيل المثال، rabv_run1)، واختر نوع خلية التدفق من القائمة المنسدلة.
- استمر في اختيار المجموعة واختر مجموعة تسلسل الربط ذات الصلة ومجموعة (مجموعات) الترميز الشريطي الأصلية المستخدمة.
- استمر في تشغيل الخيارات. احتفظ بالإعدادات الافتراضية ، ما لم يكن من المرغوب فيه أن يتوقف التشغيل تلقائيا بعد عدد معين من الساعات (يمكن إيقاف التشغيل يدويا في أي وقت).
- تابع إلى الاتصال الأساسي. اختر تشغيل Basecalling أو إيقاف تشغيله وفقا لموارد الحوسبة (انظر إعداد الكمبيوتر). اختر تحرير الخيارات ضمن الترميز الشريطي وتأكد من تشغيل الرمز الشريطي كلا الطرفين . احفظ وتابع إلى قسم الإخراج.
- اقبل الإعدادات الافتراضية وتابع المراجعة النهائية وتحقق من الإعدادات وسجل التفاصيل في ورقة العمل (الملف التكميلي 3). انقر فوق ابدأ.
ملاحظة: إذا كان يتم إعادة استخدام خلية التدفق ، فاضبط جهد البدء (في القسم المتقدم من خيارات التشغيل) ، كما هو موضح في المخطط في الملف التكميلي 3. - سجل القنوات النشطة الأولية - إذا كان هذا أقل بكثير من فحص مراقبة الجودة (QC) ، فأعد تشغيل برنامج التسلسل. إذا كان لا يزال أقل ، فقم بإعادة تشغيل الكمبيوتر.
- سجل القنوات الأولية في الخصلة مقابل المسام المفردة لإعطاء إشغال تقريبي للمسام. سوف يتقلب هذا الرقم ، لذا أعط تقديرا تقريبيا.
- راقب الجري أثناء تقدمه.
14. الاتصال الأساسي المباشر وغير المتصل
ملاحظة: تفترض هذه التعليمات أن بنية الدليل الموجودة مسبقا والمقدمة في مستودع artic-rabv وأنه تم اتباع القسمين 1 و 3 من المتطلبات الأساسية من البروتوكول.
- على نظام الملفات المحلي الخاص بك ، قم بإنشاء دليل جديد يسمى التحليل ، حيث ستقوم بتخزين جميع مخرجات التحليل الخاصة بك. لمزيد من التنظيم: قم بإنشاء دليل فرعي باسم مشروعك وداخله دليل جديد للتشغيل ، باستخدام نموذج المعرف المقدم إلى MinKNOW ك run_name. قم بذلك في أمر واحد على النحو التالي:
مكدير -P
تحليل/project_name/run_name
ثم انتقل إلى موقعه:
سي دي
المسار / التحليل / project_name / run_name - الاتصال الأساسي المباشر
ملاحظة: لإجراء استدعاء أساسي نانوي المسام في الوقت الفعلي ، تتطلب أجهزة الكمبيوتر المحمولة وحدة معالجة رسومات (GPU) متوافقة مع NVIDIA CUDA. تأكد من تنفيذ التعليمات الخاصة بإعداد الاتصال الأساسي لوحدة معالجة الرسومات باستخدام بروتوكول guppy56.- أثناء إعداد التشغيل، قم بتشغيل الاتصال الأساسي المباشر.
- استخدم RAMPART لمراقبة تغطية التسلسل في الوقت الفعلي ، وفقا للتعليمات أدناه.
- في محطة الكمبيوتر ، قم بتنشيط بيئة artic-rabv conda:
كوندا تفعيل القطب الشمالي رابف - قم بإنشاء دليل جديد لإخراج السور داخل دليل run_name وانتقل إليه:
القرص المضغوط / المسار / التحليل / project_name / run_name
مكدير rampart_output
rampart_output القرص المضغوط - قم بإنشاء ملف .csv الرموز الشريطية لإقران الرموز الشريطية وأسماء العينات. يجب أن يحتوي على سطر واحد لكل رمز شريطي وأن يحدد فقط الرموز الشريطية الموجودة في المكتبة ، مع العناوين "الباركود" و "العينة". اتبع المثال في دليل artic-rabv:
تحليل / example_project / example_run / rampart_output / الباركود.csv - بدء تشغيل RAMPART عن طريق توفير مجلد البروتوكول ذي الصلة والمسار إلى المجلد fastq_pass في إخراج MinKNOW للتشغيل:
rampart - بروتوكول / مسار / متشعب / scheme_name_V1_protocol - مسار أساسي يسمى < إدراج مسار ل Fastqpassfolder> - افتح نافذة متصفح وانتقل إلى localhost:3000 في مربع URL. انتظر حتى يتم استدعاء بيانات كافية قبل ظهور النتائج على الشاشة.
- الاتصال الأساسي دون اتصال (يتم إجراؤه بعد التشغيل)
- إذا لم يتم تعيين basecalling المباشر ، فسيكون الإخراج من MinKNOW عبارة عن بيانات إشارة أولية (ملفات fast5). لن يتمكن المرء من استخدام RAMPART أثناء التشغيل. قم بتحويل ملفات fast5 إلى بيانات أساسية تسمى (ملفات fastq) بعد التشغيل باستخدام guppy (انظر الإعداد في الخطوة 1.1.1 من المتطلبات الأساسية). قم بتشغيل RAMPART بعد التخصيص على البيانات الأساسية.
- قم بتشغيل المتصل الأساسي غوبي:
guppy_basecaller -c dna_r9.4.1_450bps_fast.cfg -i / path / to / reads / fast5_ * -s / path / analysis / project_name / run_name -x auto -r
-c هو ملف التكوين لتحديد نموذج basecalling ، -i هو مسار الإدخال ، -s هو مسار الحفظ ، -x يحدد basecalling بواسطة جهاز GPU (استبعاد في حالة استخدام إصدار الكمبيوتر من guppy) ، و -r يحدد للبحث في ملفات الإدخال بشكل متكرر.
ملاحظة: يمكن تغيير ملف التكوين (.cfg) إلى basecaller عالية الدقة عن طريق استبدال _fast ب _hac ، على الرغم من أن هذا سيستغرق وقتا أطول بكثير.
15. غسل خلايا التدفق
- يمكن غسل خلايا التدفق وإعادة استخدامها لتسلسل مكتبات جديدة إذا كانت المسام لا تزال قابلة للحياة. انظر تعليمات الغسيل في بروتوكول غسل خلايا التدفقONT 57.
16. التحليل والتفسير
- توليد تسلسل الإجماع مع خط أنابيب المعلوماتية الحيوية ARTIC
- اتبع الإرشادات المفصلة في مستودع artic-rabv GitHub40 في المجلد rabv_protocols لإنشاء تسلسلات إجماع من ملفات fast5 الأولية أو ملفات fastq الأساسية.
ملاحظة: راجع خط أنابيب القطب الشمالي - خط الأنابيب الأساسي58 للحصول على مزيد من الإرشادات.
- اتبع الإرشادات المفصلة في مستودع artic-rabv GitHub40 في المجلد rabv_protocols لإنشاء تسلسلات إجماع من ملفات fast5 الأولية أو ملفات fastq الأساسية.
- اختياري: تحليل متوسط عمق القراءة لكل أمبليكون.
- قم بتكييف البرامج النصية المتاحة من مستودع artic-rabv ، بالإشارة إلى الملف التكميلي 1. باختصار ، يتم إنشاء إحصاءات متعمقة باستخدام SAMtools59 والتغطية لكل نيوكليوتيد مرسوم في R.
- تحليل النشوء والتطور باستخدام الغراء
- منRABV_GLUE 42 ، حدد التحليل > التنميط الجيني والتفسير علامة التبويب > إضافة ملفات، وتحديد ملف fasta لتسلسلات الإجماع.
- انقر على إرسال وانتظر. بمجرد اكتمال التحليلات ، سيكون الزر إظهار التحليل متاحا للنقر ، ويظهر تعيينات clade و subclade ، والتغطية لكل جين ، والاختلاف عن التسلسلات المرجعية ، وأقرب قريب.
- يمكن أيضا تحديد التسلسلات السياقية ذات الصلة في بيانات التسلسل > تسلسلات NCBI بواسطة قسم Clade .
- حدد المجموعة المحددة أو انقر فوق فيروس داء (RABV) لرؤية جميع التسلسلات المتاحة.
- تصفية التسلسلات ذات الصلة (على سبيل المثال ، بلد المنشأ).
- قم بتنزيل هذه التسلسلات والبيانات الوصفية المقابلة للتحليل والمقارنة.
- تعيين النسب باستخدام MADDOG41
- اسحب مستودع MADDOG من GitHub للتأكد من أنك تعمل مع أحدث إصدار.
- قم بإنشاء مجلد مهمة داخل مستودع MADDOG المحلي (تم إنشاؤه مسبقا في قسم المتطلبات الأساسية) يسمى اسم التشغيل.
- داخل المجلد ، أضف ملف fasta الذي يحتوي على تسلسلات الإجماع.
- أضف ملف بيانات تعريف إلى المجلد.
ملاحظة: يجب أن يكون هذا الملف ملف csv يحتوي على 4 أعمدة تسمى "المعرف" و "البلد" و "السنة" و "التعيين" ، مع تفصيل معرفات التسلسل وبلد أخذ العينات وسنة جمع العينات ، بينما يجب أن يكون عمود "المهمة" فارغا. - في واجهة سطر الأوامر ، قم بتنشيط بيئة conda: conda قم بتنشيط MADDOG.
- في واجهة سطر الأوامر ، انتقل إلى مجلد مستودع MADDOG.
- في البداية ، قم بإجراء تعيين النسب على التسلسلات للتحقق من أي تشوهات محتملة ولتحديد ما إذا كان تشغيل خطوة تعيين النسب الأطول سيكون مناسبا. لهذا ، اكتب هذا في سطر الأوامر: sh assignment.sh.
- عند المطالبة ، أدخل Y للإشارة إلى أنك قمت بسحب المستودع وتعمل مع أحدث إصدار من MADDOG.
- عند المطالبة ، أدخل اسم المجلد داخل مجلد مستودع MADDOG الذي يحتوي على ملف fasta.
- عند اكتمال تعيين النسب، تحقق من ملف الإخراج في مجلدك. إذا كان الإخراج كما هو متوقع وكان هناك تسلسلات متعددة تم تعيينها لنفس النسب، فقم بتشغيل تعيين النسب.
- في حالة تشغيل تعيين النسب، احذف ملف إخراج المهمة الذي تم إنشاؤه للتو.
- في المحطة الطرفية ، داخل مجلد مستودع MADDOG ، قم بتشغيل الأمر sh designation.sh.
- عند مطالبتك ، أدخل Y للإشارة إلى أنك قمت بسحب المستودع وتعمل مع أحدث إصدار من MADDOG.
- عند المطالبة ، أدخل اسم المجلد داخل مجلد مستودع MADDOG الذي يحتوي على ملف fasta والبيانات الوصفية. ينتج هذا معلومات النسب حول كل تسلسل ، وسلالة من التسلسلات السابقة الجديدة وذات الصلة (من 16.3.6) ، ومعلومات هرمية حول الأنساب ، وتفاصيل السلالات الناشئة المحتملة ومناطق أخذ العينات الناقصة.
ملاحظة: يمكن العثور على التفاصيل الكاملة للبروتوكول والاستخدام والمخرجات في Campbell et al.60. - عند اكتمال التحليل الأولي ، إذا طلب منك أيضا اختبار السلالات الناشئة والعينات غير المتجانسة ، أدخل Y ، إذا كان ذلك مطلوبا. وإلا، فأدخل N.
- إذا طلب منك تأكيد الأنساب التي تم العثور عليها حديثا، فأدخل Y واتبع التعليمات في ملف NEXT_STEPS.eml الناتج. وإلا، فأدخل N.
النتائج
تم استخدام سير عمل العينة إلى التسلسل إلى التفسير ل RABV الموصوف في هذا البروتوكول بنجاح في ظروف مختبرية مختلفة في البلدان الموبوءة ، مثل تنزانيا وكينيا ونيجيريا والفلبين (الشكل 4). تم استخدام البروتوكول على أنواع وظروف مختلفة من العينات (الجدول 6): أنسجة المخ الطازجة والمجمدة ، ومستخلصات cDNA و RNA من أنسجة المخ المنقولة تحت سلسلة التبريد لفترات طويلة ، وبطاقات FTA مع مسحات أنسجة المخ.
يظهر الاتصال الأساسي المباشر باستخدام RAMPART (الشكل 5) إنشاء القراءات في الوقت الفعلي تقريبا والنسبة المئوية للتغطية لكل عينة. هذا مفيد بشكل خاص في تحديد وقت إيقاف التشغيل وحفظ خلية التدفق لإعادة استخدامها. لوحظ اختلاف في وقت التشغيل ، حيث انتهى بعضها في 2 ساعة ، بينما قد يستغرق البعض الآخر أكثر من 12 ساعة للوصول إلى عمق تغطية كاف (x100). يمكننا أيضا عرض المناطق ذات التضخيم الضعيف ؛ على سبيل المثال ، يوضح الشكل 6 لقطة من تشغيل تسلسل واحد حيث تظهر ملفات تعريف التغطية بعض الأمبليكونات ذات التضخيم المنخفض جدا ، مما يشير إلى وجود مواد أولية يحتمل أن تكون إشكالية. من خلال التحقيق في هذه المناطق ذات التضخيم الضعيف بشكل أكثر شمولا ، تمكنا من تحديد عدم تطابق التمهيدي ، مما سيمكننا من إعادة تصميم وتحسين البرايمات الفردية. أظهرت بعض مخططات التمهيدي عدم تطابق أكثر من غيرها. ويلاحظ ذلك في المخطط التمهيدي لشرق أفريقيا، مقارنة بالفلبين، تمشيا مع التنوع المستهدف، حيث يهدف مخطط شرق أفريقيا إلى التقاط تنوع أوسع بكثير.
تم استخدام RABV-GLUE42 ، وهو مورد للأغراض العامة لإدارة بيانات جينوم RABV ، و MADDOG60 ، وهو نظام تصنيف وتسميات النسب ، لتجميع وتفسير تسلسلات RABV الناتجة. ويبين الجدول 7 الكتل الرئيسية والثانوية المتداولة في كل بلد باستخدام RABV-GLUE. يظهر أيضا تصنيف أعلى دقة للأنساب المحلية بعد تعيين MADDOG.
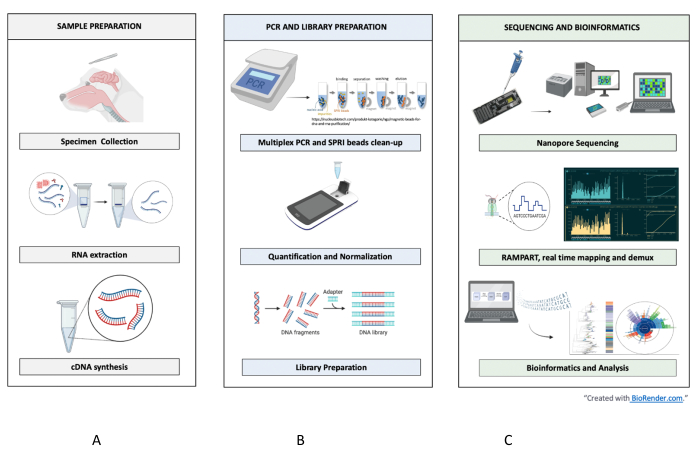
الشكل 1: سير عمل العينة إلى التسلسل إلى التفسير ل RABV. يتم عرض الخطوات الملخصة ل (أ) إعداد العينات ، (ب) PCR وإعداد المكتبة ، و (ج) التسلسل والمعلوماتية الحيوية حتى التحليل والتفسير. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
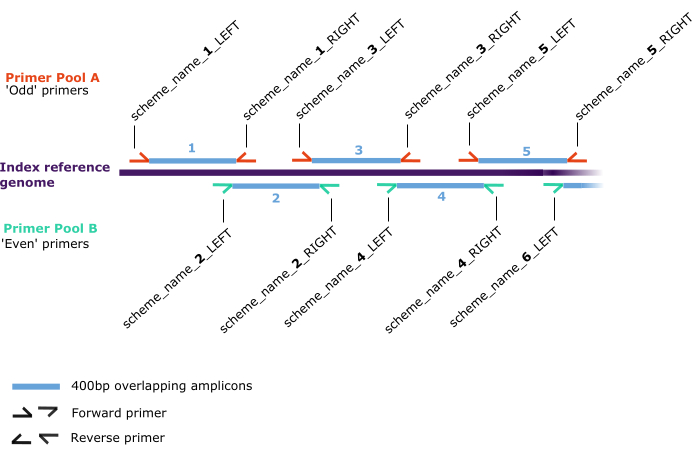
الشكل 2: مخطط التمهيدي التخطيطي. مواضع التلدين على طول "الجينوم المرجعي للفهرس" (أرجواني غامق) لأزواج من البادئات الأمامية والخلفية (نصف الأسهم) ، والتي يتم تعيينها في مجموعتين منفصلتين: A (أحمر) و B (أخضر). تولد أزواج التمهيدي 400 bp amplicons متداخلة (زرقاء) والتي يتم ترقيمها بالتتابع على طول الجينوم المرجعي للفهرس بالتنسيق "scheme_name_X_DIRECTION" ، حيث "X" هو رقم يشير إلى amplicon الناتج عن التمهيدي و "DIRECTION" إما "LEFT" أو "RIGHT" ، يصف الأمام أو الخلف على التوالي. تحدد القيم الفردية أو الزوجية ل "X" التجمع (A أو B ، على التوالي). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
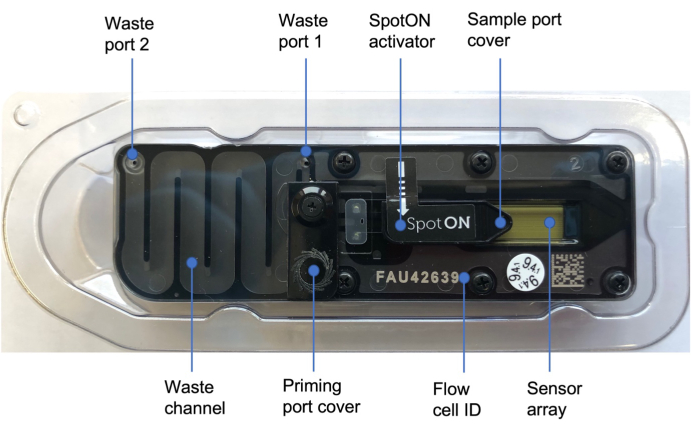
الشكل 3: خلية تدفق النانوبور48. توضح الملصقات الزرقاء الأجزاء المختلفة لخلية التدفق ، بما في ذلك غطاء منفذ التحضير الذي يغطي منفذ التحضير حيث يتم إضافة محلول التحضير ، وغطاء منفذ عينة SpotON الذي يغطي منفذ العينة حيث تتم إضافة العينة بطريقة قطرة ، ومنافذ النفايات 1 و 2 ، ومعرف خلية التدفق. يرجى النقر هنا لعرض نسخة أكبر من هذا الشكل.
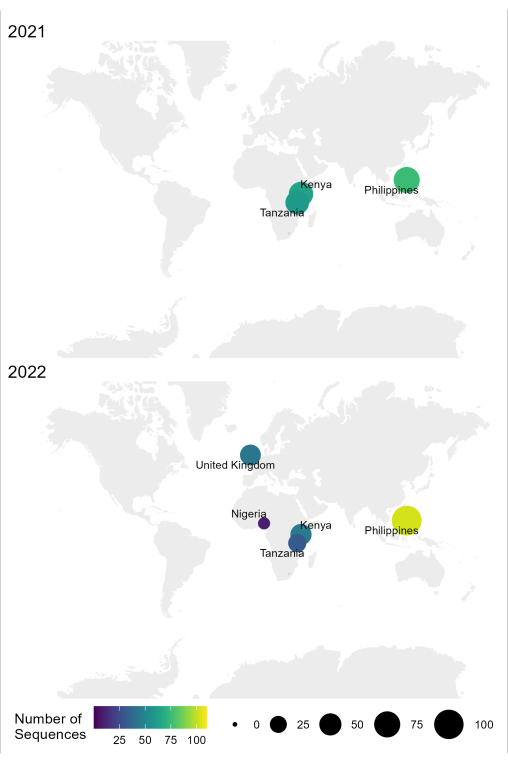
الشكل 4: خريطة توضح الموقع الذي تم فيه إجراء تسلسل RABV باستخدام سير العمل الأمثل في عامي 2021 و 2022. يتوافق حجم الفقاعة ولونها مع عدد التسلسلات لكل موقع ، حيث يكون الأصغر والأغمق أقل ، بينما الأكبر والأفتح أكثر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
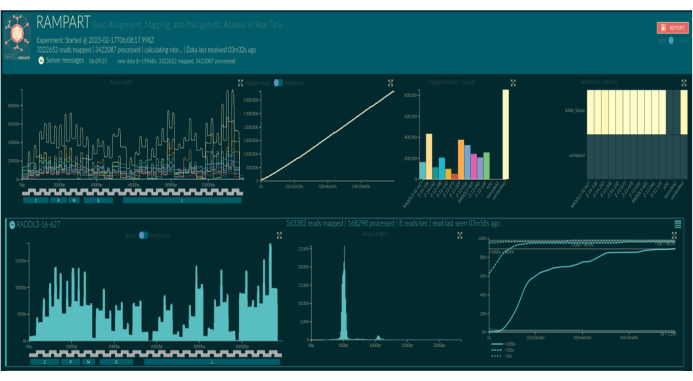
الشكل 5: لقطة شاشة لتصور RAMPART في متصفح الويب. يتم استبدال أسماء الباركود بأسماء العينات وفقا لإعداد المعلوماتية الحيوية. تعرض اللوحات الثلاث الأولى مخططات موجزة للتشغيل بأكمله: عمق تغطية القراءات المعينة لكل رمز شريطي لكل موضع نيوكليوتيد على الجينوم المرجعي للفهرس (أعلى اليسار ، ملون بالرمز الشريطي) ، والقراءات المعينة المجمعة من جميع الرموز الشريطية بمرور الوقت (أعلى الوسط) ، والقراءات المعينة لكل رمز شريطي (أعلى اليمين ، ملونة بالرمز الشريطي). تظهر اللوحات السفلية صفوفا من قطع الأراضي لكل رمز شريطي. من اليسار إلى اليمين: عمق تغطية القراءات المعينة لكل موضع نيوكليوتيد على الجينوم المرجعي للفهرس (يسار) ، وتوزيع طول القراءات المعينة (وسط) ، ونسبة مواضع النوكليوتيدات على الجينوم المرجعي للفهرس التي حصلت على تغطية 10x و 100x و 1000x للقراءات المعينة بمرور الوقت (يمين). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
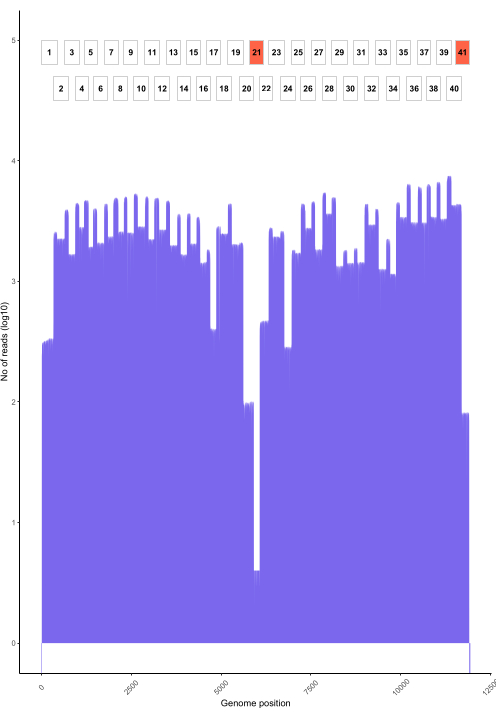
الشكل 6: مثال على التغطية عبر الجينوم لعينة فيروس داء من الفلبين متسلسلة باستخدام البروتوكول. تظهر تغطية القراءة في كل موضع نيوكليوتيدات في الجينوم ، جنبا إلى جنب مع موضع الأمبليكونات المتداخلة (1-41) المستخدمة لإنشاء المكتبة. تتوافق المسامير في عمق التغطية مع مناطق تداخل amplicon. تتوافق Amplicons ذات عمق التغطية المنخفض مع مناطق تداخل amplicon. يتم تمييز Amplicons ذات عمق التغطية المنخفض باللون الأحمر للإشارة إلى المناطق الإشكالية التي قد تتطلب التحسين. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الجدول 1: المزيج الرئيسي وظروف التدوير الحراري لإعداد cDNA. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 2: ظروف المزيج الرئيسي والدورة الحرارية لتفاعل البوليميراز المتسلسل المتعدد. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 3: المزيج الرئيسي وظروف التدوير الحراري لتفاعل الإعدادية النهائية. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 4: المزيج الرئيسي وظروف التدوير الحراري للترميز الشريطي. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 5: المزيج الرئيسي لربط المحول والتخفيف النهائي للمكتبة للتسلسل. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 6: عدد تسلسلات الجينوم الكامل لفيروس داء المتولدة ونوع العينات المستخدمة في مختلف البلدان باستخدام سير عمل العينة إلى التسلسل إلى التفسير. الرجاء الضغط هنا لتنزيل هذا الجدول.
الجدول 7: تعيينات clade الرئيسية والثانوية من RABV-GLUE وتعيينات النسب من MADDOG للتسلسلات التي تم إنشاؤها باستخدام سير العمل. الرجاء الضغط هنا لتنزيل هذا الجدول.
الملف التكميلي 1: تصميم مخطط التمهيدي وتحسينه ، وتحليل عمق قراءة amplicon. الرجاء الضغط هنا لتنزيل هذا الملف.
الملف التكميلي 2: الإعداد الحسابي الرجاء النقر هنا لتنزيل هذا الملف.
الملف التكميلي 3: ورقة عمل بروتوكول RABV WGS الرجاء النقر هنا لتنزيل هذا الملف.
Discussion
تم تطوير سير عمل تسلسل الجينوم الكامل RABV ، القائم على المسام النانوية ، بواسطة Brunker et al.61 ، باستخدام موارد من شبكة ARTIC46. هنا ، نقدم سير عمل محدثا ، مع خطوات كاملة من عينة إلى تسلسل إلى تفسير. يفصل سير العمل إعداد عينات أنسجة المخ لتسلسل الجينوم الكامل ، ويقدم خط أنابيب المعلوماتية الحيوية لمعالجة القراءات وإنشاء تسلسلات إجماع ، ويسلط الضوء على أداتين خاصتين بداء لأتمتة تعيين النسب وتحديد سياق التطور. يوفر سير العمل المحدث أيضا تعليمات شاملة لإعداد مساحات العمل الحسابية والمختبرية المناسبة ، مع اعتبارات للتنفيذ في سياقات مختلفة (بما في ذلك الإعدادات منخفضة الموارد). لقد أثبتنا التنفيذ الناجح لسير العمل في كل من الإعدادات الأكاديمية والبحثية في أربعة بلدان منخفضة ومتوسطة الدخل مستوطنة في RABV مع عدم وجود قدرة مراقبة جينومية أو محدودة. أثبت سير العمل مرونته للتطبيق عبر إعدادات متنوعة ، ومفهومه من قبل المستخدمين ذوي الخبرات المختلفة.
سير العمل هذا لتسلسل RABV هو البروتوكول الأكثر شمولا المتاح للجمهور (يغطي خطوات العينة إلى التسلسل إلى التفسير) وتم تكييفه خصيصا لتقليل تكاليف بدء التشغيل والتشغيل. يتم تقليل الوقت والتكلفة اللازمين لإعداد المكتبة وتسلسلها باستخدام تقنية المسام النانوية بشكل كبير مقارنة بالمنصات الأخرى ، مثل Illumina61 ، وتعمل التطورات التكنولوجية المستمرة على تحسين جودة التسلسل ودقته لتكون قابلة للمقارنة مع Illumina62.
تم تصميم هذا البروتوكول ليكون مرنا في سياقات متنوعة منخفضة الموارد. من خلال الرجوع إلى إرشادات استكشاف الأخطاء وإصلاحها والتعديلات المقدمة جنبا إلى جنب مع البروتوكول الأساسي ، يتم دعم المستخدمين لتكييف سير العمل مع احتياجاتهم. تشكل إضافة أدوات المعلوماتية الحيوية سهلة الاستخدام إلى سير العمل تطورا كبيرا للبروتوكول الأصلي ، مما يوفر طرقا سريعة وموحدة يمكن تطبيقها من قبل المستخدمين الذين لديهم الحد الأدنى من الخبرة السابقة في المعلوماتية الحيوية لتفسير بيانات التسلسل في السياقات المحلية. غالبا ما تكون القدرة على القيام بذلك في الموقع محدودة بسبب الحاجة إلى مهارات محددة في البرمجة والتطور ، والتي تتطلب استثمارا مكثفا وطويل الأجل في التدريب على المهارات. في حين أن مجموعة المهارات هذه مهمة لتفسير بيانات التسلسل بدقة ، فإن أدوات الترجمة الفورية الأساسية والتي يمكن الوصول إليها مرغوبة بنفس القدر من أجل تمكين "أبطال التسلسل" المحليين ، الذين قد تكون خبرتهم الأساسية قائمة على المختبر الرطب ، مما يمكنهم من تفسير بياناتهم وتولي ملكيتها.
نظرا لأن البروتوكول قد تم تنفيذه لعدد من السنوات في العديد من البلدان ، يمكننا الآن تقديم إرشادات حول كيفية تحسين مخططات التمهيدي المتعدد لتحسين التغطية والتعامل مع التنوع المتراكم. كما بذلت جهود لمساعدة المستعملين على تحسين فعالية التكلفة أو السماح بسهولة الشراء في منطقة معينة، وهو ما يشكل عادة تحديا لاستدامة النهجالجزيئية(63). على سبيل المثال ، في إفريقيا (تنزانيا وكينيا ونيجيريا) ، اخترنا المزيج الرئيسي للربط الحاد / TA في خطوة ربط المحول ، والذي كان متاحا بسهولة أكبر من الموردين المحليين وبديلا أرخص لكواشف الربط الأخرى.
من التجربة ، هناك عدة طرق لتقليل التكلفة لكل عينة ولكل تشغيل. يمكن أن يؤدي تقليل عدد العينات لكل عملية (على سبيل المثال ، من 24 إلى 12 عينة) إلى إطالة عمر خلايا التدفق على مدار عمليات تشغيل متعددة ، في حين أن زيادة عدد العينات لكل تشغيل يزيد من الوقت والكواشف. في أيدينا ، تمكنا من غسل وإعادة استخدام خلايا التدفق لواحد من كل ثلاث عمليات تسلسل ، مما مكن من تسلسل 55 عينة إضافية. يبدو أن غسل خلية التدفق مباشرة بعد الاستخدام ، أو إذا لم يكن ذلك ممكنا ، إزالة سائل النفايات من قناة النفايات بعد كل تشغيل ، يحافظ على عدد المسام المتاحة للتشغيل الثاني. مع الأخذ في الاعتبار العدد الأولي للمسام المتاحة في خلية التدفق ، يمكن أيضا تحسين تشغيل واحد لتخطيط عدد العينات التي سيتم تشغيلها في خلية تدفق معينة.
على الرغم من أن سير العمل يهدف إلى أن يكون شاملا قدر الإمكان ، مع إضافة إرشادات مفصلة وموارد إرشادية ، إلا أن الإجراء لا يزال معقدا ويمكن أن يكون شاقا بالنسبة للمستخدم الجديد. يتم تشجيع المستخدم على طلب التدريب والدعم الشخصي ، من الناحية المثالية محليا ، أو بدلا من ذلك من خلال المتعاونين الخارجيين. ففي الفلبين على سبيل المثال، أدى مشروع لبناء القدرات داخل المختبرات الإقليمية للترصد الجينومي لفيروس كورونا-سارس-2 باستخدام ONT إلى تطوير الكفاءات الأساسية بين أخصائيي تشخيص الرعاية الصحية التي يمكن نقلها بسهولة إلى تسلسل فيروس التهاب الكبد الوبائي. قد يكون من الصعب إتقان الخطوات المهمة ، مثل تنظيف حبة SPRI ، دون تدريب عملي ، ويمكن أن يؤدي التنظيف غير الفعال إلى إتلاف خلية التدفق وتعريض الجري للخطر. يعد تلوث العينة دائما مصدر قلق كبير عند معالجة الأمبليكونات في المختبر وقد يكون من الصعب التخلص منها. على وجه الخصوص ، من الصعب للغاية اكتشاف التلوث المتبادل بين العينات أثناء المعلوماتية الحيوية بعد التشغيل. تعتبر التقنيات والممارسات المختبرية الجيدة ، مثل الحفاظ على أسطح العمل النظيفة ، وفصل مناطق ما قبل وبعد تفاعل البوليميراز المتسلسل ، ودمج الضوابط السلبية ، ضرورية لضمان مراقبة الجودة. تعد الوتيرة السريعة لتطورات تسلسل المسام النانوية ميزة وعيوبا للمراقبة الجينومية الروتينية ل RABV. تعمل التحسينات المستمرة على دقة المسام النانوية وإمكانية الوصول إليها وذخيرة البروتوكول على توسيع وتحسين نطاق تطبيقها. ومع ذلك ، فإن نفس التطورات تجعل من الصعب الحفاظ على إجراءات التشغيل القياسية وخطوط أنابيب المعلوماتية الحيوية. في هذا البروتوكول ، نقدم وثيقة تساعد في الانتقال من مجموعات إعداد مكتبة المسام النانوية القديمة إلى الحالية (جدول المواد).
ومن العوائق الشائعة أمام التسلسل في البلدان المنخفضة والمتوسطة الدخل إمكانية الوصول، بما في ذلك ليس فقط التكلفة ولكن أيضا القدرة على شراء المواد الاستهلاكية في الوقت المناسب (ولا سيما كواشف التسلسل، وهي جديدة نسبيا لفرق المشتريات والموردين) والموارد الحسابية، فضلا عن مجرد الوصول إلى الطاقة المستقرة والإنترنت. يساعد استخدام تقنية تسلسل المسام النانوية المحمولة كأساس لسير العمل هذا في العديد من مشكلات إمكانية الوصول هذه ، وقد أثبتنا استخدام بروتوكولنا عبر مجموعة من الإعدادات ، وإجراء البروتوكول الكامل والتحليل داخل البلد. من المسلم به أن شراء المعدات وتسلسل المواد الاستهلاكية في الوقت المناسب لا يزال يمثل تحديا ، وفي كثير من الحالات ، اضطررنا إلى حمل أو شحن الكواشف من المملكة المتحدة. ومع ذلك ، في بعض المناطق ، تمكنا من الاعتماد كليا على طرق الإمداد المحلية للكواشف ، مستفيدين من الاستثمار في تسلسل SARS-CoV-2 (على سبيل المثال ، الفلبين) الذي بسط عمليات الشراء وبدأ في تطبيع تطبيق علم الجينوم الممرض.
يتم تقليل الحاجة إلى اتصال إنترنت مستقر عن طريق عمليات التثبيت لمرة واحدة فقط ؛ على سبيل المثال ، لا تتطلب مستودعات GitHub وتنزيل البرامج وتسلسل المسام النانوية نفسها سوى الوصول إلى الإنترنت لبدء التشغيل (وليس طوال الوقت) أو يمكن إجراؤها في وضع عدم الاتصال تماما بموافقة الشركة. في حالة توفر بيانات الجوال، يمكن استخدام الهاتف كنقطة اتصال للكمبيوتر المحمول لبدء تشغيل التسلسل، قبل قطع الاتصال طوال مدة التشغيل. عند معالجة العينات بشكل روتيني ، يمكن أن تنمو متطلبات تخزين البيانات بسرعة ، ومن الناحية المثالية سيتم تخزين البيانات على الخادم. خلاف ذلك ، فإن محركات الأقراص الصلبة ذات الحالة الصلبة (SSD) رخيصة نسبيا للمصدر.
وبينما ندرك أنه لا تزال هناك حواجز أمام المراقبة الجينومية في البلدان المنخفضة والمتوسطة الدخل، فإن زيادة الاستثمار في بناء إمكانية الوصول إلى علم الجينوم والخبرة (على سبيل المثال، مبادرة علم الجينوم الممرض في أفريقيا [Africa PGI])64 تشير إلى أن هذا الوضع سيتحسن. يعد الترصد الجينومي أمرا بالغ الأهمية للتأهب للجوائح6 ، ويمكن بناء القدرات من خلال روتين المراقبة الجينومية لمسببات الأمراض المتوطنة مثل RABV. يجب أن تكون التفاوتات العالمية في قدرات التسلسل التي تم تسليط الضوء عليها خلال جائحة SARS-CoV-2 محركا للتغيير التحفيزي لمعالجة هذه التفاوتات الهيكلية.
إن سير العمل هذا من العينة إلى التسلسل إلى التفسير ل RABV ، بما في ذلك أدوات المعلوماتية الحيوية التي يمكن الوصول إليها ، لديه القدرة على استخدامه لتوجيه تدابير المكافحة التي تستهدف هدف القضاء على الوفيات البشرية الناجمة عن داء بوساطة بحلول عام 2030 ، وفي نهاية المطاف للقضاء على متغيرات RABV. وإلى جانب البيانات الوصفية ذات الصلة، تسهل البيانات الجينومية الناتجة عن هذا البروتوكول التوصيف السريع لفيروس التهاب الكبد الوبائي أثناء تحريات الفاشية وفي تحديد السلالات المتداولة في بلد أو منطقة60،61،65. نوضح خط الأنابيب الخاص بنا في الغالب باستخدام أمثلة من داء بوساطة. ومع ذلك ، فإن سير العمل ينطبق مباشرة على داء في الحياة البرية. تقلل قابلية النقل هذه والتكلفة المنخفضة من التحديات في جعل التسلسل الروتيني متاحا بسهولة ، ليس فقط لداء ولكن أيضا لمسببات الأمراضالأخرى 46،66،67 ، لتحسين إدارة المرض ومكافحته.
Disclosures
ليس لدى المؤلفين ما يكشفون عنه.
Acknowledgements
تم دعم هذا العمل من قبل Wellcome [207569 / Z / 17 / Z ، 224670 / Z / 21 / Z] ، وتمويل نيوتن من مجلس البحوث الطبية [MR / R025649 / 1] ووزارة العلوم والتكنولوجيا الفلبينية (DOST) ، والجهد العالمي للبحث والابتكار في المملكة المتحدة بشأن COVID-19 [MR / V035444 / 1] ، وصندوق الدعم الاستراتيجي المؤسسي بجامعة غلاسكو [204820] ، وجائزة الباحث الجديد لمجلس البحوث الطبية (KB) [MR / X002047 / 1] ، وصندوق تنمية الشراكة الدولية ، ومنحة المجلس الثقافي البريطاني في الفلبين (CB) ، والمعهد الوطني للبحوث الصحية [17/63/82] منحة GemVi (GJ) ، ومنح جامعة غلاسكو من MVLS DTP (KC) [125638-06] ، EPSRC DTP (RD) [EP / T517896 / 1] ، و Wellcome IIB DTP (HF) [218518 / Z / 19 / Z]. نحن ممتنون للزملاء والمتعاونين الذين دعموا هذا العمل: دانيال سترايكر ، أليس بروس ، إليزابيث ميراندا ، DVM ، داريا مانالو ، DVM ، ثومبي موانجي ، كينيدي لوشاسي ، تشارلز كايوكي ، جود كارلو بوليفار ، جيرومير بوندوك ، إستفين بالبين ، رونيل تونغوهان ، أجاثا أوكاندي ، ديفيس كوتشاكا ، مومبوا موتونغا ، لويتيكو سيكانا ، وآنا زوبرينا.
Materials
| Name | Company | Catalog Number | Comments |
| Brand name | |||
| Software | |||
| Sequencing software (MinKnow) | Oxford Nanopore Technologies | https://community.nanoporetech.com/downloads | |
| Bioinformatics tool kit (Guppy) | Oxford Nanopore Technologies | https://community.nanoporetech.com/docs/prepare/library_prep_protocols/Guppy-protocol/v/gpb_2003_v1_revao_14dec2018 | |
| Equipment | |||
| Thermal cycler (miniPCR™ mini16 thermal cycler) | Cambio | MP-QP-1016-01 | |
| Homogenizer (Precellys Evolution Touch Homogenizer) | Bertin Instruments | EQ02520-300 | |
| Cold Racks (0.2-0.5mL) (PCR Mini-cooler with transparent lid) | BRAND | 781260 | |
| Pipettor | |||
| (Pipetman L Fixed F1000L, 1000 uL) | Gilson | SKU: FA10030 | |
| (Pipetman L Fixed F100L, 100 uL) | Gilson | SKU: FA10024 | |
| (Pipetman L Fixed F10L, 10 uL) | Gilson | SKU: FA10020 | |
| (Pipetman L Fixed F1L, 1 uL) | Gilson | SKU: FA10025 | |
| (Pipetman L Fixed F20L, 20 uL) | Gilson | SKU: FA10021 | |
| (Pipetman L Fixed F250L, 250 uL) | Gilson | SKU: FA10026 | |
| Fluorometer (Qubit 4 Fluorometer) | Thermofisher scientific/Fisher scientific | Q33238 | |
| Laptop (Any brand with ~2 GB of drive space, minimum of 512 GB storage space, msi installer [GPU]) | |||
| Microcentrifuge (Refrigerated centrifuge) | Thermofisher scientific/Fisher scientific | 75004081 | |
| Vortex mixer (Basic vortex mixer) | Thermofisher scientific/Fisher scientific | 88882011 | |
| Magnetic rack (DynaMag -2 Magnet) | Thermofisher scientific/Fisher scientific | 12321D | |
| Sequencing device (MinION) | Oxford Nanopore Technologies | MinION Mk1B | |
| RNA Extraction | |||
| RNA extraction kit (Qiagen RNEasy Mini Kit 250) | Qiagen | 74106 | |
| RNA stabilizing reagents | |||
| (RNA later) | Invitrogen | AM7020 | |
| (DNA/RNA Shield) | Zymo Research | R1100-50 | |
| PCR | |||
| Nuclease-free Water (Nuclease-free Water [not DEPC-treated]) | Thermofisher scientific/Fisher scientific | AM9937 | |
| Master mix for first strand cDNA synthesis (LunaScript RT SuperMix Kit) | New England Biolabs | E3010S | |
| DNA amplification master mix (Q5® Hot Start High-Fidelity 2X Master Mix [NEB]) | New England Biolabs | M0494L | |
| Primer (Scheme) (Custom DNA oligos) | Invitrogen | ||
| SPRI Bead Clean-up | |||
| SPRI beads (Aline Biosciences PCR Clean DX ) | Cambio | AL-AC1003-50 | |
| Ethanol, Pure Absolute, >99.8% (GC) [Riedel-De Haen] | Merck | 818760 | |
| Short Fragment buffer (SFB expansion pack) | Oxford Nanopore Technologies | EXP-SFB001 | |
| DNA Quantification | |||
| DNA quantification kit (Qubit® dsDNA HS Assay Kit) | Thermofisher scientific/Fisher scientific | Q32854 | |
| DNA quantification assay tubes (Qubit™ Assay Tubes) | Thermofisher scientific/Fisher scientific | Q32856 | |
| End Prep and barcoding (Qubit™ Assay Tubes) | |||
| End Prep master mix (NEBNext Ultra End Repair/dA-Tailing Module) | New England Biolabs | E7546L | |
| Barcoding kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 | |
| (Native Barcoding Expansion 1-12) | EXP-NBD104 | ||
| (Native Barcoding Expansion 13-24) | EXP-NBD114 | ||
| (Native Barcoding Expansion 96) | EXP-NBD196 | ||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 | |
| (not compatible) | (not compatible) | ||
| (Native Barcoding Kit 24 V14) | SQK-NBD114.24 | ||
| (Native Barcoding Kit 96 V14) | SQK-NBD114.96 | ||
| Ligation mastermix (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter Ligation | |||
| Adapter ligation master mix | |||
| (NEBNext Quick Ligation Module) | New England Biolabs | E6056S | |
| (NEBNext Ultra II Ligation Module) | New England Biolabs | E7595S | |
| (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter mix | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-AMII001 | |
| (Adapter Mix II [AMII]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-NBA114 | |
| (Native adapter [NA]) | |||
| Sequencing | |||
| Flowcell priming kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-FLP002 | |
| (Flush Buffer [FB]) | |||
| (Flush Tether [FT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-FLP004 | |
| (Flow Cell Flush [FCF]) | |||
| (Flow Cell Tether [FCT]) | |||
| Ligation Sequencing Kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 SQK-LSK109 | |
| Adapter Mix (Adapter Mix [AMX]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SQB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LB]) | |||
| Sequencing Tether (Sequencing Tether [SQT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 SQK-LSK114 | |
| Adapter Mix (Ligation Adapter [LA]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LIB]) | |||
| Sequencing Tether (Flow Cell Tether [FCT]) | |||
| Library solution (Library solution [LIS]) | |||
| Flush buffer (Flow Cell Flush [FCF]) | |||
| Flow Cell | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 FLO-MIN106D | |
| (Flow Cell [R9.4.1]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 FLO-MIN114 | |
| (Flow Cell [R10.4.1]) | |||
| Flow Cell wash | |||
| Flowcell wash kit (Flow cell wash kit) | Oxford Nanopore Technologies | EXP-WSH004 | |
| Consummables | |||
| Surface decontaminant | |||
| (DNA Away Surface Decontaminant, Squeeze Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7010PK | |
| (RNase Away Surface Decontaminant, Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7002PK | |
| PCR 8-Tube Strip 0.2ml, individual cap (PCR 8-Tube Strip 0.2ml, with Individual attached Flat Caps, Sterile, DNAse/RNAse, Pyrogen Free,Natural [Greiner]) | Greiner | 608281 | |
| PCR Tube 0.2ml (PCR Tube 0.2ml, Natural [Domed Cap] Bagged in 500s, Non-Sterile [Greiner]) | Greiner | 671201 | |
| 1000µL Filter Tips (500) (Stacked 1000µL Filter Tips [500]) | Thermofisher scientific/Fisher scientific | 11977724 | |
| 100µL Filter Tips (1000) | Thermofisher scientific/Fisher scientific | 11947724 | |
| 10µL Filter Tips (1000) (Stacked 100µL Filter Tips [1000]) | Thermofisher scientific/Fisher scientific | 11907724 | |
| Reinforced tubes tubes (2ml) with screw caps and o-rings (Fisherbrand™ Bulk tubes) | Thermofisher scientific/Fisher scientific | 15545809 | |
| Microcentrifuge tube (1.5ml) (1.5 ml Eppendorf Tubes [500]) | Eppendorf | 1229888 | |
| DNA LoBind Tubes (1.5ml) (DNA LoBind Tubes) | Thermofisher scientific/Fisher scientific | 10051232 | |
| Cryobabies labels | |||
| Gloves (S/M/L) | |||
| Paper towel |
References
- Rupprecht, C. E. Rhabdoviruses: rabies virus. Medical Microbiology. , University of Texas Medical Branch. Galveston, TX. (1996).
- Rabies. World Health Organization. , Available from: https://www.who.int/news-room/fact-sheets/detail/rabies (2023).
- Health Organization, W. orld WHO Expert Consultation on Rabies: WHO TRS N°1012. World Health Organization. , Available from: https://www.who.int/publications-detail-redirect/WHO-TRS-1012 (2018).
- Benavides, J. A., et al. Defining new pathways to manage the ongoing emergence of bat rabies in Latin America. Viruses. 12 (9), 1002(2020).
- Hampson, K. Estimating the global burden of endemic canine rabies. PLoS Neglected Tropical Diseases. 9 (4), e0003709(2015).
- Global genomic surveillance strategy for pathogens with pandemic and epidemic potential, 2022-2032. World Health Organization. , Available from: https://www.who.int/publications-detail-redirect/978924004679 (2022).
- Tsai, K. J., et al. Emergence of a sylvatic enzootic formosan ferret badger-associated rabies in Taiwan and the geographical separation of two phylogenetic groups of rabies viruses. Veterinary Microbiology. 182, 28-34 (2016).
- Chiou, H. -Y., et al. Molecular characterization of cryptically circulating rabies virus from ferret badgers, Taiwan. Emerging Infectious Diseases. 20 (5), 790-798 (2014).
- Sabeta, C. T., Mansfield, K. L., McElhinney, L. M., Fooks, A. R., Nel, L. H. Molecular epidemiology of rabies in bat-eared foxes (Otocyon megalotis) in South Africa. Virus Research. 129 (1), 1-10 (2007).
- Scott, T. P. Complete genome and molecular epidemiological data infer the maintenance of rabies among kudu (Tragelaphus strepsiceros) in Namibia. PLoS One. 8 (3), e58739(2013).
- Lembo, T., et al. Exploring reservoir dynamics: a case study of rabies in the Serengeti ecosystem. The Journal of Applied Ecology. 45 (4), 1246-1257 (2008).
- Coetzee, P., Nel, L. H. Emerging epidemic dog rabies in coastal South Africa: a molecular epidemiological analysis. Virus Research. 126 (1-2), 186-195 (2007).
- Ou de Munnink, B. B. First molecular analysis of rabies virus in Qatar and clinical cases imported into Qatar, a case report. International Journal of Infectious Diseases. 96, 323-326 (2020).
- Smith, J., et al. Case report: Rapid ante-mortem diagnosis of a human case of rabies imported into the UK from the Philippines. Journal of Medical Virology. 69, 150-155 (2003).
- Mahardika, G. N. K., et al. Phylogenetic analysis and victim contact tracing of rabies virus from humans and dogs in Bali, Indonesia. Epidemiology and Infection. 142 (6), 1146-1154 (2014).
- Tohma, K., et al. Molecular and mathematical modeling analyses of inter-island transmission of rabies into a previously rabies-free island in the Philippines. Infection, Genetics and Evolution. 38, 22-28 (2016).
- Tohma, K., et al. Phylogeographic analysis of rabies viruses in the Philippines. Infection, Genetics and Evolution. 23, 86-94 (2014).
- Saito, M., et al. Genetic diversity and geographic distribution of genetically distinct rabies viruses in the Philippines. PLoS Neglected Tropical Diseases. 7 (4), e2144(2013).
- Biek, R., Henderson, J. C., Waller, L. A., Rupprecht, C. E., Real, L. A. A high-resolution genetic signature of demographic and spatial expansion in epizootic rabies virus. Proceedings of the National Academy of Sciences. 104 (19), 7993-7998 (2007).
- Reddy, G. B., et al. Molecular characterization of Indian rabies virus isolates by partial sequencing of nucleoprotein (N) and phosphoprotein (P) genes. Virus Genes. 43, 13-17 (2011).
- David, D., Dveres, N., Yakobson, B. A., Davidson, I. Emergence of dog rabies in the northern region of Israel. Epidemiology and Infection. 137 (4), 544-548 (2009).
- Benjathummarak, S. Molecular genetic characterization of rabies virus glycoprotein gene sequences from rabid dogs in Bangkok and neighboring provinces in Thailand, 2013-2014. Archives of Virology. 161 (5), 1261-1271 (2016).
- Denduangboripant, J., et al. Transmission dynamics of rabies virus in Thailand: implications for disease control. BMC Infectious Diseases. 5, 52(2005).
- Talbi, C., et al. Phylodynamics and human-mediated dispersal of a zoonotic virus. PLoS Pathogens. 6 (10), e1001166(2010).
- Bourhy, H., et al. Revealing the micro-scale signature of endemic zoonotic disease transmission in an African urban setting. PLoS Pathogens. 12 (4), e1005525(2016).
- Zinsstag, J., et al. Vaccination of dogs in an African city interrupts rabies transmission and reduces human exposure. Science Translational Medicine. 9 (421), (2017).
- Yakovleva, A., et al. Tracking SARS-COV-2 variants using Nanopore sequencing in Ukraine in 2021. Scientific Reports. 12, 15749(2022).
- Mannsverk, S., et al. SARS-CoV-2 variants of concern and spike protein mutational dynamics in a Swedish cohort during 2021, studied by Nanopore sequencing. Virology Journal. 19, 164(2022).
- Soufi, M., et al. Fast and easy nanopore sequencing workflow for rapid genetic testing of familial Hypercholesterolemia. Frontiers in Genetics. 13, 836231(2022).
- Cabibbe, A. M. Application of targeted next-generation sequencing assay on a portable sequencing platform for culture-free detection of drug-resistant tuberculosis from clinical samples. Journal of Clinical Microbiology. 58 (10), 00632(2020).
- Xu, Y., et al. Nanopore metagenomic sequencing of influenza virus directly from respiratory samples: diagnosis, drug resistance and nosocomial transmission, United Kingdom, 2018/19 influenza season. Euro Surveillance. 26 (27), 2000004(2021).
- Stubbs, S. C. B., et al. Assessment of a multiplex PCR and Nanopore-based method for dengue virus sequencing in Indonesia. Virology Journal. 17, 24(2020).
- Croville, G., et al. Rapid whole-genome based typing and surveillance of avipoxviruses using nanopore sequencing. Journal of Virological Methods. 261, 34-39 (2018).
- Theuns, S., et al. Nanopore sequencing as a revolutionary diagnostic tool for porcine viral enteric disease complexes identifies porcine kobuvirus as an important enteric virus. Scientific Reports. 8, 9830(2018).
- O'Donnell, V. K., et al. Rapid sequence-based characterization of African swine fever virus by use of the Oxford Nanopore MinION sequence sensing device and a companion analysis software tool. Journal of Clinical Microbiology. 58, 01104-01119 (2019).
- Brito, A. F. Global disparities in SARS-CoV-2 genomic surveillance. Nature Communications. 13, 7003(2022).
- ONT login/register. Oxford Nanopore Technology. , Available from: https://nanoporetech.com/login-register (2023).
- Software Downloads. Oxford Nanopore Technology. , Available from: https://community.nanoporetech.com/downloads (2023).
- GitHub. , Available from: https://github.com/ (2023).
- Brunker, K. Artic-rabv. , Available from: https://github.com/kirstyn/artic-rabv (2022).
- Campbell, K. MADDOG: Method for Assignment, Definition and Designation of Global Lineages. , Available from: https://github.com/KathrynCampbell/MADDOG (2022).
- Campbell, K. RABV-GLUE. Centre for Virus Research. , Available from: https://github.com/KathrynCampbell/MADDOG (2022).
- Itokawa, K., Sekizuka, T., Hashino, M., Tanaka, R., Kuroda, M. Disentangling primer interactions improves SARS-CoV-2 genome sequencing by multiplex tiling PCR. PLoS ONE. 15 (9), e0239403(2020).
- Davis, M. W., Jorgensen, E. M. ApE, A plasmid editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 818619(2022).
- Döring, M., Pfeifer, N. openPrimeR: Multiplex PCR primer design and analysis. , (2023).
- Quick, J. Multiplex PCR method for MiniON and Illumina sequencing of Zika and other virus genomes directly from clinical samples. Nature Protocols. 12 (6), 1261-1276 (2017).
- Laboratory Techniques in Rabies. World Health Organization. 1, Available from: https://apps.who.int/iris/bitstream/handle/10665/310836/9789241515153-eng.pdf (2018).
- Lembo, T. Partners for Rabies Prevention. The blueprint for rabies prevention and control: a novel operational toolkit for rabies elimination. PLoS Neglected Tropical Diseases. 6 (2), e1388(2012).
- Terrestrial Manual Online Access. World Organization for Animal Health. , Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals/terrestrial-manual-online-access/ (2023).
- Mauti, S. Field postmortem rabies rapid immunochromatographic diagnostic test for resource-limited settings with further molecular applications. Journal of Visualized Experiments. (160), e60008(2020).
- Patrick, E., et al. Enhanced rabies surveillance using a direct rapid immunohistochemical test. Journal of Visualized Experiments. (146), e59416(2019).
- Lembo, T., et al. Evaluation of a direct, rapid immunohistochemical test for rabies diagnosis. Emerging Infectious Diseases. 12 (2), 310-313 (2006).
- Marston, D. A., et al. Pan-lyssavirus real time RT-PCR for rabies diagnosis. Journal of Visualized Experiments. (149), e59709(2019).
- Brunker, K. DNA quantification using the Qubit fluorometer. , Available from: https://www.protocols.io/view/dna-quantification-using-the-qubit-fluorometer-bc6vize6 (2020).
- Quick, J. DNA quantification using the Quantus fluorometer. , Available from: https://www.protocols.io/view/dna-quantification-using-the-quantus-fluorometer-7pzhmp6 (2020).
- Guppy protocol. Nanopore Community. , Available from: https://community.nanoporetech.com/protocols/Guppy-protocol/v/gpb_2003_v1_revaq_14dec2018 (2023).
- Flow Cell Wash Kit (EXP-WSH004). Nanopore Community. , Available from: https://community.nanoporetech.com/protocols/flow-cell-wash-kit-exp-wsh004/v/wfc_9120_v1_revh_08dec2020 (2023).
- Core Pipeline - arctic pipeline. , Available from: https://artic.readthedocs.io/en/latest/minion/ (2023).
- Samtools. , Available from: https://www.htslib.org (2023).
- Campbell, K., et al. Making genomic surveillance deliver: A lineage classification and nomenclature system to inform rabies elimination. PLoS Pathogens. 18 (5), e1010023(2022).
- Brunker, K., et al. Rapid in-country sequencing of whole virus genomes to inform rabies elimination programmes. Wellcome Open Research. 5, 3(2020).
- Bull, R. A., et al. Analytical validity of nanopore sequencing for rapid SARS-CoV-2 genome analysis. Nature Communications. 11, 6272(2020).
- Okeke, I. N., Ihekweazu, C. The importance of molecular diagnostics for infectious diseases in low-resource settings. Nature Reviews. Microbiology. 19 (9), 547-548 (2021).
- Inzaule, S. C., Tessema, S. K., Kebede, Y., Ouma, A. E. O., Nkengasong, J. N. Genomic-informed pathogen surveillance in Africa: opportunities and challenges. The Lancet Infectious Diseases. 21 (9), 281-289 (2021).
- Kennedy, L. Integrating contact tracing and whole-genome sequencing to track the elimination of dog-mediated rabies: an observational and genomic study. eLife. , (2023).
- Pallerla, S. R. Diagnosis of pathogens causing bacterial meningitis using Nanopore sequencing in a resource-limited setting. Annals of Clinical Microbiology and Antimicrobials. 21, 39(2022).
- Quick, J. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved