Method Article
使用纳米孔技术快速表征狂犬病病毒的全基因组测序
摘要
在这里,我们提出了一种使用纳米孔技术表征狂犬病病毒(RABV)基因组的快速且具有成本效益的工作流程。该工作流程旨在支持地方层面的基因组学监测,提供有关循环RABV谱系及其在区域系统发育中的位置的信息,以指导狂犬病控制措施。
摘要
基因组数据可用于追踪传染病的传播和地理传播。然而,在许多低收入和中等收入国家,基因组监测所需的测序能力仍然有限,在这些国家,狗介导的狂犬病和/或由吸血蝙蝠等野生动物传播的狂犬病构成了重大的公共卫生和经济问题。我们在这里介绍一种使用纳米孔技术的快速且经济实惠的从样品到序列再到解释的工作流程。简要介绍了狂犬病的样本采集和诊断方案,然后详细介绍了优化的全基因组测序工作流程,包括多重聚合酶链反应 (PCR) 的引物设计和优化、改良的低成本测序文库制备、活体和离线碱基检出测序、遗传谱系指定和系统发育分析。演示了工作流程的实施,并强调了本地部署的关键步骤,例如管道验证、引物优化、纳入阴性对照,以及使用公开可用的数据和基因组工具(GLUE、MADDOG)在区域和全球系统发育中进行分类和定位。工作流程的周转时间为 2-3 天,成本从 96 次样品运行的每个样品 25 美元到 12 次样品运行的每个样品 80 美元不等。我们的结论是,在中低收入国家建立狂犬病病毒基因组监测是可行的,可以支持实现到2030年实现零狗介导的人类狂犬病死亡的全球目标,并加强对野生动物狂犬病传播的监测。此外,该平台可以适用于其他病原体,有助于建立多功能基因组能力,有助于流行病和大流行的准备。
引言
狂犬病病毒 (RABV) 是弹状病毒科中的一种裂解 病毒, 可引起哺乳动物致命的神经系统疾病1。尽管狂犬病可以通过接种疫苗100%预防,但在狂犬病流行国家,狂犬病仍然是一个主要的公共卫生和经济问题。据估计,每年有60,000例人类狂犬病死亡,其中95%以上发生在非洲和亚洲,其中狗是主要宿主2。相比之下,狗的疫苗接种已经导致西欧、北美和拉丁美洲大部分地区消除了狗介导的狂犬病。在这些地区,狂犬病的宿主现在仅限于野生动物,如蝙蝠、浣熊、臭鼬和野生犬科动物3.在整个拉丁美洲,普通吸血蝙蝠是狂犬病的一个有问题的来源,因为在夜间喂血期间,蝙蝠经常溢出到人类和牲畜4.据估计,狂犬病每年对全球经济的影响为86亿美元,其中牲畜损失占6%5。
来自病毒病原体的序列数据与有关感染时间和来源的元数据相结合,可以提供可靠的流行病学见解6.对于RABV,测序已被用于调查疫情的起源7,8,确定宿主与野生动物或家犬的关联8,9,10,11,12,并追踪人类病例的来源13,14。使用系统发育分析进行的暴发调查表明,狂犬病是通过从加里曼丹或苏拉威西岛附近的流行地区15的单一传入而出现在印度尼西亚巴厘岛以前没有狂犬病的省份。与此同时,在菲律宾,朗布隆省塔布拉斯岛的疫情被证明是从吕宋岛16主岛引入的。病毒基因组数据还被用于更好地了解在地理上靶向控制措施所需的病原体传播动态。例如,RABV 的基因组特征说明了分支 17、18、19、谱系20、21、22、人类介导的病毒运动 17、23、24 和元种群动力学 25,26 的地理聚类。
疾病监测是基因组监测的一项重要功能,随着全球测序能力的提高,以应对SARS-CoV-2大流行,基因组监测得到了加强。基因组监测支持实时跟踪关注的SARS-COV-2变异株27,28和相关对策6。可及测序技术(如纳米孔技术)的进步导致了人类29、30、31、32 和动物33、34、35 病原体快速测序的改进和更实惠的方案。然而,在许多狂犬病流行国家,开展病原体基因组监测仍存在障碍,SARS-CoV-2测序能力的全球差异表明了这一点36。实验室基础设施、供应链和技术知识的局限性使得基因组监测的建立和常规化具有挑战性。在本文中,我们展示了如何在资源有限的环境中部署优化、快速且经济实惠的全基因组测序工作流程进行 RABV 监测。
研究方案
该研究得到了国家医学研究所医学研究协调委员会的批准(NIMR/HQ/R.8a/vol.坦桑尼亚地区行政和地方政府部(AB.81/288/01)和伊法卡拉卫生研究所机构审查委员会(IHI/IRB/No:22-2014);肯尼亚内罗毕大学热带和传染病研究所(P947/11/2019)和肯尼亚医学研究所(KEMRI-SERU;第3268号议定书);以及菲律宾卫生部热带医学研究所(RITM)(2019-023)。对来自尼日利亚的样本进行了测序,对作为国家监测的一部分收集的存档诊断材料进行了测序。
注意:第 1-4 节是先决条件。第 5-16 节描述了 RABV 纳米孔测序的从样本到序列再到解释的工作流程(图 1)。对于方案中需要脉冲离心的后续步骤,以10-15,000× g 离心5-15秒。
1. 用于排序和数据分析的计算环境设置
- 打开 Oxford Nanopore Technology (ONT) 网站37 并创建一个帐户以访问纳米孔特定资源。
- 登录并安装ONT测序和碱基检出软件38。
- 打开 GitHub39 并创建一个帐户。
- 转到 artic-rabv40 和 MADDOG 存储库41 并按照安装说明进行操作。
2. 设计或更新多重引物方案
注:现有的 RABV 方案可在 artic-rabv 存储库40 中找到。当瞄准一个新的地理区域时,应设计一个新的方案,或者修改现有的方案,以纳入更多的多样性。
- 选择基因组参考集来表示研究区域的多样性;这通常是一组公开可用的序列(例如,来自 NCBI GenBank)或初步的内部数据。按照步骤 2.1.1 使用 RABV-GLUE42(一种 RABV 序列数据资源)过滤和下载 NCBI 序列和相关元数据。
注:选择具有完整基因组的参考序列(即,没有间隙和掩蔽碱基)。建议选择多达 10 个序列作为引物设计的参考集。如果可用的序列数据不完整或不能代表研究区域,请参阅补充文件 1 中的建议43、44、45。- 从 RABV-GLUE 的序列数据下拉菜单中导航到 NCBI RABV Sequences by Clade 页面。单击狂犬病病毒 (RABV) 链接以访问所有可用数据或选择感兴趣的特定分支。使用过滤器选项添加符合所需条件(例如,原产国、序列长度)的过滤器。下载序列和元数据。
- 按照原始方案46 提供的说明生成多重聚合酶链反应 (PCR) 的引物方案。建议使用 400 bp 方案和 50 bp 重叠来对低质量样品进行测序。下载并保存所有输出(不要编辑文件或引物名称)。
注意:该方案将被索引到输入fasta中的第一个序列,此后称为"索引引用"(图2)。参见 补充文件 1 ,了解优化引物性能的选项。
3. 设置 RAMPART 和 ARTIC 生物信息学管道
- 请参阅补充文件 2 以设置目录结构以管理 RAMPART 和 ARTIC 生物信息学管道的输入/输出文件。
4. 生物安全和实验室设置
- 在生物安全等级 (BSL) 2 或 3 条件下处理潜在的狂犬病阳性样本。
- 确保实验室工作人员已完成狂犬病暴露前疫苗接种,并根据世界卫生组织 (WHO) 建议3 接受免疫监测。
- 确保实验室遵循国家或国际指南,制定专门的标准操作程序和风险评估。
- 所需的实验室设置:通过保持PCR前和PCR后区域之间的物理隔离,最大限度地减少污染。在空间有限的实验室或现场实验室环境中,使用便携式手套箱或临时实验室站,以尽量减少污染。
- 在此协议中,请确保为以下各项指定单独的区域:
- 样品提取:设置BSL2/3柜/手套箱,用于处理生物材料并进行灭活和RNA提取。
- 模板区域:设置BSL1柜/手套箱,用于将模板(RNA/cDNA)添加到预制备的反应预混液中。
- 预混液区:设置指定的洁净区(BSL1柜/手套箱),用于制备试剂预混液。此区域不应有模板。
- PCR后区域:设置一个单独的区域,用于扩增子和测序文库制备。
注意: 所有区域均应在使用前后使用表面去污剂和紫外线 (UV) 消毒进行清洁。
5. 现场样本采集与诊断
注意:样本必须由经过培训和免疫接种的人员收集,他们穿着个人防护设备并遵循参考标准程序47、48、49。
- 通过枕骨大孔(即枕骨途径)收集样本,如Mauti等人50中详细描述的那样。
- 在现场使用快速诊断测试诊断狂犬病,并使用推荐的程序47在实验室进行确认,例如直接荧光抗体检测(DFA)、直接快速免疫组织化学检测(DRIT)51,52或实时逆转录(RT)-PCR53。
- 使用确认的阳性脑样本进行RNA提取,或在-20°C的冰箱中储存2-3个月或-80°C更长时间。使用合适的 DNA/RNA 稳定培养基保存 RNA 以进行储存和运输。
6. 样品制备和RNA提取(3小时)
注:使用适合样品类型的基于离心柱的病毒RNA提取试剂盒。
- 用装满 1.4 mm 陶瓷珠的约 200 μL 管填充 2 mL PCR 管,制备两个陶瓷珠管,并标记管。
- 将RNA提取试剂盒中提供的推荐体积的裂解缓冲液加入标记的PCR管中。
- 使用木制涂抹器从确认患有狂犬病感染的脑样本中取出约3mm立方体,并将其放入带有样品ID的标记管中,并将100μL无核酸酶水放入标记阴性对照的管中。
注意:使用基于封闭管微珠的均质化来限制样品暴露。如果不可能,请使用其他合适的机械干扰器(例如,基于转子)或手动微型研杵。然而,这些方法可能不如在坚硬表面上敲打珠子来破坏组织有效(组织样品在某些储存介质中可能会变硬)。 - 使用木制涂抹棒手动破坏脑组织,然后以最大速度涡旋,直到实现完全的组织均质化。
- 按照制造商的说明离心裂解物,并使用移液管将上清液转移到新标记的微量离心管中。仅在后续步骤中使用该上清液。
- 按照 RNA 提取试剂盒的离心柱说明获得纯化的 RNA。
- 在此处包括阴性提取对照 (NEC),并一直到测序阶段。
7. cDNA制备(20分钟)
- 在预混液区,根据待处理的样品和对照品数量,准备用于第一链cDNA合成的预混液(过量体积为10%,以保证试剂充足; 表1)。在此阶段应包括无模板控件 (NTC)。
- 标记 0.2 mL PCR 联排管,并将 5 μL 预混液分装到试管中。
- 将准备好的试管带到模板区域。将 5 μL RNA 加入每个标记管中,包括 NEC。向 NTC 中加入 5 μL 无核酸酶水 (NFW)。
- 按照 表1中提到的条件在热循环仪中孵育。
注意:可选暂停点:如有必要,cDNA可以在-20°C下储存长达1个月,但最好进行PCR。
8. 引物池原液制备(1 h)
注意:仅当从单个引物制备新储备液时,才需要此步骤,之后可以使用预先制备的储备液。
- 在预混液区制备100μM原液的引物池。
- 将冻干引物重悬于1x tris-EDTA(TE)缓冲液或NFW中,浓度为100μM。彻底涡旋并向下旋转。
注意:在以下步骤中,将单个引物分成两个引物池 - 奇数编号(称为池A)和偶数编号(称为池B) - 以避免引物与扩增子重叠之间的相互作用。这些引物池产生跨越靶基因组的重叠 400 bp 扩增子。 - 将所有奇数引物排列在试管架中。通过将每个引物中的5μL加入标有"引物方案名称 - 池A(100μM)"的1.5mL微量离心管中来生成引物池原液。
- 对所有偶数引物重复该过程,并标记为"引物方案名称 - 池B(100μM)"。
- 在分子级水中以 1:10 的比例稀释每个引物池,以生成 10 μM 引物储备液。
注:制备多个等分试样的 10 μM 引物稀释液,并在降解或污染的情况下冷冻它们。
9. 多重PCR(5小时)
- 准备两个PCR预混液,一个用于预混液区域的每个引物池。
- 每个引物的终浓度为 0.015 μM。使用以下公式计算PCR反应所需的引物池体积(表2):
引物池体积=引物数量×反应体积×0.015/引物原液浓度(μM)
- 每个引物的终浓度为 0.015 μM。使用以下公式计算PCR反应所需的引物池体积(表2):
- 将池 A 预混液和池 B 预混液各 10 μL 分装到模板区域标记的 PCR 联排管中。 对于每个样品,将 2.5 μL cDNA(来自步骤 3)添加到每个相应的标记引物池 A 和 B 反应中。过量的cDNA可以储存在-20°C。
- 轻轻轻弹混合,脉冲离心机。
- 在PCR机器上将样品与 表2 中提到的条件一起孵育。
注:由于退火时间长至5 min(由于引物数量多而需要)和扩增子的短长度(400 bp),因此该程序不包括特定的延伸步骤,这足以进行延伸。
10. PCR纯化和定量(3.5小时)
- 从现在开始,在PCR后区域执行所有工作。
- 将等分试样固相可逆固定 (SPRI) 微珠从主瓶放入微量离心管中。储存在4°C。
- 将SPRI微珠等分试样加热至室温(RT; ~20°C)并彻底涡旋,直到微珠完全重悬于溶液中。
- 在 1.5 mL 试管中,合并每个样品的引物池 A 和引物池 B PCR 产物。如有必要,加水使体积达到 25 μL。
- 向每个样品中加入 25 μL SPRI 磁珠(磁珠:样品比为 1:1)。通过上下移液或轻轻敲击试管进行混合。
- 在室温下孵育 10 分钟,偶尔倒置或轻弹试管。
- 放在磁架上,直到珠子和溶液完全分离。取出并丢弃上清液,注意不要干扰珠子沉淀。
- 用80%乙醇洗涤两次(加热至室温)。
- 向沉淀中加入 200 μL 乙醇。等待 30 秒以确保珠子正确清洗。
- 小心地取出并丢弃上清液,尽量不要接触珠子沉淀。
- 重复步骤10.8.1-10.8.2第二次洗涤颗粒。
- 使用 10 μL 吸头去除所有痕量乙醇。风干,直到痕量乙醇蒸发(使用小珠子,这发生得很快,~30 秒);发生这种情况时,颗粒应该从闪亮变为哑光。注意不要过度干燥(如果沉淀破裂,则太干),因为这会影响 DNA 回收率。
- 将珠子重悬于 15 μL NFW 中,并在室温下孵育 10 分钟(非磁架)。
- 返回磁性架,将上清液(清洁的产品)转移到新鲜的 1.5 mL 试管中。
- 在单独的试管中制备每个样品的 1:10 稀释液(2 μL 产物 + 18 μL NFW)。
注意:在此阶段要非常小心,以避免交叉污染。一次只能打开一个扩增子管。首先将 18 μL 水分装到试管中(在干净的预混液区域)。 - 使用高灵敏度和特异性荧光计测量每个稀释样品的 DNA 浓度,如 protocols.io54,55 中所述。
11. 归一化(30 分钟)
- 使用每个样品的归一化模板(补充文件3)和DNA浓度(ng / μL)计算每个样品在总体积为5μL时200fmol所需的稀释(或纯)样品体积。
- 标记新的PCR管并添加计算体积的NFW和样品以获得标准化的DNA。
- 如果需要超过 5 μL 的稀释样品才能获得 200 fmol,则使用未稀释(纯)样品的计算体积。
注意:可选暂停点:此时,清理后的PCR产物可以在4°C下储存长达1周,或在需要时置于-20°C下进行长期储存。
12. 末端准备和条形码(1.5 小时)
注:接下来的步骤假设使用纳米孔特异性条形码和连接测序试剂盒中的特定试剂(有关详细信息,请参阅 材料表 )。该方案可在不同的化学版本之间转移,但用户应注意根据制造商的说明使用兼容的试剂盒。
- 端部修复和 dA 拖尾
- 为 表3中提到的每个样品设置末端制备反应。根据样品数量(加上 10% 的过量)制备预混液。移液时要小心,因为试剂是粘稠的。
- 将 5 μL 预混液加入每管标准化 DNA (5 μL) 中。总反应混合物应为 10 μL。 每次更换吸头,一次只打开一根管子。
- 在 表3中提到的条件下在热循环仪中孵育。
- 条形码
- 以 1.25 μL/管的速度将条形码从条形码试剂盒分装到 PCR 联排管中,并记录分配给每个样品的条形码。
- 将 0.75 μL 末端制备的样品添加到其指定的条形码等分试样中。
- 根据样品数量(加上10%过量)制备连接预混液(表4)。
- 向末端制备的样品 + 条形码中加入 8 μL 连接预混液,总反应量为 10 μL。
- 使用 表4中提到的条件在热循环仪中孵育。
- SPRI 磁珠纯化和 DNA 定量
- 在室温下解冻短片段缓冲液 (SFB),涡旋混合,脉冲离心,然后置于冰上。
- 将所有带条形码的样品汇集在 1.5 mL lobind 微量离心管中。为了避免净化体积太大而无法使用,从每个非变性条形码反应中混合 12-24 个样品 (10 μL/样品)、最多 48 个样品 (5 μL/样品) 或最多 96 个样品 (2.5 μL/样品)。
- 将 0.4 倍体积的 SPRI 珠子添加到条形码池中。轻轻混合(轻弹或移液)并在室温下孵育 5 分钟。
- 将样品放在磁铁上,直到珠子沉淀并且上清液完全透明(~2分钟)。取出并丢弃上清液。注意不要打扰珠子。
- 用 250 μL SFB 洗涤两次。
- 从磁铁上取下试管,将沉淀完全重悬于250μLSFB中。孵育30秒,脉冲离心,然后返回磁体。
- 取出上清液并丢弃。
- 重复步骤 12.3.5 以执行第二次 SFB 洗涤。
- 脉冲离心机并除去任何残留的 SFB。
- 加入 200 μL 80% (RT) 乙醇对沉淀进行沐浴。取出并丢弃乙醇,注意不要干扰珠子颗粒。风干 30 秒或直到颗粒失去光泽。
- 在室温下重悬于 22 μL NFW 中 10 分钟。
- 放在磁铁上,静置~2分钟,然后小心地取出溶液并转移到干净的1.5mL微量离心管中。
- 如前所述,使用1μL获得DNA浓度(步骤10.13)。
注:可选暂停点:此时,文库可以在4°C下储存长达1周,或在-20°C下长期储存,但最好继续进行接头连接和测序。
13. 测序(最长 48 小时)
- 准备计算机(另请参阅先决条件第 1-4 节)。
- 检查是否有足够的空间来存储新数据(至少 150 GB),在删除之前将旧运行的数据备份/移动到服务器,以及安装最新版本的 MinKNOW。
- 从冰箱中取出储存的流通池,使其达到室温。
- 适配器连接(1小时)
- 脉冲离心,将适配器混合物和连接酶放在冰上。
- 在室温下解冻洗脱缓冲液 (EB)、SFB 和连接缓冲液,涡旋混合,脉冲离心,然后置于冰上。
- 制备接头连接预混液(表5),在低结合管中按指定顺序合并试剂。
注:根据实验室的可用性,可以使用接头连接预混液试剂(表 5)的替代品。有关替代方案列表,请参阅 补充文件 3 和 材料表 。使用 补充文件 3 工作表中的计算来获得相当于 200 fmol的 DNA 文库体积。如果计算出的浓度低于 20 μL,则添加 NFW 以补足 20 μL。 - 通过轻弹和脉冲离心机混合。在室温下孵育20分钟。
注意:在孵育过程中,开始准备流通池(第 13.5 节)。
- 使用SPRI珠子进行清理(不要像早期的清理那样使用乙醇)。
- 向样品中加入 0.4 倍体积的 SPRI 微球 (RT)。在室温下孵育 10 分钟,间歇性地轻轻轻弹以帮助混合。
- 放在磁铁上,直到珠子和溶液完全分离(~5分钟)。取出并弃去上清液;注意不要打扰珠子颗粒。
- 用 125 μL SFB 洗涤两次。
- 用移液管混合,用125μLSFB完全重悬沉淀。静置孵育 30 秒。
- 脉冲离心机将液体收集在管底并放置在磁铁上。取出上清液并丢弃。
- 重复步骤13.4.4-13.4.5第二次洗涤颗粒。
- 脉冲离心机并除去多余的 SFB。
- 重悬于 15 μL EB 中,并在室温下孵育 10 分钟。
- 返回磁体 ~2 分钟,然后小心地将溶液转移到干净的 1.5 mL 微量离心管中。
- 定量 1 μL 洗脱文库,如前面的步骤 10.13 所述
注意:为获得最佳结果,请直接进行 MinION 测序;然而,如果需要,最终文库可以在4°C的EB中储存长达1周。
- 运行流通池质量检查。
- 将测序设备连接到笔记本电脑并打开测序软件。
- 选择流通池类型,然后单击 "检查流通池 "并开始 测试。
- 完成后,将显示活动(即活)孔的总数。新的流通池应具有 >800 个活性孔;如果没有,请联系制造商进行更换。
- 灌注和加载流通池(20分钟)
- 在室温下解冻以下试剂,然后将测序缓冲液、冲洗系绳、冲洗缓冲液和上样微球置于冰上。
- 涡旋测序缓冲液并冲洗缓冲液,脉冲离心机,并置于冰上。
- 脉冲离心冲洗系绳并通过移液混合;然后放在冰上。
- 通过将 30 μL 冲洗系绳直接添加到流通池启动试剂盒的冲洗缓冲液管中来制备流通池引液混合物,并通过移液混合。
- 在使用前立即通过移液混合上样珠,因为它们会迅速沉淀。
- 在新鲜试管中,制备用于测序的最终文库稀释液,如 表5所述。
注意:使用 补充文件 3 工作表中的计算来获得相当于 50 fmol 的 DNA 文库体积。如果计算出的浓度小于 12 μL,则添加 EB 以达到 12 μL。 - 向后翻转排序设备盖并顺时针滑动启动端口盖,使启动端口可见(图 3)
- 通过将 P1000 移液器设置为 200 μL 小心地去除气泡,将吸头插入灌注口,然后转动转轮,直到看到进入移液器吸头的小体积(最大转动至 230 μL)。
- 通过灌注端口将 800 μL 流通池灌注混合物加载到流通池中,注意避免产生气泡。
- 静置 5 分钟。
- 轻轻提起样品端口盖,并使用 P1000 移液器 通过 启动端口将 200 μL 引物混合物加载到流通池中。
- 上样前上下移液库混合物,确保在上样前重悬预混液中的上样微珠。
- 将 75 μL 文库混合物 通过 样品端口以滴加方式加载到流通池中。在添加下一个滴水之前,确保每个滴水都流入端口。
- 轻轻盖上样品端口盖,确保塞子进入样品端口。
- 关闭启动端口并盖上测序装置盖。
- 测序运行(最长 48 小时)
- 将测序设备连接到笔记本电脑并打开测序软件。
- 单击 " 开始",然后单击" 开始排序"。
- 单击 "新建实验 ",然后按照测序软件图形用户界面 (GUI) 工作流程设置运行参数。
- 输入实验名称和样品ID(例如,rabv_run1),然后从下拉菜单中选择 流通池类型 。
- 继续选择试剂盒,并选择相关的连接测序试剂盒和使用的非变性条形码试剂盒。
- 继续 运行 选项。保留默认值,除非希望运行在一定小时数后自动停止(可以随时手动停止运行)。
- 继续进行碱基检出。根据计算资源选择打开或关闭 Basecalling(请参阅计算机设置)。在条形码下选择"编辑选项",并确保"条形码两端"已打开。保存并继续输出部分。
- 接受默认值并继续进行最终审核,检查设置,并将详细信息记录在工作表(补充文件 3)中。单击 "开始"。
注意: 如果重复使用流通池,请调整启动电压(在运行选项的高级部分),如补充文件中的方案所示 文件 3. - 记录初始活动通道 - 如果明显低于质量控制 (QC) 检查,请重新启动测序软件。如果仍然较低,则重新启动计算机。
- 记录股状与单孔的初始通道,以给出近似的孔隙占用率。这个数字会波动,所以给出一个近似值。
- 监视运行的进展。
14. 实时和离线基地呼叫
注意:这些说明假定 artic-rabv 存储库中提供的预先存在的目录结构,并且已遵循协议的先决条件第 1 节和第 3 节。
- 在本地文件系统上,创建一个名为 analysis 的新目录,您将在其中存储所有分析输出。要进一步组织:使用提供给 MinKNOW 的示例 ID 作为run_name,创建一个包含项目名称的子目录,并在该子目录中创建一个用于运行的新目录。在一个命令中执行此操作,如下所示:
mkdir -p
分析/project_name/run_name
然后导航到其位置:
光盘
路径/分析/project_name/run_name - 实时碱基呼叫
注意:要实时执行纳米孔碱基检出,笔记本电脑需要兼容 NVIDIA CUDA 的图形处理单元 (GPU)。确保已使用 guppy 协议56 执行了 GPU 碱基调用设置的指令。- 在运行设置期间,打开实时碱基调用。
- 按照以下说明,使用 RAMPART 实时监控测序覆盖率。
- 在计算机的终端中,激活 artic-rabv conda 环境:
Conda 激活 ARTIC-RABV - 在 run_name 目录中为 rampart 输出创建一个新目录并导航到该目录:
cd /path/analysis/project_name/run_name
mkdir rampart_output
光盘rampart_output - 创建一个条形码 .csv 文件以配对条形码和样品名称。每个条形码应有一行,并且仅指定库中存在的条形码,标题为"条形码"和"样品"。按照 artic-rabv 目录中的示例操作:
分析/example_project/example_run/rampart_output/条形码.csv - 通过在运行的 MinKNOW 输出中提供相关的协议文件夹和 fastq_pass 文件夹的路径来启动 RAMPART:
rampart --protocol /path/rampart/scheme_name_V1_protocol - basecalledPath - 打开浏览器窗口并导航到 URL 框中的 localhost:3000。等待足够的数据被调用,然后结果才会出现在屏幕上。
- 离线碱基检出(运行后执行)
- 如果未设置实时碱基调用,MinKNOW的输出将是原始信号数据(fast5文件)。在运行过程中将无法使用 RAMPART。在运行后使用 guppy 将 fast5 文件转换为 basecalled 数据(fastq 文件)(请参阅先决条件步骤 1.1.1 中的设置)。在称为 basecalled 的数据上运行 RAMPART 事后。
- 运行 guppy basecaller:
guppy_basecaller -c dna_r9.4.1_450bps_fast.cfg -i /path/to/reads/fast5_* -s /path/analysis/project_name/run_name -x auto -r
-c 是指定 basecalling 模型的配置文件,-i 是输入路径,-s 是保存路径,-x 指定 GPU 设备的 basecalling(如果使用 guppy 的计算机版本则除外),-r 指定递归搜索输入文件。
注意:配置文件 (.cfg) 可以通过将 _fast 替换为 _hac 来更改为高精度 basecaller,尽管这需要更长的时间。
15. 清洗流通池
- 如果孔隙仍然有效,可以洗涤流通池并重复使用以对新文库进行测序。参见 ONT 流通池清洗方案57 中的洗涤说明。
16. 分析与解释
- 使用ARTIC生物信息学流程生成共识序列
- 按照 rabv_protocols 文件夹中的 artic-rabv GitHub 存储库40 中详述的说明,从原始的 fast5 或称为 basecalled 的 fastq 文件生成共识序列。
注意:有关进一步指导,请参阅 Artic 管道 - 核心管道58 。
- 按照 rabv_protocols 文件夹中的 artic-rabv GitHub 存储库40 中详述的说明,从原始的 fast5 或称为 basecalled 的 fastq 文件生成共识序列。
- 可选:分析每个扩增子的平均读取深度。
- 改编 artic-rabv 存储库中提供的脚本,参考 补充文件 1。简而言之,使用 SAMtools59 生成深入的统计数据,并在 R 中绘制每个核苷酸的覆盖率。
- 使用GLUE进行系统发育分析
- 从RABV_GLUE42 中,选择 "分析">"基因分型和解释 "选项卡> "添加文件",选择共识序列的 fasta 文件。
- 单击 "提交 "并等待。分析完成后,可单击 "显示分析 "按钮,显示分支和亚分支分配、每个基因的覆盖率、参考序列的变异和最接近的亲缘关系。
- 相关的上下文序列也可以在 按分支划分的序列数据> NCBI 序列 部分进行识别。
- 选择已识别的分支或单击 狂犬病病毒 (RABV) 以查看所有可用序列。
- 筛选相关序列(例如,原产国)。
- 下载这些序列和相应的元数据进行分析和比较。
- 使用 MADDOG41 进行血统分配
- 从 GitHub 拉取 MADDOG 存储库,以确保您使用的是最新版本。
- 在本地 MADDOG 存储库(之前在"先决条件"部分中创建)中创建一个名为运行名称的分配文件夹。
- 在文件夹内,添加包含共识序列的 fasta 文件。
- 将元数据文件添加到文件夹。
注意:此文件必须是 csv,其中包含 4 列,分别称为"ID"、"国家/地区"、"年份"和"分配",详细说明序列 ID、采样国家/地区和样品采集年份,而"分配"列应为空。 - 在命令行界面中,激活 conda 环境: conda activate MADDOG。
- 在命令行界面中,导航到 MADDOG 存储库文件夹。
- 最初,对序列进行谱系分配,以检查任何潜在的异常,并确定运行较长的谱系指定步骤是否合适。为此,请在命令行中键入以下内容: sh assignment.sh。
- 出现提示时,输入 Y 表示您已拉取存储库,并且正在使用最新版本的 MADDOG。
- 出现提示时,输入包含 fasta 文件的 MADDOG 存储库文件夹中的文件夹名称。
- 世系分配完成后,检查文件夹中的输出文件。如果输出符合预期,并且有多个序列分配给同一谱系,请运行谱系指定。
- 如果运行世系指定,请删除刚刚创建的分配输出文件。
- 在终端的 MADDOG 存储库文件夹中,运行命令 sh designation.sh。
- 出现提示时,输入 Y 表示您已拉取存储库,并且正在使用最新版本的 MADDOG。
- 出现提示时,在包含 fasta 文件和元数据的 MADDOG 存储库文件夹中输入文件夹名称。这将输出有关每个序列的谱系信息、新的和相关的先前序列的系统发育(从 16.3.6 开始)、有关谱系的层次结构信息,以及潜在新兴谱系和欠采样区域的详细信息。
注:有关协议、用法和输出的完整详细信息,请参见 Campbell et al.60。 - 初始分析完成后,如果系统提示同时测试新出现的和采样不足的谱系,请输入 Y(如果需要)。否则,请输入 N。
- 如果系统提示您确认新找到的世系,请输入 Y 并按照生成的 NEXT_STEPS.eml 文件中的说明进行操作。否则,请输入 N。
结果
该协议中描述的RABV的样本到序列到解释工作流程已成功用于坦桑尼亚,肯尼亚,尼日利亚和菲律宾等流行国家的不同实验室条件(图4)。该方案用于不同的样品类型和条件(表6):新鲜和冷冻的脑组织,在冷链下长时间运输的脑组织的cDNA和RNA提取物,以及带有脑组织涂片的FTA卡。
使用 RAMPART 的实时碱基检出(图 5)显示了几乎实时生成的读数和每个样本的覆盖率百分比。这在决定何时停止运行并保存流通池以供重复使用时特别有用。观察到运行时间的变化,有些在 2 小时内完成,而另一些可能需要超过 12 小时才能达到足够的覆盖深度 (x100)。我们还可以看到放大效果较差的区域;例如,图 6 显示了一次测序运行的快照,其中覆盖率图显示了一些扩增子的扩增子,扩增子的扩增子非常低,表明引物可能存在问题。通过更彻底地研究这些扩增不良的区域,我们已经能够识别引物错配,这将使我们能够重新设计和改进单个引物。一些引物方案比其他方案显示出更多的错配。与菲律宾相比,东非入门计划与目标多样性一致,因为东非计划旨在捕捉更广泛的多样性。
RABV-GLUE42(一种用于 RABV 基因组数据管理的通用资源)和 MADDOG60(一种谱系分类和命名系统)用于编译和解释生成的 RABV 序列。 表7 显示了使用RABV-GLUE分配的每个国家中流通的主要和次要分支。还显示了 MADDOG 分配后本地谱系的更高分辨率分类。
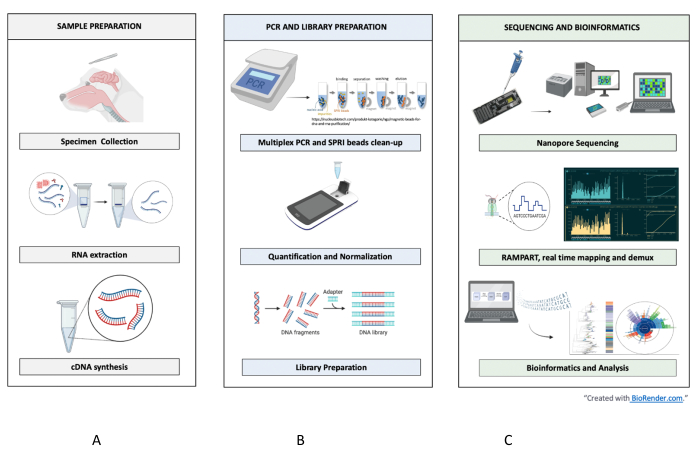
图 1:RABV 从样本到序列再到解释的工作流程。 总结了(A)样品制备,(B)PCR和文库制备,以及(C)测序和生物信息学直至分析和解释的步骤。 请点击这里查看此图的较大版本.
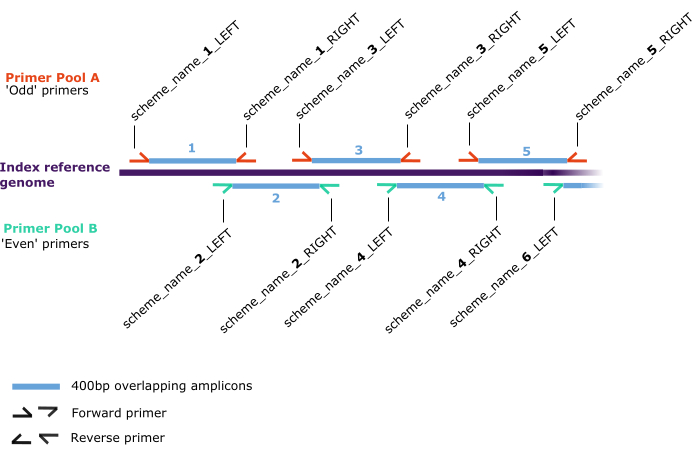
图 2:引物方案示意图。 正向和反向引物对(半箭头)沿"索引参考基因组"(深紫色)的退火位置,它们被分配在两个单独的池中: A (红色)和 B (绿色)。引物对产生 400 bp 重叠扩增子(蓝色),这些扩增子以"scheme_name_X_DIRECTION"格式沿索引参考基因组依次编号,其中"X"是指引物产生的扩增子的数字,"DIRECTION"是"LEFT"或"RIGHT",分别描述正向或反向。"X"的奇数或偶数值决定池(分别为 A 或 B)。 请点击这里查看此图的较大版本.
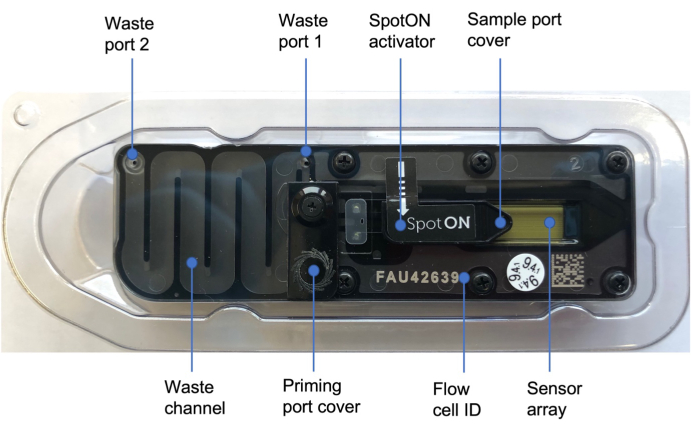
图 3:纳米孔流通池48。 蓝色标签表示流通池的不同部分,包括覆盖添加灌注溶液的灌注端口盖、覆盖以滴加样品的样品端口的 SpotON 样品端口盖、废液端口 1 和 2 以及流通池 ID。 请点击这里查看此图的较大版本.
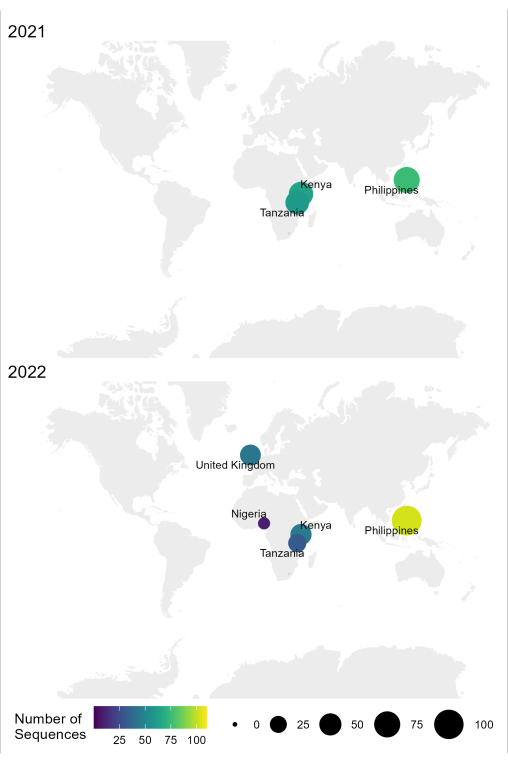
图 4:显示了 2021 年和 2022 年使用优化工作流程进行 RABV 测序的位置的地图。 气泡大小和颜色对应于每个位置的序列数,其中越小越暗越少,而越大越亮越多。 请点击这里查看此图的较大版本.
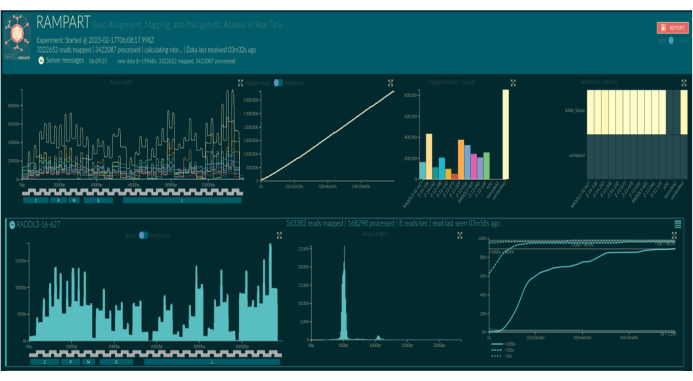
图 5:Web 浏览器中 RAMPART 可视化的屏幕截图。 根据生物信息学设置,条形码名称被样品名称取代。前三个面板显示了整个运行的汇总图:索引参考基因组上每个核苷酸位置的每个条形码的映射读长覆盖深度(左上,由条形码着色)、所有条形码随时间推移的映射读长总和(上上中)和每个条形码的映射读长(右上,按条形码着色)。下方面板显示每个条形码的绘图行。从左到右:索引参考基因组上每个核苷酸位置的映射读段覆盖深度(左)、映射读段的长度分布(中)以及索引参考基因组上核苷酸位置的比例,这些比例随着时间的推移获得了 10 倍、100 倍和 1,000 倍的映射读段覆盖率(右)。 请点击这里查看此图的较大版本.
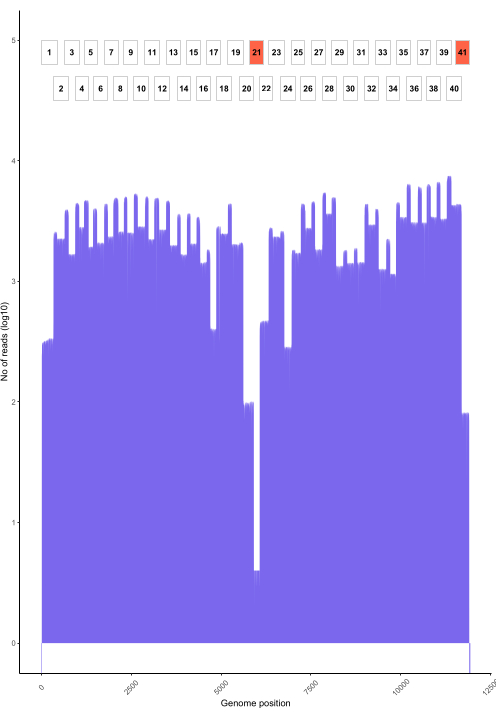
图6:使用该方案测序的菲律宾狂犬病病毒样本的基因组读取覆盖率示例。图中显示了基因组中每个核苷酸位置的读取覆盖率,以及用于生成文库的重叠扩增子 (1-41) 的位置。覆盖深度的峰值对应于扩增子重叠的区域。覆盖深度低的扩增子对应于扩增子重叠的区域。覆盖深度低的扩增子以红色突出显示,表示可能需要优化的问题区域。 请点击这里查看此图的较大版本.
表1:用于cDNA制备的预混液和热循环仪条件。请按此下载此表格。
表2:用于多重PCR的预混液和热循环仪条件。请按此下载此表格。
表3:预混液和热循环仪在制备结束反应中的条件。请按此下载此表格。
表 4:用于条形码的预混液和热循环仪条件。请按此下载此表格。
表 5:用于测序的接头连接预混液和最终文库稀释。请按此下载此表格。
表6:不同国家使用从样本到测序再到解释的工作流程生成的狂犬病病毒全基因组序列数量和使用的样本类型。请按此下载此表格。
表 7:RABV-GLUE 的主要和次要分支分配以及 MADDOG 对使用工作流程生成的序列的谱系分配。请按此下载此表格。
补充文件1:引物方案设计与优化,扩增子读取深度分析。请点击这里下载此文件。
补充文件2:计算设置 请点击这里下载此文件。
补充文件3:RABV WGS协议工作表 请点击此处下载此文件。
讨论
Brunker 等人 61 利用 ARTIC 网络46 的资源开发了一种可访问的 RABV、基于纳米孔的全基因组测序工作流程。在这里,我们介绍了一个更新的工作流程,包括从样本到序列再到解释的完整步骤。该工作流程详细介绍了用于全基因组测序的脑组织样本的制备,提供了用于处理读取和生成共识序列的生物信息学管道,并重点介绍了两种狂犬病特异性工具,用于自动谱系分配和确定系统发育背景。更新后的工作流程还为设置适当的计算和实验室工作区提供了全面的说明,并考虑了在不同上下文(包括低资源设置)中实施的注意事项。我们已经证明,在四个没有或基因组监测能力有限的RABV流行中低收入国家的学术和研究机构环境中成功实施了该工作流程。事实证明,该工作流程能够适应各种设置的应用,并且具有不同专业知识的用户也可以理解。
这种用于 RABV 测序的工作流程是最全面的公开协议(涵盖从样本到测序再到解释的步骤),专门用于降低启动和运行成本。与其他平台(如Illumina61)相比,使用纳米孔技术进行文库制备和测序所需的时间和成本大大降低,并且持续的技术发展正在提高序列质量和准确性,使其与Illumina62相媲美。
该协议旨在在各种低资源环境中具有弹性。通过参考核心协议随附的故障排除和修改指南,支持用户根据自己的需求调整工作流程。在工作流程中增加用户友好的生物信息学工具是对原始协议的重大发展,它提供了快速和标准化的方法,可以由具有最少生物信息学经验的用户应用这些方法来解释本地环境中的序列数据。 就地 进行这项工作的能力往往受到需要具备特定编程和系统发育技能的限制,这需要密集和长期的技能培训投资。虽然这种技能对于彻底解释序列数据很重要,但基本和可访问的解释工具同样是可取的,以便能够培养当地的"测序冠军",他们的核心专业知识可能是基于湿实验室的,使他们能够解释并拥有数据的所有权。
由于该方案已在多个国家实施多年,我们现在可以就如何优化多重引物方案以提高覆盖率和处理累积的多样性提供指导。还努力帮助用户提高成本效益或便于在特定地区进行采购,这通常是对分子方法可持续性的挑战63。例如,在非洲(坦桑尼亚、肯尼亚和尼日利亚),我们在接头连接步骤选择了钝/TA 连接酶预混液,这更容易从当地供应商处获得,并且是其他连接试剂的更便宜的替代品。
根据经验,有几种方法可以降低每个样品和每次运行的成本。减少每次运行的样品数量(例如,从 24 个样品减少到 12 个样品)可以延长流通池在多次运行中的使用寿命,而增加每次运行的样品数量可以最大限度地延长时间和试剂。在我们手中,我们每三次测序运行中就有一次能够清洗和重复使用流通池,从而能够对另外 55 个样品进行测序。使用后立即清洗流通池,或者如果不可能,则在每次运行后从废物通道中去除废液,似乎可以保留可用于第二次运行的孔隙数量。考虑到流通池中可用的初始孔数,还可以优化一次运行,以计划在特定流通池中运行多少样品。
尽管工作流程旨在尽可能全面,并添加了详细的指导和路标资源,但该过程仍然很复杂,对于新用户来说可能令人生畏。鼓励用户寻求面对面的培训和支持,最好是在当地,或者通过外部合作者。例如,在菲律宾,一个关于使用ONT进行SARS-CoV-2基因组监测的区域实验室能力建设项目已经培养了卫生保健诊断医生的核心能力,这些能力很容易转移到RABV测序中。如果没有动手培训,SPRI磁珠清理等重要步骤可能很难掌握,而无效的清理可能会损坏流通池并影响运行。在实验室中处理扩增子时,样品污染始终是一个主要问题,并且可能难以消除。特别是,在运行后的生物信息学中,样品之间的交叉污染极难检测。良好的实验室技术和实践,如保持清洁的工作表面、分离PCR前后区域以及纳入阴性对照,对于确保质量控制至关重要。纳米孔测序的快速发展是常规RABV基因组监测的优点和缺点。纳米孔的精度、可及性和协议库的不断改进拓宽并提高了其应用范围。然而,同样的发展使得维护标准操作程序和生物信息学管道具有挑战性。在该协议中,我们提供了一份文件,帮助从旧的纳米孔库制备试剂盒过渡到当前的纳米孔文库制备试剂盒(材料表)。
中低收入国家测序的一个常见障碍是可及性,不仅包括成本,还包括及时采购耗材(特别是测序试剂,对采购团队和供应商来说相对较新的测序试剂)和计算资源的能力,以及简单地获得稳定的电力和互联网。使用便携式纳米孔测序技术作为该工作流程的基础有助于解决许多这些可访问性问题,我们已经演示了在一系列环境中使用我们的方案,在国内进行完整的方案和分析。诚然,及时采购设备和测序耗材仍然是一个挑战,在许多情况下,我们被迫从英国携带或运送试剂。然而,在某些地区,我们能够完全依赖当地的试剂供应路线,这得益于对SARS-CoV-2测序的投资(例如菲律宾),这些投资简化了采购流程,并开始使病原体基因组学的应用正常化。
通过一次性安装,最大限度地减少了对稳定互联网连接的需求;例如,GitHub 存储库、软件下载和纳米孔测序本身只需要互联网访问即可开始运行(而不是整个运行),或者可以在公司同意的情况下完全离线执行。如果移动数据可用,则可以将手机用作笔记本电脑的热点,以开始测序运行,然后在运行期间断开连接。在常规处理样品时,数据存储需求可能会迅速增长,理想情况下,数据将存储在服务器上。否则,固态硬盘 (SSD) 硬盘的采购成本相对较低。
虽然我们认识到中低收入国家的基因组监测仍然存在障碍,但增加对建立基因组学可及性和专业知识的投资(例如,非洲病原体基因组学倡议[Africa PGI])64 表明这种情况将得到改善。基因组监测对于大流行防范至关重要6,可以通过对RABV等地方病原体进行基因组监测来建立能力。SARS-CoV-2大流行期间强调的全球测序能力差异应成为推动变革的驱动力,以解决这些结构性不平等问题。
这种从样本到序列再到解释的RABV工作流程,包括可访问的生物信息学工具,有可能用于指导控制措施,目标是到2030年实现狗介导的狂犬病导致人类零死亡的目标,并最终消除RABV变异株。结合相关元数据,该协议产生的基因组数据有助于在疫情调查期间快速鉴定RABV和鉴定一个国家或地区的流行谱系60,61,65。我们主要使用狗介导的狂犬病的例子来说明我们的管道;但是,该工作流程直接适用于野生动物狂犬病。这种可转移性和低成本最大限度地减少了常规测序的难题,不仅针对狂犬病,而且针对其他病原体46,66,67,以改善疾病管理和控制。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了惠康 [207569/Z/17/Z、224670/Z/21/Z]、医学研究委员会 [MR/R025649/1] 和菲律宾科学技术部 (DOST) 的牛顿资助、英国研究与创新全球 COVID-19 努力 [MR/V035444/1]、格拉斯哥大学机构战略支持基金 [204820]、医学研究委员会新研究者奖 (KB) [MR/X002047/1] 的支持, 国际伙伴关系发展基金、DOST 英国文化协会菲律宾学生奖学金 (CB)、国家卫生研究所 [17/63/82] GemVi 奖学金 (GJ) 以及格拉斯哥大学 MVLS DTP (KC) [125638-06]、EPSRC DTP (RD) [EP/T517896/1] 和 Wellcome IIB DTP (HF) [218518/Z/19/Z] 的学生奖学金。我们感谢支持这项工作的同事和合作者:Daniel Streicker、Alice Broos、Elizabeth Miranda、DVM、Daria Manalo、DVM、Thumbi Mwangi、Kennedy Lushasi、Charles Kayuki、Jude Karlo Bolivar、Jeromir Bondoc、Esteven Balbin、Ronnel Tongohan、Agatha Ukande、Davis Kuchaka、Mumbua Mutunga、Lwitiko Sikana 和 Anna Czupryna。
材料
| Name | Company | Catalog Number | Comments |
| Brand name | |||
| Software | |||
| Sequencing software (MinKnow) | Oxford Nanopore Technologies | https://community.nanoporetech.com/downloads | |
| Bioinformatics tool kit (Guppy) | Oxford Nanopore Technologies | https://community.nanoporetech.com/docs/prepare/library_prep_protocols/Guppy-protocol/v/gpb_2003_v1_revao_14dec2018 | |
| Equipment | |||
| Thermal cycler (miniPCR™ mini16 thermal cycler) | Cambio | MP-QP-1016-01 | |
| Homogenizer (Precellys Evolution Touch Homogenizer) | Bertin Instruments | EQ02520-300 | |
| Cold Racks (0.2-0.5mL) (PCR Mini-cooler with transparent lid) | BRAND | 781260 | |
| Pipettor | |||
| (Pipetman L Fixed F1000L, 1000 uL) | Gilson | SKU: FA10030 | |
| (Pipetman L Fixed F100L, 100 uL) | Gilson | SKU: FA10024 | |
| (Pipetman L Fixed F10L, 10 uL) | Gilson | SKU: FA10020 | |
| (Pipetman L Fixed F1L, 1 uL) | Gilson | SKU: FA10025 | |
| (Pipetman L Fixed F20L, 20 uL) | Gilson | SKU: FA10021 | |
| (Pipetman L Fixed F250L, 250 uL) | Gilson | SKU: FA10026 | |
| Fluorometer (Qubit 4 Fluorometer) | Thermofisher scientific/Fisher scientific | Q33238 | |
| Laptop (Any brand with ~2 GB of drive space, minimum of 512 GB storage space, msi installer [GPU]) | |||
| Microcentrifuge (Refrigerated centrifuge) | Thermofisher scientific/Fisher scientific | 75004081 | |
| Vortex mixer (Basic vortex mixer) | Thermofisher scientific/Fisher scientific | 88882011 | |
| Magnetic rack (DynaMag -2 Magnet) | Thermofisher scientific/Fisher scientific | 12321D | |
| Sequencing device (MinION) | Oxford Nanopore Technologies | MinION Mk1B | |
| RNA Extraction | |||
| RNA extraction kit (Qiagen RNEasy Mini Kit 250) | Qiagen | 74106 | |
| RNA stabilizing reagents | |||
| (RNA later) | Invitrogen | AM7020 | |
| (DNA/RNA Shield) | Zymo Research | R1100-50 | |
| PCR | |||
| Nuclease-free Water (Nuclease-free Water [not DEPC-treated]) | Thermofisher scientific/Fisher scientific | AM9937 | |
| Master mix for first strand cDNA synthesis (LunaScript RT SuperMix Kit) | New England Biolabs | E3010S | |
| DNA amplification master mix (Q5® Hot Start High-Fidelity 2X Master Mix [NEB]) | New England Biolabs | M0494L | |
| Primer (Scheme) (Custom DNA oligos) | Invitrogen | ||
| SPRI Bead Clean-up | |||
| SPRI beads (Aline Biosciences PCR Clean DX ) | Cambio | AL-AC1003-50 | |
| Ethanol, Pure Absolute, >99.8% (GC) [Riedel-De Haen] | Merck | 818760 | |
| Short Fragment buffer (SFB expansion pack) | Oxford Nanopore Technologies | EXP-SFB001 | |
| DNA Quantification | |||
| DNA quantification kit (Qubit® dsDNA HS Assay Kit) | Thermofisher scientific/Fisher scientific | Q32854 | |
| DNA quantification assay tubes (Qubit™ Assay Tubes) | Thermofisher scientific/Fisher scientific | Q32856 | |
| End Prep and barcoding (Qubit™ Assay Tubes) | |||
| End Prep master mix (NEBNext Ultra End Repair/dA-Tailing Module) | New England Biolabs | E7546L | |
| Barcoding kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 | |
| (Native Barcoding Expansion 1-12) | EXP-NBD104 | ||
| (Native Barcoding Expansion 13-24) | EXP-NBD114 | ||
| (Native Barcoding Expansion 96) | EXP-NBD196 | ||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 | |
| (not compatible) | (not compatible) | ||
| (Native Barcoding Kit 24 V14) | SQK-NBD114.24 | ||
| (Native Barcoding Kit 96 V14) | SQK-NBD114.96 | ||
| Ligation mastermix (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter Ligation | |||
| Adapter ligation master mix | |||
| (NEBNext Quick Ligation Module) | New England Biolabs | E6056S | |
| (NEBNext Ultra II Ligation Module) | New England Biolabs | E7595S | |
| (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter mix | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-AMII001 | |
| (Adapter Mix II [AMII]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-NBA114 | |
| (Native adapter [NA]) | |||
| Sequencing | |||
| Flowcell priming kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-FLP002 | |
| (Flush Buffer [FB]) | |||
| (Flush Tether [FT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-FLP004 | |
| (Flow Cell Flush [FCF]) | |||
| (Flow Cell Tether [FCT]) | |||
| Ligation Sequencing Kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 SQK-LSK109 | |
| Adapter Mix (Adapter Mix [AMX]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SQB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LB]) | |||
| Sequencing Tether (Sequencing Tether [SQT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 SQK-LSK114 | |
| Adapter Mix (Ligation Adapter [LA]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LIB]) | |||
| Sequencing Tether (Flow Cell Tether [FCT]) | |||
| Library solution (Library solution [LIS]) | |||
| Flush buffer (Flow Cell Flush [FCF]) | |||
| Flow Cell | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 FLO-MIN106D | |
| (Flow Cell [R9.4.1]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 FLO-MIN114 | |
| (Flow Cell [R10.4.1]) | |||
| Flow Cell wash | |||
| Flowcell wash kit (Flow cell wash kit) | Oxford Nanopore Technologies | EXP-WSH004 | |
| Consummables | |||
| Surface decontaminant | |||
| (DNA Away Surface Decontaminant, Squeeze Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7010PK | |
| (RNase Away Surface Decontaminant, Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7002PK | |
| PCR 8-Tube Strip 0.2ml, individual cap (PCR 8-Tube Strip 0.2ml, with Individual attached Flat Caps, Sterile, DNAse/RNAse, Pyrogen Free,Natural [Greiner]) | Greiner | 608281 | |
| PCR Tube 0.2ml (PCR Tube 0.2ml, Natural [Domed Cap] Bagged in 500s, Non-Sterile [Greiner]) | Greiner | 671201 | |
| 1000µL Filter Tips (500) (Stacked 1000µL Filter Tips [500]) | Thermofisher scientific/Fisher scientific | 11977724 | |
| 100µL Filter Tips (1000) | Thermofisher scientific/Fisher scientific | 11947724 | |
| 10µL Filter Tips (1000) (Stacked 100µL Filter Tips [1000]) | Thermofisher scientific/Fisher scientific | 11907724 | |
| Reinforced tubes tubes (2ml) with screw caps and o-rings (Fisherbrand™ Bulk tubes) | Thermofisher scientific/Fisher scientific | 15545809 | |
| Microcentrifuge tube (1.5ml) (1.5 ml Eppendorf Tubes [500]) | Eppendorf | 1229888 | |
| DNA LoBind Tubes (1.5ml) (DNA LoBind Tubes) | Thermofisher scientific/Fisher scientific | 10051232 | |
| Cryobabies labels | |||
| Gloves (S/M/L) | |||
| Paper towel |
参考文献
- Rupprecht, C. E. Rhabdoviruses: rabies virus. Medical Microbiology. , University of Texas Medical Branch. Galveston, TX. (1996).
- Rabies. World Health Organization. , Available from: https://www.who.int/news-room/fact-sheets/detail/rabies (2023).
- Health Organization, W. orld WHO Expert Consultation on Rabies: WHO TRS N°1012. World Health Organization. , Available from: https://www.who.int/publications-detail-redirect/WHO-TRS-1012 (2018).
- Benavides, J. A., et al. Defining new pathways to manage the ongoing emergence of bat rabies in Latin America. Viruses. 12 (9), 1002(2020).
- Hampson, K. Estimating the global burden of endemic canine rabies. PLoS Neglected Tropical Diseases. 9 (4), e0003709(2015).
- Global genomic surveillance strategy for pathogens with pandemic and epidemic potential, 2022-2032. World Health Organization. , Available from: https://www.who.int/publications-detail-redirect/978924004679 (2022).
- Tsai, K. J., et al. Emergence of a sylvatic enzootic formosan ferret badger-associated rabies in Taiwan and the geographical separation of two phylogenetic groups of rabies viruses. Veterinary Microbiology. 182, 28-34 (2016).
- Chiou, H. -Y., et al. Molecular characterization of cryptically circulating rabies virus from ferret badgers, Taiwan. Emerging Infectious Diseases. 20 (5), 790-798 (2014).
- Sabeta, C. T., Mansfield, K. L., McElhinney, L. M., Fooks, A. R., Nel, L. H. Molecular epidemiology of rabies in bat-eared foxes (Otocyon megalotis) in South Africa. Virus Research. 129 (1), 1-10 (2007).
- Scott, T. P. Complete genome and molecular epidemiological data infer the maintenance of rabies among kudu (Tragelaphus strepsiceros) in Namibia. PLoS One. 8 (3), e58739(2013).
- Lembo, T., et al. Exploring reservoir dynamics: a case study of rabies in the Serengeti ecosystem. The Journal of Applied Ecology. 45 (4), 1246-1257 (2008).
- Coetzee, P., Nel, L. H. Emerging epidemic dog rabies in coastal South Africa: a molecular epidemiological analysis. Virus Research. 126 (1-2), 186-195 (2007).
- Ou de Munnink, B. B. First molecular analysis of rabies virus in Qatar and clinical cases imported into Qatar, a case report. International Journal of Infectious Diseases. 96, 323-326 (2020).
- Smith, J., et al. Case report: Rapid ante-mortem diagnosis of a human case of rabies imported into the UK from the Philippines. Journal of Medical Virology. 69, 150-155 (2003).
- Mahardika, G. N. K., et al. Phylogenetic analysis and victim contact tracing of rabies virus from humans and dogs in Bali, Indonesia. Epidemiology and Infection. 142 (6), 1146-1154 (2014).
- Tohma, K., et al. Molecular and mathematical modeling analyses of inter-island transmission of rabies into a previously rabies-free island in the Philippines. Infection, Genetics and Evolution. 38, 22-28 (2016).
- Tohma, K., et al. Phylogeographic analysis of rabies viruses in the Philippines. Infection, Genetics and Evolution. 23, 86-94 (2014).
- Saito, M., et al. Genetic diversity and geographic distribution of genetically distinct rabies viruses in the Philippines. PLoS Neglected Tropical Diseases. 7 (4), e2144(2013).
- Biek, R., Henderson, J. C., Waller, L. A., Rupprecht, C. E., Real, L. A. A high-resolution genetic signature of demographic and spatial expansion in epizootic rabies virus. Proceedings of the National Academy of Sciences. 104 (19), 7993-7998 (2007).
- Reddy, G. B., et al. Molecular characterization of Indian rabies virus isolates by partial sequencing of nucleoprotein (N) and phosphoprotein (P) genes. Virus Genes. 43, 13-17 (2011).
- David, D., Dveres, N., Yakobson, B. A., Davidson, I. Emergence of dog rabies in the northern region of Israel. Epidemiology and Infection. 137 (4), 544-548 (2009).
- Benjathummarak, S. Molecular genetic characterization of rabies virus glycoprotein gene sequences from rabid dogs in Bangkok and neighboring provinces in Thailand, 2013-2014. Archives of Virology. 161 (5), 1261-1271 (2016).
- Denduangboripant, J., et al. Transmission dynamics of rabies virus in Thailand: implications for disease control. BMC Infectious Diseases. 5, 52(2005).
- Talbi, C., et al. Phylodynamics and human-mediated dispersal of a zoonotic virus. PLoS Pathogens. 6 (10), e1001166(2010).
- Bourhy, H., et al. Revealing the micro-scale signature of endemic zoonotic disease transmission in an African urban setting. PLoS Pathogens. 12 (4), e1005525(2016).
- Zinsstag, J., et al. Vaccination of dogs in an African city interrupts rabies transmission and reduces human exposure. Science Translational Medicine. 9 (421), (2017).
- Yakovleva, A., et al. Tracking SARS-COV-2 variants using Nanopore sequencing in Ukraine in 2021. Scientific Reports. 12, 15749(2022).
- Mannsverk, S., et al. SARS-CoV-2 variants of concern and spike protein mutational dynamics in a Swedish cohort during 2021, studied by Nanopore sequencing. Virology Journal. 19, 164(2022).
- Soufi, M., et al. Fast and easy nanopore sequencing workflow for rapid genetic testing of familial Hypercholesterolemia. Frontiers in Genetics. 13, 836231(2022).
- Cabibbe, A. M. Application of targeted next-generation sequencing assay on a portable sequencing platform for culture-free detection of drug-resistant tuberculosis from clinical samples. Journal of Clinical Microbiology. 58 (10), 00632(2020).
- Xu, Y., et al. Nanopore metagenomic sequencing of influenza virus directly from respiratory samples: diagnosis, drug resistance and nosocomial transmission, United Kingdom, 2018/19 influenza season. Euro Surveillance. 26 (27), 2000004(2021).
- Stubbs, S. C. B., et al. Assessment of a multiplex PCR and Nanopore-based method for dengue virus sequencing in Indonesia. Virology Journal. 17, 24(2020).
- Croville, G., et al. Rapid whole-genome based typing and surveillance of avipoxviruses using nanopore sequencing. Journal of Virological Methods. 261, 34-39 (2018).
- Theuns, S., et al. Nanopore sequencing as a revolutionary diagnostic tool for porcine viral enteric disease complexes identifies porcine kobuvirus as an important enteric virus. Scientific Reports. 8, 9830(2018).
- O'Donnell, V. K., et al. Rapid sequence-based characterization of African swine fever virus by use of the Oxford Nanopore MinION sequence sensing device and a companion analysis software tool. Journal of Clinical Microbiology. 58, 01104-01119 (2019).
- Brito, A. F. Global disparities in SARS-CoV-2 genomic surveillance. Nature Communications. 13, 7003(2022).
- ONT login/register. Oxford Nanopore Technology. , Available from: https://nanoporetech.com/login-register (2023).
- Software Downloads. Oxford Nanopore Technology. , Available from: https://community.nanoporetech.com/downloads (2023).
- GitHub. , Available from: https://github.com/ (2023).
- Brunker, K. Artic-rabv. , Available from: https://github.com/kirstyn/artic-rabv (2022).
- Campbell, K. MADDOG: Method for Assignment, Definition and Designation of Global Lineages. , Available from: https://github.com/KathrynCampbell/MADDOG (2022).
- Campbell, K. RABV-GLUE. Centre for Virus Research. , Available from: https://github.com/KathrynCampbell/MADDOG (2022).
- Itokawa, K., Sekizuka, T., Hashino, M., Tanaka, R., Kuroda, M. Disentangling primer interactions improves SARS-CoV-2 genome sequencing by multiplex tiling PCR. PLoS ONE. 15 (9), e0239403(2020).
- Davis, M. W., Jorgensen, E. M. ApE, A plasmid editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 818619(2022).
- Döring, M., Pfeifer, N. openPrimeR: Multiplex PCR primer design and analysis. , (2023).
- Quick, J. Multiplex PCR method for MiniON and Illumina sequencing of Zika and other virus genomes directly from clinical samples. Nature Protocols. 12 (6), 1261-1276 (2017).
- Laboratory Techniques in Rabies. World Health Organization. 1, Available from: https://apps.who.int/iris/bitstream/handle/10665/310836/9789241515153-eng.pdf (2018).
- Lembo, T. Partners for Rabies Prevention. The blueprint for rabies prevention and control: a novel operational toolkit for rabies elimination. PLoS Neglected Tropical Diseases. 6 (2), e1388(2012).
- Terrestrial Manual Online Access. World Organization for Animal Health. , Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals/terrestrial-manual-online-access/ (2023).
- Mauti, S. Field postmortem rabies rapid immunochromatographic diagnostic test for resource-limited settings with further molecular applications. Journal of Visualized Experiments. (160), e60008(2020).
- Patrick, E., et al. Enhanced rabies surveillance using a direct rapid immunohistochemical test. Journal of Visualized Experiments. (146), e59416(2019).
- Lembo, T., et al. Evaluation of a direct, rapid immunohistochemical test for rabies diagnosis. Emerging Infectious Diseases. 12 (2), 310-313 (2006).
- Marston, D. A., et al. Pan-lyssavirus real time RT-PCR for rabies diagnosis. Journal of Visualized Experiments. (149), e59709(2019).
- Brunker, K. DNA quantification using the Qubit fluorometer. , Available from: https://www.protocols.io/view/dna-quantification-using-the-qubit-fluorometer-bc6vize6 (2020).
- Quick, J. DNA quantification using the Quantus fluorometer. , Available from: https://www.protocols.io/view/dna-quantification-using-the-quantus-fluorometer-7pzhmp6 (2020).
- Guppy protocol. Nanopore Community. , Available from: https://community.nanoporetech.com/protocols/Guppy-protocol/v/gpb_2003_v1_revaq_14dec2018 (2023).
- Flow Cell Wash Kit (EXP-WSH004). Nanopore Community. , Available from: https://community.nanoporetech.com/protocols/flow-cell-wash-kit-exp-wsh004/v/wfc_9120_v1_revh_08dec2020 (2023).
- Core Pipeline - arctic pipeline. , Available from: https://artic.readthedocs.io/en/latest/minion/ (2023).
- Samtools. , Available from: https://www.htslib.org (2023).
- Campbell, K., et al. Making genomic surveillance deliver: A lineage classification and nomenclature system to inform rabies elimination. PLoS Pathogens. 18 (5), e1010023(2022).
- Brunker, K., et al. Rapid in-country sequencing of whole virus genomes to inform rabies elimination programmes. Wellcome Open Research. 5, 3(2020).
- Bull, R. A., et al. Analytical validity of nanopore sequencing for rapid SARS-CoV-2 genome analysis. Nature Communications. 11, 6272(2020).
- Okeke, I. N., Ihekweazu, C. The importance of molecular diagnostics for infectious diseases in low-resource settings. Nature Reviews. Microbiology. 19 (9), 547-548 (2021).
- Inzaule, S. C., Tessema, S. K., Kebede, Y., Ouma, A. E. O., Nkengasong, J. N. Genomic-informed pathogen surveillance in Africa: opportunities and challenges. The Lancet Infectious Diseases. 21 (9), 281-289 (2021).
- Kennedy, L. Integrating contact tracing and whole-genome sequencing to track the elimination of dog-mediated rabies: an observational and genomic study. eLife. , (2023).
- Pallerla, S. R. Diagnosis of pathogens causing bacterial meningitis using Nanopore sequencing in a resource-limited setting. Annals of Clinical Microbiology and Antimicrobials. 21, 39(2022).
- Quick, J. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。