Method Article
Nanopor Teknolojisi Kullanılarak Kuduz Virüsünün Hızlı Karakterizasyonu için Tüm Genom Dizilimi
Bu Makalede
Özet
Burada, nanopor teknolojisini kullanarak kuduz virüsü (RABV) genomlarını karakterize etmek için hızlı ve uygun maliyetli bir iş akışı sunuyoruz. İş akışı, kuduz kontrol önlemlerine rehberlik etmek için dolaşımdaki RABV soyları ve bunların bölgesel filogeniler içindeki yerleşimleri hakkında bilgi sağlayarak yerel düzeyde genomik bilgili sürveyansı desteklemeyi amaçlamaktadır.
Özet
Genomik veriler, bulaşıcı hastalıkların bulaşmasını ve coğrafi yayılımını izlemek için kullanılabilir. Bununla birlikte, genomik sürveyans için gereken dizileme kapasitesi, köpek aracılı kuduzun ve/veya vampir yarasalar gibi vahşi yaşam yoluyla bulaşan kuduzun önemli halk sağlığı ve ekonomik kaygılar oluşturduğu birçok düşük ve orta gelirli ülkede (LMIC'ler) sınırlı kalmaktadır. Burada, nanopor teknolojisini kullanarak numuneden diziye ve yorumlamaya hızlı ve uygun maliyetli bir iş akışı sunuyoruz. Numune toplama ve kuduz teşhisi için protokoller kısaca açıklanır, ardından multipleks polimeraz zincir reaksiyonu (PCR) için primer tasarımı ve optimizasyonu, modifiye edilmiş, düşük maliyetli bir dizileme kütüphanesi hazırlığı, canlı ve çevrimdışı baz çağrısı ile dizileme, genetik soy ataması ve filogenetik analiz dahil olmak üzere optimize edilmiş tüm genom dizileme iş akışının ayrıntıları gelir. İş akışının uygulanması gösterilir ve boru hattı doğrulaması, primer optimizasyonu, negatif kontrollerin dahil edilmesi ve bölgesel ve küresel filogenilerde sınıflandırma ve yerleştirme için halka açık verilerin ve genomik araçların (GLUE, MADDOG) kullanımı gibi yerel dağıtım için kritik adımlar vurgulanır. İş akışının geri dönüş süresi 2-3 gündür ve maliyet, 96 örnek çalıştırma için örnek başına 25 ABD Doları ile 12 örnek çalıştırma için örnek başına 80 ABD Doları arasında değişir. LMIC'lerde kuduz virüsü genomik sürveyansının kurulmasının uygulanabilir olduğu ve 2030 yılına kadar köpek aracılı insan kuduz ölümlerinin küresel hedefine doğru ilerlemeyi destekleyebileceği ve ayrıca yaban hayatı kuduzunun yayılmasının daha iyi izlenmesini destekleyebileceği sonucuna vardık. Ayrıca platform, diğer patojenler için uyarlanabilir ve salgın ve pandemi hazırlığına katkıda bulunan çok yönlü bir genomik kapasite oluşturmaya yardımcı olur.
Giriş
Kuduz virüsü (RABV), Rhabdoviridae familyasından memelilerde ölümcül bir nörolojik hastalığa neden olan bir lyssavirüstür1. Kuduz aşılama ile %100 önlenebilir olmasına rağmen, endemik ülkelerde önemli bir halk sağlığı ve ekonomik sorun olmaya devam etmektedir. Her yıl meydana geldiği tahmin edilen 60.000 insan kuduz ölümünün% 95'inden fazlası, köpeklerin birincil rezervuar olduğu Afrika ve Asya'dadır2. Buna karşılık, köpek aşılaması, Batı Avrupa, Kuzey Amerika ve Latin Amerika'nın çoğunda köpek aracılı kuduzun ortadan kaldırılmasına yol açmıştır. Bu bölgelerde, kuduz rezervuarları artık yarasalar, rakunlar, kokarcalar ve yabani köpekgiller gibi vahşi yaşamla sınırlıdır3. Latin Amerika'da, yaygın vampir yarasa, gece kanla besleme sırasında yarasalardan hem insanlara hem de çiftlik hayvanlarına düzenli yayılma nedeniyle sorunlu bir kuduz kaynağıdır4. Kuduzun yıllık küresel ekonomik etkisinin 8,6 milyar dolar olduğu tahmin edilmektedir ve hayvan kayıpları %6'dır5.
Viral patojenlerden elde edilen dizi verileri, enfeksiyonların zamanlaması ve kaynağına ilişkin meta verilerle birleştirildiğinde sağlam epidemiyolojik içgörüler sağlayabilir6. RABV için, salgınlarınkökenini araştırmak için sıralama kullanılmıştır 7,8, yaban hayatı veya evcil köpeklerlekonakçı ilişkilerini belirlemek 8,9,10,11,12 ve insan vakalarının kaynaklarını izlemek13,14. Filogenetik analiz kullanılarak yapılan salgın araştırmaları, kuduzun Endonezya'nın daha önce kuduzsuz olan Bali eyaletinde, yakındaki endemik bölgelerden Kalimantan veya Sulawesi15'ten tek bir girişle ortaya çıktığını göstermiştir. Bu arada, Filipinler'de, Romblon Eyaleti, Tablas Adası'ndaki bir salgının ana Luzon16 adasından tanıtıldığı kanıtlandı. Viral genomik veriler, kontrol önlemlerini coğrafi olarak hedeflemek için gerekli olan patojen bulaşma dinamiklerini daha iyi anlamak için de kullanılmıştır. Örneğin, RABV'nin genomik karakterizasyonu, 17,18,19 sınıflarının coğrafi kümelenmesini, soyların20,21,22 birlikte dolaşımını, insan aracılı viral hareketi 17,23,24 ve metapopülasyon dinamiklerini 25,26 göstermektedir.
Hastalık izleme, SARS-CoV-2 pandemisine yanıt olarak dizileme kapasitesindeki küresel artışla geliştirilen genomik sürveyansın önemli bir işlevidir. Genomik sürveyans, endişe verici SARS-COV-2 varyantlarının gerçek zamanlı izlenmesinidesteklemiştir 27,28 ve ilgili karşı önlemler6. Nanopor teknolojisi gibi erişilebilir dizileme teknolojisindeki gelişmeler, hem insan 29,30,31,32 hem de hayvan 33,34,35 patojenlerinin hızlı dizilimi için geliştirilmiş ve daha uygun fiyatlı protokollere yol açmıştır. Bununla birlikte, kuduz endemik birçok ülkede, SARS-CoV-2 dizileme kapasitesindeki küresel eşitsizliklerin gösterdiği gibi, patojen genomik sürveyansının operasyonel hale getirilmesinin önünde hala engeller vardır36. Laboratuvar altyapısındaki, tedarik zincirlerindeki ve teknik bilgideki sınırlamalar, genomik sürveyansın kurulmasını ve rutinleştirilmesini zorlaştırmaktadır. Bu yazıda, kaynakların sınırlı olduğu ortamlarda RABV sürveyansı için optimize edilmiş, hızlı ve uygun fiyatlı bir tüm genom dizileme iş akışının nasıl uygulanabileceğini gösteriyoruz.
Protokol
Çalışma, Ulusal Tıbbi Araştırma Enstitüsü Tıbbi Araştırma Koordinasyon Komitesi tarafından onaylanmıştır (NIMR/HQ/R.8a/vol. IX/2788), Tanzanya'da Bölgesel İdare ve Yerel Yönetim Bakanlığı (AB.81/288/01) ve Ifakara Sağlık Enstitüsü Kurumsal İnceleme Kurulu (IHI/IRB/No:22-2014); Kenya'daki Nairobi Üniversitesi Tropikal ve Bulaşıcı Hastalıklar Enstitüsü (P947/11/2019) ve Kenya Tıbbi Araştırma Enstitüsü (KEMRI-SERU; protokol No. 3268); ve Filipinler'deki Tropikal Tıp Araştırma Enstitüsü (RITM), Sağlık Bakanlığı (2019-023). Nijerya menşeli örneklerin dizilimi, ulusal sürveyansın bir parçası olarak toplanan arşivlenmiş teşhis materyali üzerinde gerçekleştirilmiştir.
NOT: Bölüm 1-4 önkoşuldur. Bölüm 5-16, RABV nanopor dizilimi için numuneden diziye ve yorumlamaya iş akışını açıklar (Şekil 1). Protokolde darbeli santrifüjleme gerektiren sonraki adımlar için, 5-15 s boyunca 10-15.000 x g'da santrifüjleyin.
1. Sıralama ve veri analizi için hesaplama ortamı kurulumu
- Oxford Nanopore Technology (ONT) web sitesini37 açın ve nanopora özgü kaynaklara erişmek için bir hesap oluşturun.
- Oturum açın ve ONT sıralama ve temel arama yazılımını kurun38.
- GitHub39'u açın ve bir hesap oluşturun.
- artic-rabv40 ve MADDOG depoları41'e gidin ve kurulum talimatlarını izleyin.
2. Multipleks astar şemasını tasarlayın veya güncelleyin
NOT: Mevcut RABV şemaları artic-rabv deposu40'ta mevcuttur. Yeni bir coğrafi alanı hedeflerken, yeni bir şema tasarlanmalı veya mevcut bir şema ek çeşitliliği içerecek şekilde değiştirilmelidir.
- Çalışma alanındaki çeşitliliği temsil etmek için bir genom referans seti seçin; bu tipik olarak halka açık bir dizi dizidir (örneğin, NCBI GenBank'tan) veya ön kurum içi verilerdir. NCBI dizilerini ve ilişkili meta verileri filtrelemek ve indirmek için bir RABV dizisi veri kaynağı olan RABV-GLUE42'yi kullanmak için adım 2.1.1'i izleyin.
NOT: Tam genomlara sahip referans dizileri seçin (yani, boşluklar ve maskelenmiş bazlar olmadan). Astar tasarımı için referans seti olarak en fazla 10 sekans seçilmesi önerilir. Mevcut dizi verileri eksikse veya çalışma alanını temsil etmiyorsa, Ek Dosya 1'deki 43,44,45 numaralı tavsiyeye bakın.- RABV-GLUE'daki Dizi Verileri açılır menüsünden NCBI RABV Dizileri sayfasına gidin. Mevcut tüm verilere erişmek için Kuduz Virüsü (RABV) bağlantısını tıklayın veya ilgilendiğiniz belirli bir türü seçin. İstenen kriterlere (ör. menşe ülke, sıra uzunluğu) uyan filtreler eklemek için filtre seçeneğini kullanın. Dizileri ve meta verileri indirin.
- Primal Scheme46 tarafından sağlanan talimatları izleyerek multipleks polimeraz zincir reaksiyonu (PCR) için bir primer şeması oluşturun. Düşük kaliteli numuneleri sıralamak için 50 bp örtüşmeye sahip 400 bp'lik bir şema önerilir. Tüm çıktıları indirin ve kaydedin (dosya veya astar adlarını düzenlemeyin).
NOT: Şema, bundan böyle 'indeks referansı' olarak anılacak olan giriş fasta'daki ilk diziye indekslenecektir (Şekil 2). Primer performansını optimize etme seçenekleri için Ek Dosya 1'e bakın.
3. RAMPART ve ARTIC biyoinformatik boru hatlarını kurun
- RAMPART ve ARTIC biyoinformatik ardışık düzen için giriş/çıkış dosyalarını yönetmek üzere bir dizin yapısı ayarlamak için Ek Dosya 2'ye bakın.
4. Biyogüvenlik ve laboratuvar kurulumu
- Potansiyel olarak kuduz pozitif numuneleri biyogüvenlik seviyesi (BSL) 2 veya 3 koşullarında kullanın.
- Laboratuvar personelinin maruziyet öncesi kuduz aşısını tamamladığından ve Dünya Sağlık Örgütü (WHO) tavsiyelerine göre bağışıklığın izlenmesinden geçtiğinden emin olun3.
- Laboratuvar için ulusal veya uluslararası yönergelere uygun özel standart çalışma prosedürlerinin ve risk değerlendirmelerinin yapıldığından emin olun.
- Gerekli laboratuvar kurulumu: PCR öncesi ve sonrası alanlar arasında fiziksel ayrımı koruyarak kontaminasyonu en aza indirin. Sınırlı alana sahip laboratuvarlarda veya saha içi laboratuvar ortamlarında, kontaminasyonu en aza indirmek için taşınabilir torpido gözü veya geçici laboratuvar istasyonları kullanın.
- Bu protokolde, aşağıdakiler için ayrı alanlar belirlendiğinden emin olun:
- Numune ekstraksiyonu: Biyolojik materyali işlemek ve inaktivasyon ve RNA ekstraksiyonu gerçekleştirmek için bir BSL2/3 kabini/torpido gözü kurun.
- Şablon alanı: Önceden hazırlanmış reaksiyon ana karışımına şablon (RNA/cDNA) eklenmesi için bir BSL1 kabini/torpido gözü kurun.
- Ana karışım alanı: Reaktif ana karışımlarının hazırlanması için belirlenmiş bir temiz alan (BSL1 kabini/torpido gözü) kurun. Bu alanda şablon olmamalıdır.
- PCR sonrası alan: Amplikonlar ve dizileme kitaplığı hazırlığı üzerinde çalışmak için ayrı bir alan oluşturun.
NOT: Tüm alanlar kullanımdan önce ve sonra bir yüzey dekontaminantı ile temizlenmeli ve ultraviyole (UV) ile sterilize edilmelidir.
5. Saha numunesi toplama ve teşhis
NOT: Numuneler, kişisel koruyucu ekipman giyen ve atıfta bulunulan standart prosedürler47,48,49 izlenerek eğitimli ve aşılanmış personel tarafından toplanmalıdır.
- Numuneyi, Mauti ve ark.50'de ayrıntılı olarak açıklandığı gibi foramen magnum (yani oksipital yol) yoluyla toplayın.
- Hızlı tanı testleri ile sahada kuduz teşhisi koyun ve doğrudan floresan antikor testi (DFA), doğrudan hızlı immünohistokimyasal test (DRIT)51,52 veya gerçek zamanlı ters transkripsiyon (RT)-PCR 53 gibi önerilen prosedürleri47 kullanarak laboratuvarda onaylayın.
- RNA ekstraksiyonu için doğrulanmış pozitif beyin örnekleri kullanın veya 2-3 ay boyunca -20 ° C'de veya daha uzun süre -80 ° C'de bir dondurucuda saklayın. Uygun bir DNA/RNA stabilizasyon ortamı kullanarak depolama ve taşıma için RNA'yı koruyun.
6. Numune hazırlama ve RNA ekstraksiyonu (3 saat)
NOT: Numune tipine uygun bir spin kolon bazlı viral RNA ekstraksiyon kiti kullanın.
- 2 mL'lik bir PCR tüpünü 1,4 mm seramik boncuklarla dolu yaklaşık 200 μL'lik bir tüple doldurarak iki seramik boncuk tüpü hazırlayın ve tüpü etiketleyin.
- RNA ekstraksiyon kitinde sağlanan önerilen lizis tamponu hacmini etiketli PCR tüpüne ekleyin.
- Ahşap bir aplikatör kullanarak kuduz enfeksiyonu ile doğrulanmış beyin örneğinden yaklaşık 3 mm'lik bir küp alın ve numune kimliği ve 100 μL nükleaz içermeyen su ile etiketli bir tüpe negatif kontrol etiketli tüpe koyun.
NOT: Numune maruziyetini sınırlamak için kapalı tüp boncuk bazlı homojenizasyon kullanın. Mümkün değilse, diğer uygun mekanik bozucular (örn. rotor tabanlı) veya manuel bir mikro tokmak kullanın. Bununla birlikte, bunlar dokuyu bozmak için sert yüzeyde boncuk dövmekten daha az etkili olabilir (doku örnekleri belirli depolama ortamlarında sertleşebilir). - Tahta bir aplikatör çubuğu kullanarak beyin dokusunu manuel olarak parçalayın ve ardından tam doku homojenizasyonu sağlanana kadar maksimum hızda girdap yapın.
- Lizatı üreticinin talimatlarına göre santrifüjleyin ve süpernatanı yeni bir etiketli mikrosantrifüj tüpüne aktarmak için bir pipet kullanın. Bu süpernatanı yalnızca sonraki adımlarda kullanın.
- Saflaştırılmış RNA elde etmek için RNA ekstraksiyon kitinin spin kolonu talimatlarını izleyin.
- Buraya bir negatif ekstraksiyon kontrolü (NEC) ekleyin ve sıralama aşamasına kadar ilerleyin.
7. cDNA hazırlığı (20 dk)
- Ana karışım alanında, işlenecek numune ve kontrol sayısına göre ilk iplikçik cDNA sentezi için bir ana karışım hazırlayın (yeterli reaktifi sağlamak için %10'luk fazla hacimle; Tablo 1). Bu aşamada şablonsuz bir denetim (NTC) eklenmelidir.
- 0.2 mL PCR şerit tüplerini etiketleyin ve ana karışımın 5 μL'sini tüplere ayırın.
- Hazırlanan tüpleri şablon alanına alın. NEC dahil olmak üzere etiketli her tüpe 5 μL RNA ekleyin. NTC'ye 5 μL nükleaz içermeyen su (NFW) ekleyin.
- Tablo 1'de belirtilen koşulları izleyerek bir termal döngüleyicide inkübe edin.
NOT: İsteğe bağlı duraklama noktası: cDNA, gerekirse -20 °C'de 1 aya kadar saklanabilir, ancak PCR'ye geçmek tercih edilir.
8. Primer havuz stoğu hazırlama (1 saat)
NOT: Bu adım, yalnızca tek tek primerlerden yeni stoklar yapılırsa gereklidir, bundan sonra önceden hazırlanmış stok çözeltileri kullanılabilir.
- Ana karışım alanında 100 μM stoktan oluşan bir astar havuzu hazırlayın.
- Liyofilize primerleri, her biri 100 μM konsantrasyonda 1x tris-EDTA (TE) tamponunda veya NFW'de yeniden süspanse edin. İyice girdap yapın ve aşağı doğru döndürün.
NOT: Aşağıdaki adımlarda, amplikon örtüşmelerini çevreleyen primerler arasındaki etkileşimleri önlemek için tek tek primerler (Havuz A olarak adlandırılır) ve çift numaralı (Havuz B olarak adlandırılır) olmak üzere iki primer havuzuna ayrılır. Bu primer havuzları, hedef genomu kapsayan üst üste binen 400 bp amplikon üretir. - Tüm tek sayılı astarları bir tüp rafına yerleştirin. "Primer şema adı - Havuz A (100 μM)" etiketli 1,5 mL'lik bir mikrosantrifüj tüpüne her primerden 5 μL ekleyerek bir primer havuzu stoğu oluşturun.
- İşlemi tüm çift numaralı primerler için tekrarlayın ve "primer şema adı - Havuz B (100 μM)" olarak etiketleyin.
- 10 μM primer stokları oluşturmak için her bir primer havuzunu moleküler dereceli suda 1:10 oranında seyreltin.
NOT: 10 μM primer seyreltmelerinin birden fazla alikotunu yapın ve bozunma veya kontaminasyon durumunda bunları dondurun.
9. Multipleks PCR (5 saat)
- Ana karışım alanındaki her primer havuzu için bir tane olmak üzere iki PCR ana karışımı hazırlayın.
- Astar başına 0.015 μM'lik bir son konsantrasyon kullanın. Aşağıdaki formülü kullanarak PCR reaksiyonu için gerekli primer havuzu hacmini hesaplayın (Tablo 2):
Primer havuzu hacmi = primer sayısı x reaksiyon hacmi x 0.015/konsantrasyon (μM) primer stoğu
- Astar başına 0.015 μM'lik bir son konsantrasyon kullanın. Aşağıdaki formülü kullanarak PCR reaksiyonu için gerekli primer havuzu hacmini hesaplayın (Tablo 2):
- Şablon alanındaki etiketli PCR şerit tüplerine Havuz A ana karışımı ve Havuz B ana karışımının her biri 10 μL'dir. Her numune için, karşılık gelen etiketli primer Havuz A ve B reaksiyonlarının her birine 2.5 μL cDNA (3. adımdan itibaren) ekleyin. Fazla cDNA -20 °C'de saklanabilir.
- Hafifçe vurarak ve nabız santrifüjü ile karıştırın.
- Numuneleri bir PCR makinesinde Tablo 2'de belirtilen koşullarla inkübe edin.
NOT: Program, 5 dakikalık uzun tavlama süresi (yüksek primer sayısı nedeniyle gereklidir) ve uzatma için yeterli olan amplikonların kısa uzunluğu (400 bp) nedeniyle belirli bir uzatma adımı içermez.
10. PCR temizleme ve miktar tayini (3,5 saat)
- Bu noktadan itibaren tüm çalışmaları PCR sonrası alanda gerçekleştirin.
- Aliquot katı fazlı tersinir immobilizasyon (SPRI) boncuklarını ana şişeden mikrosantrifüj tüplerine yerleştirin. 4 °C'de saklayın.
- Bir SPRI boncuğu alikotunu oda sıcaklığına (RT; ~ 20 ° C) ısıtın ve boncuklar çözelti içinde tamamen yeniden süspanse olana kadar iyice girdap yapın.
- 1,5 mL'lik tüplerde, her numune için primer Pool A ve primer Pool B PCR ürünlerini birleştirin. Gerekirse, hacmi 25 μL'ye getirmek için su ekleyin.
- Her numuneye 25 μL SPRI boncuk ekleyin (1:1 boncuk: numune oranı). Yukarı ve aşağı pipetleyerek veya tüpe hafifçe vurarak karıştırın.
- RT'de 10 dakika inkübe edin, ara sıra tüpleri ters çevirin veya hafifçe vurun.
- Boncuklar ve çözelti tamamen ayrılana kadar manyetik bir rafa yerleştirin. Süpernatanı çıkarın ve atın, boncuk peletini rahatsız etmemeye dikkat edin.
- İki kez% 80 etanol ile yıkayın (RT'ye ısıtılır).
- Pelete 200 μL etanol ekleyin. Boncukların düzgün bir şekilde yıkandığından emin olmak için 30 saniye bekleyin.
- Boncuk peletine dokunmamaya çalışarak süpernatanı dikkatlice çıkarın ve atın.
- Peleti ikinci kez yıkamak için 10.8.1-10.8.2 adımlarını tekrarlayın.
- 10 μL'lik bir uç kullanarak tüm etanol izlerini çıkarın. İz etanol buharlaşana kadar havayla kurutun (küçük boncuklarla bu hızlı bir şekilde gerçekleşir, ~ 30 s); Bu olduğunda, pelet parlaktan mata gitmelidir. Aşırı kurutmamaya dikkat edin (pelet çatlıyorsa, çok kurudur), çünkü bu DNA iyileşmesini etkileyecektir.
- Boncukları 15 μL NFW'de yeniden süspanse edin ve RT'de (manyetik rafın dışında) 10 dakika inkübe edin.
- Manyetik rafa geri dönün ve süpernatanı (temizlenmiş ürün) 1,5 mL'lik yeni bir tüpe aktarın.
- Her numunenin ayrı bir tüpte (2 μL ürün + 18 μL NFW) 1:10 seyreltmesini hazırlayın.
NOT: Çapraz bulaşmayı önlemek için bu aşamada çok dikkatli olun. Bir seferde yalnızca bir amplikon tüpü açık olsun. Önce tüplere 18 μL su koyun (temiz bir ana karışım alanında). - protocols.io 54,55'te açıklandığı gibi, oldukça hassas ve spesifik bir florometre kullanarak seyreltilmiş her numunenin DNA konsantrasyonunu ölçün.
11. Normalleştirme (30 dk)
- Toplam 5 μL hacminde her bir numunenin 200 fmol'ü için gereken seyreltilmiş (veya saf) numune hacmini hesaplamak için her numunenin normalizasyon şablonunu (Ek Dosya 3) ve DNA konsantrasyonunu (ng/μL) kullanın.
- Yeni PCR tüplerini etiketleyin ve normalleştirilmiş DNA elde etmek için hesaplanmış NFW hacimleri ve numune ekleyin.
- 200 fmol elde etmek için 5 μL'den fazla seyreltilmiş numune gerekiyorsa, seyreltilmemiş (saf) numuneler için hesaplanan hacmi kullanın.
NOT: İsteğe bağlı duraklama noktası: Bu noktada, temizlenmiş PCR ürünü 4 °C'de 1 haftaya kadar saklanabilir veya gerekirse daha uzun süreli saklama için -20 °C'ye yerleştirilebilir.
12. Son hazırlık ve barkodlama (1,5 saat)
NOT: Sonraki adımlar, nano gözeneklere özgü barkodlama ve ligasyon dizileme kitlerinden belirli reaktiflerin kullanıldığını varsayar (ayrıntılar için Malzeme Tablosuna bakın). Protokol farklı kimya sürümleri arasında aktarılabilir, ancak kullanıcılar üretici talimatlarına göre uyumlu kitleri kullanmaya özen göstermelidir.
- Son onarım ve dA-tailing
- Tablo 3'te belirtilen her numune için son hazırlık reaksiyonunu ayarlayın. Numune sayısına göre bir ana karışım hazırlayın (artı %10 fazlalık). Reaktifler viskoz olduğundan pipetleme yaparken dikkatli olun.
- Normalize DNA'nın her tüpüne (5 μL) 5 μL ana karışım ekleyin. Toplam reaksiyon karışımı 10 μL olmalıdır. Uçları her seferinde değiştirin ve bir seferde yalnızca bir tüpü açık tutun.
- Tablo 3'te belirtilen koşullar altında bir termal döngüleyicide inkübe edin.
- Barkodlama
- Barkodlama kitindeki barkodları 1,25 μL/tüpte PCR şerit tüplerine aktarın ve her numuneye atanan barkodu kaydedin.
- 0.75 μL uç hazırlanmış numuneyi atanmış barkod alikotuna ekleyin.
- Numune sayısına göre (artı %10 fazla) bir ligasyon ana karışımı hazırlayın (Tablo 4).
- Sonuna kadar hazırlanmış numune + barkodlara 8 μL ligasyon ana karışımı ekleyerek toplam 10 μL reaksiyon verin.
- Tablo 4'te belirtilen koşulları kullanarak bir termal döngüleyicide inkübe edin.
- SPRI boncuk temizleme ve DNA miktar tayini
- Kısa parça tamponunu (SFB) RT'de çözün, girdaplama, darbe santrifüjü ile karıştırın ve buz üzerine yerleştirin.
- Tüm barkodlu numuneleri 1,5 mL'lik bir lobind mikrosantrifüj tüpünde bir araya getirin. Temizleme hacmini kullanılamayacak kadar büyük hale getirmemek için, her bir yerel barkodlama reaksiyonundan 12-24 numune (10 μL/numune), 48 numuneye kadar (5 μL/numune) veya 96 numuneye (2,5 μL/numune) kadar havuzlayın.
- Barkodlu havuza 0,4x hacimde SPRI boncuk ekleyin. Hafifçe karıştırın (hafifçe vurarak veya pipetleyerek) ve RT'de 5 dakika inkübe edin.
- Boncuklar peletlenene ve süpernatan tamamen berraklaşana kadar (~2 dakika) numuneleri bir mıknatıs üzerine yerleştirin. Süpernatanı çıkarın ve atın. Boncukları rahatsız etmemeye dikkat edin.
- 250 μL SFB ile iki kez yıkayın.
- Tüpü mıknatıstan çıkarın ve peleti 250 μL SFB'de tamamen yeniden süspanse edin. 30 saniye inkübe edin, darbe santrifüjü yapın ve mıknatısa geri dönün.
- Süpernatanı çıkarın ve atın.
- İkinci bir SFB yıkaması gerçekleştirmek için 12.3.5 adımını tekrarlayın.
- Darbe santrifüjü yapın ve kalan SFB'yi çıkarın.
- Peleti yıkamak için 200 μL %80 (RT) etanol ekleyin. Etanolü çıkarın ve atın, boncuk peletini rahatsız etmemeye dikkat edin. 30 saniye boyunca veya pelet parlaklığını kaybedene kadar havayla kurutun.
- RT'de 22 μL NFW'de 10 dakika boyunca yeniden süspanse edin.
- Mıknatısın üzerine yerleştirin, ~ 2 dakika bekletin, ardından çözeltiyi dikkatlice çıkarın ve temiz bir 1.5 mL mikrosantrifüj tüpüne aktarın.
- Daha önce tarif edildiği gibi DNA konsantrasyonunu elde etmek için 1 μL kullanın (adım 10.13).
NOT: İsteğe bağlı duraklama noktası: Bu noktada, kitaplık 4 °C'de 1 haftaya kadar veya daha uzun süreli depolama için -20 °C'de saklanabilir, ancak adaptör ligasyonu ve sıralamasına devam edilmesi tercih edilir.
13. Sıralama (maksimum 48 saat)
- Bilgisayarı hazırlayın (Önkoşullar bölüm 1-4'e de bakın).
- Yeni verileri depolamak için yeterli alan olup olmadığını (en az 150 GB), eski çalıştırmalardan elde edilen verilerin silinmeden önce yedeklendiğini/bir sunucuya taşındığını ve MinKNOW'un en son sürümünün yüklendiğini kontrol edin.
- Depolanan akış hücresini buzdolabından çıkarın ve RT'ye ulaşmasına izin verin.
- Adaptör ligasyonu (1 saat)
- Adaptör karışımını ve ligaz darbesini santrifüjleyin ve buzun üzerine yerleştirin.
- RT'de çözünme elüsyon tamponu (EB), SFB ve ligasyon tamponu. Girdap, nabız santrifüjü ile karıştırın ve buz üzerine yerleştirin.
- Adaptör ligasyon ana karışımını hazırlayın (Tablo 5), reaktifleri belirtilen sırayla düşük bağlamalı bir tüpte birleştirerek.
NOT: Adaptör ligasyonu ana karışım reaktifleri için alternatifler (Tablo 5), laboratuvardaki mevcudiyete bağlı olarak kullanılabilir. Alternatiflerin listesi için Ek Dosya 3'e ve Malzeme Tablosuna bakın. 200 fmol'e eşdeğer DNA kitaplığı hacmini elde etmek için Ek Dosya 3 çalışma sayfasındaki hesaplamayı kullanın. 20 μL'den az hesaplanırsa, 20 μL'ye kadar NFW ekleyin. - Hafifçe vurma ve nabız santrifüjü ile karıştırın. RT'de 20 dakika inkübe edin.
NOT: İnkübasyon sırasında akış hücresini hazırlamaya başlayın (bölüm 13.5).
- SPRI boncukları kullanarak temizleyin (daha önceki temizliklerde olduğu gibi etanol kullanmayın).
- Örneklere 0,4x hacimde SPRI boncukları (RT) ekleyin. RT'de 10 dakika inkübe edin, karıştırmaya yardımcı olmak için aralıklı olarak hafifçe hafifçe vurun.
- Boncuklar ve çözelti tamamen ayrılana kadar (~5 dakika) mıknatısın üzerine yerleştirin. Süpernatanı çıkarın ve atın; Boncuk peletini rahatsız etmemeye dikkat edin.
- 125 μL SFB ile iki kez yıkayın.
- Pelet, bir pipetle karıştırarak 125 μL SFB ile tamamen yeniden süspanse edin. 30 saniye inkübe etmeye bırakın.
- Tüp tabanında sıvı toplamak ve mıknatıs üzerine yerleştirmek için darbe santrifüjü. Süpernatanı çıkarın ve atın.
- Peleti ikinci kez yıkamak için 13.4.4-13.4.5 adımlarını tekrarlayın.
- Nabız santrifüjü yapın ve fazla SFB'yi çıkarın.
- 15 μL EB'de tekrar süspanse edin ve RT'de 10 dakika inkübe edin.
- ~ 2 dakika boyunca mıknatısa geri dönün ve ardından çözeltiyi dikkatlice 1.5 mL'lik temiz bir mikrosantrifüj tüpüne aktarın.
- Daha önce adım 10.13'te açıklandığı gibi, ayrıştırılmış kitaplığın 1 μL'sini ölçün
NOT: En iyi sonuçlar için doğrudan MinION sıralamasına geçin; ancak son kütüphane gerekirse EB'de 4 °C'de 1 haftaya kadar saklanabilir.
- Bir akış hücresi kalite denetimi çalıştırın.
- Sıralama cihazını bir dizüstü bilgisayara bağlayın ve sıralama yazılımını açın.
- Akış hücresi türünü seçin ve ardından Akış Hücresini Denetle ve Testi Başlat'ı tıklatın.
- Tamamlandığında, toplam aktif (yani canlı) gözenek sayısı görüntülenecektir. Yeni bir akış hücresi >800 aktif gözeneğe sahip olmalıdır; Olmazsa, değiştirme için üreticiyle iletişime geçin.
- Akış hücresinin hazırlanması ve yüklenmesi (20 dk)
- Aşağıdaki reaktifleri RT'de çözün ve ardından sıralama tamponu, bir yıkama bağlantısı, yıkama tamponu ve yükleme boncuklarını buz üzerine yerleştirin.
- Sıralama tamponunu girdap haline getirin ve tamponu yıkayın, darbe santrifüjü yapın ve buz üzerine yerleştirin.
- Yıkama bağını darbeli santrifüjleyin ve pipetleme ile karıştırın; Sonra buzun üzerine yerleştirin.
- Bir akış hücresi hazırlama kitinden doğrudan yıkama tamponu tüpüne 30 μL yıkama ipi ekleyerek akış hücresi hazırlama karışımını hazırlayın ve pipetleme ile karıştırın.
- Yükleme boncuklarını, hızlı bir şekilde yerleştikleri için kullanmadan hemen önce pipetleyerek karıştırın.
- Yeni bir tüpte, Tablo 5'te belirtildiği gibi sıralama için son kitaplık seyreltmesini hazırlayın.
NOT: DNA kitaplığının hacmini 50 fmol'e eşdeğer olarak elde etmek için Ek Dosya 3 çalışma sayfasındaki hesaplamayı kullanın. 12 μL'den az hesaplanırsa, 12 μL'ye kadar EB ekleyin. - Sıralama cihazı kapağını geri çevirin ve hazırlama portu kapağını, hazırlama portu görünecek şekilde saat yönünde kaydırın (Şekil 3)
- Bir P1000 pipeti 200 μL'ye ayarlayarak hava kabarcıklarını dikkatlice çıkarın, ucu hazırlama portuna yerleştirin ve pipet ucuna giren küçük bir hacim görülene kadar tekerleği çevirin (maksimum dönüş 230 μL'ye kadar).
- Kabarcıkları önlemeye dikkat ederek, 800 μL akış hücresi hazırlama karışımını hazırlama portu aracılığıyla akış hücresine yükleyin.
- 5 dakika bekletin.
- Numune portu kapağını yavaşça kaldırın ve bir P1000 pipet kullanarak 200 μL astarlama karışımını astarlama portu üzerinden akış hücresine yükleyin.
- Yüklemeden önce kitaplık karışımını yukarı ve aşağı pipetleyin, ana karışımdaki yükleme boncuklarının yüklemeden önce yeniden askıya alındığından emin olun.
- 75 μL kütüphane karışımını numune portu üzerinden akış hücresine damla damla yükleyin. Bir sonrakini eklemeden önce her damlamanın bağlantı noktasına aktığından emin olun.
- S'yi yerine takınampport kapağı nazikçe yerleştirin ve pung'un s'ye girdiğinden emin olun.ampport.
- Doldurma portunu kapatın ve sıralama cihazı kapağını değiştirin.
- Sıralama çalışması (maksimum 48 saat)
- Sıralama cihazını dizüstü bilgisayara bağlayın ve sıralama yazılımını açın.
- Başlat'a tıklayın ve ardından Sıralamayı Başlat'a tıklayın.
- Yeni Deneme'yi tıklatın ve çalıştırma parametrelerini ayarlamak için sıralama yazılımı grafik kullanıcı arabirimi (GUI) iş akışını izleyin.
- Deney adını ve numune kimliğini (ör. rabv_run1) yazın ve açılır menüden Akış Hücresi Tipi'ni seçin.
- Kit seçimine devam edin ve kullanılan ilgili ligasyon sıralama kitini ve yerel barkodlama kitini seçin.
- Seçenekleri Çalıştırmaya Devam Edin. Çalıştırmanın belirli bir saat sayısından sonra otomatik olarak durdurulması istenmediği sürece varsayılanları koruyun (çalıştırmalar herhangi bir zamanda manuel olarak durdurulabilir).
- Basecalling'e devam edin. Bilgi işlem kaynaklarına göre Basecalling'i Açma veya Kapatma seçeneğini belirleyin (bkz. bilgisayar kurulumu). Barkodlama altında Seçenekleri Düzenle'yi seçin ve Barkodun Her İki Ucunun açık olduğundan emin olun. Kaydedin ve çıktı bölümüne devam edin.
- Varsayılanları kabul edin ve son gözden geçirmeye devam edin, ayarları kontrol edin ve ayrıntıları çalışma sayfasına kaydedin (Ek Dosya 3). Başlat'a tıklayın.
NOT: Akış hücresi yeniden kullanılıyorsa, başlangıç voltajını (çalıştırma seçeneklerinin gelişmiş bölümünde), Ek Dosya 3'teki şemada gösterildiği gibi ayarlayın. - İlk aktif kanalları kaydedin - bu, kalite kontrol (QC) kontrolünden önemli ölçüde düşükse, sıralama yazılımını yeniden başlatın. Hala düşükse, bilgisayarı yeniden başlatın.
- Yaklaşık bir gözenek doluluğu sağlamak için ilk kanalları tek gözeneklere karşı iplikçik halinde kaydedin. Bu sayı dalgalanacaktır, bu nedenle yaklaşık bir değer verin.
- İlerledikçe koşuyu izleyin.
14. Canlı ve çevrimdışı temel arama
NOT: Bu talimatlar, artic-rabv deposunda önceden var olan dizin yapısının sağlandığını ve protokolün Önkoşullar bölüm 1 ve 3'e uyulduğunu varsayar.
- Yerel dosya sisteminizde, tüm analiz çıktılarınızı depolayacağınız analiz adlı yeni bir dizin oluşturun. Daha fazla düzenlemek için: MinKNOW'a run_name olarak sağlanan örnek kimliği kullanarak projenizin adıyla bir alt dizin oluşturun ve bunun içinde çalıştırma için yeni bir dizin oluşturun. Bunu tek bir komutla aşağıdaki gibi yapın:
mkdir -p
Analiz/project_name/run_name
Ardından konumuna gidin:
CD
yol/analiz/project_name/run_name - Canlı temel arama
NOT: Nanopor taban çağrısını gerçek zamanlı olarak gerçekleştirmek için dizüstü bilgisayarlar NVIDIA CUDA uyumlu bir grafik işleme birimi (GPU) gerektirir. GPU temel arama kurulumu için talimatların guppy protokolü56 kullanılarak gerçekleştirildiğinden emin olun.- Çalıştırma kurulumu sırasında canlı temel çağrıyı açın.
- Aşağıdaki talimata göre sıralama kapsamını gerçek zamanlı olarak izlemek için RAMPART'ı kullanın.
- Bilgisayarın terminalinde artic-rabv conda ortamını etkinleştirin:
Conda Artic-RABV'yi etkinleştirin - run_name dizini içinde sur çıktısı için yeni bir dizin oluşturun ve içine gidin:
cd / yol / analiz / project_name / run_name
mkdir rampart_output
cd rampart_output - Barkodları ve numune adlarını eşleştirmek için bir barkod.csv dosyası oluşturun. Her barkod için bir satır olmalı ve yalnızca "barkod" ve "örnek" başlıklarıyla kitaplıkta bulunan barkodları belirtmelidir. artic-rabv dizinindeki örneği izleyin:
analiz / example_project / example_run / rampart_output / barkodlar.csv - Çalıştırma için MinKNOW çıkışında ilgili protokol klasörünü ve fastq_pass klasörün yolunu sağlayarak RAMPART'ı başlatın:
rampart --protocol /path/rampart/scheme_name_V1_protocol - basecalledPath - Bir tarayıcı penceresi açın ve URL kutusunda localhost:3000 adresine gidin. Sonuçlar ekranda görünmeden önce yeterli verinin temel çağrılmasını bekleyin.
- Çevrimdışı temel arama (çalıştırma sonrası gerçekleştirilir)
- Canlı temel arama ayarlanmadıysa, MinKNOW'dan gelen çıktı ham sinyal verisi (fast5 dosyaları) olacaktır. Koşu sırasında RAMPART kullanılamayacaktır. fast5 dosyalarını guppy kullanarak çalıştırma sonrası temel verilere (fastq dosyaları) dönüştürün (Önkoşullar adım 1.1.1'deki kuruluma bakın). RAMPART'ı tabanda post-hoc çalıştırın veri denir.
- Lepistes basecaller'ı çalıştırın:
guppy_basecaller -c dna_r9.4.1_450bps_fast.cfg -i /path/to/reads/fast5_* -s /path/analysis/project_name/run_name -x auto -r
-c, temel çağırma modelini belirtmek için yapılandırma dosyasıdır, -i giriş yoludur, -s kaydetme yoludur, -x, GPU aygıtı tarafından temel çağrıyı belirtir (guppy'nin bilgisayar sürümünü kullanıyorsanız hariç tutun) ve -r, giriş dosyalarını özyinelemeli olarak aramayı belirtir.
NOT: Yapılandırma dosyası (.cfg), _fast _hac ile değiştirilerek yüksek doğrulukta bir temel çağırıcıya değiştirilebilir, ancak bu çok daha uzun sürecektir.
15. Akış hücrelerinin yıkanması
- Akış hücreleri, gözenekler hala canlıysa, yeni kitaplıkları sıralamak için yıkanabilir ve yeniden kullanılabilir. ONT akış hücresi yıkama protokolü57'de yıkama talimatlarına bakın.
16. Analiz ve yorumlama
- ARTIC biyoinformatik boru hattı ile konsensüs dizisi oluşturma
- Ham fast5 veya basecalled fastq dosyalarından konsensüs dizileri oluşturmak için rabv_protocols klasöründeki artic-rabv GitHub deposu40'ta ayrıntılı olarak açıklanan talimatları izleyin.
NOT: Daha fazla rehberlik için Artic boru hattı - Çekirdek boru hattı58'e bakın.
- Ham fast5 veya basecalled fastq dosyalarından konsensüs dizileri oluşturmak için rabv_protocols klasöründeki artic-rabv GitHub deposu40'ta ayrıntılı olarak açıklanan talimatları izleyin.
- İsteğe bağlı: Amplikon başına ortalama okuma derinliğini analiz edin.
- Ek Dosya 1'e başvurarak artic-rabv deposunda bulunan komut dosyalarını uyarlayın. Kısaca, SAMtools59 ve R'de çizilen nükleotid başına kapsam kullanılarak derinlemesine istatistikler oluşturulur.
- GLUE kullanarak filogenetik analiz
- RABV_GLUE 42'den itibaren, Analiz > Genotipleme ve Yorumlama sekmesini seçin > Dosya Ekle'yi seçin ve konsensüs dizilerinin fasta dosyasını seçin.
- Gönder'i tıklayın ve bekleyin. Analizler tamamlandıktan sonra, sınıf ve alt sınıf atamalarını, gen başına kapsamı, referans dizilerinden varyasyonu ve en yakın akrabayı gösteren Analizi Göster düğmesi tıklanabilecektir.
- İlgili bağlamsal diziler, Clade'e Göre Dizi Verileri > NCBI Dizileri bölümünde de tanımlanabilir.
- Tanımlanan sınıfı seçin veya mevcut tüm dizileri görmek için Kuduz Virüsü'ne (RABV) tıklayın.
- İlgili diziler için filtreleyin (ör. menşe ülke).
- Analiz ve karşılaştırma için bu dizileri ve ilgili meta verileri indirin.
- MADDOG41 kullanarak soy ataması
- En güncel sürümle çalıştığınızdan emin olmak için MADDOG deposunu GitHub'dan çekin.
- Yerel MADDOG deposunda (daha önce Önkoşullar bölümünde oluşturulmuş) çalıştırma adı olarak adlandırılan bir atama klasörü oluşturun.
- Klasörün içine, konsensüs dizilerini içeren fasta dosyasını ekleyin.
- Klasöre bir meta veri dosyası ekleyin.
NOT: Bu dosya, 'ID', 'country', 'year' ve 'assignment' adlı 4 sütundan oluşan, sıra kimliklerini, örneklemenin yapıldığı ülkeyi ve numunenin toplandığı yılı detaylandıran bir csv olmalı, 'assignment' sütunu ise boş olmalıdır. - Komut satırı arayüzünde conda ortamını etkinleştirin: conda MADDOG'u etkinleştirin.
- Komut satırı arayüzünde MADDOG depo klasörüne gidin.
- Başlangıçta, olası anormallikleri kontrol etmek ve daha uzun köken belirleme adımını çalıştırmanın uygun olup olmayacağını belirlemek için dizilerde köken ataması yapın. Bunun için komut satırına şunu yazın: sh assignment.sh.
- İstendiğinde, depoyu çektiğinizi ve MADDOG'un en güncel sürümüyle çalıştığınızı belirtmek için Y girin.
- İstendiğinde, fasta dosyasını içeren MADDOG depo klasöründeki klasörün adını girin.
- Köken ataması tamamlandığında klasörünüzdeki çıkış dosyasını denetleyin. Çıktı beklendiği gibiyse ve aynı kökene atanmış birden çok dizi varsa, köken atamasını çalıştırın.
- Köken ataması çalıştırıyorsanız, yeni oluşturulan atama çıktı dosyasını silin.
- Terminalde, MADDOG depo klasörünün içinde sh designation.sh komutunu çalıştırın.
- İstendiğinde, depoyu çektiğinizi ve MADDOG'un en güncel sürümüyle çalıştığınızı belirtmek için Y girin.
- İstendiğinde, fasta dosyasını ve meta verileri içeren MADDOG depo klasörüne klasör adını girin. Bu, her dizi hakkında soy bilgilerini, yeni ve ilgili önceki dizilerin bir filogenisini (16.3.6'dan itibaren), soylar hakkında hiyerarşik bilgileri ve potansiyel olarak ortaya çıkan soyların ve yetersiz örnekleme alanlarının ayrıntılarını verir.
NOT: Protokolün, kullanımın ve çıktıların tüm ayrıntıları Campbell ve ark.60'ta bulunabilir. - İlk analiz tamamlandığında, ortaya çıkan ve az örneklenmiş kökenleri de test etmeniz istenirse, gerekirse Y girin. Aksi takdirde, N girin.
- Yeni bulunan kökenleri onaylamanız istenirse, Y girin ve ortaya çıkan NEXT_STEPS.eml dosyasındaki yönergeleri izleyin. Aksi takdirde, N girin.
Sonuçlar
Bu protokolde açıklanan RABV için numuneden diziye ve yorumlama iş akışı, Tanzanya, Kenya, Nijerya ve Filipinler gibi endemik ülkelerde farklı laboratuvar koşullarında başarıyla kullanılmıştır (Şekil 4). Protokol farklı numune türleri ve koşulları üzerinde kullanıldı (Tablo 6): taze ve donmuş beyin dokusu, uzun süre soğuk zincir altında taşınan beyin dokusundan cDNA ve RNA ekstraktları ve beyin dokusu yaymalarına sahip FTA kartları.
RAMPART (Şekil 5) kullanılarak canlı temel arama, neredeyse gerçek zamanlı okuma oluşturma ve örnek başına kapsama yüzdesini gösterir. Bu, özellikle çalıştırmanın ne zaman durdurulacağına ve akış hücresinin yeniden kullanım için ne zaman kaydedileceğine karar vermede kullanışlıdır. Çalışma süresinde değişiklik gözlemlendi, bazıları 2 saatte tamamlandı, diğerleri ise yeterli kapsama derinliğine (x100) ulaşılması 12 saatten fazla sürebilir. Amplifikasyonun zayıf olduğu bölgeleri de görebiliriz; örneğin, Şekil 6 , kapsama profillerinin çok düşük amplifikasyona sahip bazı amplikonları gösterdiği ve potansiyel olarak sorunlu primerleri gösteren bir dizileme çalışmasının anlık görüntüsünü göstermektedir. Bu zayıf amplifiye edici bölgeleri daha kapsamlı bir şekilde araştırarak, tek tek primerleri yeniden tasarlamamızı ve geliştirmemizi sağlayacak primer uyumsuzluklarını belirleyebildik. Bazı primer şemaları diğerlerinden daha fazla uyumsuzluk göstermiştir. Bu, Doğu Afrika programı çok daha geniş bir çeşitliliği yakalamayı amaçladığından, hedeflenen çeşitliliğe uygun olarak Filipinler'e kıyasla Doğu Afrika primer şemasında gözlemlenmektedir.
Elde edilen RABV dizilerini derlemek ve yorumlamak için RABV genom veri yönetimi için genel amaçlı bir kaynak olan RABV-GLUE42 ve bir soy sınıflandırma ve isimlendirme sistemi olan MADDOG60 kullanıldı. Tablo 7 , RABV-GLUE kullanılarak atanan her ülkede dolaşan majör ve minör dalları göstermektedir. Ayrıca, MADDOG atamasını takiben yerel soyların daha yüksek çözünürlüklü bir sınıflandırması da gösterilmiştir.
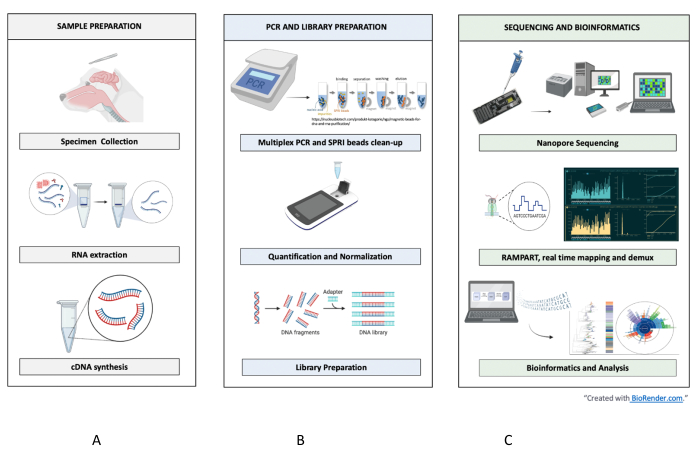
Şekil 1: RABV için örnekten diziye ve yorumlamaya iş akışı. (A) numune hazırlama, (B) PCR ve kütüphane hazırlama ve (C) analiz ve yorumlamaya kadar dizileme ve biyoinformatik için özet adımlar gösterilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
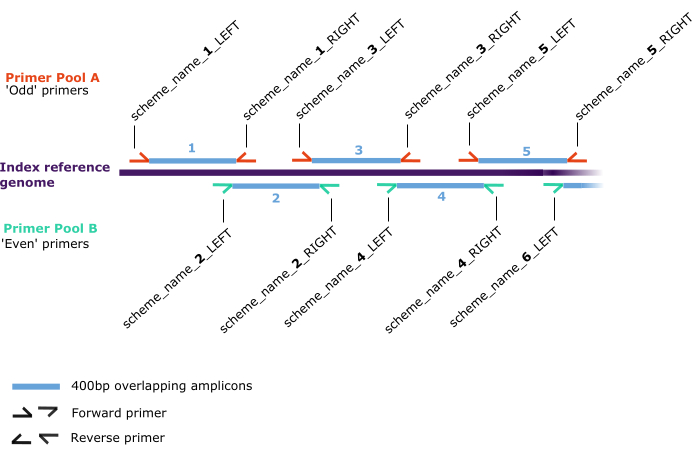
Şekil 2: Astar şeması. A (kırmızı) ve B (yeşil) olmak üzere iki ayrı havuza atanan ileri ve geri primer çiftleri (yarım oklar) için 'indeks referans genomu' (koyu mor) boyunca tavlama konumları. Primer çiftleri, indeks referans genomu boyunca 'scheme_name_X_DIRECTION' formatında sırayla numaralandırılan 400 bp örtüşen amplikon (mavi) üretir, burada 'X', primer tarafından üretilen amplikona atıfta bulunan bir sayıdır ve 'DIRECTION', sırasıyla ileri veya geri olanı tanımlayan 'SOL' veya 'SAĞ'dır. 'X'in tek veya çift değerleri havuzu belirler (sırasıyla A veya B). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
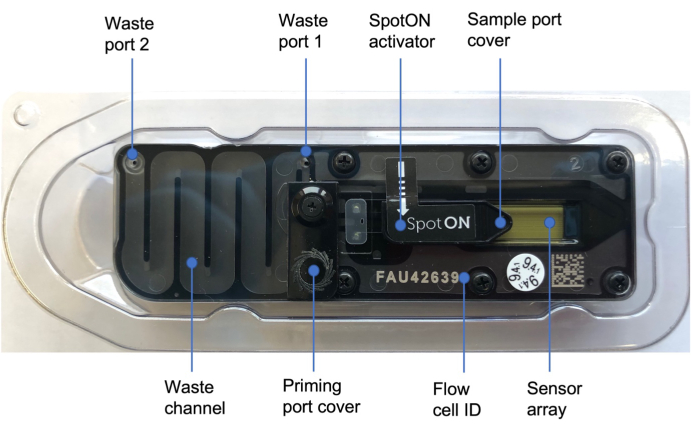
Şekil 3: Nanopor akış hücresi48. Mavi etiketler, hazırlama çözeltisinin eklendiği hazırlama portunu kaplayan hazırlama portu kapağı, numunenin damla damla eklendiği numune portunu kaplayan SpotON numune portu kapağı, atık portları 1 ve 2 ve akış hücresi kimliği dahil olmak üzere akış hücresinin farklı parçalarını gösterir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
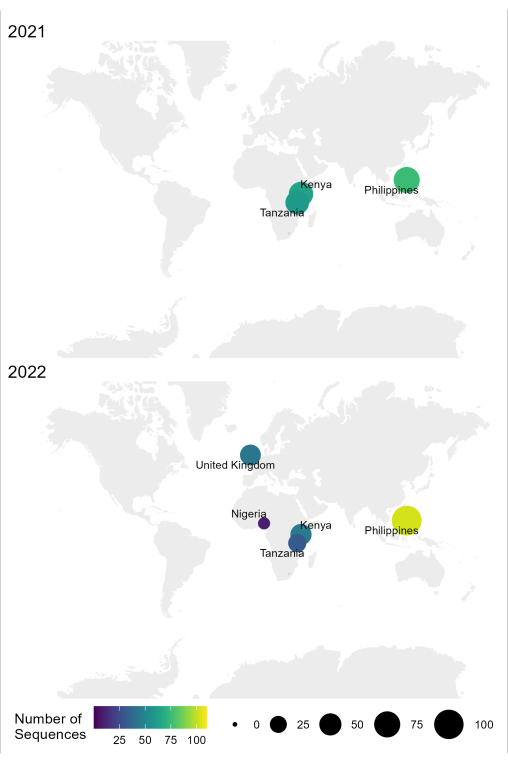
Şekil 4: 2021 ve 2022'de optimize edilmiş iş akışı kullanılarak RABV sıralamasının gerçekleştirildiği konumu gösteren harita. Kabarcık boyutu ve rengi, daha küçük ve daha koyu daha az, daha büyük ve daha açık olanın daha fazla olduğu konum başına dizi sayısına karşılık gelir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
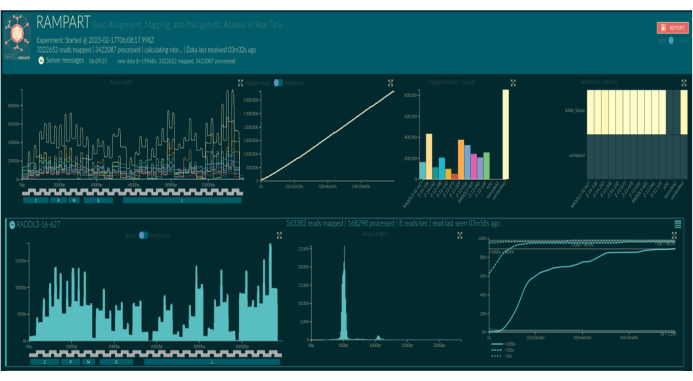
Şekil 5: Web tarayıcısında RAMPART görselleştirmesinin ekran görüntüsü. Barkod adları, biyoinformatik düzeneğe göre örnek adlarla değiştirilir. En üstteki üç panel, tüm çalışma için özet grafikleri gösterir: indeks referans genomundaki nükleotid konumu başına her barkod için eşlenen okumaların kapsama derinliği (sol üstte, barkodla renklendirilmiş), zaman içinde tüm barkodlardan toplanan eşlenmiş okumalar (üst orta) ve barkod başına eşlenmiş okumalar (sağ üst, barkodla renklendirilmiş). Alt paneller, barkod başına çizim satırlarını gösterir. Soldan sağa: indeks referans genomunda nükleotid pozisyonu başına eşlenmiş okumaların kapsama derinliği (solda), haritalanmış okumaların uzunluk dağılımı (ortada) ve zaman içinde 10x, 100x ve 1.000x eşlenmiş okuma kapsamı elde eden indeks referans genomundaki nükleotid konumlarının oranı (sağda). Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
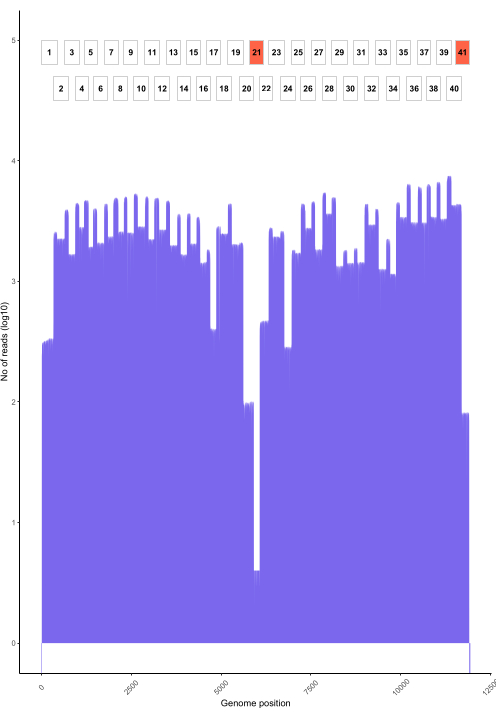
Şekil 6: Protokol kullanılarak dizilenen Filipinler'den bir kuduz virüsü örneği için genom boyunca bir örnek okuma kapsamı. Genomdaki her nükleotid pozisyonundaki okuma kapsamı, kütüphaneyi oluşturmak için kullanılan üst üste binen amplikonların (1-41) pozisyonunun yanı sıra gösterilir. Kapsama derinliğindeki sivri uçlar, amplikon örtüşme alanlarına karşılık gelir. Düşük kapsama derinliğine sahip amplikonlar, amplikon örtüşme alanlarına karşılık gelir. Düşük kapsama derinliğine sahip amplikonlar, optimizasyon gerektirebilecek sorunlu bölgeleri gösteren kırmızı renkle vurgulanır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tablo 1: cDNA hazırlığı için ana karışım ve termal döngüleyici koşulları. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 2: Multipleks PCR için ana karışım ve termal döngüleyici koşulları. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 3: Son hazırlık reaksiyonu için ana karışım ve termal döngüleyici koşulları. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 4: Barkodlama için ana karışım ve termal döngüleyici koşulları. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 5: Dizileme için adaptör ligasyon ana karışımı ve son kitaplık seyreltmesi. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 6: Üretilen kuduz virüsü tüm genom dizilerinin sayısı ve örneklemden diziye yorumlama iş akışı kullanılarak farklı ülkelerde kullanılan örneklerin türü. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Tablo 7: İş akışı kullanılarak oluşturulan diziler için RABV-GLUE'dan büyük ve küçük sınıf atamaları ve MADDOG'dan köken atamaları. Bu Tabloyu indirmek için lütfen buraya tıklayın.
Ek Dosya 1: Primer şema tasarımı ve optimizasyonu ve amplikon okuma derinliği analizi. Bu Dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 2: Hesaplamalı kurulum Bu Dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 3: RABV WGS protokol çalışma sayfası Bu Dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Erişilebilir bir RABV, nanopor tabanlı, tüm genom dizileme iş akışı, Brunker ve ark.61 tarafından, ARTIC ağından46 kaynaklar kullanılarak geliştirilmiştir. Burada, eksiksiz örneklemeden diziye ve yorumlamaya adımlarla güncellenmiş bir iş akışı sunuyoruz. İş akışı, tüm genom dizilimi için beyin dokusu örneklerinin hazırlanmasını detaylandırır, okumaları işlemek ve konsensüs dizileri oluşturmak için bir biyoinformatik boru hattı sunar ve soy atamasını otomatikleştirmek ve filogenetik bağlamı belirlemek için kuduza özgü iki aracı vurgular. Güncellenen iş akışı ayrıca, farklı bağlamlarda (düşük kaynak ayarları dahil) uygulamaya yönelik hususlarla birlikte uygun hesaplama ve laboratuvar çalışma alanlarının kurulumu için kapsamlı talimatlar sağlar. İş akışının hem akademik hem de araştırma enstitüsü ortamlarında, genomik sürveyans kapasitesi olmayan veya sınırlı olan dört RABV endemik LMIC'de başarılı bir şekilde uygulandığını gösterdik. İş akışının, çeşitli ayarlarda uygulamaya dayanıklı olduğu ve farklı uzmanlıklara sahip kullanıcılar tarafından anlaşılabilir olduğu kanıtlanmıştır.
RABV dizileme için bu iş akışı, halka açık en kapsamlı protokoldür (örneklemden diziye ve yorumlamaya kadar olan adımları kapsar) ve hem başlatma hem de çalıştırma maliyetlerini azaltmak için özel olarak uyarlanmıştır. Nanopor teknolojisi ile kütüphane hazırlama ve dizileme için gereken zaman ve maliyet, Illumina61 gibi diğer platformlara göre büyük ölçüde azalır ve sürekli teknoloji gelişmeleri, Illumina62 ile karşılaştırılabilir olacak şekilde dizi kalitesini ve doğruluğunu iyileştirmektedir.
Bu protokol, çeşitli düşük kaynak bağlamlarında dayanıklı olacak şekilde tasarlanmıştır. Çekirdek protokolle birlikte sağlanan sorun giderme ve değişiklik kılavuzuna başvurarak, kullanıcıların iş akışını ihtiyaçlarına göre uyarlamaları desteklenir. Kullanıcı dostu biyoinformatik araçların iş akışına eklenmesi, orijinal protokolde büyük bir gelişme teşkil eder ve dizi verilerini yerel bağlamlarda yorumlamak için minimum biyoinformatik deneyimine sahip kullanıcılar tarafından uygulanabilecek hızlı ve standartlaştırılmış yöntemler sağlar. Bunu yerinde yapma kapasitesi, genellikle yoğun ve uzun vadeli bir beceri eğitimi yatırımı gerektiren belirli programlama ve filogenetik becerilere sahip olma ihtiyacı ile sınırlıdır. Bu beceri seti, dizi verilerini kapsamlı bir şekilde yorumlamak için önemli olsa da, temel uzmanlıkları ıslak laboratuvar tabanlı olabilen ve verilerini yorumlamalarına ve sahiplenmelerine olanak tanıyan yerel "sıralama şampiyonlarını" kapasitif hale getirmek için temel ve erişilebilir yorumlama araçları da aynı derecede arzu edilir.
Protokol birkaç ülkede birkaç yıldır uygulandığından, artık kapsamı iyileştirmek ve birikmiş çeşitlilikle başa çıkmak için multipleks astar şemalarının nasıl optimize edileceği konusunda rehberlik sağlayabiliriz. Ayrıca, kullanıcıların maliyet etkinliğini iyileştirmelerine yardımcı olmak veya belirli bir bölgede tedarik kolaylığı sağlamak için çaba sarf edilmiştir, bu da tipik olarak moleküler yaklaşımların sürdürülebilirliği için bir zorluktur63. Örneğin, Afrika'da (Tanzanya, Kenya ve Nijerya), adaptör ligasyon adımında, yerel tedarikçilerden daha kolay temin edilebilen ve diğer ligasyon reaktiflerine daha ucuz bir alternatif olan künt / TA ligaz ana karışımını seçtik.
Deneyimlere göre, numune başına ve çalışma başına maliyeti azaltmanın birkaç yolu vardır. Çalışma başına numune sayısını azaltmak (örneğin, 24'ten 12 örneğe düşürmek), akış hücrelerinin ömrünü birden fazla çalışmada uzatabilirken, çalışma başına numune sayısını artırmak, zamanı ve reaktifleri en üst düzeye çıkarır. Elimizde, her üç dizileme çalışmasından biri için akış hücrelerini yıkayıp yeniden kullanabildik, bu da ek 55 numunenin dizilenmesini sağladı. Akış hücresini kullanımdan hemen sonra yıkamak veya mümkün değilse, her çalıştırmadan sonra atık sıvıyı atık kanalından çıkarmak, ikinci bir çalışma için mevcut gözenek sayısını koruyor gibi görünüyordu. Bir akış hücresinde bulunan ilk gözenek sayısı dikkate alınarak, belirli bir akış hücresinde kaç numunenin çalıştırılacağını planlamak için bir çalışma da optimize edilebilir.
İş akışı, ayrıntılı rehberlik ve işaretli kaynakların eklenmesiyle mümkün olduğunca kapsamlı olmayı amaçlasa da, prosedür hala karmaşıktır ve yeni bir kullanıcı için göz korkutucu olabilir. Kullanıcının, ideal olarak yerel olarak veya alternatif olarak harici işbirlikçiler aracılığıyla yüz yüze eğitim ve destek alması teşvik edilir. Örneğin Filipinler'de, ONT kullanarak SARS-CoV-2 genomik sürveyansı için bölgesel laboratuvarlarda kapasite geliştirme üzerine bir proje, sağlık hizmetleri teşhis uzmanları arasında RABV dizilimine kolayca aktarılabilen temel yetkinlikler geliştirmiştir. SPRI boncuk temizleme gibi önemli adımlarda uygulamalı eğitim olmadan ustalaşmak zor olabilir ve etkisiz temizleme akış hücresine zarar verebilir ve çalışmayı tehlikeye atabilir. Numune kontaminasyonu, amplikonlar laboratuvarda işlenirken her zaman büyük bir endişe kaynağıdır ve ortadan kaldırılması zor olabilir. Özellikle, numuneler arasındaki çapraz kontaminasyonun, çalışma sonrası biyoinformatik sırasında tespit edilmesi son derece zordur. Temiz çalışma yüzeylerinin korunması, PCR öncesi ve sonrası alanların ayrılması ve negatif kontrollerin dahil edilmesi gibi iyi laboratuvar tekniği ve uygulamaları, kalite kontrolünü sağlamak için zorunludur. Nanopor dizileme gelişmelerinin hızlı temposu, rutin RABV genomik sürveyansı için hem bir avantaj hem de dezavantajdır. Nanopor'un doğruluğunda, erişilebilirliğinde ve protokol repertuarında devam eden iyileştirmeler, uygulama kapsamını genişletir ve geliştirir. Bununla birlikte, aynı gelişmeler standart işletim prosedürlerinin ve biyoinformatik boru hatlarının sürdürülmesini zorlaştırmaktadır. Bu protokolde, eski nanopor kütüphane hazırlama kitlerinden güncel nanopor kütüphane hazırlama kitlerine geçişe yardımcı olan bir doküman sunuyoruz (Table of Materials).
LMIC'lerde sıralamanın önündeki yaygın bir engel, yalnızca maliyet değil, aynı zamanda sarf malzemelerini zamanında tedarik etme yeteneği (özellikle tedarik ekipleri ve tedarikçiler için nispeten yeni olan sıralama reaktifleri) ve hesaplama kaynakları da dahil olmak üzere erişilebilirliktir. Bu iş akışının temeli olarak taşınabilir nanopor dizileme teknolojisini kullanmak, bu erişilebilirlik sorunlarının çoğuna yardımcı olur ve protokolümüzün bir dizi ayarda kullanıldığını gösterdik, ülke içinde tam protokol ve analiz gerçekleştirdik. Kuşkusuz, ekipman tedarik etmek ve sarf malzemelerini zamanında sıralamak bir zorluk olmaya devam ediyor ve çoğu durumda Birleşik Krallık'tan reaktifleri taşımak veya göndermek zorunda kaldık. Bununla birlikte, bazı bölgelerde, satın alma süreçlerini kolaylaştıran ve patojen genomiklerinin uygulanmasını normalleştirmeye başlayan SARS-CoV-2 dizilimine (örneğin Filipinler) yapılan yatırımdan yararlanarak, reaktifler için tamamen yerel tedarik yollarına güvenebildik.
İstikrarlı bir internet bağlantısına duyulan ihtiyaç, yalnızca bir kerelik kurulumlarla en aza indirilir; örneğin, GitHub depoları, yazılım indirme ve nanopor diziliminin kendisi, çalıştırmayı başlatmak için yalnızca internet erişimi gerektirir (baştan sona değil) veya şirketin anlaşmasıyla tamamen çevrimdışı olarak gerçekleştirilebilir. Mobil veri varsa, çalışma süresi boyunca bağlantıyı kesmeden önce sıralama çalıştırmasını başlatmak için bir telefon dizüstü bilgisayarın etkin noktası olarak kullanılabilir. Örnekleri rutin olarak işlerken, veri depolama gereksinimleri hızla artabilir ve ideal olarak veriler bir sunucuda depolanır. Aksi takdirde, katı hal sürücüsü (SSD) sabit sürücülerinin kaynağı nispeten ucuzdur.
LMIC'lerde genomik sürveyansın önünde hala engeller olduğunu kabul etsek de, genomik erişilebilirlik ve uzmanlık oluşturmaya yönelik artan yatırımlar (örneğin, Afrika Patojen Genomik Girişimi [Afrika PGI])64 bu durumun iyileşeceğini göstermektedir. Genomik sürveyans, pandemiye hazırlık için kritik öneme sahiptir6 ve kapasite, RABV gibi endemik patojenlerin genomik sürveyansının rutinleştirilmesi yoluyla oluşturulabilir. SARS-CoV-2 salgını sırasında vurgulanan sıralama kapasitelerindeki küresel eşitsizlikler, bu yapısal eşitsizlikleri ele almak için katalizör değişimin itici gücü olmalıdır.
Erişilebilir biyoinformatik araçları da dahil olmak üzere RABV için bu örneklemeden diziye ve yorumlama iş akışı, 2030 yılına kadar köpek aracılı kuduzdan sıfır insan ölümü hedefini hedefleyen kontrol önlemlerine rehberlik etmek ve nihayetinde RABV varyantlarının ortadan kaldırılması için kullanılma potansiyeline sahiptir. İlgili meta verilerle birleştirildiğinde, bu protokolden üretilen genomik veriler, salgın araştırmaları sırasında ve bir ülke veya bölgede dolaşan soyların tanımlanmasında hızlı RABV karakterizasyonunu kolaylaştırır60,61,65. Boru hattımızı çoğunlukla köpek aracılı kuduzdan örnekler kullanarak gösteriyoruz; Bununla birlikte, iş akışı doğrudan yaban hayatı kuduzlarına uygulanabilir. Bu aktarılabilirlik ve düşük maliyet, hastalık yönetimini ve kontrolünü iyileştirmek için sadece kuduz için değil, aynı zamanda diğer patojenler için de rutin dizilemeyi kolayca erişilebilir hale getirmedeki zorlukları en aza indirir46,66,67.
Açıklamalar
Yazarların açıklayacak hiçbir şeyi yok.
Teşekkürler
Bu çalışma Wellcome [207569/Z/17/Z, 224670/Z/21/Z], Tıbbi Araştırma Konseyi [MR/R025649/1] ve Filipinler Bilim ve Teknoloji Departmanı'ndan (DOST) Newton finansmanı, Birleşik Krallık COVID-19 Araştırma ve İnovasyon Küresel çabası [MR/V035444/1], Glasgow Üniversitesi Kurumsal Stratejik Destek Fonu [204820], Tıbbi Araştırma Konseyi Yeni Araştırmacı Ödülü (KB) [MR/X002047/1] tarafından desteklenmiştir. ve Uluslararası Ortaklık Geliştirme Fonu, bir DOST British Council-Filipinler öğrenciliği (CB), Ulusal Sağlık Araştırmaları Enstitüsü [17/63/82] GemVi bursu (GJ) ve Glasgow Üniversitesi MVLS DTP (KC) [125638-06], EPSRC DTP (RD) [EP/T517896/1] ve Wellcome IIB DTP (HF) [218518/Z/19/Z]. Bu çalışmayı destekleyen meslektaşlarımıza ve işbirlikçilerimize minnettarız: Daniel Streicker, Alice Broos, Elizabeth Miranda, DVM, Daria Manalo, DVM, Thumbi Mwangi, Kennedy Lushasi, Charles Kayuki, Jude Karlo Bolivar, Jeromir Bondoc, Esteven Balbin, Ronnel Tongohan, Agatha Ukande, Davis Kuchaka, Mumbua Mutunga, Lwitiko Sikana ve Anna Czupryna.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Brand name | |||
| Software | |||
| Sequencing software (MinKnow) | Oxford Nanopore Technologies | https://community.nanoporetech.com/downloads | |
| Bioinformatics tool kit (Guppy) | Oxford Nanopore Technologies | https://community.nanoporetech.com/docs/prepare/library_prep_protocols/Guppy-protocol/v/gpb_2003_v1_revao_14dec2018 | |
| Equipment | |||
| Thermal cycler (miniPCR™ mini16 thermal cycler) | Cambio | MP-QP-1016-01 | |
| Homogenizer (Precellys Evolution Touch Homogenizer) | Bertin Instruments | EQ02520-300 | |
| Cold Racks (0.2-0.5mL) (PCR Mini-cooler with transparent lid) | BRAND | 781260 | |
| Pipettor | |||
| (Pipetman L Fixed F1000L, 1000 uL) | Gilson | SKU: FA10030 | |
| (Pipetman L Fixed F100L, 100 uL) | Gilson | SKU: FA10024 | |
| (Pipetman L Fixed F10L, 10 uL) | Gilson | SKU: FA10020 | |
| (Pipetman L Fixed F1L, 1 uL) | Gilson | SKU: FA10025 | |
| (Pipetman L Fixed F20L, 20 uL) | Gilson | SKU: FA10021 | |
| (Pipetman L Fixed F250L, 250 uL) | Gilson | SKU: FA10026 | |
| Fluorometer (Qubit 4 Fluorometer) | Thermofisher scientific/Fisher scientific | Q33238 | |
| Laptop (Any brand with ~2 GB of drive space, minimum of 512 GB storage space, msi installer [GPU]) | |||
| Microcentrifuge (Refrigerated centrifuge) | Thermofisher scientific/Fisher scientific | 75004081 | |
| Vortex mixer (Basic vortex mixer) | Thermofisher scientific/Fisher scientific | 88882011 | |
| Magnetic rack (DynaMag -2 Magnet) | Thermofisher scientific/Fisher scientific | 12321D | |
| Sequencing device (MinION) | Oxford Nanopore Technologies | MinION Mk1B | |
| RNA Extraction | |||
| RNA extraction kit (Qiagen RNEasy Mini Kit 250) | Qiagen | 74106 | |
| RNA stabilizing reagents | |||
| (RNA later) | Invitrogen | AM7020 | |
| (DNA/RNA Shield) | Zymo Research | R1100-50 | |
| PCR | |||
| Nuclease-free Water (Nuclease-free Water [not DEPC-treated]) | Thermofisher scientific/Fisher scientific | AM9937 | |
| Master mix for first strand cDNA synthesis (LunaScript RT SuperMix Kit) | New England Biolabs | E3010S | |
| DNA amplification master mix (Q5® Hot Start High-Fidelity 2X Master Mix [NEB]) | New England Biolabs | M0494L | |
| Primer (Scheme) (Custom DNA oligos) | Invitrogen | ||
| SPRI Bead Clean-up | |||
| SPRI beads (Aline Biosciences PCR Clean DX ) | Cambio | AL-AC1003-50 | |
| Ethanol, Pure Absolute, >99.8% (GC) [Riedel-De Haen] | Merck | 818760 | |
| Short Fragment buffer (SFB expansion pack) | Oxford Nanopore Technologies | EXP-SFB001 | |
| DNA Quantification | |||
| DNA quantification kit (Qubit® dsDNA HS Assay Kit) | Thermofisher scientific/Fisher scientific | Q32854 | |
| DNA quantification assay tubes (Qubit™ Assay Tubes) | Thermofisher scientific/Fisher scientific | Q32856 | |
| End Prep and barcoding (Qubit™ Assay Tubes) | |||
| End Prep master mix (NEBNext Ultra End Repair/dA-Tailing Module) | New England Biolabs | E7546L | |
| Barcoding kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 | |
| (Native Barcoding Expansion 1-12) | EXP-NBD104 | ||
| (Native Barcoding Expansion 13-24) | EXP-NBD114 | ||
| (Native Barcoding Expansion 96) | EXP-NBD196 | ||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 | |
| (not compatible) | (not compatible) | ||
| (Native Barcoding Kit 24 V14) | SQK-NBD114.24 | ||
| (Native Barcoding Kit 96 V14) | SQK-NBD114.96 | ||
| Ligation mastermix (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter Ligation | |||
| Adapter ligation master mix | |||
| (NEBNext Quick Ligation Module) | New England Biolabs | E6056S | |
| (NEBNext Ultra II Ligation Module) | New England Biolabs | E7595S | |
| (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter mix | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-AMII001 | |
| (Adapter Mix II [AMII]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-NBA114 | |
| (Native adapter [NA]) | |||
| Sequencing | |||
| Flowcell priming kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-FLP002 | |
| (Flush Buffer [FB]) | |||
| (Flush Tether [FT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-FLP004 | |
| (Flow Cell Flush [FCF]) | |||
| (Flow Cell Tether [FCT]) | |||
| Ligation Sequencing Kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 SQK-LSK109 | |
| Adapter Mix (Adapter Mix [AMX]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SQB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LB]) | |||
| Sequencing Tether (Sequencing Tether [SQT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 SQK-LSK114 | |
| Adapter Mix (Ligation Adapter [LA]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LIB]) | |||
| Sequencing Tether (Flow Cell Tether [FCT]) | |||
| Library solution (Library solution [LIS]) | |||
| Flush buffer (Flow Cell Flush [FCF]) | |||
| Flow Cell | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 FLO-MIN106D | |
| (Flow Cell [R9.4.1]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 FLO-MIN114 | |
| (Flow Cell [R10.4.1]) | |||
| Flow Cell wash | |||
| Flowcell wash kit (Flow cell wash kit) | Oxford Nanopore Technologies | EXP-WSH004 | |
| Consummables | |||
| Surface decontaminant | |||
| (DNA Away Surface Decontaminant, Squeeze Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7010PK | |
| (RNase Away Surface Decontaminant, Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7002PK | |
| PCR 8-Tube Strip 0.2ml, individual cap (PCR 8-Tube Strip 0.2ml, with Individual attached Flat Caps, Sterile, DNAse/RNAse, Pyrogen Free,Natural [Greiner]) | Greiner | 608281 | |
| PCR Tube 0.2ml (PCR Tube 0.2ml, Natural [Domed Cap] Bagged in 500s, Non-Sterile [Greiner]) | Greiner | 671201 | |
| 1000µL Filter Tips (500) (Stacked 1000µL Filter Tips [500]) | Thermofisher scientific/Fisher scientific | 11977724 | |
| 100µL Filter Tips (1000) | Thermofisher scientific/Fisher scientific | 11947724 | |
| 10µL Filter Tips (1000) (Stacked 100µL Filter Tips [1000]) | Thermofisher scientific/Fisher scientific | 11907724 | |
| Reinforced tubes tubes (2ml) with screw caps and o-rings (Fisherbrand™ Bulk tubes) | Thermofisher scientific/Fisher scientific | 15545809 | |
| Microcentrifuge tube (1.5ml) (1.5 ml Eppendorf Tubes [500]) | Eppendorf | 1229888 | |
| DNA LoBind Tubes (1.5ml) (DNA LoBind Tubes) | Thermofisher scientific/Fisher scientific | 10051232 | |
| Cryobabies labels | |||
| Gloves (S/M/L) | |||
| Paper towel |
Referanslar
- Rupprecht, C. E. Rhabdoviruses: rabies virus. Medical Microbiology. , University of Texas Medical Branch. Galveston, TX. (1996).
- Rabies. World Health Organization. , Available from: https://www.who.int/news-room/fact-sheets/detail/rabies (2023).
- Health Organization, W. orld WHO Expert Consultation on Rabies: WHO TRS N°1012. World Health Organization. , Available from: https://www.who.int/publications-detail-redirect/WHO-TRS-1012 (2018).
- Benavides, J. A., et al. Defining new pathways to manage the ongoing emergence of bat rabies in Latin America. Viruses. 12 (9), 1002(2020).
- Hampson, K. Estimating the global burden of endemic canine rabies. PLoS Neglected Tropical Diseases. 9 (4), e0003709(2015).
- Global genomic surveillance strategy for pathogens with pandemic and epidemic potential, 2022-2032. World Health Organization. , Available from: https://www.who.int/publications-detail-redirect/978924004679 (2022).
- Tsai, K. J., et al. Emergence of a sylvatic enzootic formosan ferret badger-associated rabies in Taiwan and the geographical separation of two phylogenetic groups of rabies viruses. Veterinary Microbiology. 182, 28-34 (2016).
- Chiou, H. -Y., et al. Molecular characterization of cryptically circulating rabies virus from ferret badgers, Taiwan. Emerging Infectious Diseases. 20 (5), 790-798 (2014).
- Sabeta, C. T., Mansfield, K. L., McElhinney, L. M., Fooks, A. R., Nel, L. H. Molecular epidemiology of rabies in bat-eared foxes (Otocyon megalotis) in South Africa. Virus Research. 129 (1), 1-10 (2007).
- Scott, T. P. Complete genome and molecular epidemiological data infer the maintenance of rabies among kudu (Tragelaphus strepsiceros) in Namibia. PLoS One. 8 (3), e58739(2013).
- Lembo, T., et al. Exploring reservoir dynamics: a case study of rabies in the Serengeti ecosystem. The Journal of Applied Ecology. 45 (4), 1246-1257 (2008).
- Coetzee, P., Nel, L. H. Emerging epidemic dog rabies in coastal South Africa: a molecular epidemiological analysis. Virus Research. 126 (1-2), 186-195 (2007).
- Ou de Munnink, B. B. First molecular analysis of rabies virus in Qatar and clinical cases imported into Qatar, a case report. International Journal of Infectious Diseases. 96, 323-326 (2020).
- Smith, J., et al. Case report: Rapid ante-mortem diagnosis of a human case of rabies imported into the UK from the Philippines. Journal of Medical Virology. 69, 150-155 (2003).
- Mahardika, G. N. K., et al. Phylogenetic analysis and victim contact tracing of rabies virus from humans and dogs in Bali, Indonesia. Epidemiology and Infection. 142 (6), 1146-1154 (2014).
- Tohma, K., et al. Molecular and mathematical modeling analyses of inter-island transmission of rabies into a previously rabies-free island in the Philippines. Infection, Genetics and Evolution. 38, 22-28 (2016).
- Tohma, K., et al. Phylogeographic analysis of rabies viruses in the Philippines. Infection, Genetics and Evolution. 23, 86-94 (2014).
- Saito, M., et al. Genetic diversity and geographic distribution of genetically distinct rabies viruses in the Philippines. PLoS Neglected Tropical Diseases. 7 (4), e2144(2013).
- Biek, R., Henderson, J. C., Waller, L. A., Rupprecht, C. E., Real, L. A. A high-resolution genetic signature of demographic and spatial expansion in epizootic rabies virus. Proceedings of the National Academy of Sciences. 104 (19), 7993-7998 (2007).
- Reddy, G. B., et al. Molecular characterization of Indian rabies virus isolates by partial sequencing of nucleoprotein (N) and phosphoprotein (P) genes. Virus Genes. 43, 13-17 (2011).
- David, D., Dveres, N., Yakobson, B. A., Davidson, I. Emergence of dog rabies in the northern region of Israel. Epidemiology and Infection. 137 (4), 544-548 (2009).
- Benjathummarak, S. Molecular genetic characterization of rabies virus glycoprotein gene sequences from rabid dogs in Bangkok and neighboring provinces in Thailand, 2013-2014. Archives of Virology. 161 (5), 1261-1271 (2016).
- Denduangboripant, J., et al. Transmission dynamics of rabies virus in Thailand: implications for disease control. BMC Infectious Diseases. 5, 52(2005).
- Talbi, C., et al. Phylodynamics and human-mediated dispersal of a zoonotic virus. PLoS Pathogens. 6 (10), e1001166(2010).
- Bourhy, H., et al. Revealing the micro-scale signature of endemic zoonotic disease transmission in an African urban setting. PLoS Pathogens. 12 (4), e1005525(2016).
- Zinsstag, J., et al. Vaccination of dogs in an African city interrupts rabies transmission and reduces human exposure. Science Translational Medicine. 9 (421), (2017).
- Yakovleva, A., et al. Tracking SARS-COV-2 variants using Nanopore sequencing in Ukraine in 2021. Scientific Reports. 12, 15749(2022).
- Mannsverk, S., et al. SARS-CoV-2 variants of concern and spike protein mutational dynamics in a Swedish cohort during 2021, studied by Nanopore sequencing. Virology Journal. 19, 164(2022).
- Soufi, M., et al. Fast and easy nanopore sequencing workflow for rapid genetic testing of familial Hypercholesterolemia. Frontiers in Genetics. 13, 836231(2022).
- Cabibbe, A. M. Application of targeted next-generation sequencing assay on a portable sequencing platform for culture-free detection of drug-resistant tuberculosis from clinical samples. Journal of Clinical Microbiology. 58 (10), 00632(2020).
- Xu, Y., et al. Nanopore metagenomic sequencing of influenza virus directly from respiratory samples: diagnosis, drug resistance and nosocomial transmission, United Kingdom, 2018/19 influenza season. Euro Surveillance. 26 (27), 2000004(2021).
- Stubbs, S. C. B., et al. Assessment of a multiplex PCR and Nanopore-based method for dengue virus sequencing in Indonesia. Virology Journal. 17, 24(2020).
- Croville, G., et al. Rapid whole-genome based typing and surveillance of avipoxviruses using nanopore sequencing. Journal of Virological Methods. 261, 34-39 (2018).
- Theuns, S., et al. Nanopore sequencing as a revolutionary diagnostic tool for porcine viral enteric disease complexes identifies porcine kobuvirus as an important enteric virus. Scientific Reports. 8, 9830(2018).
- O'Donnell, V. K., et al. Rapid sequence-based characterization of African swine fever virus by use of the Oxford Nanopore MinION sequence sensing device and a companion analysis software tool. Journal of Clinical Microbiology. 58, 01104-01119 (2019).
- Brito, A. F. Global disparities in SARS-CoV-2 genomic surveillance. Nature Communications. 13, 7003(2022).
- ONT login/register. Oxford Nanopore Technology. , Available from: https://nanoporetech.com/login-register (2023).
- Software Downloads. Oxford Nanopore Technology. , Available from: https://community.nanoporetech.com/downloads (2023).
- GitHub. , Available from: https://github.com/ (2023).
- Brunker, K. Artic-rabv. , Available from: https://github.com/kirstyn/artic-rabv (2022).
- Campbell, K. MADDOG: Method for Assignment, Definition and Designation of Global Lineages. , Available from: https://github.com/KathrynCampbell/MADDOG (2022).
- Campbell, K. RABV-GLUE. Centre for Virus Research. , Available from: https://github.com/KathrynCampbell/MADDOG (2022).
- Itokawa, K., Sekizuka, T., Hashino, M., Tanaka, R., Kuroda, M. Disentangling primer interactions improves SARS-CoV-2 genome sequencing by multiplex tiling PCR. PLoS ONE. 15 (9), e0239403(2020).
- Davis, M. W., Jorgensen, E. M. ApE, A plasmid editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 818619(2022).
- Döring, M., Pfeifer, N. openPrimeR: Multiplex PCR primer design and analysis. , (2023).
- Quick, J. Multiplex PCR method for MiniON and Illumina sequencing of Zika and other virus genomes directly from clinical samples. Nature Protocols. 12 (6), 1261-1276 (2017).
- Laboratory Techniques in Rabies. World Health Organization. 1, Available from: https://apps.who.int/iris/bitstream/handle/10665/310836/9789241515153-eng.pdf (2018).
- Lembo, T. Partners for Rabies Prevention. The blueprint for rabies prevention and control: a novel operational toolkit for rabies elimination. PLoS Neglected Tropical Diseases. 6 (2), e1388(2012).
- Terrestrial Manual Online Access. World Organization for Animal Health. , Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals/terrestrial-manual-online-access/ (2023).
- Mauti, S. Field postmortem rabies rapid immunochromatographic diagnostic test for resource-limited settings with further molecular applications. Journal of Visualized Experiments. (160), e60008(2020).
- Patrick, E., et al. Enhanced rabies surveillance using a direct rapid immunohistochemical test. Journal of Visualized Experiments. (146), e59416(2019).
- Lembo, T., et al. Evaluation of a direct, rapid immunohistochemical test for rabies diagnosis. Emerging Infectious Diseases. 12 (2), 310-313 (2006).
- Marston, D. A., et al. Pan-lyssavirus real time RT-PCR for rabies diagnosis. Journal of Visualized Experiments. (149), e59709(2019).
- Brunker, K. DNA quantification using the Qubit fluorometer. , Available from: https://www.protocols.io/view/dna-quantification-using-the-qubit-fluorometer-bc6vize6 (2020).
- Quick, J. DNA quantification using the Quantus fluorometer. , Available from: https://www.protocols.io/view/dna-quantification-using-the-quantus-fluorometer-7pzhmp6 (2020).
- Guppy protocol. Nanopore Community. , Available from: https://community.nanoporetech.com/protocols/Guppy-protocol/v/gpb_2003_v1_revaq_14dec2018 (2023).
- Flow Cell Wash Kit (EXP-WSH004). Nanopore Community. , Available from: https://community.nanoporetech.com/protocols/flow-cell-wash-kit-exp-wsh004/v/wfc_9120_v1_revh_08dec2020 (2023).
- Core Pipeline - arctic pipeline. , Available from: https://artic.readthedocs.io/en/latest/minion/ (2023).
- Samtools. , Available from: https://www.htslib.org (2023).
- Campbell, K., et al. Making genomic surveillance deliver: A lineage classification and nomenclature system to inform rabies elimination. PLoS Pathogens. 18 (5), e1010023(2022).
- Brunker, K., et al. Rapid in-country sequencing of whole virus genomes to inform rabies elimination programmes. Wellcome Open Research. 5, 3(2020).
- Bull, R. A., et al. Analytical validity of nanopore sequencing for rapid SARS-CoV-2 genome analysis. Nature Communications. 11, 6272(2020).
- Okeke, I. N., Ihekweazu, C. The importance of molecular diagnostics for infectious diseases in low-resource settings. Nature Reviews. Microbiology. 19 (9), 547-548 (2021).
- Inzaule, S. C., Tessema, S. K., Kebede, Y., Ouma, A. E. O., Nkengasong, J. N. Genomic-informed pathogen surveillance in Africa: opportunities and challenges. The Lancet Infectious Diseases. 21 (9), 281-289 (2021).
- Kennedy, L. Integrating contact tracing and whole-genome sequencing to track the elimination of dog-mediated rabies: an observational and genomic study. eLife. , (2023).
- Pallerla, S. R. Diagnosis of pathogens causing bacterial meningitis using Nanopore sequencing in a resource-limited setting. Annals of Clinical Microbiology and Antimicrobials. 21, 39(2022).
- Quick, J. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır