A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
قياس النشاط الأنزيمي لإنزيمات deubiquitylating المرتبطة باضطراب النمو العصبي عن طريق مقايسة انقسام سلسلة Ubiquitin في المختبر
In This Article
Summary
يشرح هذا البروتوكول كيفية قياس تأثير التباين الجيني المرتبط باضطرابات النمو العصبي على نشاط إنزيم deubiquitylating من خلال الجمع بين تنقية البروتين المؤتلف وفحوصات انقسام سلسلة ubiquitin.
Abstract
ترتبط اضطرابات النمو العصبي (NDDs) بضعف في وظيفة الجهاز العصبي ولكنها غالبا ما تظل غير مفهومة جيدا على المستوى الجزيئي. توفر الاضطرابات المنفصلة التي تسببها الجينات المفردة نماذج للتحقيق في الآليات التي تقود النمو العصبي غير النمطي. ترتبط متغيرات الجينات التي تشفر بروتينات عائلة إنزيم deubiquitylating (DUB) بالعديد من NDDs ، ولكن هناك حاجة لتحديد الآليات المسببة للأمراض للاضطرابات التي تحركها هذه المتغيرات الجينية. يمكن تحديد تأثير المتغيرات الجينية على نشاط DUB تجريبيا باستخدام مقايسة انقسام اليوبيكويتين المستقلة عن الركيزة. لا يتطلب هذا الاختبار معرفة الركائز النهائية لقياس النشاط التحفيزي مباشرة. هنا ، تم نمذجة بروتوكول تحديد تأثير المتغيرات الجينية على النشاط الأنزيمي باستخدام DUB Ubiquitin Specific Protease 27 ، X-linked (USP27X) ، والذي تم تحوره في الإعاقة الذهنية المرتبطة ب X 105 (XLID105). يمكن استخدام خط الأنابيب التجريبي هذا لتوضيح الآليات الكامنة وراء اضطرابات النمو العصبي التي تحركها المتغيرات في جينات DUB.
Introduction
تنشأ اضطرابات النمو العصبي (NDDs) من مسببات متنوعة ذات محددات بيئية أو وراثية تدفع نمو الجهاز العصبي غير النمطي1. ربط الجيل التالي من الاختبارات الجينية لتسلسل الجيل التالي عددا متزايدا من المتغيرات في الجينات المرتبطة بنظام اليوبيكويتين مع NDDsالوراثية 2. يحفز نظام اليوبيكويتين ربط معدل البروتين الصغير يوبيكويتين إلى بقايا اللايسين في المقام الأول في ركائز البروتين لإحداث تغييرات في السلوك الخلوي ، بما في ذلك التوطين أو الاستقرار أو تفاعلات البروتين والبروتين أو النشاط3. يتم التوسط في كل مكان عن طريق تنشيط E1 ، واقتران E2 ، وإنزيمات E3 ligase4 ويمكن عكسها من خلال نشاط إنزيمات deubiquitylating (DUBs) التي تحفز الانقسام وإزالة اليوبيكويتين من ركائز البروتين5. يمكن ربط Ubiquitin بالركيزة كمونومر (monoubiquitylation) أو سلاسل بوليمرية (polyubiquitylation) التي تتشكل على أي من بقايا اللايسين السبعة (K6 ، K11 ، K27 ، K29 ، K33 ، K48 ، K63) أو بقايا M1 من يوبيكويتين. تنشئ طبولوجيا سلسلة يوبيكويتين المختلفة ومجموعاتها رمزا خلويا يعد مفتاحا لنقل الإشارة6.
تم ربط DUBs مثل USP27X و USP7 و USP9X و USP48 و STAMBP و OTUD4 و OTUD6B و OTUD5 ب NDDs2،7،8،9،10،11. بالنسبة لمعظم NDDs ، تظل الآليات الجزيئية التي تدفع التسبب في المرض غير محددة تجريبيا. بعض DUBs التي تقود الاضطرابات الموصوفة مؤخرا غير مفهومة جيدا وتفتقر إلى القراءات الخلوية المعروفة التي يمكن استخدامها لتقييم تأثير التباين الجيني على وظيفة البروتين. في المختبر ، تتغلب فحوصات انقسام سلسلة يوبيكويتين على هذا القيد كقراءات لنشاط DUB المستقلة عن الركيزة والتي يمكنها قياس تأثير المتغيرات الجينية على النشاط الأنزيمي12.
في المختبر تم استخدام فحوصات انقسام ubiquitin منذ الثمانينيات. سمحت هذه المقايسات باستخدام ركائز ذات علامات إشعاعية باكتشاف أول DUBs ، بما في ذلك الأيزوببتيداز ، الذي تم تحديده لقدرته على إزالة الطابع هيستون H2A13 ، وهيدرولاز الكربوكسيل الطرفي يوبيكويتين (UCH) ، الذي تم تحديده من خلال قدرته على تحلل اليوبيكويتين من مجموعة متنوعة من المترافقات الكيميائية14،15،16. علاوة على ذلك ، تم استخدام البروتينات أو الببتيدات كاملة الطول متعددة اليوبيكيتيل ذات العلامات المشعة لتحديد isopeptidase T والعديد من UCHs والبروتياز الخاص باليوبيكويتين (UBPs) من كريات الدم الحمراء والعضلات الهيكلية ، على التوالي17،18،19،20. تم استخدام سلاسل Ubiquitin ذات الطول المحدد ونوع الارتباط (tetra-ubiquitin المرتبط ب K48) لأول مرة لقياس نشاط DUB ل Isopeptidase T21. منذ ذلك الحين ، أصبح هذا الاختبار هو المعيار الذهبي لقياس نشاط DUB في التحليلات الطفرية22،23. يسمح صقل هذا الاختبار حاليا بتصور انقسام اليوبيكويتين عن طريق الرحلان الكهربائي وبقع الهلام التقليدية مثل صبغة Coomassie blue و SYPRO البرتقالية والياقوت والفضة أو الكشف الفلوري أو القائم على النشافالمناعي 12،24. يمكن توضيح الجوانب الجزيئية لنشاط DUB ، مثل الحد الأدنى لطول السلسلة وخصوصيةالارتباط 25،26،27،28 ، باستخدام سلاسل يوبيكويتين بأطوال مختلفة (على سبيل المثال ، di- ، tri- ، tetra-ubiquitin) والروابط (K6 ، K11 ، K27 ، K29 ، K33 ، K48 ، K63 ، خطي) في المقايسات الوظيفية. يمكن للمتغيرات المرتبطة ب NDD أن تؤدي إلى عيوب نشاط DUB الخاصة بنوع ربط سلسلةubiquitin 11.
يمكن لمقايسة انقسام ثنائي يوبيكويتين باستخدام بروتينات DUB المؤتلفة المنقاة قياس تأثير متغيرات NDD بشكل مباشر على نشاط DUB. USP27X ، الذي تم تحوره في اضطراب الإعاقة الذهنية المرتبط ب NDD X 105 (XLID105) 7،28 نموذج العملية المعروضة هنا. يسمح هذا النهج بتحديد كيفية تعطيل نشاط DUB بواسطة المتغيرات الجينية في NDDs الحالية وغير المعروفة المرتبطة ب DUB.
Access restricted. Please log in or start a trial to view this content.
Protocol
يمكن تكييف البروتوكول التالي للبروتينات المؤتلفة باستخدام علامات تقارب مختلفة يتم التعبير عنها في سلالات مختلفة من الخلايا المختصة. اعتمادا على البروتين الذي يتم التعبير عنه ، قد تتطلب ظروف الاستزراع والتعبير بين عشية وضحاها تحسين OD600 عند تحريض التعبير ووقت التعبير ودرجة حرارة التعبير وتركيز IPTG. يتم توضيح نظرة عامة على البروتوكول في الشكل 1. تفاصيل الكواشف والمعدات المستخدمة في هذه الدراسة مدرجة في جدول المواد.
1. تحويل خلايا الإشريكية القولونية Rosetta 2 المختصة مع بلازميد التعبير المؤتلف GST-USP27X
ملاحظة: للحفاظ على عقم المزرعة البكتيرية ، قم بتنفيذ الخطوات التي تكون فيها حاويات الوسائط مفتوحة تحت لهب موقد بنسن. للسماح بنقل الأكسجين الأمثل ، قم بإجراء اهتزاز المزرعة البكتيرية في شاكر يتم التحكم في درجة حرارته على الطاولة بمدار من 19 مم إلى 50 مم وسرعة 200 دورة في الدقيقة29.
- قم بإذابة 20 ميكرولتر من خلايا روزيتا 2 بكتريا قولونية المختصة كيميائيا على الثلج مباشرة قبل الاستخدام. أضف 1-10 نانوغرام من بلازميد التعبير pGEX6P1-USP27X (ترميز الجلوتاثيون-S-transferase الطرفي N (GST) 30 -الموسومة USP27X ويحتوي على مقاومة الأمبيسلين) واحتضان 5 دقائق على الجليد.
- صدمة حرارية لمدة 30 ثانية عند 42 درجة مئوية في حمام جاف. احتضن على الثلج لمدة 2 دقيقة.
- أضف 80 ميكرولتر من وسط SOC بدرجة حرارة الغرفة (RT). احتضن لمدة 60 دقيقة عند 37 درجة مئوية مع دوران 200 دورة في الدقيقة في شاكر مدور 19 مم يتم التحكم في درجة حرارته.
- صفيحة 50 ميكرولتر من المزرعة على صفيحة أجار LB مكملة ب 25 ميكروغرام / لتر كلورامفينيكول و 50 ميكروغرام / لتر أمبيسيلين. احتضن لمدة 20 ساعة عند 37 درجة مئوية بحيث يكون جانب الغطاء لأسفل في حاضنة يتم التحكم في درجة حرارتها.
2. التعبير البكتيري بين عشية وضحاها عن البروتين المؤتلف من بلازميد التعبير
ملاحظات: للحفاظ على عقم الثقافة البكتيرية ، قم بتنفيذ الخطوات التي تكون فيها حاويات الوسائط مفتوحة تحت لهب موقد بنسن. للسماح بنقل الأكسجين الأمثل ، قم بإجراء اهتزاز المزرعة البكتيرية في شاكر يتم التحكم في درجة حرارته على الطاولة بمدار من 19 مم إلى 50 مم وسرعة 200 دورة في الدقيقة29. قم بقياس الثقافة OD600 باستخدام مقياس الطيف الضوئي.
- قم بإعداد 1 لتر من وسط المرق الرائع (TB) عن طريق إضافة 47.6 جم من مسحوق السل إلى 1 لتر من الماء فائق النقاء مع 0.4٪ جلسرين في قارورة مزرعة محيرة سعة 2 لتر. الأوتوكلاف وسط لمدة 30 دقيقة ويبرد إلى RT.
- اختر مستعمرة واحدة من البكتيريا المحولة وأضفها إلى 10 مل من وسط LB المعقم المكمل ب 25 ميكروغرام / لتر من الكلورامفينيكول و 50 ميكروغرام / لتر أمبيسلين. احتضان لمدة 20 ساعة عند 37 درجة مئوية مع دوران 200 دورة في الدقيقة في شاكر مدور 19 مم يتم التحكم في درجة حرارته.
- يضاف 10 مل من المزرعة الليلية إلى 1 لتر من وسط السل مكمل ب 25 ميكروغرام/لتر كلورامفينيكول و50 ميكروغرام/لتر أمبيسلين (نسبة 1:100 من مزرعة البداية إلى مزرعة الشفط التعبيرية). احتضان عند 37 درجة مئوية مع دوران 200 دورة في الدقيقة في شاكر على الطاولة يتم التحكم في درجة حرارته بمدار 19 مم حتى يصبح ثقافة OD600 بين 0.5-0.6.
- أضف 50 ميكرومتر من IPTG إلى المزرعة للحث على التعبير ، وتبرد إلى 16 درجة مئوية ، واحتضانها لمدة 20 ساعة عند 16 درجة مئوية مع دوران 200 دورة في الدقيقة.
- خلايا الحبيبات من ثقافة التعبير عن طريق الطرد المركزي لمدة 20 دقيقة عند ≥3,000 × جم و 4 درجات مئوية. قم بتخزين الحبيبات عند -80 درجة مئوية لمدة 1 ساعة على الأقل.
ملاحظة: في هذه المرحلة ، يمكن إيقاف التجربة مؤقتا وإعادة تشغيلها لاحقا (يفضل أن يكون ذلك في نفس الأسبوع). يمكن تخزين الحبيبات على المدى الطويل عند -80 درجة مئوية.
3. تنقية البروتين عن طريق عمود تقارب تدفق الجاذبية
ملاحظة: تعتمد مخازن الراتنج والربط والغسيل والشطف والتخزين المناسبة لكل تنقية على البروتين المؤتلف الذي يتم تنقيةه. اجمع عينات من حبيبات الخلية ، والطاف ، والتدفق ، وكسور الغسيل ، وكسور الشطف في المخزن المؤقت SDS-PAGE. قم بإجراء تلطيخ SDS-PAGE و Coomassie للعينات لتقييم نجاح التنقية. قم بإجراء التنقية عند 4 درجات مئوية والتعامل مع الكسور الموجودة على الجليد. يمكن إجراء انقسام علامات البروتين داخل العمود أو خارجه باستخدام البروتياز المناسب لاستهداف موقع الانقسام الخاص بالبروتياز.
- قم بتأمين عمود تدفق الجاذبية الفارغ في حامل المعوجة واملأه براتنج الجلوتاثيون الاغاروز. استخدم 2-3 مل من الراتنج لتنقية حبيبات الخلية التي تم جمعها من 1 لتر من ثقافة التعبير.
- اغسل العمود بحجم سرير راتنج واحد بنسبة 20٪ من الإيثانول. اغسل العمود 3 مرات بحجم واحد من الراتنج من المخزن المؤقت MS500 (20 ملي مولار من درجة الحموضة ثلاثية 7.5 ، 500 ملي مولار من كلوريد الصوديوم ، 0.5 ملي مولار من TCEP). أوقف العمود حتى يظل الراتنج مغطى بمخزن مؤقت لمنع الجفاف أثناء تحضير حبيبات الخلية.
- قم بإذابة حبيبات الخلية عند 4 درجات مئوية. أضف 30 مل من المخزن المؤقت MS500 (20 ملي من درجة الحموضة Tris 7.5 ، 500 ملي من كلوريد الصوديوم ، 0.5 ملي مولار من TCEP ، 60 مجم من الليزوزيم ، وقرص مثبط للبروتياز واحد) إلى الحبيبات المذابة. Lyse لمدة 30 دقيقة عند 4 درجات مئوية مع دوران لطيف من طرف إلى طرف.
- تقوم بتحويل الخلايا المحللة إما في أنبوب طرد مركزي سعة 50 مل أو دورق معدني على الجليد حتى يتدفق المحلل بحرية عند الاستغناء عنه من طرف الماصة. جهاز طرد مركزي لمدة 30 دقيقة عند 4 درجات مئوية و ≥ 20,000 × جم لمسح المادة الطافية.
ملاحظة: تحديد إعدادات الصوتنة تجريبيا. اضبط الصوتنة بحيث يكون الوقت الإجمالي 120 ثانية كافيا لتقليل المحللة اللزجة إلى سائل شفاف وشفاف يتدفق بحرية. - صب المادة الطافية في دورق وتحميل المحللة التي تم تطهيرها على العمود. قم بتشغيل المحللة عبر العمود عن طريق تدفق الجاذبية أثناء جمع التدفق من خلال. قم بتحميل العمود مع التدفق الذي تم جمعه من خلاله وقم بتشغيله عبر العمود.
- اغسل العمود بحجمين على الأقل من طبقة الراتنج من المخزن المؤقت للغسيل MS500 (20 ملي مولار من درجة الحموضة ثلاثية 7.5 ، 500 ملي مولار من كلوريد الصوديوم ، 0.5 ملي مولار من TCEP) 5 مرات. اجمع تدفق الغسيل في أجزاء سعة 5 مل. أضف 1 ميكرولتر من كل جزء إلى 100 ميكرولتر من كاشف برادفورد للتحقق بصريا من وجود البروتين. اغسلها حتى يختفي البروتين غير المرتبط في الغسيل الأخير ، مع إضافة خطوات غسيل إضافية إذا لزم الأمر.
- استرجع البروتين عن طريق تشغيل المخزن المؤقت للشطف MS500 (المخزن المؤقت MS500 مكمل ب 10 ملي مولار من الجلوتاثيون و 10 ملي مولار من هيدروكسيد الصوديوم) من خلال العمود ، وجمع 5 مل من كسور الشطف. تحقق من وجود البروتين عن طريق إضافة 1 ميكرولتر من الشطف إلى 100 ميكرولتر من كاشف برادفورد. توقف عن جمع الكسور عندما لا يشير كاشف برادفورد إلى وجود البروتينات.
- إجراء تبادل المخزن المؤقت عن طريق الترسيب والطرد المركزي. قم بترسيب البروتين عن طريق إضافة مجلدين من 4 M من كبريتات الأمونيوم إلى الشطف ، مع قلبها برفق حتى تصبح غائمة ، ثم الطرد المركزي لمدة 30 دقيقة عند 4 درجات مئوية و ≥20,000 × جم ومرة أخرى لمدة 5 دقائق أخرى. قم بإزالة المادة الطافية بعد كل طرد مركزي دون إزعاج حبيبات البروتين.
- أعد إذابة البروتين وتخزينه في MS500 المكمل بنسبة 25٪ من الجلسرين. قم بتخزين كريات البروتين أو البروتين في مخزن مؤقت عند -80 درجة مئوية.
4. في المختبر اختبار انقسام سلسلة يوبيكويتين
ملاحظة: حدد طول سلسلة يوبيكويتين وأنواع الربط بناء على خصوصية DUB الموضحة في التقاريرالسابقة 31 أو المحددة تجريبيا. إذا لزم الأمر ، يمكن استخدام هذا البروتوكول لاختبار نشاط DUB من النوع البري ذي الأهمية على لوحة من سلاسل اليوبيكويتين المتاحة تجاريا ذات الطول المحدد ونوع الربط. يمكن استخدام كمية سلسلة di-ubiquitin من 375-750 ng وتركيز DUB من 1-2 ميكرومتر كنقاط انطلاق للمقايسة27.
- قم بإعداد 10x DUB المخزن المؤقت لتنشيط DUB (500 ملي مولار من Tris-HCl pH 7.5 ، و 500 ملي مولار من كلوريد الصوديوم ، و 100 ملي مولار من TCEP).
- لكل نقطة زمنية لكل DUB ، قم بإعداد 10 ميكرولتر إجمالي 2 ميكرومتر من GST-USP27X المنقى في 1x مخزن مؤقت لتنشيط DUB (مزيج DUB). قم بإعداد الخلطات الرئيسية وتقسيمها إلى نقاط زمنية.
- احتضن مزيج DUB لمدة 10 دقائق في RT.
- أضف 7 ميكرولتر من المخزن المؤقت لتحميل SDS-PAGE إلى الوقت 0 قبل إضافة سلاسل يوبيكويتين لمنع تفاعل deubiquitylation من البدء.
- إلى كل نقطة زمنية لكل DUB ، أضف 375 نانوغرام من سلاسل K-63 di-ubiquitin المخففة في 10 ميكرولتر 1x مخزن مؤقت لتنشيط DUB. الحجم الكلي هو 20 ميكرولتر لكل تفاعل.
- احتضان الأنابيب عند 30 درجة مئوية ، وتوقف كل نقطة زمنية مع 7 ميكرولتر من المخزن المؤقت للتحميل SDS-PAGE.
- قم بإجراء SDS-PAGE باستخدام هلام متدرج بنسبة 4٪ -12٪ وبقعة مناعية7،32 ل ubiquitin و USP27X لتحليل التغيير في وجود أحادي يوبيكويتين عبر نقاط زمنية محددة.
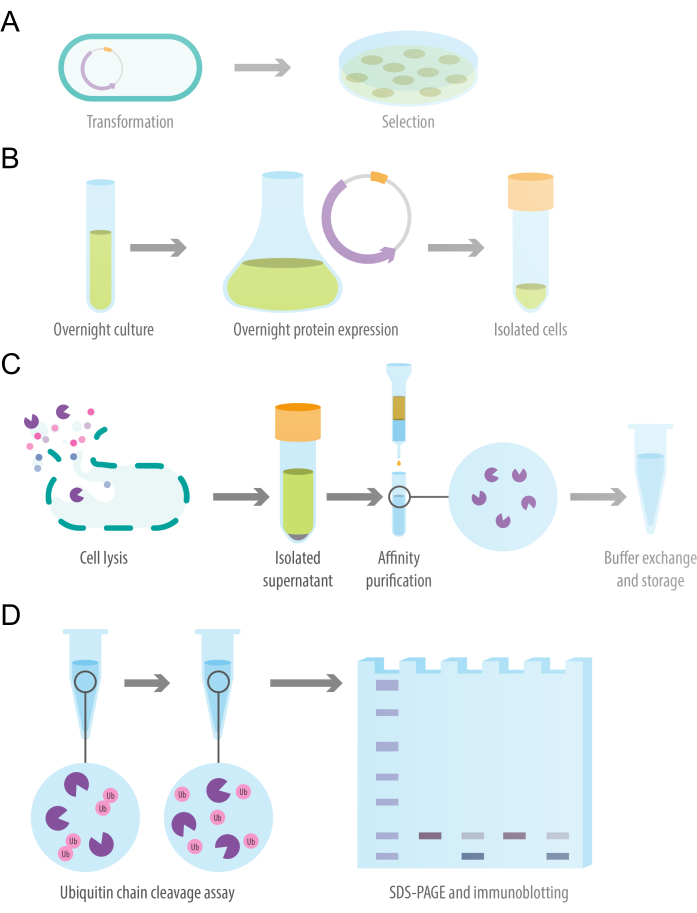
الشكل 1: تخطيطي لتصميم الدراسة. (أ) تحويل الخلايا الإشريكية القولونية المختصة مع بلازميد التعبير عن البروتين المؤتلف. (ب) التعبير البكتيري بين عشية وضحاها لبروتين ديوبيكيتيلاز المؤتلف. (ج) تنقية البروتين من الديوبيكويتيلاز المؤتلف باستخدام عمود تقارب تدفق الجاذبية. (د) مقايسة الانقسام المتسلسل في المختبر لتقييم نشاط deubiquitylating. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
Access restricted. Please log in or start a trial to view this content.
النتائج
لتحديد تأثير المتغيرات المرتبطة XLID105 على النشاط التحفيزي USP27X ، تم تنقية بروتينات USP27X من النوع البري الموسوم بضريبة السلع والخدمات والمتغير المرتبط ب XLID105 F313V و Y381H و S404N USP27X من البكتيريا. توجد هذه المتغيرات داخل المجال التحفيزي USP ل USP27X (الشكل 2 أ). نظرا لأنه ...
Access restricted. Please log in or start a trial to view this content.
Discussion
تقدم هذه المقالة بروتوكولا للتعبير عن وتنقية USP27X DUBs المؤتلفة ومقايسة انقسام سلسلة يوبيكويتين في المختبر لمقارنة نشاط إزالة الهوبيكيتيل للبروتينات المتغيرة المرتبطة ب USP27X من النوع البري و NDD. حدد هذا الاختبار أن المتغيرات المرتبطة XLID105 تعطل النشاط التحفيزي USP27X
Access restricted. Please log in or start a trial to view this content.
Disclosures
يعلن أصحاب البلاغ عدم وجود تضارب في المصالح.
Acknowledgements
تم تمويل هذا العمل من قبل صناديق بدء التشغيل Sanford Research إلى FB ومنحة المعاهد الوطنية للصحة R01CA233700 إلى MJS. تم عمل العمل الفني بواسطة فيليبي جي سيرانو (www.illustrative-science.com). نشكر الدكتور جريج فيندلي (جامعة دندي) على بلازميد GST-USP27X.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Amersham Protran 0.45 NC 200 mm × 200 mm 25/PK | Cytiva | 10600041 | |
| Ammonium sulfate | Fisher Scientific | AC205872500 | |
| Ampicillin | Fisher Scientific | BP1760 25 | |
| Anti- Ubiquitin (Mouse monoclonal) | Biolegend | Cat# 646302, RRID:AB_1659269 | (WB: 1:1000) |
| Anti-GST (Sheep polyclonal) | MRC-PPU Reagents and Services | Cat# S902A Third bleed | (WB: 1:1000) https://mrcppureagents.dundee.ac.uk/ |
| Baffled Culture Flasks 2 L | Fisher Scientific | 10-042-5N | |
| Bradford Reagent | Millipore Sigma | B6916-500ML | |
| Chloramphenicol | Gold Biotechnology | C-105-25 | |
| Complete, Protease Inhibitor tablets | Millipore Sigma | 5056489001 | |
| Econo-Column 1.5 × 5 cm | Bio-Rad | 7371507 | |
| Eppendorf ThermoMixer F1.5 | Eppendorf | 5384000020 | |
| Excel | Microsoft | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Glycerol | Genesee Scientific | 18-205 | |
| Illustrator | Adobe | https://www.adobe.com/products/illustrator.html | |
| Image Studio | LI-COR Biosciences | https://www.licor.com/bio/image-studio/ | |
| Inkscape | Inkscape | https://inkscape.org/ | |
| Invitrogen 4-12% NuPAGE 1mm 12 well gel | Thermo Fisher Scientific | NP0322BOX | |
| IPTG (Isopropyl-b-D-Thiogalactopyranoside) | Genesee Scientific | 20-109 | |
| IRDye 800CW Donkey anti-Goat IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32214 | (WB: 1:10000) |
| IRDye 800CW Donkey anti-Mouse IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32212 | (WB: 1:10000) |
| Isotemp Digital Dry Bath | Fisher Scientific | 88860022 | |
| K63 Di-Ubiquitin | South Bay Bio LLC | SBB-UP0072 | |
| LB Agar | Genesee Scientific | 11-119 | |
| LB Broth | Genesee Scientific | 11-118 | |
| Lysozyme | Gold Biotechnology | L-040-100 | |
| MaxQ 4000 Benchtop Orbital Shaker | Thermo Fisher Scientific | SHKE4000-7 | |
| MES-SDS Running Buffer | Boston Bioproducts Inc | BP-177 | |
| Mini Tube Rotator | Fisher Scientific | 88-861-051 | |
| NuPage LDS Sample buffer 4x | Thermo Fisher Scientific | NP0007 | |
| Odyssey Fc Imager | LI-COR Biosciences | 43214 | |
| PageRuler Plus Ladder | Thermo Fisher Scientific | 26620 | |
| pGEX6P1 human USP27X | MRC-PPU Reagents and Services | DU21193 | https://mrcppureagents.dundee.ac.uk/ |
| pGEX6P1 human USP27X F313V | Addgene | 225715 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X S404N | Addgene | 225717 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X Y381H | Addgene | 225716 | Koch et at 2024 (PMID: 38182161) |
| Pierce Glutathione Agarose | Thermo Fisher Scientific | 16100 | |
| PMSF (Phenylmethylsulfonyl fluoride) | Gold Biotechnology | P-470-10 | |
| Polysorbate 20 (Tween 20) | Fisher Scientific | AC233360010 | |
| Rosetta 2 Competent Cells | Millipore Sigma | 71402-M | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6060 | |
| SmartSpec 3000 | Bio-Rad | 170-2501 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Sodium chloride | Genesee Scientific | 18-216 | |
| Sonifier 250 | Branson | 100-132-135 | |
| Sorvall RC 6 Plus Centrifuge | Thermo Fisher Scientific | 46910 | |
| TCEP (Tris-(carboxyethyl) phosphine hydrochloride) | Gold Biotechnology | TCEP10 | |
| Terrific Broth Powder | Genesee Scientific | 18-225 | |
| Tris Base | Genesee Scientific | 18-146 | |
| XCell SureLock Mini-Cell and XCell II Blot Module | Thermo Fisher Scientific | EI0002 |
References
- Morris-Rosendahl, D. J., Crocq, M. -A. Neurodevelopmental disorders: the history and future of a diagnostic concept. Dialogues Clin Neurosci. 22 (1), 65-72 (2020).
- Ebstein, F., Küry, S., Papendorf, J. J., Krüger, E. Neurodevelopmental disorders (NDD) caused by genomic alterations of the ubiquitin-proteasome system (UPS): the possible contribution of immune dysregulation to disease pathogenesis. Front Mol Neurosci. 14, 733012(2021).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annu Rev Biochem. 67 (10), 425-479 (1998).
- Pickart, C. M. Mechanisms underlying ubiquitination. Annu Rev Biochem. 70 (1), 503-533 (2001).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Yau, R., Rape, M. The increasing complexity of the ubiquitin code. Nat Cell Biol. 18 (6), 579-586 (2016).
- Koch, I., et al. USP27X variants underlying X-linked intellectual disability disrupt protein function via distinct mechanisms. Life Sci Alliance. 7 (3), e202302258(2024).
- Hao, Y. -H., et al. USP7 acts as a molecular rheostat to promote WASH-dependent endosomal protein recycling and is mutated in a human neurodevelopmental disorder. Mol Cell. 59 (6), 956-969 (2015).
- Fountain, M. D., et al. Pathogenic variants in USP7 cause a neurodevelopmental disorder with speech delays, altered behavior, and neurologic anomalies. Genet Med. 21 (8), 1797-1807 (2019).
- Santiago-Sim, T., et al. Biallelic variants in OTUD6B cause an intellectual disability syndrome associated with seizures and dysmorphic features. Am J Hum Genet. 100 (4), 676-688 (2017).
- Beck, D. B., et al. Linkage-specific deubiquitylation by OTUD5 defines an embryonic pathway intolerant to genomic variation. Sci Adv. 7 (4), eabe2116(2021).
- Cho, J., Park, J., Kim, E. E., Song, E. J. Assay systems for profiling deubiquitinating activity. Int J Mol Sci. 21 (16), 1-16 (2020).
- Matsui, S., Sandberg, A. A., Negoro, S., Seon, B. K., Goldstein, G. Isopeptidase: A novel eukaryotic enzyme that cleaves isopeptide bonds. Proc Natl Acad Sci USA. 79 (5), 1535-1539 (1982).
- Rose, I. A., Warms, J. V. B. An enzyme with ubiquitin carboxy-terminal esterase activity from reticulocytes. Biochemistry. 22 (18), 4234-4237 (1983).
- Wilkinson, K. D., Cox, M. J., Mayer, A. N., Frey, T. Synthesis and characterization of ubiquitin ethyl ester, a new substrate for ubiquitin carboxyl-terminal hydrolase. Biochemistry. 25 (21), 6644-6649 (1986).
- Pickart, C. M., Rose, I. A. Ubiquitin carboxyl-terminal hydrolase acts on ubiquitin carboxyl-terminal amides. J Biol Chem. 260 (13), 7903-7910 (1985).
- Hadari, T., Warms, J. V. B., Rose, I. A., Hershko, A. A ubiquitin C-terminal isopeptidase that acts on polyubiquitin chains: Role in protein degradation. J Biol Chem. 267 (2), 719-727 (1992).
- Woo, S. K., et al. Multiple ubiquitin C-terminal hydrolases from chick skeletal muscle. J Biol Chem. 270 (32), 18766-18773 (1995).
- Woo, S. K., et al. Purification and characterization of a new ubiquitin C-terminal hydrolase (UCH-1) with isopeptidase activity from chick skeletal muscle. J Biochem. 121 (4), 684-689 (1997).
- Baek, S. H., et al. Molecular cloning of a novel ubiquitin-specific protease, UBP41, with isopeptidase activity in chick skeletal muscle. J Biol Chem. 272 (41), 25560-25565 (1997).
- Wilkinson, K. D., et al. Metabolism of the polyubiquitin degradation signal: Structure, mechanism, and role of isopeptidase T. Biochemistry. 34 (44), 14535-14546 (1995).
- Hu, M., et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell. 111 (7), 1041-1054 (2002).
- Hu, M., et al. Structure and mechanisms of the proteasome-associated deubiquitinating enzyme USP14. EMBO J. 24 (21), 3747-3756 (2005).
- Gorka, M., Magnussen, H. M., Kulathu, Y. Chemical biology tools to study deubiquitinases and UBL proteases. Semin Cell Dev Biol. 132, 86-96 (2022).
- Faesen, A. C., et al. The differential modulation of USP activity by internal regulatory domains, interactors, and eight ubiquitin chain types. Chem Biol. 18 (12), 1550-1561 (2011).
- Mevissen, T. E. T., et al. OTU deubiquitinases reveal mechanisms of linkage specificity and enable ubiquitin chain restriction analysis. Cell. 154 (1), 169-184 (2013).
- Kwasna, D., et al. Discovery and characterization of ZUFSP/ZUP1, a distinct deubiquitinase class important for genome stability. Mol Cell. 70 (1), 150-164.e6 (2018).
- Hu, H., et al. X-exome sequencing of 405 unresolved families identifies seven novel intellectual disability genes. Mol Psychiatry. 21 (1), 133-148 (2016).
- Hartmann, I. Shaker Orbit - Revolving in Space Around the Samples? Eppendorf Lab Academy. Eppendorf Lab Academy. , at https://www.eppendorf.com/us-en/lab-academy/lab-solutions/others/shaker-orbit-revolving-in-space-around-the-samples (2020).
- Smith, D. B., Johnson, K. S. Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene. 67 (1), 31-40 (1988).
- Ritorto, M. S., et al. Screening of DUB activity and specificity by MALDI-TOF mass spectrometry. Nat Commun. 5, 4763(2014).
- Bustos, F., Bustos, F. Immunoblotting using precast gels. , protocols.io (2024).
- Wingfield, P. T. Overview of the purification of recombinant proteins. Curr Protoc Protein Sci. 80, 6.1.1-6.1.35 (2015).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved