需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过体外泛素链切割测定法测量神经发育障碍相关去泛素化酶的酶活性
摘要
该方案解释了如何通过将重组蛋白纯化与泛素链切割测定相结合来测量与神经发育障碍相关的遗传变异对去泛素化酶活性的影响。
摘要
神经发育障碍 (NDD) 与神经系统功能受损有关,但在分子水平上往往知之甚少。由单个基因引起的离散疾病为研究驱动非典型神经发育的机制提供了模型。编码去泛素化酶 (DUB) 家族蛋白的基因变体与多种 NDD 相关,但需要确定由这些基因变体驱动的疾病的致病机制。基因变异对 DUB 活性的影响可以使用不依赖底物的 体外 泛素切割测定法通过实验确定。该测定不需要了解下游底物即可直接测量催化活性。在这里,确定基因变异对酶活性影响的方案使用 DUB 泛素特异性蛋白酶 27,X 连锁 (USP27X) 进行建模,该蛋白酶在 X 连锁智力障碍 105 (XLID105) 中发生突变。该实验管道可用于阐明由 DUB 基因变异驱动的神经发育障碍的潜在机制。
引言
神经发育障碍 (NDD) 由多种病因引起,环境或遗传决定因素驱动非典型神经系统发育1。下一代测序基因检测已将泛素系统相关基因中越来越多的变异与遗传 NDD 联系起来2。泛素系统催化小蛋白修饰物泛素主要与蛋白质底物中的赖氨酸残基连接,以驱动细胞行为的变化,包括定位、稳定性、蛋白质-蛋白质相互作用或活性3。泛素化由 E1 激活、E2 偶联和 E3 连接酶介导4 ,并且可通过去泛素化酶 (DUB) 的活性进行可逆,去泛素化酶 (DUB) 催化蛋白质底物中泛素的裂解和去除5。泛素可以作为单体(单泛素化)或聚合物链(多泛素化)连接到底物上,这些单体在泛素的七个赖氨酸残基(K6、K11、K27、K29、K33、K48、K63)或泛素的 M1 残基上形成。这些不同的泛素链拓扑结构及其组合创造了一个细胞密码,这是信号转导的关键6。
USP27X、USP7、USP9X、USP48、STAMBP、OTUD4、OTUD6B 和 OTUD5 等 DUB 与 NDD2、7、8、9、10、11 有关。对于大多数 NDD,驱动发病机制的分子机制在实验中仍未明确。一些驱动最近描述的疾病的 DUB 知之甚少,并且缺乏可用于评估遗传变异对蛋白质功能影响的已知细胞读数。 在体外,泛素链切割测定克服了这一限制,因为不依赖底物的 DUB 活性读数可以测量基因变异对酶活性的影响12。
自 1980 年代以来,体外泛素切割测定一直在使用。这些使用放射性标记底物的测定发现了第一批 DUB,包括异肽酶,其去泛素化组蛋白 H2A的能力 13,以及泛素羧基末端水解酶 (UCH),其水解各种化学偶联物中的泛素的能力 14,15,16.此外,放射性标记的多泛素化全长蛋白或肽分别用于鉴定红细胞和骨骼肌中的异肽酶 T 和几种 UCH 和泛素特异性蛋白酶 (UBP)17、18、19、20。定义长度和键类型的泛素链 (K48-linked tetra-ubiquitin) 首先用于测量异肽酶 T21 的 DUB 活性。从那时起,该测定已成为在突变分析中测量 DUB 活性的金标准22,23。该检测的改进目前允许通过电泳和常规凝胶染色(如考马斯蓝、SYPRO 橙、红宝石和银染)或基于荧光或免疫印迹的检测来观察泛素切割12,24。DUB 活性的分子方面,例如最小链长和键特异性 25,26,27,28,可以通过在功能测定中使用不同长度的泛素链(例如,二、三、四泛素)和键(K6、K11、K27、K29、K33、K48、K63、线性)来阐明。NDD 相关变体可驱动泛素链键类型特异性11 的 DUB 活性缺陷。
使用纯化的重组 DUB 蛋白的二泛素切割测定可以直接测量 NDD 变体对 DUB 活性的影响。USP27X 在 NDD X 连锁智力障碍 105 (XLID105)7,28 中发生突变,模拟了此处介绍的过程。这种方法允许确定 DUB 活性如何被现有和未知 DUB 相关 NDD 中的基因变异破坏。
Access restricted. Please log in or start a trial to view this content.
研究方案
以下方案可适用于使用在不同感受态细胞菌株中表达的各种亲和标签的重组蛋白。根据表达的蛋白质,培养和过夜表达条件可能需要在表达诱导、表达时间、表达温度和 IPTG 浓度方面优化 OD600 。该协议的概述如图 1 所示。本研究中使用的试剂和设备的详细信息列在 材料表中。
1. 使用重组 GST-USP27X 表达质粒转化感受态 Rosetta 2 大肠杆菌细胞
注:为了保持细菌培养物的无菌性,请执行在本生灯火焰下打开培养基容器的步骤。为了实现最佳的氧气转移,在轨道为 19 mm 至 50 mm、速度为 200 rpm29 的台式温控摇床中进行细菌培养振荡。
- 使用前立即在冰上解冻 20 μL 化学感受态 Rosetta 2 大肠杆菌 细胞。加入 1-10 ng pGEX6P1-USP27X 表达质粒(编码 N 末端谷胱甘肽-S-转移酶 (GST)30 标记的 USP27X,含有氨苄青霉素抗性),并在冰上孵育 5 分钟。
- 在 42 °C 下在干浴中热休克 30 秒。在冰上孵育 2 分钟。
- 添加 80 μL 室温 (RT) SOC 培养基。在 37 °C 下以 200 rpm 的转速在 19 mm 轨道台式温控摇床中孵育 60 分钟。
- 在补充有 25 μg/L 氯霉素和 50 μg/L 氨苄青霉素的 LB 琼脂平板上接种 50 μL 培养物。在 37 °C 下在温控培养箱中孵育 20 小时。
2. 来自表达质粒的重组蛋白的过夜细菌表达
注意:为了保持细菌培养物的无菌性,请执行在本生灯火焰下打开培养基容器的步骤。为了实现最佳的氧气转移,在轨道为 19 mm 至 50 mm、速度为 200 rpm29 的台式温控摇床中进行细菌培养振荡。使用分光光度计测量培养物 OD600 。
- 在 2 L 带挡板的培养瓶中,将 47.6 g TB 粉末加入 1 L 含 0.4% 甘油的超纯水中,制备 1 L Terrific Broth (TB) 培养基。将培养基高压灭菌 30 分钟并冷却至 RT。
- 挑选一个转化细菌菌落,加入 10 mL 补充有 25 μg/L 氯霉素和 50 μg/L 氨苄青霉素的无菌 LB 培养基中。在 37 °C 下以 200 rpm 的转速在 19 mm 轨道台式温控振荡器中孵育 20 小时。
- 将 10 mL 过夜培养物添加到 1 L 补充有 25 μg/L 氯霉素和 50 μg/L 氨苄青霉素的 TB 培养基中(起始培养物与表达培养物的比例为 1:100)。在 37 °C 下在 19 mm 轨道台式温控振荡器中以 200 rpm 旋转孵育,直到培养物 OD600 在 0.5-0.6 之间。
- 向培养物中加入 50 μM IPTG 以诱导表达,冷却至 16 °C,并在 16 °C 下以 200 rpm 旋转孵育 20 小时。
- 通过在 ≥3,000 x g 和 4 °C 下离心 20 分钟,从表达培养物中沉淀细胞。 将沉淀在 -80 °C 下储存至少 1 小时。
注意:此时,可以暂停实验并稍后重新启动(最好是同一周)。颗粒可在 -80 °C 下长期储存。
3. 通过重力流亲和柱纯化蛋白质
注:适用于每种纯化的填料、结合、洗涤、洗脱和储存缓冲液将取决于被纯化的重组蛋白。在 SDS-PAGE 缓冲液中从细胞沉淀、上清液、流出液、洗涤组分和洗脱组分中收集样品。对样品进行 SDS-PAGE 和考马斯染色,以评估纯化的成功。在 4 °C 下进行纯化并在冰上处理馏分。可以在柱上或柱下使用适当的蛋白酶靶向相关的蛋白酶特异性切割位点进行蛋白质标签的切割。
- 将空的重力流柱固定在蒸馏架中,并用谷胱甘肽琼脂糖树脂填充。使用 2-3 mL 树脂纯化从 1 L 表达培养物中收集的细胞沉淀。
- 用一个树脂床体积的 20% 乙醇清洗色谱柱。用一个树脂床体积的 MS500 缓冲液(20 mM Tris pH 7.5、500 mM NaCl、0.5 mM TCEP)洗涤柱 3 次。停止色谱柱,使树脂保持被缓冲液覆盖,以防止在制备细胞沉淀时干燥。
- 在 4 °C 下解冻细胞沉淀。 向解冻的沉淀中加入 30 mL MS500 缓冲液(20 mM Tris pH 7.5、500 mM NaCl、0.5 mM TCEP、60 mg 溶菌酶和一片蛋白酶抑制剂片剂)。在 4 °C 下裂解 30 分钟,轻轻地端到端旋转。
- 在 50 mL 离心管或冰上金属烧杯中对裂解细胞进行超声处理,直到裂解物从移液器吸头分配时自由流动。在 4 °C 和 ≥20,000 x g 下离心 30 分钟以清除上清液。
注意:根据经验确定超声仪设置。设置超声仪,使 120 秒的总时间足以将粘性裂解物减少为自由流动的透明液体。 - 将上清液倒入烧杯中,将澄清的裂解物加载到色谱柱上。通过重力流使裂解液流经色谱柱,同时收集流过的液流。将收集的流经填充到色谱柱中,然后使其穿过色谱柱。
- 用至少两个树脂床体积的 MS500 洗涤缓冲液(20 mM Tris pH 7.5、500 mM NaCl、0.5 mM TCEP)洗涤色谱柱 5 次。以 5 mL 馏分收集洗涤液流出。将 1 μL 每种馏分添加到 100 μL Bradford 试剂中,目视检查是否存在蛋白质。洗涤直到最后一次洗涤中不再存在未结合的蛋白质,必要时增加额外的洗涤步骤。
- 通过色谱柱运行 MS500 洗脱缓冲液(补充有 10 mM 谷胱甘肽和 10 mM NaOH 的 MS500 缓冲液),收集 5 mL 洗脱馏分来回收蛋白质。向 100 μL Bradford 试剂中加入 1 μL 洗脱液,检查是否存在蛋白质。当 Bradford 试剂不再指示存在蛋白质时,停止收集馏分。
- 通过沉淀和离心进行缓冲液置换。向洗脱液中加入 2 体积的 4 M 硫酸铵,轻轻倒置至浑浊,然后在 4 °C 和 ≥20,000 x g 下离心 30 分钟,然后再离心 5 分钟,从而沉淀蛋白质。每次离心后除去上清液,不要干扰蛋白质沉淀。
- 将蛋白质重新溶解并储存在补充有 25% 甘油的 MS500 中。将蛋白质沉淀或蛋白质储存在 -80 °C 的储存缓冲液中。
4. 体外泛 素链切割测定
注:根据先前报告31 中描述的 DUB 特异性或根据经验确定的泛素链长度和连锁类型。如有必要,该方案可用于在一组具有确定长度和键类型的市售泛素链上测试感兴趣的野生型 DUB 的活性。375-750 ng 的双泛素链量和 1-2 μM 的 DUB 浓度可用作测定的起点27。
- 准备 10x DUB 活化缓冲液(500 mM Tris-HCl pH 7.5、500 mM NaCl 和 100 mM TCEP)。
- 对于每个 DUB 的每个时间点,在 1x DUB 激活缓冲液(DUB 混合物)中制备 10 μL 共 2 μM 纯化的 GST-USP27X。准备预混液并将其分成多个时间点。
- 在 RT 下将 DUB 混合物孵育 10 分钟。
- 在添加泛素链之前,将 7 μL SDS-PAGE 上样缓冲液添加到时间 0 中,以防止去泛素化反应开始。
- 对于每个 DUB 的每个时间点,加入 375 ng 在 10 μL 1x DUB 活化缓冲液中稀释的 K-63 二泛素链。每个反应的总体积为 20 μL。
- 将试管在 30 °C 下孵育,用 7 μL SDS-PAGE 上样缓冲液停止每个时间点。
- 使用 4%-12% 梯度凝胶和免疫印迹 7,32 对泛素和 USP27X 进行 SDS-PAGE,以分析选定时间点上单泛素存在的变化。
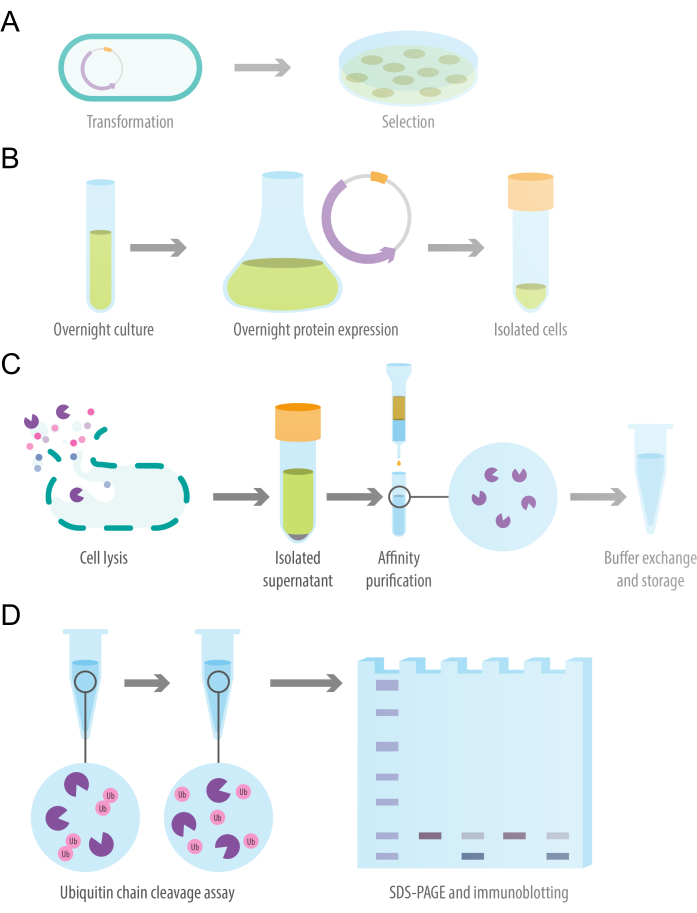
图 1:研究设计示意图。(A) 用重组蛋白表达质粒转化感受态大肠杆菌细胞。(B) 重组去泛素化酶蛋白的过夜细菌表达。(C) 使用重力流亲和柱对重组去泛素酶进行蛋白质纯化。(D) 体外泛素链切割测定,以评估去泛素化活性。请单击此处查看此图的较大版本。
Access restricted. Please log in or start a trial to view this content.
结果
为了确定 XLID105 相关变体对 USP27X 催化活性的影响,从细菌中纯化了 GST 标记的野生型 USP27X 和 XLID105 相关变体 F313V、Y381H 和 S404N USP27X 蛋白。这些变体位于 USP27X 的 USP 催化结构域内(图 2A)。由于先前报道 USP27X 可裂解 K63 泛素链31,因此将野生型 USP27X 和 XLID105 相关变体 F313V、Y381H 和 S404N USP27X 蛋白与 K63 连接的双泛素链孵育 1 小时...
Access restricted. Please log in or start a trial to view this content.
讨论
本文介绍了一种重组 USP27X DUB 的表达和纯化方案以及 体外 泛素链切割测定,以比较野生型 USP27X 和 NDD 相关变体蛋白的去泛素化活性。该测定确定 XLID105 相关变体会破坏 USP27X 催化活性7。这种机制见解帮助我们将 XLID105定义为 USP27X 功能破坏障碍。
该方案可以适用于与分子机制知之甚少的遗传疾病相关的其他 DUB。针对特定目?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者声明没有利益冲突。
致谢
这项工作由 Sanford Research 启动基金资助给 FB 和 NIH 赠款R01CA233700 MJS。这幅画是由 Felipe G. Serrano (www.illustrative-science.com) 完成的。我们感谢 Greg Findlay 博士(邓迪大学)的 GST-USP27X 质粒。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Amersham Protran 0.45 NC 200 mm × 200 mm 25/PK | Cytiva | 10600041 | |
| Ammonium sulfate | Fisher Scientific | AC205872500 | |
| Ampicillin | Fisher Scientific | BP1760 25 | |
| Anti- Ubiquitin (Mouse monoclonal) | Biolegend | Cat# 646302, RRID:AB_1659269 | (WB: 1:1000) |
| Anti-GST (Sheep polyclonal) | MRC-PPU Reagents and Services | Cat# S902A Third bleed | (WB: 1:1000) https://mrcppureagents.dundee.ac.uk/ |
| Baffled Culture Flasks 2 L | Fisher Scientific | 10-042-5N | |
| Bradford Reagent | Millipore Sigma | B6916-500ML | |
| Chloramphenicol | Gold Biotechnology | C-105-25 | |
| Complete, Protease Inhibitor tablets | Millipore Sigma | 5056489001 | |
| Econo-Column 1.5 × 5 cm | Bio-Rad | 7371507 | |
| Eppendorf ThermoMixer F1.5 | Eppendorf | 5384000020 | |
| Excel | Microsoft | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Glycerol | Genesee Scientific | 18-205 | |
| Illustrator | Adobe | https://www.adobe.com/products/illustrator.html | |
| Image Studio | LI-COR Biosciences | https://www.licor.com/bio/image-studio/ | |
| Inkscape | Inkscape | https://inkscape.org/ | |
| Invitrogen 4-12% NuPAGE 1mm 12 well gel | Thermo Fisher Scientific | NP0322BOX | |
| IPTG (Isopropyl-b-D-Thiogalactopyranoside) | Genesee Scientific | 20-109 | |
| IRDye 800CW Donkey anti-Goat IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32214 | (WB: 1:10000) |
| IRDye 800CW Donkey anti-Mouse IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32212 | (WB: 1:10000) |
| Isotemp Digital Dry Bath | Fisher Scientific | 88860022 | |
| K63 Di-Ubiquitin | South Bay Bio LLC | SBB-UP0072 | |
| LB Agar | Genesee Scientific | 11-119 | |
| LB Broth | Genesee Scientific | 11-118 | |
| Lysozyme | Gold Biotechnology | L-040-100 | |
| MaxQ 4000 Benchtop Orbital Shaker | Thermo Fisher Scientific | SHKE4000-7 | |
| MES-SDS Running Buffer | Boston Bioproducts Inc | BP-177 | |
| Mini Tube Rotator | Fisher Scientific | 88-861-051 | |
| NuPage LDS Sample buffer 4x | Thermo Fisher Scientific | NP0007 | |
| Odyssey Fc Imager | LI-COR Biosciences | 43214 | |
| PageRuler Plus Ladder | Thermo Fisher Scientific | 26620 | |
| pGEX6P1 human USP27X | MRC-PPU Reagents and Services | DU21193 | https://mrcppureagents.dundee.ac.uk/ |
| pGEX6P1 human USP27X F313V | Addgene | 225715 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X S404N | Addgene | 225717 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X Y381H | Addgene | 225716 | Koch et at 2024 (PMID: 38182161) |
| Pierce Glutathione Agarose | Thermo Fisher Scientific | 16100 | |
| PMSF (Phenylmethylsulfonyl fluoride) | Gold Biotechnology | P-470-10 | |
| Polysorbate 20 (Tween 20) | Fisher Scientific | AC233360010 | |
| Rosetta 2 Competent Cells | Millipore Sigma | 71402-M | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6060 | |
| SmartSpec 3000 | Bio-Rad | 170-2501 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Sodium chloride | Genesee Scientific | 18-216 | |
| Sonifier 250 | Branson | 100-132-135 | |
| Sorvall RC 6 Plus Centrifuge | Thermo Fisher Scientific | 46910 | |
| TCEP (Tris-(carboxyethyl) phosphine hydrochloride) | Gold Biotechnology | TCEP10 | |
| Terrific Broth Powder | Genesee Scientific | 18-225 | |
| Tris Base | Genesee Scientific | 18-146 | |
| XCell SureLock Mini-Cell and XCell II Blot Module | Thermo Fisher Scientific | EI0002 |
参考文献
- Morris-Rosendahl, D. J., Crocq, M. -A. Neurodevelopmental disorders: the history and future of a diagnostic concept. Dialogues Clin Neurosci. 22 (1), 65-72 (2020).
- Ebstein, F., Küry, S., Papendorf, J. J., Krüger, E. Neurodevelopmental disorders (NDD) caused by genomic alterations of the ubiquitin-proteasome system (UPS): the possible contribution of immune dysregulation to disease pathogenesis. Front Mol Neurosci. 14, 733012(2021).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annu Rev Biochem. 67 (10), 425-479 (1998).
- Pickart, C. M. Mechanisms underlying ubiquitination. Annu Rev Biochem. 70 (1), 503-533 (2001).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Yau, R., Rape, M. The increasing complexity of the ubiquitin code. Nat Cell Biol. 18 (6), 579-586 (2016).
- Koch, I., et al. USP27X variants underlying X-linked intellectual disability disrupt protein function via distinct mechanisms. Life Sci Alliance. 7 (3), e202302258(2024).
- Hao, Y. -H., et al. USP7 acts as a molecular rheostat to promote WASH-dependent endosomal protein recycling and is mutated in a human neurodevelopmental disorder. Mol Cell. 59 (6), 956-969 (2015).
- Fountain, M. D., et al. Pathogenic variants in USP7 cause a neurodevelopmental disorder with speech delays, altered behavior, and neurologic anomalies. Genet Med. 21 (8), 1797-1807 (2019).
- Santiago-Sim, T., et al. Biallelic variants in OTUD6B cause an intellectual disability syndrome associated with seizures and dysmorphic features. Am J Hum Genet. 100 (4), 676-688 (2017).
- Beck, D. B., et al. Linkage-specific deubiquitylation by OTUD5 defines an embryonic pathway intolerant to genomic variation. Sci Adv. 7 (4), eabe2116(2021).
- Cho, J., Park, J., Kim, E. E., Song, E. J. Assay systems for profiling deubiquitinating activity. Int J Mol Sci. 21 (16), 1-16 (2020).
- Matsui, S., Sandberg, A. A., Negoro, S., Seon, B. K., Goldstein, G. Isopeptidase: A novel eukaryotic enzyme that cleaves isopeptide bonds. Proc Natl Acad Sci USA. 79 (5), 1535-1539 (1982).
- Rose, I. A., Warms, J. V. B. An enzyme with ubiquitin carboxy-terminal esterase activity from reticulocytes. Biochemistry. 22 (18), 4234-4237 (1983).
- Wilkinson, K. D., Cox, M. J., Mayer, A. N., Frey, T. Synthesis and characterization of ubiquitin ethyl ester, a new substrate for ubiquitin carboxyl-terminal hydrolase. Biochemistry. 25 (21), 6644-6649 (1986).
- Pickart, C. M., Rose, I. A. Ubiquitin carboxyl-terminal hydrolase acts on ubiquitin carboxyl-terminal amides. J Biol Chem. 260 (13), 7903-7910 (1985).
- Hadari, T., Warms, J. V. B., Rose, I. A., Hershko, A. A ubiquitin C-terminal isopeptidase that acts on polyubiquitin chains: Role in protein degradation. J Biol Chem. 267 (2), 719-727 (1992).
- Woo, S. K., et al. Multiple ubiquitin C-terminal hydrolases from chick skeletal muscle. J Biol Chem. 270 (32), 18766-18773 (1995).
- Woo, S. K., et al. Purification and characterization of a new ubiquitin C-terminal hydrolase (UCH-1) with isopeptidase activity from chick skeletal muscle. J Biochem. 121 (4), 684-689 (1997).
- Baek, S. H., et al. Molecular cloning of a novel ubiquitin-specific protease, UBP41, with isopeptidase activity in chick skeletal muscle. J Biol Chem. 272 (41), 25560-25565 (1997).
- Wilkinson, K. D., et al. Metabolism of the polyubiquitin degradation signal: Structure, mechanism, and role of isopeptidase T. Biochemistry. 34 (44), 14535-14546 (1995).
- Hu, M., et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell. 111 (7), 1041-1054 (2002).
- Hu, M., et al. Structure and mechanisms of the proteasome-associated deubiquitinating enzyme USP14. EMBO J. 24 (21), 3747-3756 (2005).
- Gorka, M., Magnussen, H. M., Kulathu, Y. Chemical biology tools to study deubiquitinases and UBL proteases. Semin Cell Dev Biol. 132, 86-96 (2022).
- Faesen, A. C., et al. The differential modulation of USP activity by internal regulatory domains, interactors, and eight ubiquitin chain types. Chem Biol. 18 (12), 1550-1561 (2011).
- Mevissen, T. E. T., et al. OTU deubiquitinases reveal mechanisms of linkage specificity and enable ubiquitin chain restriction analysis. Cell. 154 (1), 169-184 (2013).
- Kwasna, D., et al. Discovery and characterization of ZUFSP/ZUP1, a distinct deubiquitinase class important for genome stability. Mol Cell. 70 (1), 150-164.e6 (2018).
- Hu, H., et al. X-exome sequencing of 405 unresolved families identifies seven novel intellectual disability genes. Mol Psychiatry. 21 (1), 133-148 (2016).
- Hartmann, I. Shaker Orbit - Revolving in Space Around the Samples? Eppendorf Lab Academy. Eppendorf Lab Academy. , at https://www.eppendorf.com/us-en/lab-academy/lab-solutions/others/shaker-orbit-revolving-in-space-around-the-samples (2020).
- Smith, D. B., Johnson, K. S. Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene. 67 (1), 31-40 (1988).
- Ritorto, M. S., et al. Screening of DUB activity and specificity by MALDI-TOF mass spectrometry. Nat Commun. 5, 4763(2014).
- Bustos, F., Bustos, F. Immunoblotting using precast gels. , protocols.io (2024).
- Wingfield, P. T. Overview of the purification of recombinant proteins. Curr Protoc Protein Sci. 80, 6.1.1-6.1.35 (2015).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。