A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מדידת הפעילות האנזימטית של אנזימים דאוביקוויטילציה הקשורים להפרעה נוירו-התפתחותית באמצעות בדיקת מחשוף שרשרת יוביקוויטין במבחנה
In This Article
Summary
פרוטוקול זה מסביר כיצד למדוד את ההשפעה של שונות גנטית הקשורה להפרעות נוירו-התפתחותיות על פעילות האנזים דאוביקוויטילציה על ידי שילוב טיהור חלבונים רקומביננטיים עם מבחני מחשוף שרשרת יוביקוויטין.
Abstract
הפרעות נוירו-התפתחותיות (NDDs) קשורות לליקויים בתפקוד מערכת העצבים, אך לעתים קרובות נותרות מובנות בצורה גרועה ברמה המולקולרית. הפרעות בדידות הנגרמות על ידי גנים בודדים מספקות מודלים לחקר מנגנונים המניעים התפתחות עצבית לא טיפוסית. גרסאות של גנים המקודדים חלבונים ממשפחת האנזימים deubiquitylating (DUB) קשורות למספר NDDs, אך יש צורך לקבוע את המנגנונים הפתוגניים של הפרעות המונעות על ידי גרסאות גנים אלה. ההשפעה של וריאנטים גנטיים על פעילות DUB ניתנת לקביעה ניסיונית באמצעות בדיקת מחשוף אוביקוויטין חוץ-גופית שאינה תלויה במצע. בדיקה זו אינה דורשת ידע במצעים במורד הזרם כדי למדוד ישירות את הפעילות הקטליטית. כאן, הפרוטוקול לקביעת ההשפעה של וריאנטים גנטיים על פעילות אנזימטית מעוצב באמצעות DUB Ubiquitin Specific Protease 27, X-linked (USP27X), אשר מוטציה במוגבלות אינטלקטואלית X-linked 105 (XLID105). ניתן להשתמש בצנרת ניסויית זו כדי להבהיר את המנגנונים העומדים בבסיס הפרעות נוירו-התפתחותיות המונעות על ידי וריאנטים בגנים של DUB.
Introduction
הפרעות נוירו-התפתחותיות (NDDs) נובעות מאטיולוגיות מגוונות עם גורמים סביבתיים או גנטיים המניעים התפתחות לא טיפוסית של מערכת העצבים1. בדיקות גנטיות של ריצוף הדור הבא קישרו מספר גדל והולך של וריאנטים בגנים הקשורים למערכת יוביקוויטין עם NDDs גנטיים2. מערכת האוביקוויטין מזרזת את קשירת החלבון הקטן יוביקוויטין לשאריות ליזין בעיקר במצעי חלבונים כדי להניע שינויים בהתנהגות התאית, כולל לוקליזציה, יציבות, אינטראקציות חלבון-חלבון, או פעילות3. Ubiquitylation מתווך על ידי E1 activating, E2 conjugating, ו E3 ligase אנזימים4 והוא הפיך על ידי הפעילות של אנזימים deubiquitylating (DUBs) אשר לזרז את הפיצול ואת ההסרה של ubiquitin ממצעים חלבונים5. ניתן לקשור את האוביקוויטין למצע כמונומר (מונוביקוויטילציה) או שרשראות פולימריות (פוליוביקוויטילציה) הנוצרות על כל אחת משבעת שאריות הליזין (K6, K11, K27, K29, K33, K48, K63) או כשאריות M1 של יוביקוויטין. טופולוגיות שרשרת יוביקוויטין שונות אלה והשילובים ביניהן יוצרים קוד תאי שהוא המפתח להעברת אותות6.
DUBs כגון USP27X, USP7, USP9X, USP48, STAMBP, OTUD4, OTUD6B ו- OTUD5 שויכו ל- NDDs 2,7,8,9,10,11. עבור רוב NDDs, המנגנונים המולקולריים המניעים פתוגנזה נותרים בלתי מוגדרים בניסוי. חלק מה-DUBs המניעים הפרעות שתוארו לאחרונה אינם מובנים היטב וחסרים קריאות תאיות ידועות שניתן להשתמש בהן כדי להעריך את ההשפעה של שונות גנטית על תפקוד החלבון. במבחנה, מבחני מחשוף שרשרת יוביקוויטין מתגברים על מגבלה זו כקריאות פעילות DUB בלתי תלויות במצע שיכולות למדוד את ההשפעה של וריאנטים גנטיים על פעילות אנזימטית12.
בדיקות מחשוף יוביקוויטין במבחנה נמצאות בשימוש מאז שנות השמונים. בדיקות אלה באמצעות מצעים מסומנים ברדיו אפשרו את גילוי ה-DUBs הראשונים, כולל איזופפטידאז, שזוהה בשל יכולתו לנטרל את היסטון H2A13, ויוביקוויטין קרבוקסיל-טרמינל הידרולאז (UCH), שזוהה על ידי יכולתו לבצע הידרוליזה של יוביקוויטין ממגוון מצומדים כימיים 14,15,16 . יתר על כן, חלבונים או פפטידים באורך מלא שסומנו ברדיו, שימשו לזיהוי איזופפטידאז T וכמה UCHs ופרוטאזות ספציפיות ליוביקוויטין (UBPs) מאריתרוציטים ושרירי השלד, בהתאמה 17,18,19,20. שרשראות יוביקוויטין באורך מוגדר ובסוג קישור (K48-linked tetra-ubiquitin) שימשו לראשונה למדידת פעילות DUB של איזופפטידאז T21. מאז, בדיקה זו הפכה לתקן הזהב למדידת פעילות DUB בניתוחי מוטציות22,23. העידון של בדיקה זו מאפשר כיום הדמיה של מחשוף יוביקוויטין באמצעות אלקטרופורזה וכתמי ג'ל קונבנציונליים כגון כחול קומאסי, כתום SYPRO, אודם וכתם כסף או זיהוי פלואורסצנטי או מבוסס חיסון12,24. ניתן להבהיר היבטים מולקולריים של פעילות DUB, כגון אורך שרשרת מינימלי וספציפיות קישור 25,26,27,28, באמצעות שרשראות יוביקוויטין באורכים שונים (למשל, di-, tri-, tetra-ubiquitin) וקישורים (K6, K11, K27, K29, K33, K48, K63, ליניארי) בבדיקות פונקציונליות. וריאנטים הקשורים ל- NDD יכולים לגרום לפגמים בפעילות DUB שהם ספציפיים לסוג קישור שרשרת יוביקוויטין11.
בדיקת מחשוף די-יוביקוויטין באמצעות חלבוני DUB רקומביננטיים מטוהרים יכולה למדוד ישירות את ההשפעה של גרסאות NDD על פעילות DUB. USP27X, אשר מוטציה בהפרעת המוגבלות האינטלקטואלית NDD X מקושרת 105 (XLID105)7,28 מודלים התהליך המוצג כאן. גישה זו מאפשרת לקבוע כיצד פעילות DUB מופרעת על ידי וריאנטים גנטיים ב- NDDs קיימים ולא ידועים הקשורים ל- DUB.
Access restricted. Please log in or start a trial to view this content.
Protocol
ניתן להתאים את הפרוטוקול הבא לחלבונים רקומביננטיים באמצעות תגי זיקה שונים המבוטאים בזנים שונים של תאים מוסמכים. בהתאם לחלבון המובע, תנאי ההתרבות והביטוי בן הלילה עשויים לדרוש אופטימיזציה של OD600 בהשראת ביטוי, זמן ביטוי, טמפרטורת ביטוי וריכוז IPTG. סקירה כללית של הפרוטוקול מודגמת באיור 1. פרטי הריאגנטים והציוד ששימש במחקר זה מפורטים בטבלת החומרים.
1. טרנספורמציה של תאי רוזטה 2 E. coli מוכשרים עם פלסמיד ביטוי GST-USP27X רקומביננטי
הערה: כדי לשמור על סטריליות תרבית החיידקים, בצע שלבים שבהם מיכלי מדיה פתוחים תחת להבת מבער בונזן. כדי לאפשר העברת חמצן אופטימלית, בצעו ניעור תרבית חיידקים בשייקר מבוקר טמפרטורה עם מסלול של 19 מ"מ עד 50 מ"מ ומהירות של 200 סל"ד29.
- הפשירו 20 מיקרוליטר של תאי רוזטה 2 E. coli בעלי כשירות כימית על קרח מיד לפני השימוש. הוסף 1-10 ng של פלסמיד ביטוי pGEX6P1-USP27X (קידוד עבור N-terminal Glutathione-S-transferase (GST)30 -מתויג USP27X ומכיל עמידות Ampicillin) ודגור 5 דקות על קרח.
- הלם חום במשך 30 שניות ב 42 ° C באמבטיה יבשה. דוגרים על קרח במשך 2 דקות.
- הוסף 80 μL של טמפרטורת החדר (RT) SOC בינוני. דגירה במשך 60 דקות ב-37°C עם סיבוב של 200 סל"ד בשייקר מבוקר טמפרטורה של 19 מ"מ במסלול.
- צלחת 50 μL של תרבית על צלחת אגר LB בתוספת 25 מיקרוגרם / ליטר Chloramphenicol ו 50 מיקרוגרם / L אמפיצילין. יש לדגור במשך 20 שעות בטמפרטורה של 37°C כלפי מטה באינקובטור מבוקר טמפרטורה.
2. ביטוי חיידקי בן לילה של חלבון רקומביננטי מפלסמיד ביטוי
הערות: כדי לשמור על סטריליות תרבית החיידקים, בצעו שלבים שבהם מיכלי המדיה פתוחים תחת להבת מבער בונזן. כדי לאפשר העברת חמצן אופטימלית, בצעו ניעור תרבית חיידקים בשייקר מבוקר טמפרטורה עם מסלול של 19 מ"מ עד 50 מ"מ ומהירות של 200 סל"ד29. מדוד תרבית OD600 באמצעות ספקטרופוטומטר.
- הכינו 1 ליטר של מרק נהדר (שחפת) בינוני על ידי הוספת 47.6 גרם אבקת שחפת לליטר אחד של מים טהורים במיוחד עם 0.4% גליצרול בצלוחית תרבית מבולבלת בנפח 2 ליטר. Autoclave בינוני במשך 30 דקות וקריר RT.
- בחרו מושבה אחת של חיידקים מותמרים והוסיפו ל-10 מ"ל של LB בינוני סטרילי בתוספת 25 מיקרוגרם/ליטר כלוראמפניקול ו-50 מיקרוגרם/ליטר אמפיצילין. דגירה במשך 20 שעות ב-37°C עם סיבוב של 200 סל"ד בשייקר מבוקר טמפרטורה של ספסל מסלול 19 מ"מ.
- הוסף תרבית לילה של 10 מ"ל ל-1 ליטר של שחפת בינונית בתוספת 25 מיקרוגרם/ליטר כלוראמפניקול ו-50 מיקרוגרם/ליטר אמפיצילין (יחס של 1:100 בין תרבית התחלתית לתרבות ביטוי). יש לדגור ב-37°C עם סיבוב של 200 סל"ד בשייקר מבוקר טמפרטורה של 19 מ"מ עד שהתרבית OD600 תהיה בין 0.5-0.6.
- הוסיפו 50 מיקרומטר של IPTG לתרבית כדי לגרום לביטוי, התקררו ל-16°C ודגרו במשך 20 שעות ב-16°C עם סיבוב של 200 סל"ד.
- תאי גלולה מתרבית ביטוי על ידי צנטריפוגה במשך 20 דקות ב ≥3,000 x גרם ו 4 ° C. יש לאחסן את הגלולה בטמפרטורה של -80°C למשך שעה אחת לפחות.
הערה: בשלב זה, ניתן להשהות את הניסוי ולהתחיל אותו מחדש מאוחר יותר (רצוי באותו שבוע). ניתן לאחסן את הגלולה לטווח ארוך ב -80 מעלות צלזיוס.
3. טיהור חלבונים על ידי עמודת זיקה של זרימת כבידה
הערה: מאגרי השרף, הקשירה, השטיפה, ההדבקה והאחסון המתאימים לכל טיהור תלויים בחלבון הרקומביננטי המטוהר. אסוף דגימות מכדורית התא, מסופרנטנט, מזרימה, משברי שטיפה וממקטעי אלוציה במאגר SDS-PAGE. בצע צביעת SDS-PAGE ו- Coomassie עבור הדגימות כדי להעריך את הצלחת הטיהור. בצעו טיהור בטמפרטורה של 4°C וטפלו בשברים על קרח. ניתן לבצע פיצול של תגי חלבון על העמוד או מחוצה לו באמצעות הפרוטאז המתאים כדי להתמקד באתר המחשוף הרלוונטי הספציפי לפרוטאז.
- אבטחו את עמוד זרימת הכבידה הריק במעמד הרטורט ומלאו אותו בשרף אגרוז גלוטתיון. השתמש 2-3 מ"ל של שרף כדי לטהר גלולה התא שנאסף מ 1 L של תרבית ביטוי.
- שטפו את העמוד בנפח מיטה שרף אחד של 20% אתנול. כבס את העמודה 3 פעמים עם נפח מיטת שרף אחת של מאגר MS500 (20 mM של Tris pH 7.5, 500 mM של NaCl, 0.5 mM של TCEP). עצור את העמוד כך שהשרף יישאר מכוסה בחיץ כדי למנוע התייבשות בעת הכנת כדורית התא.
- הפשיר את גלולת התא ב 4 °C. הוסף 30 מ"ל של מאגר MS500 (20 מילימטר של Tris pH 7.5, 500 mM של NaCl, 0.5 mM של TCEP, 60 מ"ג של ליזוזים וטבליה אחת של מעכב פרוטאז) לגלולה המופשרת. ליז למשך 30 דקות ב-4°C עם סיבוב עדין מקצה לקצה.
- סוניק תאים lysed בצינור צנטריפוגה 50 מ"ל או מתכת על קרח עד הליזט זורם בחופשיות כאשר הוא מנופק מקצה פיפטה. צנטריפוגה למשך 30 דקות ב-4°C ו-≥20,000 x g כדי לנקות את הסופר-נטנט.
הערה: קבע את הגדרות הסוניקטור באופן אמפירי. הגדר את הסוניקטור כך שזמן כולל של 120 שניות יספיק כדי להפחית את הליזט הצמיג לנוזל זורם ושקוף. - דקרו את הסופרנאטנט לתוך והעמיסו ליזט מפונה על העמוד. הרץ ליזט דרך הטור על ידי זרימת כוח הכבידה תוך איסוף הזרימה דרכו. טען את העמודה עם הזרימה שנאספה והפעל אותה דרך העמודה.
- שטוף את העמוד עם לפחות שני נפחי מיטת שרף של מאגר הכביסה MS500 (20 mM של Tris pH 7.5, 500 mM של NaCl, 0.5 mM של TCEP) 5 פעמים. אספו את זרימת השטיפה בשברים של 5 מ"ל. הוסף 1 μL מכל שבר ל- 100 μL של מגיב ברדפורד כדי לבדוק חזותית נוכחות חלבון. יש לשטוף עד שהחלבון שאינו קשור כבר לא נמצא בשטיפה האחרונה, ולהוסיף שלבי שטיפה נוספים במידת הצורך.
- שחזור חלבון על-ידי הפעלת מאגר אלוציה MS500 (מאגר MS500 בתוספת 10 מילימטר גלוטתיון ו-10 מ"מ NaOH) דרך העמודה, ואיסוף שברי אלוציה של 5 מ"ל. בדוק נוכחות חלבון על ידי הוספת 1 μL של פליט ל 100 μL של מגיב ברדפורד. הפסיקו לאסוף שברים כאשר מגיב ברדפורד כבר לא מצביע על כך שחלבונים קיימים.
- בצע חילופי חיץ על ידי משקעים וצנטריפוגה. לזרז חלבון על ידי הוספת 2 כרכים של 4 M של אמוניום סולפט כדי eluate, בעדינות להפוך עד מעונן, ולאחר מכן צנטריפוגה במשך 30 דקות ב 4 ° C ו ≥20,000 x גרם ושוב במשך 5 דקות נוספות. מוציאים את הסופרנאטנט אחרי כל צנטריפוגה בלי להפריע לכדורית החלבון.
- יש להמיס מחדש ולאחסן חלבון ב-MS500 בתוספת 25% גליצרול. יש לאחסן כדורי חלבון או חלבון במאגר אחסון בטמפרטורה של -80°C.
4. בדיקת מחשוף שרשרת יוביקוויטין במבחנה
הערה: בחר אורך שרשרת יוביקוויטין וסוגי קישור בהתבסס על ספציפיות DUB שתוארה בדוחות קודמים31 או נקבעה אמפירית. במידת הצורך, פרוטוקול זה יכול לשמש לבדיקת הפעילות של DUB מסוג פראי של עניין על פאנל של שרשראות יוביקוויטין זמין מסחרית של אורך מוגדר וסוג הצמדה. כמות שרשרת די-יוביקוויטין של 375-750 ננוגרם וריכוז DUB של 1-2 מיקרומטר יכולים לשמש כנקודות התחלה לבדיקה27.
- הכן מאגר הפעלה של 10x DUB (500 mM של Tris-HCl pH 7.5, 500 mM של NaCl ו- 100 mM של TCEP).
- עבור כל נקודת זמן עבור כל DUB, הכינו 10 μL סה"כ של 2 מיקרומטר של GST-USP27X מטוהר במאגר הפעלה 1x DUB (תערובת DUB). הכינו תערובות מאסטר ופצלו אותן לנקודות זמן.
- דגרו על תערובת DUB למשך 10 דקות ב- RT.
- הוסף 7 μL של מאגר טעינת SDS-PAGE לזמן 0 לפני הוספת שרשראות יוביקוויטין כדי למנוע את התחלת תגובת הדוביקוויטילציה.
- לכל נקודת זמן עבור כל DUB, הוסף 375 ng של שרשראות K-63 di-ubiquitin מדוללות במאגר הפעלה של 10 μL 1x DUB. הנפח הכולל הוא 20 μL לכל תגובה.
- יש לדגור על הצינורות בטמפרטורה של 30°C, ולעצור בכל נקודת זמן עם 7 μL של מאגר טעינה מסוג SDS-PAGE.
- בצע SDS-PAGE עם ג'ל הדרגתי של 4%-12% ואימונובלוט 7,32 עבור יוביקוויטין ו-USP27X כדי לנתח את השינוי בנוכחות מונו-יוביקוויטין בנקודות זמן נבחרות.
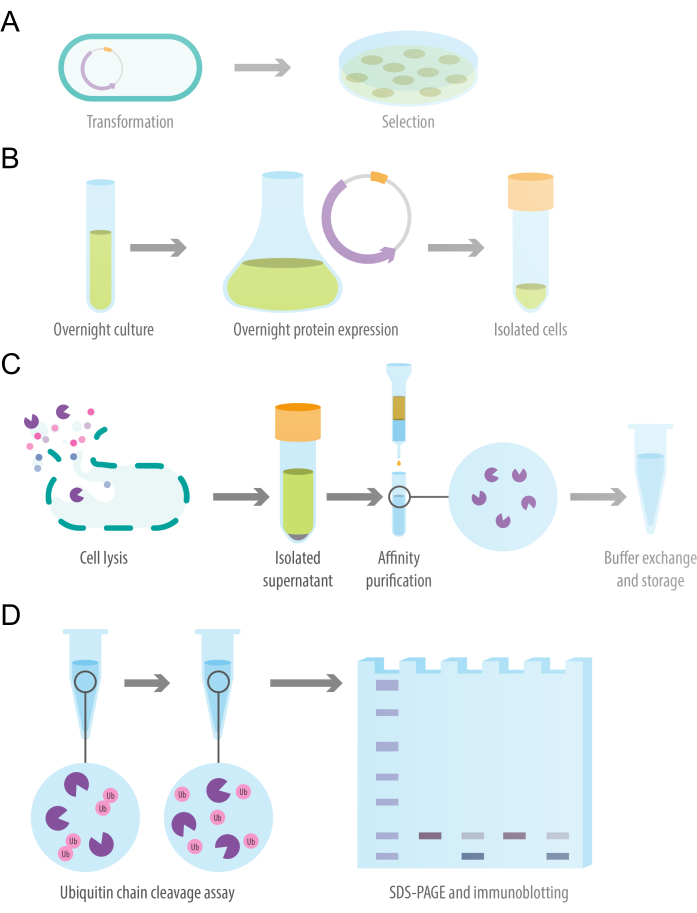
איור 1: סכמטי של תכנון המחקר. (A) טרנספורמציה של תאי E. coli מוכשרים עם פלסמיד ביטוי חלבון רקומביננטי. (B) ביטוי חיידקי בן לילה של חלבון רקומביננטי דאוביקוויטילאז. (C) טיהור חלבונים של דאוביקוויטילאז רקומביננטי באמצעות עמודת זיקה של זרימת כבידה. (D) בדיקת מחשוף שרשרת יוביקוויטין במבחנה להערכת פעילות דאוביקוויטילציה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Access restricted. Please log in or start a trial to view this content.
תוצאות
כדי לקבוע את ההשפעה של וריאנטים הקשורים ל-XLID105 על הפעילות הקטליטית של USP27X, החלבונים USP27X מסוג Wild המסומנים ב-GST והחלבונים F313V, Y381H ו-S404N USP27X הקשורים ל-XLID105 טוהרו מחיידקים. גרסאות אלה ממוקמות בתחום הקטליטי של USP של USP27X (איור 2A). מכיוון שבעבר דווח כי USP27X בקע שרשראות...
Access restricted. Please log in or start a trial to view this content.
Discussion
מאמר זה מציג פרוטוקול לביטוי וטיהור של USP27X DUBs רקומביננטיים ובדיקת מחשוף שרשרת יוביקוויטין במבחנה כדי להשוות את פעילות deubiquitylating של חלבונים מסוג פראי USP27X וחלבונים הקשורים NDD. בדיקה זו קבעה כי וריאנטים הקשורים XLID105 משבשים את הפעילות הקטליטית USP27X7. תובנה מ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
המחברים מצהירים כי אין ניגודי עניינים.
Acknowledgements
עבודה זו מומנה על ידי קרנות סטארט-אפ של סנפורד ריסרץ' ל-FB והמענק של ה-NIH R01CA233700 ל-MJS. יצירת האמנות נעשתה על ידי פליפה ג. סראנו (www.illustrative-science.com). אנו מודים לד"ר גרג פינדליי (אוניברסיטת דנדי) על פלסמיד GST-USP27X.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Amersham Protran 0.45 NC 200 mm × 200 mm 25/PK | Cytiva | 10600041 | |
| Ammonium sulfate | Fisher Scientific | AC205872500 | |
| Ampicillin | Fisher Scientific | BP1760 25 | |
| Anti- Ubiquitin (Mouse monoclonal) | Biolegend | Cat# 646302, RRID:AB_1659269 | (WB: 1:1000) |
| Anti-GST (Sheep polyclonal) | MRC-PPU Reagents and Services | Cat# S902A Third bleed | (WB: 1:1000) https://mrcppureagents.dundee.ac.uk/ |
| Baffled Culture Flasks 2 L | Fisher Scientific | 10-042-5N | |
| Bradford Reagent | Millipore Sigma | B6916-500ML | |
| Chloramphenicol | Gold Biotechnology | C-105-25 | |
| Complete, Protease Inhibitor tablets | Millipore Sigma | 5056489001 | |
| Econo-Column 1.5 × 5 cm | Bio-Rad | 7371507 | |
| Eppendorf ThermoMixer F1.5 | Eppendorf | 5384000020 | |
| Excel | Microsoft | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Glycerol | Genesee Scientific | 18-205 | |
| Illustrator | Adobe | https://www.adobe.com/products/illustrator.html | |
| Image Studio | LI-COR Biosciences | https://www.licor.com/bio/image-studio/ | |
| Inkscape | Inkscape | https://inkscape.org/ | |
| Invitrogen 4-12% NuPAGE 1mm 12 well gel | Thermo Fisher Scientific | NP0322BOX | |
| IPTG (Isopropyl-b-D-Thiogalactopyranoside) | Genesee Scientific | 20-109 | |
| IRDye 800CW Donkey anti-Goat IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32214 | (WB: 1:10000) |
| IRDye 800CW Donkey anti-Mouse IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32212 | (WB: 1:10000) |
| Isotemp Digital Dry Bath | Fisher Scientific | 88860022 | |
| K63 Di-Ubiquitin | South Bay Bio LLC | SBB-UP0072 | |
| LB Agar | Genesee Scientific | 11-119 | |
| LB Broth | Genesee Scientific | 11-118 | |
| Lysozyme | Gold Biotechnology | L-040-100 | |
| MaxQ 4000 Benchtop Orbital Shaker | Thermo Fisher Scientific | SHKE4000-7 | |
| MES-SDS Running Buffer | Boston Bioproducts Inc | BP-177 | |
| Mini Tube Rotator | Fisher Scientific | 88-861-051 | |
| NuPage LDS Sample buffer 4x | Thermo Fisher Scientific | NP0007 | |
| Odyssey Fc Imager | LI-COR Biosciences | 43214 | |
| PageRuler Plus Ladder | Thermo Fisher Scientific | 26620 | |
| pGEX6P1 human USP27X | MRC-PPU Reagents and Services | DU21193 | https://mrcppureagents.dundee.ac.uk/ |
| pGEX6P1 human USP27X F313V | Addgene | 225715 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X S404N | Addgene | 225717 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X Y381H | Addgene | 225716 | Koch et at 2024 (PMID: 38182161) |
| Pierce Glutathione Agarose | Thermo Fisher Scientific | 16100 | |
| PMSF (Phenylmethylsulfonyl fluoride) | Gold Biotechnology | P-470-10 | |
| Polysorbate 20 (Tween 20) | Fisher Scientific | AC233360010 | |
| Rosetta 2 Competent Cells | Millipore Sigma | 71402-M | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6060 | |
| SmartSpec 3000 | Bio-Rad | 170-2501 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Sodium chloride | Genesee Scientific | 18-216 | |
| Sonifier 250 | Branson | 100-132-135 | |
| Sorvall RC 6 Plus Centrifuge | Thermo Fisher Scientific | 46910 | |
| TCEP (Tris-(carboxyethyl) phosphine hydrochloride) | Gold Biotechnology | TCEP10 | |
| Terrific Broth Powder | Genesee Scientific | 18-225 | |
| Tris Base | Genesee Scientific | 18-146 | |
| XCell SureLock Mini-Cell and XCell II Blot Module | Thermo Fisher Scientific | EI0002 |
References
- Morris-Rosendahl, D. J., Crocq, M. -A. Neurodevelopmental disorders: the history and future of a diagnostic concept. Dialogues Clin Neurosci. 22 (1), 65-72 (2020).
- Ebstein, F., Küry, S., Papendorf, J. J., Krüger, E. Neurodevelopmental disorders (NDD) caused by genomic alterations of the ubiquitin-proteasome system (UPS): the possible contribution of immune dysregulation to disease pathogenesis. Front Mol Neurosci. 14, 733012(2021).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annu Rev Biochem. 67 (10), 425-479 (1998).
- Pickart, C. M. Mechanisms underlying ubiquitination. Annu Rev Biochem. 70 (1), 503-533 (2001).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Yau, R., Rape, M. The increasing complexity of the ubiquitin code. Nat Cell Biol. 18 (6), 579-586 (2016).
- Koch, I., et al. USP27X variants underlying X-linked intellectual disability disrupt protein function via distinct mechanisms. Life Sci Alliance. 7 (3), e202302258(2024).
- Hao, Y. -H., et al. USP7 acts as a molecular rheostat to promote WASH-dependent endosomal protein recycling and is mutated in a human neurodevelopmental disorder. Mol Cell. 59 (6), 956-969 (2015).
- Fountain, M. D., et al. Pathogenic variants in USP7 cause a neurodevelopmental disorder with speech delays, altered behavior, and neurologic anomalies. Genet Med. 21 (8), 1797-1807 (2019).
- Santiago-Sim, T., et al. Biallelic variants in OTUD6B cause an intellectual disability syndrome associated with seizures and dysmorphic features. Am J Hum Genet. 100 (4), 676-688 (2017).
- Beck, D. B., et al. Linkage-specific deubiquitylation by OTUD5 defines an embryonic pathway intolerant to genomic variation. Sci Adv. 7 (4), eabe2116(2021).
- Cho, J., Park, J., Kim, E. E., Song, E. J. Assay systems for profiling deubiquitinating activity. Int J Mol Sci. 21 (16), 1-16 (2020).
- Matsui, S., Sandberg, A. A., Negoro, S., Seon, B. K., Goldstein, G. Isopeptidase: A novel eukaryotic enzyme that cleaves isopeptide bonds. Proc Natl Acad Sci USA. 79 (5), 1535-1539 (1982).
- Rose, I. A., Warms, J. V. B. An enzyme with ubiquitin carboxy-terminal esterase activity from reticulocytes. Biochemistry. 22 (18), 4234-4237 (1983).
- Wilkinson, K. D., Cox, M. J., Mayer, A. N., Frey, T. Synthesis and characterization of ubiquitin ethyl ester, a new substrate for ubiquitin carboxyl-terminal hydrolase. Biochemistry. 25 (21), 6644-6649 (1986).
- Pickart, C. M., Rose, I. A. Ubiquitin carboxyl-terminal hydrolase acts on ubiquitin carboxyl-terminal amides. J Biol Chem. 260 (13), 7903-7910 (1985).
- Hadari, T., Warms, J. V. B., Rose, I. A., Hershko, A. A ubiquitin C-terminal isopeptidase that acts on polyubiquitin chains: Role in protein degradation. J Biol Chem. 267 (2), 719-727 (1992).
- Woo, S. K., et al. Multiple ubiquitin C-terminal hydrolases from chick skeletal muscle. J Biol Chem. 270 (32), 18766-18773 (1995).
- Woo, S. K., et al. Purification and characterization of a new ubiquitin C-terminal hydrolase (UCH-1) with isopeptidase activity from chick skeletal muscle. J Biochem. 121 (4), 684-689 (1997).
- Baek, S. H., et al. Molecular cloning of a novel ubiquitin-specific protease, UBP41, with isopeptidase activity in chick skeletal muscle. J Biol Chem. 272 (41), 25560-25565 (1997).
- Wilkinson, K. D., et al. Metabolism of the polyubiquitin degradation signal: Structure, mechanism, and role of isopeptidase T. Biochemistry. 34 (44), 14535-14546 (1995).
- Hu, M., et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell. 111 (7), 1041-1054 (2002).
- Hu, M., et al. Structure and mechanisms of the proteasome-associated deubiquitinating enzyme USP14. EMBO J. 24 (21), 3747-3756 (2005).
- Gorka, M., Magnussen, H. M., Kulathu, Y. Chemical biology tools to study deubiquitinases and UBL proteases. Semin Cell Dev Biol. 132, 86-96 (2022).
- Faesen, A. C., et al. The differential modulation of USP activity by internal regulatory domains, interactors, and eight ubiquitin chain types. Chem Biol. 18 (12), 1550-1561 (2011).
- Mevissen, T. E. T., et al. OTU deubiquitinases reveal mechanisms of linkage specificity and enable ubiquitin chain restriction analysis. Cell. 154 (1), 169-184 (2013).
- Kwasna, D., et al. Discovery and characterization of ZUFSP/ZUP1, a distinct deubiquitinase class important for genome stability. Mol Cell. 70 (1), 150-164.e6 (2018).
- Hu, H., et al. X-exome sequencing of 405 unresolved families identifies seven novel intellectual disability genes. Mol Psychiatry. 21 (1), 133-148 (2016).
- Hartmann, I. Shaker Orbit - Revolving in Space Around the Samples? Eppendorf Lab Academy. Eppendorf Lab Academy. , at https://www.eppendorf.com/us-en/lab-academy/lab-solutions/others/shaker-orbit-revolving-in-space-around-the-samples (2020).
- Smith, D. B., Johnson, K. S. Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene. 67 (1), 31-40 (1988).
- Ritorto, M. S., et al. Screening of DUB activity and specificity by MALDI-TOF mass spectrometry. Nat Commun. 5, 4763(2014).
- Bustos, F., Bustos, F. Immunoblotting using precast gels. , protocols.io (2024).
- Wingfield, P. T. Overview of the purification of recombinant proteins. Curr Protoc Protein Sci. 80, 6.1.1-6.1.35 (2015).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved