Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mesure de l’activité enzymatique d’enzymes désubiquitylantes associées à des troubles neurodéveloppementaux au moyen d’un test de clivage de la chaîne d’ubiquitine in vitro
Dans cet article
Résumé
Ce protocole explique comment mesurer l’effet de la variation génétique associée aux troubles neurodéveloppementaux sur l’activité de l’enzyme de désubiquitylation en combinant la purification des protéines recombinantes avec des tests de clivage de la chaîne d’ubiquitine.
Résumé
Les troubles neurodéveloppementaux (TND) sont associés à des altérations de la fonction du système nerveux, mais restent souvent mal compris au niveau moléculaire. Les troubles discrets causés par des gènes uniques fournissent des modèles pour étudier les mécanismes à l’origine du neurodéveloppement atypique. Des variants de gènes codant pour des protéines de la famille des enzymes deubiquitylantes (DUB) sont associés à plusieurs NDD, mais il est nécessaire de déterminer les mécanismes pathogènes des troubles induits par ces variants génétiques. L’impact des variantes génétiques sur l’activité de DUB peut être déterminé expérimentalement à l’aide d’un test de clivage de l’ubiquitine in vitro indépendant du substrat. Ce test ne nécessite pas de connaissance des substrats en aval pour mesurer directement l’activité catalytique. Ici, le protocole permettant de déterminer l’impact des variantes génétiques sur l’activité enzymatique est modélisé à l’aide de la protéase spécifique de l’ubiquitine DUB 27, liée à l’X (USP27X), qui est mutée dans la déficience intellectuelle liée à l’X 105 (XLID105). Ce pipeline expérimental peut être utilisé pour clarifier les mécanismes sous-jacents aux troubles neurodéveloppementaux induits par des variantes des gènes DUB.
Introduction
Les troubles neurodéveloppementaux (TND) résultent de diverses étiologies avec des déterminants environnementaux ou génétiques qui entraînent le développement atypique du système nerveux1. Les tests génétiques de séquençage de nouvelle génération ont permis d’établir un lien entre un nombre croissant de variantes des gènes liés au système ubiquitine et les NDD génétiques2. Le système ubiquitine catalyse la ligature de l’ubiquitine, un petit modificateur protéique, principalement aux résidus de lysine dans les substrats protéiques pour entraîner des changements dans le comportement cellulaire, y compris la localisation, la stabilité, les interactions protéine-protéine ou l’activité3. L’ubiquitylation est médiée par les enzymes E1 activatrices, E2 conjuguées et E3 ligases4 et est réversible par l’activité des enzymes désubiquitylantes (DUBs) qui catalysent le clivage et l’élimination de l’ubiquitine des substrats protéiques5. L’ubiquitine peut être ligaturée au substrat sous forme de monomère (monoubiquitylation) ou de chaînes polymères (polyubiquitylation) qui se forment sur l’un des sept résidus de lysine (K6, K11, K27, K29, K33, K48, K63) ou le résidu M1 de l’ubiquitine. Ces différentes topologies de chaîne d’ubiquitine et leurs combinaisons créent un code cellulaire clé pour la transductiondu signal 6.
Les DUB tels que USP27X, USP7, USP9X, USP48, STAMBP, OTUD4, OTUD6B et OTUD5 ont été associés aux NDD2, 7, 8, 9, 10, 11. Pour la plupart des NDD, les mécanismes moléculaires à l’origine de la pathogenèse restent expérimentalement indéfinis. Certains des DUB à l’origine de troubles récemment décrits sont mal compris et ne disposent pas de données cellulaires connues pouvant être utilisées pour évaluer l’impact de la variation génétique sur la fonction des protéines. In vitro, les tests de clivage de la chaîne d’ubiquitine surmontent cette limitation en tant que lectures de l’activité DUB indépendantes du substrat qui peuvent mesurer l’impact des variantes génétiques sur l’activité enzymatique12.
Les tests de clivage in vitro de l’ubiquitine sont utilisés depuis les années 1980. Ces analyses utilisant des substrats radiomarqués ont permis de découvrir les premiers DUBs, y compris l’isopeptidase, identifiée pour sa capacité à désubiquityler l’histone H2A13, et l’ubiquitine carboxyl-terminale hydrolase (UCH), identifiée par sa capacité à hydrolyser l’ubiquitine à partir d’une variété de conjugués chimiques 14,15,16. De plus, des protéines ou des peptides polyubiquitylés radiomarqués ont été utilisés pour identifier l’isopeptidase T et plusieurs UCH et protéases spécifiques à l’ubiquitine (UBP) des érythrocytes et des muscles squelettiques, respectivement 17,18,19,20. Des chaînes d’ubiquitine d’une longueur définie et d’un type de liaison (tétra-ubiquitine liée à K48) ont d’abord été utilisées pour mesurer l’activité DUB de l’isopeptidase T21. Depuis lors, ce test est devenu l’étalon-or pour mesurer l’activité de DUB dans les analyses mutationnelles22,23. Le raffinement de ce test permet actuellement de visualiser le clivage de l’ubiquitine par électrophorèse et des colorants sur gel conventionnels tels que le bleu de Coomassie, l’orange SYPRO, le rubis et la coloration argentée ou la détection fluorescente ou basée sur l’immunobuvardage12,24. Les aspects moléculaires de l’activité de la DUB, tels que la longueur minimale de la chaîne et la spécificité de liaison 25,26,27,28, peuvent être clarifiés en utilisant des chaînes d’ubiquitine de différentes longueurs (par exemple, di-, tri-, tétra-ubiquitine) et de liaisons (K6, K11, K27, K29, K33, K48, K63, linéaire) dans les essais fonctionnels. Les variantes associées à la NDD peuvent entraîner des défauts d’activité DUB spécifiques au type de liaison de la chaîne d’ubiquitine11.
Un test de clivage de la di-ubiquitine utilisant des protéines DUB recombinantes purifiées peut mesurer directement l’impact des variants de la NDD sur l’activité de la DUB. USP27X, qui est muté dans le trouble de déficience intellectuelle lié à l’X NDD 105 (XLID105)7,28, modélise le processus présenté ici. Cette approche permet de déterminer comment l’activité des DUB est perturbée par des variantes génétiques dans les NDD associés aux DUB existants et inconnus.
Access restricted. Please log in or start a trial to view this content.
Protocole
Le protocole suivant peut être adapté aux protéines recombinantes en utilisant divers marqueurs d’affinité exprimés dans différentes souches de cellules compétentes. Selon la protéine exprimée, les conditions de culture et d’expression pendant la nuit peuvent nécessiter l’optimisation de la DO600 au niveau de l’induction de l’expression, du temps d’expression, de la température d’expression et de la concentration IPTG. La figure 1 illustre une vue d’ensemble du protocole. Les détails des réactifs et de l’équipement utilisés dans cette étude sont énumérés dans la table des matériaux.
1. Transformation de cellules E. coli Rosetta 2 compétentes avec le plasmide d’expression recombinant GST-USP27X
REMARQUE : Pour maintenir la stérilité de la culture bactérienne, effectuez des étapes où les récipients de milieu sont ouverts sous une flamme de bec Bunsen. Pour permettre un transfert optimal de l’oxygène, effectuez l’agitation de la culture bactérienne dans un agitateur de paillasse à température contrôlée avec une orbite de 19 mm à 50 mm et une vitesse de 200 tr/min29.
- Décongeler 20 μL de cellules E. coli Rosetta 2 chimiquement compétentes sur de la glace immédiatement avant utilisation. Ajoutez 1 à 10 ng du plasmide d’expression pGEX6P1-USP27X (codant pour la glutathion-S-transférase N-terminale (GST)30 marquée USP27X et contenant une résistance à l’ampicilline) et incubez 5 min sur de la glace.
- Choc thermique pendant 30 s à 42 °C dans un bain sec. Incuber sur glace pendant 2 min.
- Ajouter 80 μL de fluide SOC à température ambiante (RT). Incuber pendant 60 min à 37 °C avec une rotation de 200 tr/min dans un agitateur de paillasse à température contrôlée de 19 mm.
- Plaque de 50 μL de culture sur une plaque de gélose LB complétée par 25 μg/L de chloramphénicol et 50 μg/L d’ampicilline. Incuber pendant 20 h à 37 °C, couvercle vers le bas, dans un incubateur à température contrôlée.
2. Expression bactérienne nocturne de la protéine recombinante à partir du plasmide d’expression
REMARQUES : Pour maintenir la stérilité de la culture bactérienne, effectuez des étapes où les récipients de milieu sont ouverts sous une flamme de bec Bunsen. Pour permettre un transfert optimal de l’oxygène, effectuez l’agitation de la culture bactérienne dans un agitateur de paillasse à température contrôlée avec une orbite de 19 mm à 50 mm et une vitesse de 200 tr/min29. Mesurez la culture OD600 à l’aide d’un spectrophotomètre.
- Préparez 1 L de milieu Terrific Broth (TB) en ajoutant 47,6 g de poudre de TB à 1 L d’eau ultrapure avec 0,4 % de glycérol dans une fiole de culture à chicanes de 2 L. Autoclave moyen pendant 30 min et refroidir à RT.
- Prélever une seule colonie de bactéries transformées et l’ajouter à 10 ml de milieu LB stérile complété par 25 μg/L de chloramphénicol et 50 μg/L d’ampicilline. Incuber pendant 20 h à 37 °C avec une rotation de 200 tr/min dans un agitateur de paillasse à température contrôlée de 19 mm.
- Ajouter 10 mL de culture de nuit à 1 L de milieu TB complété par 25 μg/L de chloramphénicol et 50 μg/L d’ampicilline (rapport de 1:100 entre la culture initiale et la culture d’expression). Incuber à 37 °C avec une rotation de 200 tr/min dans un agitateur de paillasse à température contrôlée de 19 mm jusqu’à ce que la quantité de culture OD600 soit comprise entre 0,5 et 0,6.
- Ajouter 50 μM d’IPTG à la culture pour induire l’expression, refroidir à 16 °C et incuber pendant 20 h à 16 °C avec une rotation de 200 tr/min.
- Cellules en granulés issues de la culture d’expression par centrifugation pendant 20 min à ≥3 000 x g et 4 °C. Conservez le granulé à -80 °C pendant au moins 1 h.
REMARQUE : À ce stade, l’expérience peut être interrompue et recommencée plus tard (de préférence la même semaine). Le granulé peut être stocké à long terme à -80 °C.
3. Purification des protéines par colonne d’affinité gravité-flux
REMARQUE : Les tampons de résine, de liaison, de lavage, d’élution et de stockage appropriés pour chaque purification dépendront de la protéine recombinante purifiée. Prélevez des échantillons de la pastille cellulaire, du surnageant, de l’écoulement, des fractions de lavage et des fractions d’élution dans le tampon SDS-PAGE. Effectuer la coloration SDS-PAGE et Coomassie des échantillons afin d’évaluer le succès de la purification. Effectuer la purification à 4 °C et manipuler les fractions sur glace. Le clivage des marqueurs protéiques peut être effectué sur ou hors de la colonne à l’aide de la protéase appropriée pour cibler le site de clivage spécifique à la protéase pertinent.
- Fixez la colonne d’écoulement par gravité vide dans le support de l’autoclave et remplissez-la de résine de glutathion et d’agarose. Utilisez 2 à 3 ml de résine pour purifier une pastille cellulaire recueillie à partir de 1 L de culture d’expression.
- Lavez la colonne avec un volume de lit de résine à 20 % d’éthanol. Laver la colonne 3 fois avec un volume de lit de résine de tampon MS500 (20 mM de Tris pH 7,5, 500 mM de NaCl, 0,5 mM de TCEP). Arrêtez la colonne pour que la résine reste recouverte d’un tampon pour éviter le dessèchement lors de la préparation de la pastille de cellule.
- Décongeler la pastille cellulaire à 4 °C. Ajouter 30 mL de tampon MS500 (20 mM de Tris pH 7,5, 500 mM de NaCl, 0,5 mM de TCEP, 60 mg de lysozyme et un comprimé d’inhibiteur de protéase) à la pastille décongelée. Laisser reposer pendant 30 min à 4 °C avec une rotation douce d’un bout à l’autre.
- Sonicate lyse les cellules dans un tube à centrifuger de 50 mL ou un bécher métallique sur de la glace jusqu’à ce que le lysat s’écoule librement lorsqu’il est distribué à partir d’une pointe de pipette. Centrifuger pendant 30 min à 4 °C et ≥20 000 x g pour éliminer le surnageant.
REMARQUE : Déterminez les paramètres du sonicateur de manière empirique. Réglez le sonicateur de telle sorte qu’un temps total de 120 s soit suffisant pour réduire le lysat visqueux en un liquide fluide et transparent. - Décantez le surnageant dans un bécher et chargez le lysat dégagé sur la colonne. Faites passer le lysat à travers la colonne par écoulement par gravité tout en recueillant le flux. Chargez la colonne avec le flux collecté et faites-le passer à travers la colonne.
- Laver la colonne avec au moins deux volumes de tampon de lavage MS500 (20 mM de Tris pH 7,5, 500 mM de NaCl, 0,5 mM de TCEP) 5 fois. Recueillir le flux de lavage en fractions de 5 ml. Ajoutez 1 μL de chaque fraction à 100 μL de réactif Bradford pour vérifier visuellement la présence de protéines. Lavez jusqu’à ce qu’il n’y ait plus de protéines non liées lors du dernier lavage, en ajoutant des étapes de lavage supplémentaires si nécessaire.
- Récupérez la protéine en faisant passer le tampon d’élution MS500 (tampon MS500 complété par 10 mM de glutathion et 10 mM de NaOH) à travers la colonne, en collectant 5 mL de fractions d’élution. Vérifiez la présence de protéines en ajoutant 1 μL d’éluat à 100 μL de réactif Bradford. Arrêtez de collecter des fractions lorsque le réactif de Bradford n’indique plus la présence de protéines.
- Effectuer l’échange de tampon par précipitation et centrifugation. Précipiter la protéine en ajoutant 2 volumes de 4 M de sulfate d’ammonium à l’éluat, en l’inversant doucement jusqu’à ce qu’il soit trouble, puis en centrifugeant pendant 30 minutes à 4 °C et ≥20 000 x g , puis à nouveau pendant 5 minutes supplémentaires. Retirer le surnageant après chaque centrifugation sans perturber la pastille de protéine.
- Redissolvez et stockez les protéines dans MS500 complété par 25% de glycérol. Conservez les granulés de protéines ou les protéines dans un tampon de stockage à -80 °C.
4. Essai in vitro de clivage de la chaîne d’ubiquitine
REMARQUE : Sélectionner la longueur de la chaîne d’ubiquitine et les types de liaison en fonction de la spécificité DUB décrite dans les rapports précédents31 ou déterminée empiriquement. Si nécessaire, ce protocole pourrait être utilisé pour tester l’activité de la DUB de type sauvage d’intérêt sur un panel de chaînes d’ubiquitine disponibles dans le commerce de longueur et de type de liaison définis. Une quantité de chaîne de di-ubiquitine de 375-750 ng et une concentration de DUB de 1-2 μM peuvent être utilisées comme points de départ pour le test27.
- Préparez 10 tampons d’activation DUB (500 mM de Tris-HCl pH 7,5, 500 mM de NaCl et 100 mM de TCEP).
- Pour chaque point temporel de chaque DUB, préparez un total de 10 μL de 2 μM de GST-USP27X purifié dans 1 tampon d’activation DUB (mélange DUB). Préparez des mixages maîtres et divisez-les en points temporels.
- Incuber le mélange DUB pendant 10 min à RT.
- Ajoutez 7 μL de tampon de chargement SDS-PAGE au temps 0 avant d’ajouter des chaînes d’ubiquitine pour empêcher la réaction de désubiquitylation de démarrer.
- À chaque point temporel de chaque DUB, ajoutez 375 ng de chaînes de di-ubiquitine K-63 diluées dans 10 μL 1x tampon d’activation DUB. Le volume total est de 20 μL par réaction.
- Incuber les tubes à 30 °C, en arrêtant chaque point temporel avec 7 μL de tampon de chargement SDS-PAGE.
- Effectuer SDS-PAGE avec un gel à gradient de 4 % à 12 % et l’immunoblot 7,32 pour l’ubiquitine et l’USP27X afin d’analyser le changement de la présence de mono-ubiquitine à des points temporels sélectionnés.
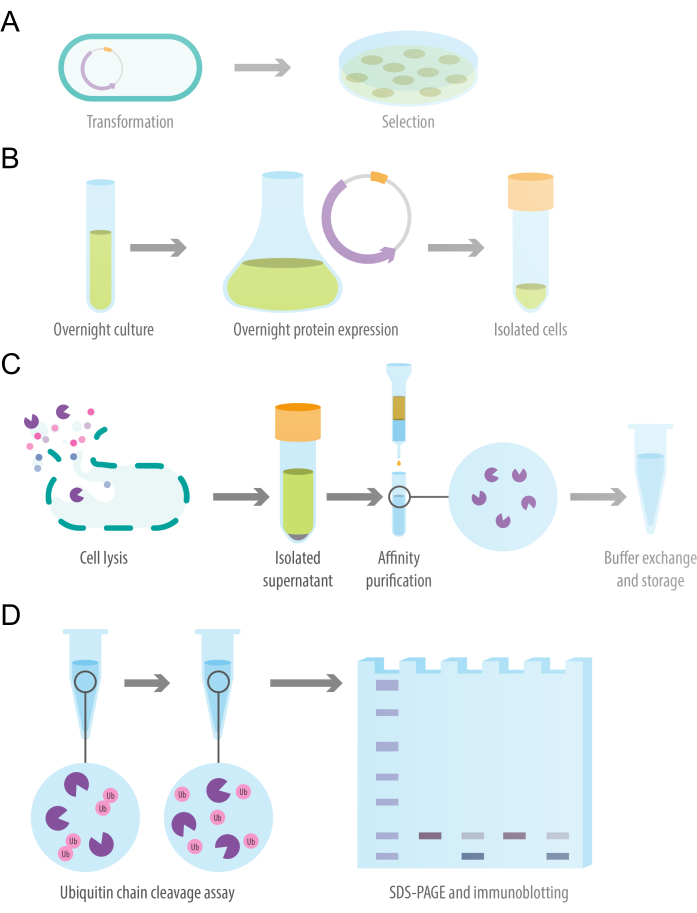
Figure 1 : Schéma du plan d’étude. (A) Transformation de cellules E. coli compétentes avec un plasmide d’expression de protéines recombinantes. (B) Expression bactérienne nocturne de la protéine deubiquitylase recombinante. (C) Purification protéique de la deubiquitylase recombinante à l’aide d’une colonne d’affinité gravité-flux. (D) Essai in vitro de clivage de la chaîne d’ubiquitine pour évaluer l’activité de désubiquitylation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
Pour déterminer l’impact des variants associés à la XLID105 sur l’activité catalytique de l’USP27X, les protéines USP27X de type sauvage marquées GST et les protéines USP27X des variants F313V, Y381H et S404N associées à la XLID105 ont été purifiées à partir de bactéries. Ces variantes sont situées dans le domaine catalytique USP de l’USP27X (Figure 2A). Étant donné qu’USP27X avait déjà été signalé pour cliver les chaînes d?...
Access restricted. Please log in or start a trial to view this content.
Discussion
Cet article présente un protocole pour l’expression et la purification de DUBs recombinants USP27X et un test de clivage de la chaîne d’ubiquitine in vitro pour comparer l’activité de désubiquitylation de l’USP27X de type sauvage et des protéines variantes associées à la NDD. Ce test a déterminé que les variants associés à XLID105 perturbent l’activité catalytique de l’USP27X7. Cette compréhension mécaniste nous a aidés à défi...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été financé par les fonds de démarrage de Sanford Research à FB et la subvention du NIH R01CA233700 à MJS. L’œuvre a été réalisée par Felipe G. Serrano (www.illustrative-science.com). Nous remercions le Dr Greg Findlay (Université de Dundee) pour le plasmide GST-USP27X.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Amersham Protran 0.45 NC 200 mm × 200 mm 25/PK | Cytiva | 10600041 | |
| Ammonium sulfate | Fisher Scientific | AC205872500 | |
| Ampicillin | Fisher Scientific | BP1760 25 | |
| Anti- Ubiquitin (Mouse monoclonal) | Biolegend | Cat# 646302, RRID:AB_1659269 | (WB: 1:1000) |
| Anti-GST (Sheep polyclonal) | MRC-PPU Reagents and Services | Cat# S902A Third bleed | (WB: 1:1000) https://mrcppureagents.dundee.ac.uk/ |
| Baffled Culture Flasks 2 L | Fisher Scientific | 10-042-5N | |
| Bradford Reagent | Millipore Sigma | B6916-500ML | |
| Chloramphenicol | Gold Biotechnology | C-105-25 | |
| Complete, Protease Inhibitor tablets | Millipore Sigma | 5056489001 | |
| Econo-Column 1.5 × 5 cm | Bio-Rad | 7371507 | |
| Eppendorf ThermoMixer F1.5 | Eppendorf | 5384000020 | |
| Excel | Microsoft | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Glycerol | Genesee Scientific | 18-205 | |
| Illustrator | Adobe | https://www.adobe.com/products/illustrator.html | |
| Image Studio | LI-COR Biosciences | https://www.licor.com/bio/image-studio/ | |
| Inkscape | Inkscape | https://inkscape.org/ | |
| Invitrogen 4-12% NuPAGE 1mm 12 well gel | Thermo Fisher Scientific | NP0322BOX | |
| IPTG (Isopropyl-b-D-Thiogalactopyranoside) | Genesee Scientific | 20-109 | |
| IRDye 800CW Donkey anti-Goat IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32214 | (WB: 1:10000) |
| IRDye 800CW Donkey anti-Mouse IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32212 | (WB: 1:10000) |
| Isotemp Digital Dry Bath | Fisher Scientific | 88860022 | |
| K63 Di-Ubiquitin | South Bay Bio LLC | SBB-UP0072 | |
| LB Agar | Genesee Scientific | 11-119 | |
| LB Broth | Genesee Scientific | 11-118 | |
| Lysozyme | Gold Biotechnology | L-040-100 | |
| MaxQ 4000 Benchtop Orbital Shaker | Thermo Fisher Scientific | SHKE4000-7 | |
| MES-SDS Running Buffer | Boston Bioproducts Inc | BP-177 | |
| Mini Tube Rotator | Fisher Scientific | 88-861-051 | |
| NuPage LDS Sample buffer 4x | Thermo Fisher Scientific | NP0007 | |
| Odyssey Fc Imager | LI-COR Biosciences | 43214 | |
| PageRuler Plus Ladder | Thermo Fisher Scientific | 26620 | |
| pGEX6P1 human USP27X | MRC-PPU Reagents and Services | DU21193 | https://mrcppureagents.dundee.ac.uk/ |
| pGEX6P1 human USP27X F313V | Addgene | 225715 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X S404N | Addgene | 225717 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X Y381H | Addgene | 225716 | Koch et at 2024 (PMID: 38182161) |
| Pierce Glutathione Agarose | Thermo Fisher Scientific | 16100 | |
| PMSF (Phenylmethylsulfonyl fluoride) | Gold Biotechnology | P-470-10 | |
| Polysorbate 20 (Tween 20) | Fisher Scientific | AC233360010 | |
| Rosetta 2 Competent Cells | Millipore Sigma | 71402-M | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6060 | |
| SmartSpec 3000 | Bio-Rad | 170-2501 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Sodium chloride | Genesee Scientific | 18-216 | |
| Sonifier 250 | Branson | 100-132-135 | |
| Sorvall RC 6 Plus Centrifuge | Thermo Fisher Scientific | 46910 | |
| TCEP (Tris-(carboxyethyl) phosphine hydrochloride) | Gold Biotechnology | TCEP10 | |
| Terrific Broth Powder | Genesee Scientific | 18-225 | |
| Tris Base | Genesee Scientific | 18-146 | |
| XCell SureLock Mini-Cell and XCell II Blot Module | Thermo Fisher Scientific | EI0002 |
Références
- Morris-Rosendahl, D. J., Crocq, M. -A. Neurodevelopmental disorders: the history and future of a diagnostic concept. Dialogues Clin Neurosci. 22 (1), 65-72 (2020).
- Ebstein, F., Küry, S., Papendorf, J. J., Krüger, E. Neurodevelopmental disorders (NDD) caused by genomic alterations of the ubiquitin-proteasome system (UPS): the possible contribution of immune dysregulation to disease pathogenesis. Front Mol Neurosci. 14, 733012(2021).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annu Rev Biochem. 67 (10), 425-479 (1998).
- Pickart, C. M. Mechanisms underlying ubiquitination. Annu Rev Biochem. 70 (1), 503-533 (2001).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Yau, R., Rape, M. The increasing complexity of the ubiquitin code. Nat Cell Biol. 18 (6), 579-586 (2016).
- Koch, I., et al. USP27X variants underlying X-linked intellectual disability disrupt protein function via distinct mechanisms. Life Sci Alliance. 7 (3), e202302258(2024).
- Hao, Y. -H., et al. USP7 acts as a molecular rheostat to promote WASH-dependent endosomal protein recycling and is mutated in a human neurodevelopmental disorder. Mol Cell. 59 (6), 956-969 (2015).
- Fountain, M. D., et al. Pathogenic variants in USP7 cause a neurodevelopmental disorder with speech delays, altered behavior, and neurologic anomalies. Genet Med. 21 (8), 1797-1807 (2019).
- Santiago-Sim, T., et al. Biallelic variants in OTUD6B cause an intellectual disability syndrome associated with seizures and dysmorphic features. Am J Hum Genet. 100 (4), 676-688 (2017).
- Beck, D. B., et al. Linkage-specific deubiquitylation by OTUD5 defines an embryonic pathway intolerant to genomic variation. Sci Adv. 7 (4), eabe2116(2021).
- Cho, J., Park, J., Kim, E. E., Song, E. J. Assay systems for profiling deubiquitinating activity. Int J Mol Sci. 21 (16), 1-16 (2020).
- Matsui, S., Sandberg, A. A., Negoro, S., Seon, B. K., Goldstein, G. Isopeptidase: A novel eukaryotic enzyme that cleaves isopeptide bonds. Proc Natl Acad Sci USA. 79 (5), 1535-1539 (1982).
- Rose, I. A., Warms, J. V. B. An enzyme with ubiquitin carboxy-terminal esterase activity from reticulocytes. Biochemistry. 22 (18), 4234-4237 (1983).
- Wilkinson, K. D., Cox, M. J., Mayer, A. N., Frey, T. Synthesis and characterization of ubiquitin ethyl ester, a new substrate for ubiquitin carboxyl-terminal hydrolase. Biochemistry. 25 (21), 6644-6649 (1986).
- Pickart, C. M., Rose, I. A. Ubiquitin carboxyl-terminal hydrolase acts on ubiquitin carboxyl-terminal amides. J Biol Chem. 260 (13), 7903-7910 (1985).
- Hadari, T., Warms, J. V. B., Rose, I. A., Hershko, A. A ubiquitin C-terminal isopeptidase that acts on polyubiquitin chains: Role in protein degradation. J Biol Chem. 267 (2), 719-727 (1992).
- Woo, S. K., et al. Multiple ubiquitin C-terminal hydrolases from chick skeletal muscle. J Biol Chem. 270 (32), 18766-18773 (1995).
- Woo, S. K., et al. Purification and characterization of a new ubiquitin C-terminal hydrolase (UCH-1) with isopeptidase activity from chick skeletal muscle. J Biochem. 121 (4), 684-689 (1997).
- Baek, S. H., et al. Molecular cloning of a novel ubiquitin-specific protease, UBP41, with isopeptidase activity in chick skeletal muscle. J Biol Chem. 272 (41), 25560-25565 (1997).
- Wilkinson, K. D., et al. Metabolism of the polyubiquitin degradation signal: Structure, mechanism, and role of isopeptidase T. Biochemistry. 34 (44), 14535-14546 (1995).
- Hu, M., et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell. 111 (7), 1041-1054 (2002).
- Hu, M., et al. Structure and mechanisms of the proteasome-associated deubiquitinating enzyme USP14. EMBO J. 24 (21), 3747-3756 (2005).
- Gorka, M., Magnussen, H. M., Kulathu, Y. Chemical biology tools to study deubiquitinases and UBL proteases. Semin Cell Dev Biol. 132, 86-96 (2022).
- Faesen, A. C., et al. The differential modulation of USP activity by internal regulatory domains, interactors, and eight ubiquitin chain types. Chem Biol. 18 (12), 1550-1561 (2011).
- Mevissen, T. E. T., et al. OTU deubiquitinases reveal mechanisms of linkage specificity and enable ubiquitin chain restriction analysis. Cell. 154 (1), 169-184 (2013).
- Kwasna, D., et al. Discovery and characterization of ZUFSP/ZUP1, a distinct deubiquitinase class important for genome stability. Mol Cell. 70 (1), 150-164.e6 (2018).
- Hu, H., et al. X-exome sequencing of 405 unresolved families identifies seven novel intellectual disability genes. Mol Psychiatry. 21 (1), 133-148 (2016).
- Hartmann, I. Shaker Orbit - Revolving in Space Around the Samples? Eppendorf Lab Academy. Eppendorf Lab Academy. , at https://www.eppendorf.com/us-en/lab-academy/lab-solutions/others/shaker-orbit-revolving-in-space-around-the-samples (2020).
- Smith, D. B., Johnson, K. S. Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene. 67 (1), 31-40 (1988).
- Ritorto, M. S., et al. Screening of DUB activity and specificity by MALDI-TOF mass spectrometry. Nat Commun. 5, 4763(2014).
- Bustos, F., Bustos, F. Immunoblotting using precast gels. , protocols.io (2024).
- Wingfield, P. T. Overview of the purification of recombinant proteins. Curr Protoc Protein Sci. 80, 6.1.1-6.1.35 (2015).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon