Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Medición de la actividad enzimática de las enzimas desubiquitilantes asociadas a trastornos del neurodesarrollo mediante un ensayo de escisión de la cadena de ubiquitina in vitro
En este artículo
Resumen
Este protocolo explica cómo medir el efecto de la variación genética asociada con los trastornos del neurodesarrollo sobre la actividad de la enzima desubiquitilante mediante la combinación de la purificación de proteínas recombinantes con ensayos de escisión de la cadena de ubiquitina.
Resumen
Los trastornos del neurodesarrollo (NDD, por sus siglas en inglés) se asocian con alteraciones en la función del sistema nervioso, pero a menudo siguen siendo poco conocidos a nivel molecular. Los trastornos discretos causados por genes individuales proporcionan modelos para investigar los mecanismos que impulsan el neurodesarrollo atípico. Las variantes de los genes que codifican proteínas de la familia de la enzima desubiquitilante (DUB) se asocian con varios NDD, pero es necesario determinar los mecanismos patogénicos de los trastornos impulsados por estas variantes genéticas. El impacto de las variantes genéticas en la actividad de DUB puede determinarse experimentalmente mediante un ensayo de escisión de ubiquitina in vitro independiente del sustrato. Este ensayo no requiere conocimiento de los sustratos aguas abajo para medir directamente la actividad catalítica. Aquí, el protocolo para determinar el impacto de las variantes genéticas en la actividad enzimática se modela utilizando la proteasa específica de ubiquitina DUB 27, ligada al cromosoma X (USP27X), que está mutada en la discapacidad intelectual ligada al cromosoma X 105 (XLID105). Esta línea experimental se puede utilizar para aclarar los mecanismos subyacentes a los trastornos del neurodesarrollo impulsados por variantes en los genes DUB.
Introducción
Los trastornos del neurodesarrollo (TND) surgen de diversas etiologías con determinantes ambientales o genéticos que impulsan el desarrollo atípico del sistema nervioso1. Las pruebas genéticas de secuenciación de próxima generación han relacionado un número cada vez mayor de variantes en genes relacionados con el sistema de ubiquitina con NDD genéticos2. El sistema de ubiquitina cataliza la ligadura del pequeño modificador de proteínas ubiquitina a residuos de lisina principalmente en sustratos de proteínas para impulsar cambios en el comportamiento celular, incluida la localización, la estabilidad, las interacciones proteína-proteína o la actividad3. La ubiquitilación está mediada por las enzimas E1 activadoras, E2 conjugadoras y E3 ligasas4 y es reversible por la actividad de las enzimas desubiquitilantes (DUB) que catalizan la escisión y la eliminación de la ubiquitina de los sustratos proteicos5. La ubiquitina se puede ligar al sustrato como un monómero (monoubiquitilación) o cadenas poliméricas (poliubiquitilación) que se forman en cualquiera de los siete residuos de lisina (K6, K11, K27, K29, K33, K48, K63) o el residuo M1 de la ubiquitina. Estas diferentes topologías de cadena de ubiquitina y sus combinaciones crean un código celular que es clave para la transducción de señales6.
Los DUB como USP27X, USP7, USP9X, USP48, STAMBP, OTUD4, OTUD6B y OTUD5 se han asociado con los NDD 2,7,8,9,10,11. Para la mayoría de los NDD, los mecanismos moleculares que impulsan la patogénesis siguen sin definirse experimentalmente. Algunos de los DUB que impulsan los trastornos recientemente descritos son poco conocidos y carecen de lecturas celulares conocidas que puedan usarse para evaluar el impacto de la variación genética en la función de las proteínas. In vitro, los ensayos de escisión de la cadena de ubiquitina superan esta limitación como lecturas de actividad DUB independientes del sustrato que pueden medir el impacto de las variantes genéticas en la actividad enzimática12.
Los ensayos de escisión de ubiquitina in vitro se han utilizado desde la década de 1980. Estos ensayos con sustratos radiomarcados permitieron el descubrimiento de los primeros DUBs, incluyendo la isopeptidasa, identificada por su capacidad de deubiquitilado la histona H2A13, y la ubiquitina carboxilo hidrolasa terminal (UCH), identificada por su capacidad de hidrolizar ubiquitina a partir de una variedad de conjugados químicos 14,15,16. Además, se utilizaron proteínas o péptidos poliubiquitilados de longitud completa radiomarcados para identificar la isopeptidasa T y varias UCHs y proteasas específicas de ubiquitina (UBPs) de eritrocitos y músculo esquelético, respectivamente 17,18,19,20. Se utilizaron por primera vez cadenas de ubiquitina de una longitud y un tipo de enlace definidos (tetra-ubiquitina ligada a K48) para medir la actividad DUB de la isopeptidasa T21. Desde entonces, este ensayo se ha convertido en el estándar de oro para medir la actividad de DUB en análisis mutacionales22,23. El refinamiento de este ensayo permite actualmente la visualización de la escisión de ubiquitina a través de electroforesis y tinciones de gel convencionales como el azul de Coomassie, el naranja SYPRO, el rubí y la tinción de plata o la detección fluorescente o basada en inmunotransferencia12,24. Los aspectos moleculares de la actividad de DUB, como la longitud mínima de la cadena y la especificidad del enlace 25,26,27,28, pueden aclararse mediante el uso de cadenas de ubiquitina de diferentes longitudes (por ejemplo, di-, tri-, tetra-ubiquitina) y enlaces (K6, K11, K27, K29, K33, K48, K63, lineales) en ensayos funcionales. Las variantes asociadas a NDD pueden conducir a defectos de actividad DUB que son específicos del tipo de enlace de la cadena de ubiquitina11.
Un ensayo de escisión de diubiquitina utilizando proteínas DUB recombinantes purificadas puede medir directamente el impacto de las variantes de NDD en la actividad de DUB. USP27X, que está mutado en el trastorno de discapacidad intelectual ligado al cromosoma X NDD 105 (XLID105)7,28 modela el proceso que aquí se presenta. Este enfoque permite determinar cómo la actividad de DUB se ve interrumpida por variantes genéticas en NDD asociados a DUB existentes y desconocidos.
Access restricted. Please log in or start a trial to view this content.
Protocolo
El siguiente protocolo se puede adaptar para proteínas recombinantes utilizando varias etiquetas de afinidad expresadas en diferentes cepas de células competentes. Dependiendo de la proteína que se exprese, las condiciones de cultivo y expresión nocturna pueden requerir la optimización de la OD600 en la inducción de la expresión, el tiempo de expresión, la temperatura de expresión y la concentración de IPTG. En la Figura 1 se ilustra una descripción general del protocolo. Los detalles de los reactivos y el equipo utilizado en este estudio se enumeran en la Tabla de Materiales.
1. Transformación de células competentes de Rosetta 2 E. coli con el plásmido de expresión recombinante GST-USP27X
NOTA: Para mantener la esterilidad del cultivo bacteriano, realice pasos en los que los recipientes de medios estén abiertos bajo una llama de mechero Bunsen. Para permitir una transferencia óptima de oxígeno, realice la agitación del cultivo bacteriano en un agitador de temperatura controlada de sobremesa con una órbita de 19 mm a 50 mm y una velocidad de 200 rpm29.
- Descongele 20 μL de células de Rosetta 2 E. coli químicamente competentes en hielo inmediatamente antes de usar. Añadir 1-10 ng del plásmido de expresión pGEX6P1-USP27X (que codifica para la glutatión-S-transferasa N-terminal (GST)30 -marcado con USP27X y que contiene resistencia a la ampicilina) e incubar 5 min en hielo.
- Choque térmico durante 30 s a 42 °C en baño seco. Incubar en hielo durante 2 min.
- Agregue 80 μL de medio SOC a temperatura ambiente (RT). Incubar durante 60 min a 37 °C con rotación de 200 rpm en un agitador de sobremesa de 19 mm con control de temperatura.
- Placa de 50 μL de cultivo en una placa de agar LB suplementada con 25 μg/L de cloranfenicol y 50 μg/L de ampicilina. Incubar durante 20 h a 37 °C con la tapa hacia abajo en una incubadora con temperatura controlada.
2. Expresión bacteriana nocturna de proteína recombinante a partir de plásmido de expresión
NOTAS: Para mantener la esterilidad del cultivo bacteriano, realice pasos donde los recipientes de medios estén abiertos bajo una llama de quemador Bunsen. Para permitir una transferencia óptima de oxígeno, realice la agitación del cultivo bacteriano en un agitador de temperatura controlada de sobremesa con una órbita de 19 mm a 50 mm y una velocidad de 200 rpm29. Mida el diámetro exterior del cultivo600 con un espectrofotómetro.
- Prepare 1 L de medio Terrific Broth (TB) añadiendo 47,6 g de TB Powder a 1 L de agua ultrapura con 0,4% de glicerol en un matraz de cultivo con deflector de 2 L. Medio de autoclave durante 30 min y enfriamiento a RT.
- Elija una sola colonia de bacterias transformadas y agregue 10 mL de medio LB estéril suplementado con 25 μg/L de cloranfenicol y 50 μg/L de ampicilina. Incubar durante 20 h a 37 °C con rotación de 200 rpm en un agitador de sobremesa de 19 mm con control de temperatura.
- Añadir 10 mL de cultivo durante la noche a 1 L de medio TB suplementado con 25 μg/L de cloranfenicol y 50 μg/L de ampicilina (proporción 1:100 entre el cultivo inicial y el cultivo de expresión). Incubar a 37 °C con una rotación de 200 rpm en un agitador de sobremesa de 19 mm de órbita con control de temperatura hasta que el diámetro exterior decultivo 600 esté entre 0,5 y 0,6.
- Añadir 50 μM de IPTG al cultivo para inducir la expresión, enfriar a 16 °C e incubar durante 20 h a 16 °C con una rotación de 200 rpm.
- Células granuladas de cultivo de expresión por centrifugación durante 20 min a ≥3.000 x g y 4 °C. Almacene el pellet a -80 °C durante al menos 1 h.
NOTA: En este punto, el experimento se puede pausar y reiniciar más tarde (preferiblemente la misma semana). El pellet se puede almacenar a largo plazo a -80 °C.
3. Purificación de proteínas por columna de afinidad de flujo por gravedad
NOTA: La resina, el aglutinante, el lavado, la elución y los tampones de almacenamiento apropiados para cada purificación dependerán de la proteína recombinante que se purifique. Recoja muestras de las fracciones de pellets de celda, sobrenadante, flujo, lavado y elución en tampón SDS-PAGE. Realice la tinción SDS-PAGE y Coomassie de las muestras para evaluar el éxito de la purificación. Realizar la purificación a 4 °C y manipular fracciones en hielo. La escisión de las etiquetas de proteínas se puede realizar dentro o fuera de la columna utilizando la proteasa adecuada para dirigirse al sitio de escisión específico de la proteasa relevante.
- Asegure la columna de flujo por gravedad vacía en el soporte de retorta y llénela con resina de glutatión agarosa. Utilice 2-3 mL de resina para purificar un pellet celular recogido de 1 L de cultivo de expresión.
- Lave la columna con un volumen de lecho de resina de etanol al 20%. Lave la columna 3 veces con un volumen de lecho de resina de tampón MS500 (20 mM de Tris pH 7,5, 500 mM de NaCl, 0,5 mM de TCEP). Detenga la columna para que la resina permanezca cubierta con tampón para evitar que se seque mientras prepara el pellet de celda.
- Descongele el pellet de celda a 4 °C. Añada 30 mL de tampón MS500 (20 mM de Tris pH 7,5, 500 mM de NaCl, 0,5 mM de TCEP, 60 mg de lisozima y un comprimido de inhibidor de la proteasa) al pellet descongelado. Lisar durante 30 min a 4 °C con una rotación suave de extremo a extremo.
- Sonicar células lisadas en un tubo de centrífuga de 50 ml o en un vaso de precipitados metálico sobre hielo hasta que el lisado fluya libremente cuando se dispense desde la punta de una pipeta. Centrifugar durante 30 min a 4 °C y ≥20.000 x g para eliminar el sobrenadante.
NOTA: Determine empíricamente los ajustes del sonicador. Configure el sonicador de manera que 120 s de tiempo total sean suficientes para reducir el lisado viscoso a un líquido transparente y de flujo libre. - Decantar el sobrenadante en un vaso de precipitados y cargar el lisado aclarado en la columna. Pase el lisado a través de la columna por flujo por gravedad mientras recoge el flujo a través. Cargue la columna con el flujo recopilado y pásela por la columna.
- Lave la columna con al menos dos volúmenes de lecho de resina de tampón de lavado MS500 (20 mM de Tris pH 7,5, 500 mM de NaCl, 0,5 mM de TCEP) 5 veces. Recoja el flujo de lavado en fracciones de 5 mL. Añada 1 μL de cada fracción a 100 μL de reactivo Bradford para comprobar visualmente la presencia de proteínas. Lavar hasta que la proteína no unida ya no esté presente en el último lavado, agregando pasos de lavado adicionales si es necesario.
- Recupere la proteína haciendo pasar el tampón de elución MS500 (tampón MS500 suplementado con 10 mM de glutatión y 10 mM de NaOH) a través de la columna, recogiendo fracciones de elución de 5 mL. Compruebe la presencia de proteínas añadiendo 1 μL de eluido a 100 μL de reactivo Bradford. Deje de recolectar fracciones cuando el reactivo de Bradford ya no indique que hay proteínas presentes.
- Realizar el intercambio de tampones por precipitación y centrifugación. Precipitar la proteína añadiendo 2 volúmenes de 4 M de sulfato de amonio al eluido, invirtiendo suavemente hasta que esté turbio, y luego centrifugando durante 30 min a 4 °C y ≥20.000 x g y de nuevo durante otros 5 min. Retire el sobrenadante después de cada centrifugación sin alterar la pastilla de proteína.
- Vuelva a disolver y almacenar proteínas en MS500 suplementado con 25% de glicerol. Almacene los gránulos de proteína o la proteína en un tampón de almacenamiento a -80 °C.
4. Ensayo de escisión de la cadena de ubiquitina in vitro
NOTA: Seleccione la longitud de la cadena de ubiquitina y los tipos de enlace en función de la especificidad de DUB descrita en informes anteriores31 o determinada empíricamente. Si fuera necesario, este protocolo podría utilizarse para probar la actividad del DUB de tipo salvaje de interés en un panel de cadenas de ubiquitina disponibles comercialmente de longitud y tipo de enlace definidos. Se puede utilizar una cantidad de cadena de diubiquitina de 375-750 ng y una concentración de DUB de 1-2 μM como puntos de partida para el ensayo27.
- Prepare 10 tampones de activación DUB (500 mM de Tris-HCl, pH 7,5, 500 mM de NaCl y 100 mM de TCEP).
- Para cada punto de tiempo para cada DUB, prepare 10 μL en total de 2 μM de GST-USP27X purificado en 1x tampón de activación DUB (mezcla DUB). Prepara mezclas maestras y divídelas en puntos de tiempo.
- Incuba la mezcla DUB durante 10 minutos en RT.
- Añada 7 μL de tampón de carga SDS-PAGE al tiempo 0 antes de añadir las cadenas de ubiquitina para evitar que se inicie la reacción de deubiquitilación.
- A cada punto de tiempo para cada DUB, agregue 375 ng de cadenas de di-ubiquitina K-63 diluidas en 10 μL 1x tampón de activación DUB. El volumen total es de 20 μL por reacción.
- Incubar los tubos a 30 °C, deteniéndose en cada punto de tiempo con 7 μL de tampón de carga SDS-PAGE.
- Realice SDS-PAGE con un gel de gradiente de 4%-12% e inmunoblot 7,32 para ubiquitina y USP27X para analizar el cambio en la presencia de mono-ubiquitina en puntos de tiempo seleccionados.
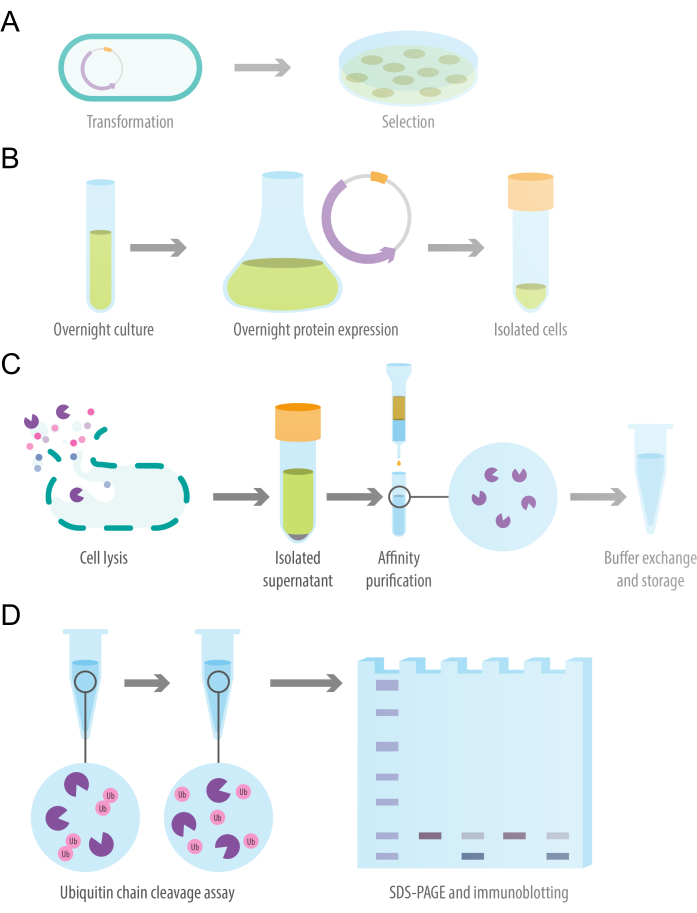
Figura 1: Esquema del diseño del estudio. (A) Transformación de células competentes de E. coli con plásmido de expresión de proteínas recombinantes. (B) Expresión bacteriana durante la noche de la proteína deubiquitilasa recombinante. (C) Purificación de proteínas de desubiquitilasa recombinante utilizando una columna de afinidad de flujo por gravedad. (D) Ensayo de escisión de la cadena de ubiquitina in vitro para evaluar la actividad desubiquitilante. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para determinar el impacto de las variantes asociadas a la XLID105 en la actividad catalítica de USP27X, se purificaron a partir de bacterias las proteínas USP27X de tipo salvaje marcadas con GST y las variantes asociadas a XLID105 F313V, Y381H y S404N USP27X. Estas variantes se encuentran dentro del dominio catalítico USP de USP27X (Figura 2A). Debido a que se informó previamente que USP27X escinde las cadenas de ubiquitinaK63 31...
Access restricted. Please log in or start a trial to view this content.
Discusión
Este artículo presenta un protocolo para la expresión y purificación de DUBs USP27X recombinantes y un ensayo in vitro de escisión de la cadena de ubiquitina para comparar la actividad desubiquitilante de USP27X de tipo salvaje y proteínas variantes asociadas a NDD. Este ensayo determinó que las variantes asociadas a XLID105 interrumpen la actividad catalítica de USP27X7. Este conocimiento mecanicista nos ayudó a definir XLID105 como un trastorno ...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Este trabajo fue financiado por los fondos de la startup de Sanford Research para FB y la subvención de los NIH R01CA233700 para MJS. La obra fue realizada por Felipe G. Serrano (www.illustrative-science.com). Agradecemos al Dr. Greg Findlay (Universidad de Dundee) por el plásmido GST-USP27X.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Amersham Protran 0.45 NC 200 mm × 200 mm 25/PK | Cytiva | 10600041 | |
| Ammonium sulfate | Fisher Scientific | AC205872500 | |
| Ampicillin | Fisher Scientific | BP1760 25 | |
| Anti- Ubiquitin (Mouse monoclonal) | Biolegend | Cat# 646302, RRID:AB_1659269 | (WB: 1:1000) |
| Anti-GST (Sheep polyclonal) | MRC-PPU Reagents and Services | Cat# S902A Third bleed | (WB: 1:1000) https://mrcppureagents.dundee.ac.uk/ |
| Baffled Culture Flasks 2 L | Fisher Scientific | 10-042-5N | |
| Bradford Reagent | Millipore Sigma | B6916-500ML | |
| Chloramphenicol | Gold Biotechnology | C-105-25 | |
| Complete, Protease Inhibitor tablets | Millipore Sigma | 5056489001 | |
| Econo-Column 1.5 × 5 cm | Bio-Rad | 7371507 | |
| Eppendorf ThermoMixer F1.5 | Eppendorf | 5384000020 | |
| Excel | Microsoft | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Glycerol | Genesee Scientific | 18-205 | |
| Illustrator | Adobe | https://www.adobe.com/products/illustrator.html | |
| Image Studio | LI-COR Biosciences | https://www.licor.com/bio/image-studio/ | |
| Inkscape | Inkscape | https://inkscape.org/ | |
| Invitrogen 4-12% NuPAGE 1mm 12 well gel | Thermo Fisher Scientific | NP0322BOX | |
| IPTG (Isopropyl-b-D-Thiogalactopyranoside) | Genesee Scientific | 20-109 | |
| IRDye 800CW Donkey anti-Goat IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32214 | (WB: 1:10000) |
| IRDye 800CW Donkey anti-Mouse IgG Secondary Antibody | LI-COR Biosciences | Cat# 926-32212 | (WB: 1:10000) |
| Isotemp Digital Dry Bath | Fisher Scientific | 88860022 | |
| K63 Di-Ubiquitin | South Bay Bio LLC | SBB-UP0072 | |
| LB Agar | Genesee Scientific | 11-119 | |
| LB Broth | Genesee Scientific | 11-118 | |
| Lysozyme | Gold Biotechnology | L-040-100 | |
| MaxQ 4000 Benchtop Orbital Shaker | Thermo Fisher Scientific | SHKE4000-7 | |
| MES-SDS Running Buffer | Boston Bioproducts Inc | BP-177 | |
| Mini Tube Rotator | Fisher Scientific | 88-861-051 | |
| NuPage LDS Sample buffer 4x | Thermo Fisher Scientific | NP0007 | |
| Odyssey Fc Imager | LI-COR Biosciences | 43214 | |
| PageRuler Plus Ladder | Thermo Fisher Scientific | 26620 | |
| pGEX6P1 human USP27X | MRC-PPU Reagents and Services | DU21193 | https://mrcppureagents.dundee.ac.uk/ |
| pGEX6P1 human USP27X F313V | Addgene | 225715 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X S404N | Addgene | 225717 | Koch et at 2024 (PMID: 38182161) |
| pGEX6P1 human USP27X Y381H | Addgene | 225716 | Koch et at 2024 (PMID: 38182161) |
| Pierce Glutathione Agarose | Thermo Fisher Scientific | 16100 | |
| PMSF (Phenylmethylsulfonyl fluoride) | Gold Biotechnology | P-470-10 | |
| Polysorbate 20 (Tween 20) | Fisher Scientific | AC233360010 | |
| Rosetta 2 Competent Cells | Millipore Sigma | 71402-M | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6060 | |
| SmartSpec 3000 | Bio-Rad | 170-2501 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Sodium chloride | Genesee Scientific | 18-216 | |
| Sonifier 250 | Branson | 100-132-135 | |
| Sorvall RC 6 Plus Centrifuge | Thermo Fisher Scientific | 46910 | |
| TCEP (Tris-(carboxyethyl) phosphine hydrochloride) | Gold Biotechnology | TCEP10 | |
| Terrific Broth Powder | Genesee Scientific | 18-225 | |
| Tris Base | Genesee Scientific | 18-146 | |
| XCell SureLock Mini-Cell and XCell II Blot Module | Thermo Fisher Scientific | EI0002 |
Referencias
- Morris-Rosendahl, D. J., Crocq, M. -A. Neurodevelopmental disorders: the history and future of a diagnostic concept. Dialogues Clin Neurosci. 22 (1), 65-72 (2020).
- Ebstein, F., Küry, S., Papendorf, J. J., Krüger, E. Neurodevelopmental disorders (NDD) caused by genomic alterations of the ubiquitin-proteasome system (UPS): the possible contribution of immune dysregulation to disease pathogenesis. Front Mol Neurosci. 14, 733012(2021).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annu Rev Biochem. 67 (10), 425-479 (1998).
- Pickart, C. M. Mechanisms underlying ubiquitination. Annu Rev Biochem. 70 (1), 503-533 (2001).
- Komander, D., Clague, M. J., Urbé, S. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol. 10 (8), 550-563 (2009).
- Yau, R., Rape, M. The increasing complexity of the ubiquitin code. Nat Cell Biol. 18 (6), 579-586 (2016).
- Koch, I., et al. USP27X variants underlying X-linked intellectual disability disrupt protein function via distinct mechanisms. Life Sci Alliance. 7 (3), e202302258(2024).
- Hao, Y. -H., et al. USP7 acts as a molecular rheostat to promote WASH-dependent endosomal protein recycling and is mutated in a human neurodevelopmental disorder. Mol Cell. 59 (6), 956-969 (2015).
- Fountain, M. D., et al. Pathogenic variants in USP7 cause a neurodevelopmental disorder with speech delays, altered behavior, and neurologic anomalies. Genet Med. 21 (8), 1797-1807 (2019).
- Santiago-Sim, T., et al. Biallelic variants in OTUD6B cause an intellectual disability syndrome associated with seizures and dysmorphic features. Am J Hum Genet. 100 (4), 676-688 (2017).
- Beck, D. B., et al. Linkage-specific deubiquitylation by OTUD5 defines an embryonic pathway intolerant to genomic variation. Sci Adv. 7 (4), eabe2116(2021).
- Cho, J., Park, J., Kim, E. E., Song, E. J. Assay systems for profiling deubiquitinating activity. Int J Mol Sci. 21 (16), 1-16 (2020).
- Matsui, S., Sandberg, A. A., Negoro, S., Seon, B. K., Goldstein, G. Isopeptidase: A novel eukaryotic enzyme that cleaves isopeptide bonds. Proc Natl Acad Sci USA. 79 (5), 1535-1539 (1982).
- Rose, I. A., Warms, J. V. B. An enzyme with ubiquitin carboxy-terminal esterase activity from reticulocytes. Biochemistry. 22 (18), 4234-4237 (1983).
- Wilkinson, K. D., Cox, M. J., Mayer, A. N., Frey, T. Synthesis and characterization of ubiquitin ethyl ester, a new substrate for ubiquitin carboxyl-terminal hydrolase. Biochemistry. 25 (21), 6644-6649 (1986).
- Pickart, C. M., Rose, I. A. Ubiquitin carboxyl-terminal hydrolase acts on ubiquitin carboxyl-terminal amides. J Biol Chem. 260 (13), 7903-7910 (1985).
- Hadari, T., Warms, J. V. B., Rose, I. A., Hershko, A. A ubiquitin C-terminal isopeptidase that acts on polyubiquitin chains: Role in protein degradation. J Biol Chem. 267 (2), 719-727 (1992).
- Woo, S. K., et al. Multiple ubiquitin C-terminal hydrolases from chick skeletal muscle. J Biol Chem. 270 (32), 18766-18773 (1995).
- Woo, S. K., et al. Purification and characterization of a new ubiquitin C-terminal hydrolase (UCH-1) with isopeptidase activity from chick skeletal muscle. J Biochem. 121 (4), 684-689 (1997).
- Baek, S. H., et al. Molecular cloning of a novel ubiquitin-specific protease, UBP41, with isopeptidase activity in chick skeletal muscle. J Biol Chem. 272 (41), 25560-25565 (1997).
- Wilkinson, K. D., et al. Metabolism of the polyubiquitin degradation signal: Structure, mechanism, and role of isopeptidase T. Biochemistry. 34 (44), 14535-14546 (1995).
- Hu, M., et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell. 111 (7), 1041-1054 (2002).
- Hu, M., et al. Structure and mechanisms of the proteasome-associated deubiquitinating enzyme USP14. EMBO J. 24 (21), 3747-3756 (2005).
- Gorka, M., Magnussen, H. M., Kulathu, Y. Chemical biology tools to study deubiquitinases and UBL proteases. Semin Cell Dev Biol. 132, 86-96 (2022).
- Faesen, A. C., et al. The differential modulation of USP activity by internal regulatory domains, interactors, and eight ubiquitin chain types. Chem Biol. 18 (12), 1550-1561 (2011).
- Mevissen, T. E. T., et al. OTU deubiquitinases reveal mechanisms of linkage specificity and enable ubiquitin chain restriction analysis. Cell. 154 (1), 169-184 (2013).
- Kwasna, D., et al. Discovery and characterization of ZUFSP/ZUP1, a distinct deubiquitinase class important for genome stability. Mol Cell. 70 (1), 150-164.e6 (2018).
- Hu, H., et al. X-exome sequencing of 405 unresolved families identifies seven novel intellectual disability genes. Mol Psychiatry. 21 (1), 133-148 (2016).
- Hartmann, I. Shaker Orbit - Revolving in Space Around the Samples? Eppendorf Lab Academy. Eppendorf Lab Academy. , at https://www.eppendorf.com/us-en/lab-academy/lab-solutions/others/shaker-orbit-revolving-in-space-around-the-samples (2020).
- Smith, D. B., Johnson, K. S. Single-step purification of polypeptides expressed in Escherichia coli as fusions with glutathione S-transferase. Gene. 67 (1), 31-40 (1988).
- Ritorto, M. S., et al. Screening of DUB activity and specificity by MALDI-TOF mass spectrometry. Nat Commun. 5, 4763(2014).
- Bustos, F., Bustos, F. Immunoblotting using precast gels. , protocols.io (2024).
- Wingfield, P. T. Overview of the purification of recombinant proteins. Curr Protoc Protein Sci. 80, 6.1.1-6.1.35 (2015).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados