需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
溴脱氧尿嘧啶核苷(BrdU)标记和文化独立的鉴定溶解有机随后的荧光激活细胞分选碳有辱人格的浮游细菌
摘要
环境浮游细菌培养与溶解有机碳(DOC)的化合物和一个DNA标记试剂,溴脱氧尿嘧啶核苷(BrdU)的一种模式。随后,商务部降解细胞分离升高BrdU使用荧光激活细胞分选(FACS)纳入散装社会。随后的分子分析这些细胞,然后确定。
摘要
微生物介导许多溶解的有机碳(DOC)基板在水生环境退化的主要代理商。然而,鉴定,改造大自然的DOC的具体池的细菌类群构成一个技术上的挑战。
在这里,我们描述了一个方法,夫妇溴脱氧尿嘧啶核苷(BrdU)掺入,荧光激活细胞分选(FACS),和16S rRNA基因的分子分析功能,使文化有辱人格的特定DOC的化合物在水生环境的浮游细菌能够独立识别。设置为接收BrdU和模型DOC化合物(DOC修订),或只BrdU(不加控制)一式三份浮游细菌的缩影。 BrdU替代品在新合成的细菌的DNA和BrdU标记的DNA的胸苷的位置可以随时immunodetected 1,2。通过一个24小时孵化,浮游细菌,可以使用添加的DOC复合预计将有选择地激活,因此有较高水平的BrdU掺入比在DOC的修订和细胞非反应细胞(您好细胞)在无除了控制(低BrdU掺入细胞,李细胞)。荧光免疫检测后,您好细胞区分和身体从李细胞分离,荧光激活细胞分选(FACS)3 。排序DOC - 反应的细胞(您好细胞)的DNA提取和分类学上通过随后的16S rRNA基因为基础的分析,包括PCR,克隆库的建设和测序鉴定。
研究方案
1。水样加工
- 通过1微米孔径的过滤膜过滤器10L环境用水,以去除大颗粒和bacteriovores。水滤液收集在一个carboy。
- 转让36毫升滤液每到3无菌Eppendorf管中含有4毫升新鲜配制的多聚甲醛溶液(PFA 10%)(50毫升)。在室温下孵育2小时,以保持细胞。收集到0.22微米孔径的白色膜过滤器真空过滤的细胞。通过真空过滤的过滤器10毫升磷酸盐缓冲液(PBS)洗过滤器。标签作为阴性对照的过滤器,并把它们存放在-20 ° C
步骤1.3-1.4建立DOC -有限的条件下是可选的。
- 添加(5微米的NH 4 CL,5微米NANO 3,和1μM的NaH 2 PO 4决赛中浓度)到carboy无机氮和磷的混合物。
- 在现场温度孵育偶尔鼓动48小时在黑暗中。
2。建立和孵化缩影
- 装满800毫升水样从第1.4步建立的缩影carboy每6个1 - L玻璃烧瓶。加入BrdU,终浓度为(10微米),每个缩影。拌匀。
- 添加1毫升模型DOC复合解决方案分为三个缩影,这些将作为商务部修订。 1毫升无菌PBS添加到剩下的三个缩影,这些将作为无除了控制。
- 在一个孵化器中孵化的缩影和孵化在现场温度在黑暗中晃动,而在100转。
- 从每一个缩影和转让到50毫升无菌Eppendorf公司在时间点0,8,16,和24小时的管收集36毫升的水样。立即加入4毫升新鲜煤灰(10%)收集管和孵育2小时在室温下保存的细胞。
- 通过0.22微米孔径的聚碳酸酯膜过滤器过滤器保存的细胞。用10 ml PBS冲洗过滤器。立即着手下一步或存放于-20 ° C的过滤器
在原位免疫检测BrdU掺入3。
- 在室温下解冻的过滤器(商务部修订的样品,不加控制和阴性对照)。
- 应用溶菌酶溶液1毫升10毫克/毫升溶菌酶蛋清在100毫米,50纳米EDTA的Tris(PH = 8)] 4覆盖过滤器上的细菌细胞。在室温下孵育30分钟。通过下吸10毫升PBS冲洗过滤器。
- 添加蛋白酶K溶液1毫升[2 mg / ml的蛋白酶K在100毫米,50纳米EDTA的Tris(PH = 8)] 4覆盖过滤器上的细菌细胞。在室温下孵育30分钟。通过下吸10毫升PBS冲洗过滤器。
- 加入1毫升的核酸外切酶的解决方案[核酸外切酶III(50 U / ml)的氯化镁 2 5毫米和50毫米的Tris - HCl] 5到过滤器上的细菌细胞。 37 ° 30分钟。通过下吸10毫升PBS冲洗过滤器。
- 根据制造商的指示组装框架密封孵化室(BIO - RAD)。
- 酒精清洁干燥的表面上使用无菌刀片切成八分过滤器。
- 使用镊子,放入一个组装框架密封孵化室(每个过滤器样品需要8帧密封腔)第八部分的过滤器。过滤器部分包含单元格方应朝上。过滤器的背面的水分,将允许过滤棒的幻灯片。如果过滤器变得干燥,适用于DIH 2澳商会中心在幻灯片上的过滤器部分一个小滴(2μL)。
步骤3.8-3.20使用罗氏原位细胞增殖试剂盒的试剂,FLUOS从制造商的指示修改的程序。 除对PBS,所有的试剂套件内提供。
- 施加足够的孵育缓冲液(0.5%,0.1%Tween20的PBS,牛血清蛋白),均匀地覆盖整个孵化室的过滤器部分,而不会造成溢出,一旦密封。
- 聚酯放置在孵化室帧帧盖。按上下紧紧密封的孵化室。以上的过滤器,避免气泡。为在室温下在黑暗中10分钟孵育密封腔。
- 删除聚酯密封,打开孵化室。 (注:开放的商会,有时会拉起来的帧从幻灯片密封在这种情况下,准备一个新腔以下步骤。)
- 吸取了孵化室的一个角落缓冲区。避免接触过滤器。
- 轻轻吹打100μLPBS冲洗过滤器和从会议厅出来的3倍。
- 准备抗BrdU - FLUOS以下步骤使用前制造商建议的工作解决方案。
- 吸取120μL抗BrdU FLUOS工作到过滤器的解决方案,照顾到均匀地覆盖整个表面。
- 与一个新的聚酯盖重新密封腔。避免过滤器顶部的气泡。室在黑暗中在37 ° C为3小时。这一步将在原地异硫氰酸荧光素(FITC)标记BrdU纳入DNA。
- 删除聚酯盖,打开孵化室。吸取了抗BrdU - FLUOS工作液。用PBS清洗过滤器的3倍。
- 将过滤器从孵化室,无菌的表面。切片成小块用消毒的刀片的过滤器“部分。
- 转移到2 ml离心管,每片含1毫升PBS的滤波器件。紧瓶盖的封口膜管和密封。在37 ° C和10分钟200转。
- 安全和最高速度为5分钟到一个vortexer涡管。吸取上清液到15毫升无菌Eppendorf管。重复5次以上的孵化和涡步骤。结合在同一15 ml Eppendorf管中,每个样品的上清液。通常情况下,80%的细胞可以恢复暂停。
- 商店取上清液,重悬细胞在4 ° C。 2天之内进行排序。
4。流式细胞仪分析
这里描述的是一个BD FACSAria流式细胞仪和相应的软件程序。
- 优化制造商所建议的流式细胞仪以下步骤设置。这就要求:调整放大控制,设置所需的片断参数和甜蜜点;优化实验鞘压力的激光延迟和面积换算因子,FSC和SSC,FSC的阈值电压,FSC荧光缩放,荧光光电倍增管电压优化设置等
- 基于FITC和侧散射(SSC)的荧光强度流式细胞仪(FCM)上运行的阴性对照样品。增加的FITC标记的阈值,直到阴性对照无细胞可以FITC - SSC采集显示的可视化。
- 运行不加控制样品和研究万细胞FITC和SSC收购的分布格局。定义一个门,附上所有的细胞,并指定为“低强度的细胞”(LIS)。
- 运行DOC修订的样品和研究基于FITC和SSC万细胞的分布格局。有些细胞会出现在预设的李门。定义另一个门附上的细胞,具有较高的荧光强度比LIS(他)。获得统计数据,以查看门细胞的相对丰度。
- 排序您好细胞,成集合,其中包含500μLPBS管“净化1滴”模式。终止排序时,他的人数达50万计数。
5。筛选PCR扩增16S rRNA基因
过滤PCR程序修改Kirchman等。
- 筛选到一个白色的排序细胞,0.22微米孔径,25毫米直径,聚碳酸酯滤膜过滤。剪掉的过滤器,不包含任何使用无菌刀片的细菌细胞的边缘。切成8个同等大小的块过滤器。
- 到PCR反应管中放置一个单一的过滤片,与细胞面临着外来的管。 PCR反应管中加入45μLPCR级水。淹没的过滤器完全在水中。
- 2罗氏插图PuReTaq阅读到进入PCR反应管PCR珠,简要旋涡。添加正向和反向的16S rRNA基因的引物(0.4微米每个引物终浓度),如27F和1492R 7各2μL,每个PCR反应管中。
- (可选)添加加入1μl牛血清白蛋白溶液(BSA,终浓度是30μg/100μL)的PCR反应,以帮助吸附放大抑制剂。如果选择不添加牛血清白蛋白,加水加入1μl。
- 进行PCR扩增热循环仪上一个。一个触摸下来PCR程序的建议,其中的退火温度,比上一季度减少62至52 ° C时为1 ° C,每循环11周期由15个循环,退火温度52 ° C。所有循环包括变性(95℃),退火(62至52℃)和扩展(72℃)50年代的持续时间步骤。 PCR程序也包括在最初的3分钟变性和最后的10分钟延长步骤。
- 溴化乙锭染色的1%琼脂糖凝胶电泳,确认PCR扩增。海关从凝胶中的PCR扩增和凯杰QIAquick凝胶提取试剂盒的清洁。
- 每个样本执行两个额外的PCR扩增,每次使用一个新的过滤器“部分。经过PCR凝胶purificati上,池同一样品的扩增产物。纯净16S rDNA的扩增产物,现在准备允许分类鉴定,如克隆库的建设和测序,分子生物学分析。
6。代表性的成果:
代表使用为例,腐胺降解菌的研究结果说明。从格鲁吉亚沿海现场采集水样,并按照上述程序进行处理。流式细胞仪分析显示,腐胺除了诱发高FITC荧光强度的细菌集团的发展,说明高BrdU掺入率(图1)。这些细胞被指定为高BrdU掺入细胞(他),预计包含大多为腐胺降解菌。他在不加控制,只包含与下级BrdU掺入(LIS)的细胞缺少。 LIS,预计主要包括浮游细菌,无法使用添加腐胺。他整理成分离的导管,然后到膜过滤器收集。获得高您好使用过滤器PCR细胞16S rRNA基因扩增产物。
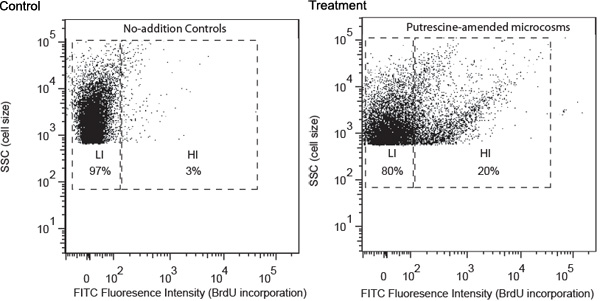
图1。流量没有除了控制和孵化24小时后收集的样品模型化合物的修订(腐胺作为一个例子)流式细胞仪分析。细胞分布的分析是基于(1)的FITC标记(X轴),这是正相关,BrdU掺入水平的荧光强度,(2)侧散射(SSC,Y轴),这是正相关的细胞大小。门符号是基于BrdU掺入,(高,高BrdU掺入,李,低BrdU的掺入)的水平。 HI和李细胞相对百分比显示在相应的门。
讨论
我们的做法夫妇BrdU掺入,流式细胞仪和16S rDNA序列分析,让物种一级的浮游细菌的代谢水环境中的个人的DOC组件的识别。 BrdU掺入检测标签的细菌细胞的代谢活动,只允许对活性细菌的分析,因此不包括休眠细胞。在我们的方法, 在原位 immunodetected和细菌有不同层次的BrdU掺入BrdU掺入随后可视化,使用流式细胞仪进行分组和排序。的BrdU纳入细胞的分析也有报道使用的BrdU标记的DNA磁珠immu...
披露声明
没有利益冲突的声明。
致谢
这个项目的经费是由国家科学基金会资助OCE1029607(XM)和MCB0702125(MAM),戈登和贝蒂摩尔基金会(MAM)的。
材料
| Name | Company | Catalog Number | Comments | |
| 名称 | 类型 | 公司 | 目录编号 | 评论 |
| BrdU | 试剂 | 西格玛 | B5002 - 5G | |
| 溶菌酶 | 试剂 | 西格玛 | L6876 - 5G | |
| 蛋白酶K | 试剂 | 西格玛 | P2308 - 25毫克 | |
| 原位细胞增殖试剂盒,FLUOS | 套件 | 罗氏公司 | 11810740001 | 抗BrdU FLUOS和反应比制造商建议的孵化每缓冲区消费。 |
| 帧密封孵化钱伯斯 | 材料 | Bio - Rad公司 | SLF - 1201 | |
| 聚碳酸酯膜过滤器(直径142毫米,1.0微米孔径) | 材料 | Millipore公司 | FALP14250 | |
| 聚碳酸酯膜过滤器(直径25毫米,0.2微米孔径) | 材料 | Millipore公司 | FGLP02500 | |
| 插图PuReTaq现成的PCR珠 | 套件 | GE医疗集团 | 27-9559-01 | |
| QIAquick凝胶提取试剂盒 | 套件 | QIAGEN公司 | 28704 | |
| 防故障PCR检测系统 | 套件 | 震中 | FS99060 |
参考文献
- Pernthaler, A., Pernthaler, J., Schattenhofer, M., Amann, R. Identification of DNA-synthesizing bacterial cells in coastal North Sea plankton. Appl. Environ. Microbiol. 68, 5728-5728 (2002).
- Urbach, E., Vergin, K. L., Giovannoni, S. J. Immunochemical detection and isolation of DNA from metabolically active bacteria. Appl. Environ. Microbiol. 65, 1207-12 (1999).
- Mou, X. Z., Hodson, R. E., Moran, M. A. Bacterioplankton assemblages transforming dissolved organic compounds in coastal seawater. Environ. Microbiol. 9, 2025-2025 (2007).
- Hodson, R. E., Dustman, W. A., Garg, R. P., Moran, M. A. In situ PCR for visualization of microscale distribution of specific genes and gene products in prokaryotic communities. Appl. Environ. Microbiol. 61, 4074-4074 (1995).
- Dinjens, W. N. Bromodeoxyuridine (BrdU) immunocytochemistry by exonuclease III (Exo III) digestion. Histochemistry. 98, 199-199 (1992).
- Kirchman, D. L., Yu, L. Y., Fuchs, B. M., Amann, R. Structure of bacterial communities in aquatic systems as revealed by filter PCR. Aquat. Microb. Ecol. 26, 13-13 (2001).
- Delong, E. F., Wickham, G. S., Pace, N. R. Phylogenetic stains: ribosomal RNA-based probes for the identification of single cells. Science. 243, 1360-1360 (1989).
- Artursson, V., Jansson, J. K. Use of bromodeoxyuridine immunocapture to identify active bacteria associated with arbuscular mycorrhizal hyphae. Appl. Environ. Microbiol. 69, 6208-6208 (2003).
- Mou, X. Z. Bacterial carbon processing by generalist species in the coastal ocean. Nature. 451, 708-708 (2008).
- Mou, X. Flow-cytometric cell sorting and subsequent molecular analyses for culture-independent identification of bacterioplankton involved in dimethylsulfoniopropionate transformations. Appl. Environ. Microbiol. 71, 1405-1405 (2005).
- Dean, F. B. Comprehensive human genome amplification using multiple displacement amplification. Proc. Natl. Acad. Sci. U.S.A. 99, 5261-5261 (2002).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。