Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Bromodeoxyuridine (BrdU) Labeling und anschließende Fluorescence Activated Cell Sorting für Kultur-unabhängige Identifizierung von gelöstem organischem Kohlenstoff abbauenden Bakterioplankton
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Environmental Bakterioplankton sind mit einem Modell des gelösten organischen Kohlenstoffs (DOC)-Verbindung und einer DNA Markierungsreagens, Bromdesoxyuridin (BrdU) inkubiert. Anschließend werden DOC-abbauenden Zellen aus dem Bulk-Community auf ihre erhöhte BrdU-Einbaus mittels Fluoreszenz-activated cell sorting (FACS) basiert getrennt. Diese Zellen werden dann durch nachfolgende molekulare Analysen identifiziert.
Zusammenfassung
Mikroben sind wichtige Mittel der Vermittlung der Abbau von zahlreichen gelösten organischen Kohlenstoffs (DOC) Substrate in der aquatischen Umwelt. Allerdings stellt Identifizierung von Bakterien-Taxa, dass bestimmte Pools von DOC in der Natur verwandeln eine technische Herausforderung.
Hier beschreiben wir einen Ansatz, Paare Bromdesoxyuridin (BrdU) Inkorporation, Fluoreszenz-activated cell sorting (FACS) und 16S rRNA-Gen-basierte molekulare Analyse, dass die Kultur-unabhängige Identifizierung Bakterioplankton, die einen Abbau einer bestimmten DOC Verbindung in der aquatischen Umwelt ermöglicht. Triplicate Bakterioplankton Mikrokosmen sind bis zu beiden BrdU und ein Modell DOC Verbindung (DOC Änderungen), oder nur BrdU (no-Neben-Steuerung) zu empfangen. BrdU ersetzt die Positionen von Thymidin in neu synthetisierte Bakterien-DNA und BrdU-markierte DNA kann leicht immunodetected 1,2 sein. Durch eine 24-Stunden-Inkubation Bakterioplankton, dass in der Lage, die hinzugefügt DOC Verbindung erwartet werden, um selektiv aktiviert werden, und haben daher ein höheres BrdU-Einbau (HALLO-Zellen) als non-responsive Zellen in der DOC Änderungen und Zellen in nicht-sind Zusätzlich steuert (low BrdU-Einbau-Zellen, LI-Zellen). Nach Fluoreszenz Immundetektion sind HALLO Zellen unterschieden und körperlich von der LI-Zellen durch Fluoreszenz-aktivierte Zellsortierung (FACS) 3 getrennt. Sortiert DOC-responsive Zellen (HALLO-Zellen) sind für die DNA extrahiert und taxonomisch durch nachträgliche 16S rRNA-Gen-Analysen, einschließlich PCR, Klon-Bibliothek Bau-und Sequenzierung identifiziert.
Protokoll
1. Wasserprobe Verarbeitung
- Filter 10L Umwelt Wasser durch 1 &mgr; m-Porengröße Membranfilter zur Entfernung großer Partikel und bacteriovores. Sammeln Sie die Wasser-Filtrat in einem Kanister.
- Transfer 36 ml Filtrat in je 3 sterile Eppendorf-Röhrchen (50 ml) mit 4 ml frisch zubereitete Paraformaldehyd-Lösung (PFA; 10%). Inkubieren für 2 Stunden bei Raumtemperatur, um Zellen zu erhalten. Sammeln Zellen auf 0,22-um-Porengröße weiß Membranfilter durch Vakuumfiltration. Waschen Sie den Filter, indem 10 ml Kochsalzlösung (PBS) Phosphat-gepufferte durch den Filter durch Vakuumfiltration. Beschriften Sie die Filter als negative Kontrollen und lagern Sie sie bei -20 ° C.
Steps 1,3-1,4 sind für die Errichtung DOC-limitierten Bedingungen optional.
- Eine Mischung von anorganischem Stickstoff und Phosphor (5 pM NH 4 Cl, 5 uM NaNO 3 und 1 &mgr; M NaH 2 PO 4 Endkonzentration) in den Kanister.
- Inkubieren im Dunkeln bei in situ Temperatur für 48 Stunden mit gelegentlichen Erschütterungen.
2. Mikrokosmos Einrichtung und Inkubation
- Füllen Sie jede der sechs 1-L Glasflaschen mit 800 ml Wasserprobe aus dem Ballon der Schritt von 1,4 bis Mikrokosmen zu etablieren. Add BrdU (10 uM, Endkonzentration) zu jeder Mikrokosmos. Gut mischen.
- 1 ml Modell DOC Verbindung Lösung in drei der Mikrokosmen, werden diese als DOC Änderungen dienen. 1 ml sterilem PBS in den verbleibenden drei Mikrokosmen, werden diese als nicht-Addition Kontrollen dienen.
- Inkubieren Sie alle Mikrokosmen in einem Inkubator Schüttler inkubieren im Dunkeln bei in situ Temperatur unter Schütteln bei 100 Umdrehungen pro Minute.
- Sammeln Sie 36 ml Wasser Probe von jeder Mikrokosmos und Transfer in 50 ml sterile Eppendorf-Röhrchen zu den Zeitpunkten 0, 8, 16, und 24 Stunden. Unmittelbar 4 ml frisches PFA (10%) auf Sammelröhrchen und Inkubation für 2 Stunden bei Raumtemperatur, um die Zellen zu erhalten.
- Filter bewahrt Zellen durch 0,22-um-Porengröße Polykarbonatmembranfilter. Waschen Sie den Filter mit 10 ml PBS. Gehen Sie sofort zum nächsten Schritt oder lagern Sie die Filter bei -20 ° C.
3. In situ Immundetektion für BrdU-Einbau
- Thaw die Filter (DOC geändert Proben, ohne zusätzlich Kontrollen und negative Kontrollen) bei Raumtemperatur.
- Es wird 1 ml Lysozym-Lösung [10 mg / ml Lysozym Eiweiß in 100 mM Tris, 50 nM EDTA (pH = 8)] 4 bis Bakterienzellen auf dem Filter zu decken. Inkubieren bei Raumtemperatur für 30 Minuten. Waschen Sie den Filter, indem 10 ml PBS durch sie unter Absaugung.
- 1 ml Proteinase K-Lösung [2 mg / ml Proteinase K in 100 mM Tris, 50 nM EDTA (pH = 8)] 4 bis Bakterienzellen auf dem Filter zu decken. Inkubieren bei Raumtemperatur für 30 Minuten. Waschen Sie den Filter, indem 10 ml PBS durch sie unter Absaugung.
- 1 ml Exonuklease-Lösung [Exonuklease III (50 U / ml) in 5 mM MgCl 2 und 50 mM Tris-HCl] 5 bis Bakterienzellen auf dem Filter zu decken. Bei 37 ° C für 30 Minuten. Waschen Sie den Filter, indem 10 ml PBS durch sie unter Absaugung.
- Montieren Sie frame-Siegel Inkubationskammern (Bio-Rad) nach den Anweisungen des Herstellers.
- Schneiden Sie die Filter in Achtel mit einer sterilen Klinge auf ein Alkohol gereinigt trockene Oberfläche.
- Mit einer Pinzette, legen ein achter Abschnitt eines Filters zu einem montierten Rahmen-Dichtung Inkubationskammer (acht frame-Siegel Kammern sind für jeden Filter Probe erforderlich). Die Seite der Filter-Sektion, die Zellen enthält, sollte nach oben zeigen. Die Feuchtigkeit an der Rückseite des Filters wird der Filter-Stick, um die Folie zu ermöglichen. Wenn der Filter trocken wird, wenden Sie einen kleinen Tropfen (2 ul) diH 2 O in der Mitte der Kammer, bevor der Filter-Sektion auf der Folie.
Steps 3,8-3,20 Verwendung der Reagenzien aus den ROCHE In Situ Cell Proliferation Kit, FLUOS folgenden Verfahren aus der Anleitung des Herstellers modifiziert. Mit Ausnahme von PBS, sind alle Reagenzien im Kit enthalten.
- Genügend Inkubationspuffer (0,5% Rinderserumalbumin, 0,1% Tween20 in PBS,) gleichmäßig auf die gesamte Filter-Sektion in der Inkubationskammer ohne Überlauf, wenn die Dichtung aufgebracht wird.
- Legen Sie eine Polyester-Frame Abdeckung über dem Brutraum Rahmen. Drücken Sie fest zu verschließen Inkubationskammer. Vermeiden Sie Luftblasen über dem Filter. Inkubieren Sie die verschlossenen Kammern für 10 min bei Raumtemperatur im Dunkeln.
- Entfernen Sie die Polyester-Siegel und öffnen Sie die Inkubationskammern. (Hinweis: Öffnen der Kammern manchmal wird hochziehen Rahmen Dichtung aus der Folie in diesem Fall, bereiten Sie einen neuen Raum für die folgenden Schritte..)
- Pipettieren Sie die Inkubationspuffer von einer Ecke der Kammer. Vermeiden Sie den Kontakt mit dem Filter.
- Waschen Sie den Filter durch leichtes Pipettieren 100 ul PBS in undaus der Kammer 3 mal.
- Bereiten anti-BrdU-FLUOS funktionierende Lösung die folgenden Schritte durch den Hersteller unmittelbar vor Gebrauch empfohlen.
- Pipet 120 ul Anti-BrdU-FLUOS funktionierende Lösung auf den Filter, wobei darauf zu gleichmäßig auf die gesamte Oberfläche.
- Verschließen Sie die Kammer mit einer neuen Polyester-Bezug. Vermeiden Sie Luftblasen auf der Oberseite des Filters. Inkubieren Sie die Kammer im Dunkeln bei 37 ° C für 3 Stunden. Dieser Schritt wird BrdU eingebaut DNA in situ mit Fluoresceinisothiocyanat (FITC)-Label.
- Entfernen Sie die Polyester-Abdeckung und öffnen Sie die Inkubationskammer. Pipettieren aus der Anti-BrdU-FLUOS funktionierende Lösung. Waschen Sie den Filter 3 mal mit PBS.
- Übertragen Sie die Filter aus dem Brutraum in eine sterile Oberfläche. Schneiden Sie die Filter-Sektion in kleine Stücke mit einer sterilen Klinge.
- Übertragen Sie die Filter Stücke in 2 ml Reaktionsgefäße mit je 1 ml PBS. Fest verschließen den Schlauch und Dichtung mit Parafilm. Bei 37 ° C und 200 rpm für 10 min.
- Befestigen Sie die Schläuche auf einem Vortexer und Vortex bei maximaler Geschwindigkeit für 5 Minuten. Pipettieren der Überstand in ein steriles 15 ml Eppendorf-Röhrchen. Wiederholen Sie die Inkubation und Wirbel Schritte für 5 weitere Male. Kombinieren Sie den Überstand in den gleichen 15 ml Eppendorf-Röhrchen für jede Probe. In der Regel können 80% Zellen in der Suspension zurückgewonnen werden.
- Bewahren Sie den Überstand mit resuspendierten Zellen bei 4 ° C. Sortieren innerhalb von 2 Tagen.
4. FACS-Analyse
Ein Verfahren für eine BD FACSAria Durchflusszytometer und entsprechender Software wird hier beschrieben.
- Optimieren Sie die Einstellung der Durchflusszytometer folgenden Schritte vom Hersteller empfohlen. Dies bedeutet: Einstellung der Verstärkung, um die gewünschte breakoff Parameter und den Sweet Spot gesetzt; Optimierung der Laser-Verzögerung und Umgebung Skalierungsfaktoren für das Experiment Mantel Druck und Optimierung der Einstellungen für FSC und SSC Spannungen, FSC Schwelle, FSC-Fluoreszenz-Skalierung, Fluoreszenz-PMT-Spannungen , etc.
- Run negativen Kontrollproben am Durchflusszytometer (FCM) auf Fluoreszenz-Intensität des FITC und side scatter (SSC) basiert. Erhöhen Sie die FITC Schwelle, bis keine Zellen in den negativen Kontrollen können visualisiert werden durch die FITC-SSC Erwerb anzuzeigen.
- Run no-Neben Kontrollproben und untersuchen das Verteilungsmuster von 10.000 Zellen auf FITC und SSC Übernahmen. Definieren Sie ein Tor, um alle Zellen umschließen und bezeichnen sie als "low intensity Zellen" (LIS).
- Run DOC-Fassung Proben und untersuchen das Verteilungsmuster von 10.000 Zellen auf FITC und SSC Basis. Einige Zellen werden in der vorgegebenen LI Tor erscheinen. Definieren Sie ein weiteres Tor zu den Zellen, die höhere Intensität der Fluoreszenz (HIS) als LIs haben umschließen. Erwerben Sie Statistiken, um die relative Fülle von gated Zellen anzuzeigen.
- Sortieren HALLO Zellen in Sammelröhrchen mit 500 ul PBS bei "reinigen 1 Tropfen"-Modus. Terminate Sortierung, wenn die Zahl seiner erreicht 500.000 zählt.
5. Filter PCR-Amplifikation von 16S rRNA-Gene
Filter PCR-Verfahren werden aus Kirchman et al geändert. 6
- Filter sortierten Zellen auf eine weiße, 0,22 um-Porengröße, 25 mm Durchmesser, Polycarbonat-Membranfilter. Schneiden Sie den Rand des Filters, dass keine bakterielle Zellen mit einer sterilen Klinge enthält. Schneiden Sie die Filter in 8 gleich große Stücke schneiden.
- Legen Sie eine einzelne Filter Stück in ein PCR-Gefäß, mit dem Zellen nach innen der Röhre. Add 45 ul PCR-Wasser in die PCR-Reaktionsgefäß. Tauchen Sie den Filter komplett im Wasser.
- Add 2 ROCHE Abbildungen PuReTaq Read-To-Go PCR Beads in die PCR-Gefäß, kurz vortexen. Add 2 ul jeder der Vorwärts-und Rückwärtsfahrt 16S rRNA-Gen-Primer (0,4 uM Endkonzentration für jedes Primer), wie 27F und 1492R 7, in jedem der PCR Reaktionsgefäße.
- (Optional) Fügen 1μl Rinderserumalbumin-Lösung (BSA, Endkonzentration 30μg/100 ul) der PCR Reaktionsmischung zu adsorbieren Verstärkung Hemmer helfen. Wenn man nicht an BSA hinzufügen wählt, fügen 1μl Wasser statt.
- Führen Sie die PCR-Amplifikation auf einem Thermocycler. A touch down PCR-Programm empfohlen wird, hat die Glühtemperatur sequenziell abnehmenden von 62 bis 52 ° C um 1 ° C pro Zyklus für 11 Zyklen von 15 Zyklen mit Annealingtemperatur von 52 ° C, gefolgt Alle Zyklen sind Denaturierung (bei 95 ° C), Annealing (at 62 bis 52 ° C) und Verlängerung (bei 72 ° C) Schritte der 50er Jahre Dauer. Eine erste 3-min Denaturierung und letzte 10-min-Erweiterung wird auch für die PCR-Programm enthalten.
- Bestätigen Sie die PCR-Amplifikation mittels Elektrophorese auf einem mit Ethidiumbromid gefärbten 1% Agarosegel. Excise der PCR-Produkte aus dem Gel und sauber mit dem QIAGEN QIAquick Gel Extraction Kit.
- Führen Sie zwei zusätzliche PCR-Amplifikationen für jede Probe, verwenden Sie jedes Mal eine neue Filter-Sektion. Nach PCR-Gel purificatiauf, bündeln die Amplikons der gleichen Probe zusammen. Gereinigtes 16S rDNA Amplikons sind nun bereit für eine Reihe von molekularen Analysen, taxonomische Identifizierung, wie Klon-Bibliothek Bau-und Sequenzierung zu ermöglichen.
6. Repräsentative Ergebnisse:
Repräsentative Ergebnisse werden anhand einer Studie von Putrescin-abbauenden Bakterien als ein Beispiel. Wasserproben wurden von einem Küstengebiet von Georgien gesammelt und verarbeitet nach dem oben beschriebenen Verfahren. FACS-Analyse ergab, dass Putrescin Neben der Entwicklung einer Gruppe von Bakterien mit hohem FITC Fluoreszenz-Intensität induziert, was eine hohe BrdU-Einbau-Rate (Abbildung 1). Diese Zellen wurden als High-BrdU-Einbau-Zellen (HIS) bezeichnet und wurde erwartet, enthalten meist Putrescin-abbauenden Bakterien. Seine waren in der nicht-Addition Kontrollen, die nur enthalten Zellen mit niedrigerem BrdU-Einbau (LIS) fehlt. LIs wurde erwartet, dass vor allem enthalten Bakterioplankton, dass nicht hinzugefügt Putrescin Einsatz waren. His wurden in getrennten Röhren sortiert und dann gesammelt auf Membranfiltern. 16S rRNA-Gen Amplikons wurden für hohe HALLO Zellen mittels Filter PCR erhalten.
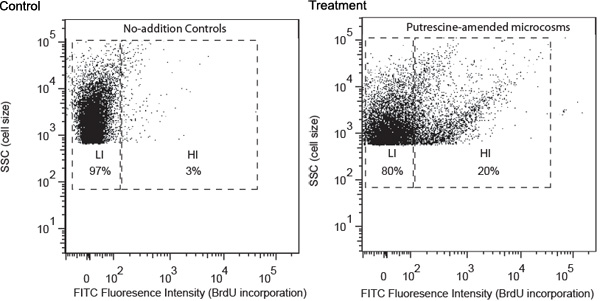
Abbildung 1. Durchflusszytometrische Analyse von nicht-zusätzlich-Steuerung und Modell-Compound-Fassung (Putrescin hier als Beispiel) Proben nach 24 h Inkubation gesammelt. Zellverteilung Analyse wurde auf (1) Fluoreszenzintensität FITC-Markierung (x-Achse), was sich positiv auf Ebene des BrdU-Einbaus ist im Zusammenhang basiert, und (2) side scatter (SSC, y-Achse), die positiv zu Zelle im Zusammenhang Größe. Tor Notation ist auf Ebene des BrdU-Einbaus, (; LI, low-BrdU-Inkorporation HALLO, High-BrdU-Inkorporation) basiert. Der relative Anteil der HALLO-und LI-Zellen sind in entsprechenden Toren gezeigt.
Diskussion
Unser Ansatz Paare BrdU-Einbau, FACS und 16S rDNA-Analyse zu ermöglichen Arten-Identifikation auf der Bakterioplankton, dass die einzelnen Komponenten DOC verstoffwechseln in der aquatischen Umwelt. Die BrdU-Einbau-Assay Etiketten Bakterienzellen auf Stoffwechselaktivitäten, die Analyse erlaubt nur auf aktive Bakterien und somit nicht enthalten ruhenden Zellen. In unserem Ansatz wird BrdU-Einbau in situ immunodetected und Bakterien, die verschiedenen Ebenen des BrdU-Einbaus haben, sind anschließend visualisi...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Finanzierung dieses Projekts wurde von der National Science Foundation gewährt OCE1029607 (XM) und MCB0702125 (MAM) und Gordon und Betty Moore Foundation (MAM) zur Verfügung gestellt.
Materialien
| Name | Company | Catalog Number | Comments | |
| Name | Typ | Firma | Katalog-Nummer | Kommentare |
| BrdU | Reagens | Sigma | B5002-5G | |
| Lysozym | Reagens | Sigma | L6876-5G | |
| Proteinase K | Reagens | Sigma | P2308-25mg | |
| In Situ Cell Proliferation Kit, FLUOS | Satz | Roche | 11810740001 | Verbrauchen Sie mehr von Anti-BrdU-FLUOS und Inkubationspuffer pro Reaktion als vom Hersteller empfohlen. |
| Frame-Seal Inkubationskammern | Material | Bio-Rad | SLF-1201 | |
| Polycarbonat-Membranfilter (142-mm-Durchmesser, 1,0 &mgr;-Porengröße) | Material | Millipore | FALP14250 | |
| Polykarbonatmembranfilter (25-mm-Durchmesser, 0,2 mu m-pore-size) | Material | Millipore | FGLP02500 | |
| Abbildungen PuReTaq Ready-To-Go PCR Beads | Satz | GE Healthcare | 27-9559-01 | |
| QIAquick Gel Extraction Kit | Satz | QIAGEN | 28704 | |
| FailSafe PCR-System | Satz | EPICENTRE | FS99060 |
Referenzen
- Pernthaler, A., Pernthaler, J., Schattenhofer, M., Amann, R. Identification of DNA-synthesizing bacterial cells in coastal North Sea plankton. Appl. Environ. Microbiol. 68, 5728-5728 (2002).
- Urbach, E., Vergin, K. L., Giovannoni, S. J. Immunochemical detection and isolation of DNA from metabolically active bacteria. Appl. Environ. Microbiol. 65, 1207-12 (1999).
- Mou, X. Z., Hodson, R. E., Moran, M. A. Bacterioplankton assemblages transforming dissolved organic compounds in coastal seawater. Environ. Microbiol. 9, 2025-2025 (2007).
- Hodson, R. E., Dustman, W. A., Garg, R. P., Moran, M. A. In situ PCR for visualization of microscale distribution of specific genes and gene products in prokaryotic communities. Appl. Environ. Microbiol. 61, 4074-4074 (1995).
- Dinjens, W. N. Bromodeoxyuridine (BrdU) immunocytochemistry by exonuclease III (Exo III) digestion. Histochemistry. 98, 199-199 (1992).
- Kirchman, D. L., Yu, L. Y., Fuchs, B. M., Amann, R. Structure of bacterial communities in aquatic systems as revealed by filter PCR. Aquat. Microb. Ecol. 26, 13-13 (2001).
- Delong, E. F., Wickham, G. S., Pace, N. R. Phylogenetic stains: ribosomal RNA-based probes for the identification of single cells. Science. 243, 1360-1360 (1989).
- Artursson, V., Jansson, J. K. Use of bromodeoxyuridine immunocapture to identify active bacteria associated with arbuscular mycorrhizal hyphae. Appl. Environ. Microbiol. 69, 6208-6208 (2003).
- Mou, X. Z. Bacterial carbon processing by generalist species in the coastal ocean. Nature. 451, 708-708 (2008).
- Mou, X. Flow-cytometric cell sorting and subsequent molecular analyses for culture-independent identification of bacterioplankton involved in dimethylsulfoniopropionate transformations. Appl. Environ. Microbiol. 71, 1405-1405 (2005).
- Dean, F. B. Comprehensive human genome amplification using multiple displacement amplification. Proc. Natl. Acad. Sci. U.S.A. 99, 5261-5261 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten