A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
Bromodeoxyuridine (BrdU) תיוג ומיון לאחר פלואורסצנטי תא פעיל עבור זיהוי, תרבות עצמאית של פחמן אורגני מומסים משפיל Bacterioplankton
* These authors contributed equally
In This Article
Summary
Bacterioplankton הסביבה מודגרת עם מודל מומס פחמן אורגני (DOC) המתחם מגיב תיוג DNA, bromodeoxyuridine (BrdU). לאחר מכן, DOC-משפיל תאים מופרדים מן הקהילה בתפזורת מבוסס על שילוב גבוהות BrdU שלהם באמצעות תא הקרינה מיון מופעל (FACS). תאים אלה מזוהים מכן על ידי מנתח מולקולרית שלאחר מכן.
Abstract
חיידקים הם סוכני התיווך העיקריים את ההשפלה של פחמן אורגני מומס רבים (DOC) מצעים בסביבות מימיים. עם זאת, זיהוי מינים חיידקים שהופכים בריכות ספציפיים של DOC בטבע מהווה אתגר טכני.
כאן אנו מתארים גישה זוגות bromodeoxyuridine (BrdU) ההתאגדות, תא הקרינה מיון מופעל (FACS), ו - 16S rRNA ניתוח מולקולרי גנטי המבוסס המאפשר תרבות עצמאית זיהוי bacterioplankton מסוגל מתחם DOC משפיל ספציפי בסביבות מימיים. Bacterioplankton מיקרוקוסמוס בשלושה עותקים מוגדרים לקבל הן BrdU ו תרכובת DOC מודל (DOC תיקונים), או רק BrdU (ללא תוספת שליטה). תחליפי BrdU עמדות thymidine ב-DNA חיידקי מסונתז החדש ו-BrdU שכותרתו DNA ניתן 1,2 immunodetected בקלות. באמצעות bacterioplankton 24 שעות דגירה, כי הם מסוגלים להשתמש במתחם הוסיף DOC צפויים להיות מופעלים באופן סלקטיבי, ולכן יש רמות גבוהות יותר של שילוב BrdU (תאים HI) מאשר אי - היענות תאים התיקונים DOC ו תאים ללא בנוסף שולטת (נמוך תאים BrdU ההתאגדות, תאים LI). לאחר immunodetection פלואורסצנטי, HI תאים נבדלים ו מופרדים פיזית על ידי מיון הקרינה התא הפעיל (FACS) 3 מתאי LI. ממוין DOC-מגיבים תאים (תאים HI) המחולצים עבור דנ"א שזוהו טקסונומית באמצעות 16S rRNA הבאים הגן מבוססי ניתוח כולל PCR, שיבוט בנייה ספריה וסדר.
Protocol
1. דגימת מים עיבוד
- מסנן מים סביבתיים 10L עד 1 מיקרומטר, נקבוביות בגודל מסננים קרום להסיר חלקיקים גדולים bacteriovores. אסוף את תסנין בקבוק מים.
- העברת 36 מ"ל תסנין לכל לתוך 3 צינורות סטרילי Eppendorf (50 מ"ל) המכילה 4 מ"ל טרי paraformaldehyde פתרון מוכן (PFA; 10%). דגירה עבור 2 שעות בטמפרטורת החדר כדי לשמר את התאים. איסוף תאים על 0.22-מיקרומטר, נקבוביות בגודל מסננים קרום לבן על ידי סינון ואקום. שטפו את המסנן על ידי העברת 10 מ"ל בופר פוספט (PBS) דרך המסנן על ידי סינון ואקום. לייבל מסננים כפקדי שלילי ולאחסן אותם -20 ° C.
צעדים 1.3-1.4 הם אופציונליים להקמת DOC מוגבלת בתנאים.
- מוסיפים תערובת של חנקן זרחן אורגניים (5 מיקרומטר NH 4 Cl, 5 מיקרומטר NANO 3, 1μM לאא 2 PO 4 בריכוז הסופי) לתוך בקבוק.
- דגירה בחושך על הטמפרטורה במשך 48 שעות באתרו עם בהסתה מזדמנים.
2. מיקרוקוסמוס הקמת הדגירה
- ממלאים כל צלוחיות של six 1-L עם זכוכית מדגם 800 מ"ל בקבוק מים של 1.4 צעד להקים מיקרוקוסמוס. הוסף BrdU (10 מיקרומטר, הריכוז הסופי) כדי מיקרוקוסמוס כל אחד. מערבבים היטב.
- הוסף 1 מ"ל מודל DOC פתרון מתחם לשלוש של מיקרוקוסמוס, אלה ישמש תיקונים DOC. הוסף 1 מ"ל סטרילי PBS לשלוש מיקרוקוסמוס הנותרים, אלה ישמש ללא בנוסף שולטת.
- דגירה כל מיקרוקוסמוס בתוך שייקר החממה דגירה בחושך בבית בטמפרטורה באתרו תוך רועד ב 100 סל"ד.
- איסוף של 36 מ"ל דגימת מים מן מיקרוקוסמוס כל העברה לתוך 50 מ"ל צינורות סטרילי Eppendorf בנקודות זמן של 0, 8, 16, ו - 24 שעות. מיד להוסיף 4 מ"ל של PFA טריים (10%) לצינורות איסוף דגירה עבור 2 שעות בטמפרטורת החדר כדי לשמר את התאים.
- סנן נשמר באמצעות תאים 0.22-מיקרומטר, נקבוביות בגודל מסננים פוליקרבונט הממברנה. שטפו את המסננים עם 10 מ"ל PBS. המשך מיד לשלב הבא או לאחסן את המסננים ב -20 ° C.
3. Immunodetection באתרו עבור התאגדות BrdU
- ההפשרה מסננים (דגימות DOC המתוקן, ללא תוספת שולט שולטת שלילי) בטמפרטורת החדר.
- החל 1 מ"ל של תמיסת ליזוזים [10 מ"ג / מ"ל ליזוזים ביצה לבן 100 מ"מ טריס, 50 ננומטר EDTA (pH = 8)] 4 תאים חיידקיים כדי לכסות על המסנן. דגירה בטמפרטורת החדר למשך 30 דקות. שטפו את המסנן על ידי העברת 10 מ"ל PBS דרכו תחת יניקה.
- הוסף 1 מ"ל של תמיסת K proteinase [2 מ"ג / מ"ל proteinase K ב 100 מ"מ טריס, 50 ננומטר EDTA (pH = 8)] 4 תאים חיידקיים כדי לכסות על המסנן. דגירה בטמפרטורת החדר למשך 30 דקות. שטפו את המסנן על ידי העברת 10 מ"ל PBS דרכו תחת יניקה.
- הוסף 1 מ"ל exonuclease פתרון [exonuclease III (50 U / ml) ב 5 מ"מ MgCl 2 ו - 50 מ"מ טריס-HCl] 5 תאים חיידקיים כדי לכסות על המסנן. לדגור על 37 מעלות צלזיוס במשך 30 דקות. שטפו את המסנן על ידי העברת 10 מ"ל PBS דרכו תחת יניקה.
- להרכיב מסגרת חותם בתאי הדגירה (Bio-Rad) על פי הוראות היצרן.
- פורסים את המסנן לתוך שמיניות באמצעות להב סטרילי על פני השטח אלכוהול לניקוי יבש.
- בעזרת מלקחיים, במקום סעיף השמיני של מסנן לתוך תא מסגרת אחת חותם התאספו הדגירה (שמונה מסגרת חותם בלשכה יש צורך במדגם כל מסנן). הצד של הסעיף לסנן המכילה תאים צריך הפנים כלפי מעלה. רטיבות בחלק האחורי של המסנן תאפשר את המקל לסנן לשקופית. אם המסנן הופך יבש, להחיל טיפה (2 μl) של diH 2 O במרכז החדר לפני הצבת בסעיף לסנן בשקופית.
צעדים 3.8-3.20 להשתמש ריאגנטים מן רוש קיט תא באתרו הפצת נשק, FLUOS שונה מן הנהלים הבאים הוראות היצרן. חוץ PBS, ריאגנטים כל מסופקות בתוך הערכה.
- החל חיץ דגירה מספיק (0.5% שור בסרום חלבון, 0.1% Tween20 ב-PBS,) כדי לכסות את החלק שווה לסנן כולו בתא דגירה מבלי לגרום הצפת פעם את החותם מוחל.
- מקום מסגרת פוליאסטר לכסות על מסגרת הקאמרית הדגירה. לחץ כלפי מטה בחוזקה כדי לאטום את תא הדגירה. הימנע בועות אוויר מעל המסנן. דגירה לתאי חתום על 10 דקות בטמפרטורת החדר בחושך.
- הסר את החותם פוליאסטר לפתוח בתאי הדגירה. (הערה: פתיחת לתאי לפעמים ימשוך את חותם מסגרת משקופית במקרה זה, להכין חדר חדש עבור לשלבים הבאים..)
- Pipet מחוץ למאגר הדגירה מפינה אחת של החדר. יש להימנע ממגע עם המסנן.
- שטפו את המסנן על ידי בעדינות pipetting 100 PBS μl פנימהמתוך החדר 3 פעמים.
- הכן אנטי BrdU-FLUOS פתרון עובד ביצוע הפעולות המומלץ על ידי היצרן מיד לפני השימוש.
- Pipet 120 μl אנטי BrdU-FLUOS עובדים על פתרון הסינון, מקפיד באופן שווה לכסות את כל פני השטח.
- Reseal תא עם כיסוי פוליאסטר חדש. הימנע בועות אוויר על גבי המסנן. דגירה הקאמרית בחושך על 37 מעלות צלזיוס במשך 3 שעות. צעד זה יהיה תווית ה-DNA BrdU שולבו באתרו עם isothiocyanate והעמסת (FITC).
- הסר את כיסוי פוליאסטר לפתוח את תא הדגירה. Pipet את פתרון האנטי BrdU-FLUOS העבודה. שטפו את המסנן 3 פעמים עם PBS.
- העברת את המסנן מתא הדגירה על משטח סטרילי. פורסים את סעיף לסנן לחתיכות קטנות בעזרת סכין סטרילית.
- מעבירים את חתיכות לסנן לתוך 2 צינורות microcentrifuge מ"ל, שכל אחת מהן מכילה 1 מ"ל PBS. כובע בחוזקה את צינור וסוגרים עם parafilm. לדגור על 37 ° C ו 200 סל"ד במשך 10 דקות.
- אבטח את הצינורות על vortexer ו מערבולת במהירות מקסימלית במשך 5 דקות. Pipet supernatant לתוך צינור סטרילי Eppendorf 15 מ"ל. חזור על צעדים הדגירה מערבולת עבור פי 5 יותר. מערבבים את supernatant באותה 15 מ"ל Eppendorf צינור עבור כל דגימה. בדרך כלל, 80% תאים ניתן לשחזר ב ההשעיה.
- חנות supernatant עם תאים resuspended ב 4 ° C. מיון בתוך 2 ימים.
4. FACS ניתוח
הליך עבור cytometer BD תזרים FACSAria ותוכנה המקביל מתואר כאן.
- מטב את ההגדרה של cytometer תזרים השלבים הבאים המומלץ על ידי היצרן. זה כרוך: התאמת בקרה הגברה כדי להגדיר את הפרמטרים הנדרשים breakoff ואת הנקודה המתוקה: אופטימיזציה של עיכוב לייזר דרוג גורמי שטח הלחץ נדן ניסוי ואופטימיזציה הגדרות עבור FSC מתח SSC, סף FSC, דרוג FSC הקרינה, הקרינה מתח PMT וכו '
- הפעל דגימות בקרה שלילית על cytometer הזרימה (FCM) מבוסס על עוצמת הקרינה של פיזור FITC צד (SSC). הגדלת סף FITC עד לא התאים שולט שלילי ניתן דמיינו ידי להציג FITC-SSC הרכישה.
- הפעלה ללא תוספת דגימות מלאה לבחון את דפוס והפצה של 10,000 תאים בהתבסס על FITC ורכישת SSC. הגדרת שער לצרף את כל התאים לייעד אותם "תאים בעצימות נמוכה" (LIS).
- הפעל DOC-המתוקן דגימות ולבחון את דפוס והפצה של 10,000 תאים בהתבסס על FITC ו SSC. תאים מסוימים יופיעו השער LI מראש. הגדרת שער נוסף לצרף את התאים יש עוצמת הקרינה גבוהה יותר (שלו) מאשר ליס. רוכשת את הסטטיסטיקה כדי לראות את השפע היחסי של תאים מגודרת.
- מיין תאים HI לתוך צינורות אוסף המכיל 500 PBS μl ב "לטהר 1 טיפה" מצב. לסיים מיון כאשר מספר שלו מגיע 500,000 שחשוב.
5. סנן הגברה PCR של 16S rRNA גנים
סנן נהלים PCR משתנים מן Kirchman et al. 6
- תאים ממוינים סנן על לבן, 0.22 מיקרומטר, נקבוביות בגודל 25 מ"מ בקוטר, פוליקרבונט הממברנה הסינון. חתוך את קצה של מסנן שאינו מכיל תאים חיידקיים באמצעות להב סטרילי. פורסים את המסנן לתוך 8 חתיכות בגודל שווה.
- מניחים חתיכת מסנן יחיד לתוך צינור תגובת PCR, עם התאים הפונים פנימה של הצינור. הוסף 45 μl מים PCR כיתה לתוך הצינור תגובת PCR. את המסנן לחלוטין במים הצף.
- הוסף 2 רוש illustra PuReTaq קראו-To-Go חרוזי PCR לתוך הצינור תגובת PCR, בקצרה מערבולת. הוסף 2 μl אחד קדימה 16S rRNA הפוך גן primers (0.4 מיקרומטר הריכוז הסופי של כל פריימר), כגון 27F ו 1492R 7, לתוך כל אחד הצינורות תגובת PCR.
- (אופציונלי) הוסף פתרון 1μl שור אלבומין בסרום (BSA, הריכוז הסופי 30μg/100 μl) לתערובת התגובה PCR לעזור מעכבי הגברה לספוג. אם מישהו בוחר שלא להוסיף BSA, להוסיף 1μl של מים במקום.
- בצע הגברה PCR על Cycler תרמית. לנחות תוכנית PCR מומלץ, יש בו את הטמפרטורה חישול ברצף הפחתת 62-52 ° C על ידי 1 ° C לכל מחזור של 11 מחזורים, ואחריו 15 מחזורים עם חישול בטמפרטורה של 52 ° C. כל המחזורים כוללים denaturing (ב 95 ° C), חישול (ב 62-52 ° C), והסיומת (ב 72 ° C) מדרגות משך ה -50. Denaturation הראשונית של 3 דקות ו - השלב הסופי של 10 דקות הארכה נכלל גם בתוכנית PCR.
- אשר הגברה PCR על ידי אלקטרופורזה על ברומיד מוכתם ethidium 1% agarose ג'ל. והבלו amplicons PCR מן הג'ל ונקי עם ערכת חילוץ QIAquick ג'ל QIAGEN.
- לבצע שתי amplifications PCR נוספים מדגם זה, בכל פעם להשתמש בסעיף מסנן חדש. אחרי ה-PCR בג'ל purificatiעל, הבריכה amplicons המדגם אותו יחד. מטוהרים 16S amplicons rDNA מוכנים כעת במשך מספר ניתוחים מולקולריים, המאפשרים זיהוי טקסונומי, כגון בניית שיבוט ספריה וסדר.
6. נציג התוצאות:
תוצאות נציג מתוארים באמצעות לימוד putrescine-חיידקים מפרקי כדוגמה. דגימות מים נאספו מאתר החוף של גאורגיה מעובד בעקבות הליכים שתוארו לעיל. ניתוח FACS גילה כי בנוסף putrescine המושרה הפיתוח של קבוצת חיידקים עם עוצמת הקרינה גבוהה FITC, המציין שיעור גבוה BrdU ההתאגדות (איור 1). תאים אלה הוגדרו גבוהה BrdU-התאגדות תאים (שלו) היו אמורים להכיל בעיקר putrescine-משפיל חיידקים. היו חסרים ללא בנוסף שולטת, אשר הכיל רק תאים עם רמות נמוכות יותר של ההתאגדות BrdU (LIS). ליס היו אמורים להכיל bacterioplankton בעיקר כי לא היו מסוגלים להשתמש putrescine הוסיף. שלו היו מיון לתוך צינורות מופרדים ולאחר מכן נאסף על מסנני הממברנה. 16S rRNA amplicons גן התקבלו גבוהה בתאים באמצעות מסנן HI-PCR.
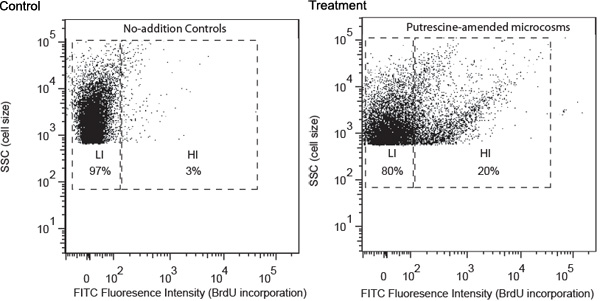
באיור 1. ניתוח תזרים cytometric של שליטה ללא תוספת מודל-תרכובת-המתוקן (putrescine כדוגמה כאן) דגימות שנאספו לאחר 24 שעות של דגירה. הפצה וניתוח Cell התבסס על עוצמה (1) הקרינה של FITC תיוג (ציר x), אשר קשורה באופן חיובי לרמת ההתאגדות BrdU, ו (2) פיזור צד (SSC, ציר y), אשר קשורה באופן חיובי לתא גודל. בסימון שער מתבססת על רמת ההתאגדות BrdU, (היי, גבוהה BrdU-ההתאגדות; LI, נמוך BrdU-ההתאגדות). האחוז היחסי של HI ו - LI תאים מוצגים שערי המקביל.
Discussion
זוגות הגישה שלנו ההתאגדות BrdU, FACS ו 16S ניתוח rDNA כדי לאפשר זיהוי ברמת המין של bacterioplankton כי לעכל רכיבים בודדים DOC בסביבות מימיים. Assay ההתאגדות BrdU תוויות תאים חיידקיים מבוסס על פעילות מטבולית, המאפשר ניתוח רק על חיידקים פעילים, ולכן אינה כוללת תאים רדומים. בגישה שלנו, ההתאגד?...
Disclosures
אין ניגודי אינטרסים הכריז.
Acknowledgements
מימון פרויקט זה סופק על ידי הקרן הלאומית למדע מענקים OCE1029607 (עד XM) ו MCB0702125 (אל MAM) ואת גורדון ובטי מור קרן (אל MAM).
Materials
| Name | Company | Catalog Number | Comments | |
| שם | סוג | חברה | מספר קטלוגי | תגובות |
| BrdU | מגיב | סיגמא | B5002-5G | |
| ליזוזים | מגיב | סיגמא | L6876-5G | |
| Proteinase K | מגיב | סיגמא | P2308-25mg | |
| באתרו התפשטות תאים קיט, FLUOS | ערכה | רוש | 11810740001 | לצרוך יותר של Anti-BrdU-FLUOS ו חיץ דגירה לכל תגובה מאשר הציע ידי היצרן. |
| מסגרות חותם דגירה צ'יימברס | חומר | Bio-Rad | SLF-1201 | |
| פוליקרבונט ממברנה מסננים (142 מ"מ, קוטר, 1.0 מיקרומטר הנקבוביות, בגודל) | חומר | Millipore | FALP14250 | |
| פוליקרבונט מסננים ממברנה (25 מ"מ, קוטר, 0.2 מיקרומטר-נקבוביות בגודל) | חומר | Millipore | FGLP02500 | |
| illustra PuReTaq Ready-To-Go-PCR חרוזים | ערכה | GE Healthcare | 27-9559-01 | |
| QIAquick ג'ל ערכת חילוץ | ערכה | QIAGEN | 28704 | |
| כשל מערכת ה-PCR | ערכה | EPICENTRE | FS99060 |
References
- Pernthaler, A., Pernthaler, J., Schattenhofer, M., Amann, R. Identification of DNA-synthesizing bacterial cells in coastal North Sea plankton. Appl. Environ. Microbiol. 68, 5728-5728 (2002).
- Urbach, E., Vergin, K. L., Giovannoni, S. J. Immunochemical detection and isolation of DNA from metabolically active bacteria. Appl. Environ. Microbiol. 65, 1207-12 (1999).
- Mou, X. Z., Hodson, R. E., Moran, M. A. Bacterioplankton assemblages transforming dissolved organic compounds in coastal seawater. Environ. Microbiol. 9, 2025-2025 (2007).
- Hodson, R. E., Dustman, W. A., Garg, R. P., Moran, M. A. In situ PCR for visualization of microscale distribution of specific genes and gene products in prokaryotic communities. Appl. Environ. Microbiol. 61, 4074-4074 (1995).
- Dinjens, W. N. Bromodeoxyuridine (BrdU) immunocytochemistry by exonuclease III (Exo III) digestion. Histochemistry. 98, 199-199 (1992).
- Kirchman, D. L., Yu, L. Y., Fuchs, B. M., Amann, R. Structure of bacterial communities in aquatic systems as revealed by filter PCR. Aquat. Microb. Ecol. 26, 13-13 (2001).
- Delong, E. F., Wickham, G. S., Pace, N. R. Phylogenetic stains: ribosomal RNA-based probes for the identification of single cells. Science. 243, 1360-1360 (1989).
- Artursson, V., Jansson, J. K. Use of bromodeoxyuridine immunocapture to identify active bacteria associated with arbuscular mycorrhizal hyphae. Appl. Environ. Microbiol. 69, 6208-6208 (2003).
- Mou, X. Z. Bacterial carbon processing by generalist species in the coastal ocean. Nature. 451, 708-708 (2008).
- Mou, X. Flow-cytometric cell sorting and subsequent molecular analyses for culture-independent identification of bacterioplankton involved in dimethylsulfoniopropionate transformations. Appl. Environ. Microbiol. 71, 1405-1405 (2005).
- Dean, F. B. Comprehensive human genome amplification using multiple displacement amplification. Proc. Natl. Acad. Sci. U.S.A. 99, 5261-5261 (2002).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved