このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ブロモデオキシウリジン(BrdU)標識と溶存有機の文化に依存しない識別に対する後続の蛍光活性化細胞選別カーボン分解Bacterioplankton
要約
環境bacterioplanktonは、有機炭素(DOC)化合物とDNA標識試薬、ブロモデオキシウリジン(BrdU)を溶解モデルとインキュベートされています。その後、DOC分解細胞は、蛍光活性化細胞選別(FACS)を使用して自身の昇格BrdUの取り込みに基づいてバルクコミュニティから分離されています。これらの細胞は、その後の分子生物学的解析によって識別されます。
要約
微生物は、水生環境における多数の溶存有機炭素(DOC)基質の分解を仲介する主要なエージェントです。しかし、自然の中でDOCの特定のプールに変換細菌の分類群の同定には、技術的な課題となっています。
ここでは、アプローチを説明しているカップルのブロモデオキシウリジン(BrdU)の取り込み、蛍光活性化細胞選別(FACS)、および16S水生環境において分解する特定のDOCの化合物の能力bacterioplanktonのカルチャに依存しない識別を可能にするrRNA遺伝子に基づく分子解析。三連のbacterioplanktonの小宇宙を、BrdUとモデルのDOCの化合物(DOC修正)、または唯一のBrdU(無添加制御)の両方を受け取るよう設定されております。 BrdUの代替は、新たに合成された細菌DNAおよびBrdU標識DNAのチミジンの位置は容易に免疫検出1,2できます。 24時間のインキュベーションを通じて、付加価値DOCの化合物を使用することができるbacterioplanktonが選択的に活性化し、それゆえにDOCの改正と細胞に非応答性細胞よりBrdU取り込み(HI細胞)のレベルが高いことが期待されているは、さらにコントロール(低BrdU取り込み細胞、LI細胞)。蛍光免疫検出した後、HIの細胞が区別され、物理的に蛍光活性化細胞選別(FACS)3でLI細胞から分離。ソートされたDOC -応答性細胞は(HI細胞)のDNAを抽出し、分類学的PCR、クローンのライブラリー構築と塩基配列決定を含め、その後の16S rRNA遺伝子ベースの解析によって識別されます。
プロトコル
1。水のサンプルの処理
- 大きな粒子とbacteriovoresを削除するには、1μmの孔径のメンブレンフィルターを通してフィルター10L環境水。カーボイの水ろ液を集める。
- 4ミリリットル、新たに調製したパラホルムアルデヒド溶液(、10%PFA)を含む3滅菌エッペンドルフチューブ(50ml)に移転36ミリリットルろ液それぞれ。細胞を維持するために、室温で2時間インキュベートする。真空濾過によって0.22μmの孔サイズの白いメンブレンフィルター上に細胞を集めます。真空濾過によってフィルターを介して生理食塩水(PBS)をリン酸緩衝10 mlを渡すことでフィルタを洗浄してください。ネガティブコントロールとしてフィルタにラベルを付けて、-20℃で保管します
ステップ1.3から1.4には、DOC -限られた条件を確立するためのオプションです。
- カーボイに無機態窒素とリン(5μMのNH 4 Clを、5μMのNaNO 3を 、及び1μMのNaH 2 PO 4、最終濃度)の混合物を追加。
- 時折扇動で48時間その場の温度で、暗所でインキュベートする。
2。縮図の設立とインキュベーション
- 小宇宙を確立するために、ステップ1.4のカーボイから800 mlの水のサンプルで6個の1リットルのガラスフラスコの各々を埋める。各縮図にBrdUを(10μM、最終濃度)を追加します。よく混ぜる。
- 小宇宙の三つに1 mlのモデルDOCの化合物溶液を追加し、これらは、DOCの改正として機能します。残りの3つの小宇宙に1 mlの滅菌PBSを追加、これらは無添加のコントロールとして機能します。
- インキュベーターシェーカーにすべての小宇宙をインキュベートし、100rpmで振とうしながらその場の温度で、暗所でインキュベートする。
- 0の時点で50 mlの滅菌エッペンドルフチューブ、8、16、および24時間にそれぞれの縮図と転送からの水のサンプルの36 mlを収集する。すぐにコレクションチューブに新鮮なPFAを4 ml(10%)を追加し、細胞を維持するために、室温で2時間インキュベートする。
- フィルタは、0.22μmのポアサイズのポリカーボネートメンブランフィルターを介して細胞を維持。 10ミリリットルのPBSでフィルターを洗う。次のステップに進んで、または-20℃でフィルターを保存する
3 BrdUの取り込みのためのその場免疫検出で
- 室温でフィルタ(DOC修正サンプル、無添加のコントロールとネガティブコントロール)を解凍。
- リゾチーム溶液1mlを適用する[10 100 mMトリス、50 nMのEDTAのmg / mlのリゾチーム卵白液(pH = 8)]フィルタで細菌細胞をカバーする4。室温で30分間インキュベートする。吸引下でそれまでの10 mlのPBSを渡すことでフィルタを洗浄してください。
- 4フィルタで細菌細胞をカバーするために[100 mMトリス、50 nMのEDTA(pHは= 8)で2 mg / mlのプロテイナーゼK]プロテイナーゼK溶液1mlを追加。室温で30分間インキュベートする。吸引下でそれまでの10 mlのPBSを渡すことでフィルタを洗浄してください。
- フィルター上の細菌細胞をカバーするために1ミリリットルエキソヌクレアーゼソリューション[5 mMのMgCl 2のエキソヌクレアーゼIII(50 U / ml)及び50mMのTris - HCl] 5を追加。 37℃で30分間。吸引下でそれまでの10 mlのPBSを渡すことでフィルタを洗浄してください。
- メーカーの指示に従って、フレームシールのインキュベーション室(Bio - Rad)を組み立てる。
- アルコール洗浄乾燥した表面に滅菌ブレードを使用してeighthsにフィルタをスライス。
- 鉗子を使用して、1組み立てたフレームシールインキュベーションチャンバー(8台のフレームシール室が、各フィルタのサンプルのために必要とされる)に、フィルタの第八節を置きます。細胞を含むフィルタセクションの側では上向きに直面しているはず。フィルターの奥の水分は、スライドにフィルタスティックをできるようになります。フィルタが乾燥した場合、スライドにフィルタセクションを配置する前に、チャンバーの中央にDIH 2 Oの小さなドロップ(2μl)を適用する。
ステップ3.8から3.20には、メーカーの指示から変更手順に従って、In Situ細胞増殖キットのロシュから試薬をFLUOS使用。 PBSを除き、すべての試薬 はキット内で提供されています。
- 均等にシールが適用されると、オーバーフローを引き起こすことなく、インキュベーション室内の全体のフィルタセクションをカバーするのに十分なインキュベーション緩衝液(0.5%ウシ血清アルブミン、PBS中の0.1%Tween20を、)を適用します。
- インキュベーションチャンバーのフレーム上にポリエステルのフレームカバーを置きます。しっかりとインキュベーションチャンバーを密封するために下に押します。フィルター上に気泡を避けてください。暗所で室温にて10分間密閉チャンバーをインキュベートする。
- ポリエステルシールを取り外して、インキュベーション室を開きます。 (注:時々チャンバーを開くには、スライドからのフレームシールをプルアップされますその場合は、次の手順のための新しいチャンバーを準備する。。)
- 室の一角からインキュベーションバッファー出力ピペット。フィルターとの接触を避ける。
- 優しくに100μlのPBSをピペッティングすることにより、フィルタを洗うとチャンバーからの3倍。
- 使用直前にメーカーが推奨する手順に従って、抗BrdU - FLUOSワーキング溶液を調製。
- ピペットで120μlの抗BrdU - FLUOSは、フィルター上に溶液を取り組んで均一に表面全体を覆うように注意してください。
- 新しいポリエステルカバーとチャンバーを再シール。フィルターの上に気泡を避けてください。 37℃暗所でチャンバーをインキュベート℃で3時間。このステップでは、フルオレセインイソチオシアネート(FITC) を用いたin situ でのBrdU法人のDNAにラベルを付けます。
- ポリエステルのカバーを取り外し、インキュベーションチャンバーを開きます。抗BrdU - FLUOSワーキングソリューションからピペット。 PBSでフィルタ3回洗浄する。
- インキュベーションチャンバーから滅菌表面にフィルターを転送する。滅菌ブレードを使用して小片にフィルタセクションをスライス。
- 2 mlのマイクロチューブにフィルター部分、それぞれを含む1mlのPBSを転送します。しっかりとパラフィルムとチューブとシールをキャップ。 37℃、10分間200rpmで。
- 5分間の最大速度でボルテックスし、渦の上にチューブを固定します。滅菌15ミリリットルのエッペンドルフチューブにピペットで上清を。さらに5回のためのインキュベーションとボルテックスの手順を繰り返します。各サンプルに対して同じ15エッペンドルフチューブに上清を組み合わせる。通常は、80%の細胞が懸濁液中に回収することができる。
- 4で再懸濁した細胞と上清℃にて保存してください。 2日以内にソート。
4。 FACS解析
BD FACSAriaフローサイトメーターと対応するソフトウェアのための手順を説明します。
- メーカーが推奨する手順に従って、フローサイトメーターの設定を最適化する。これが伴います:必要なブレークオフパラメータとスイートスポットを設定するには、増幅コントロールを調整し、実験のシース圧のためにレーザーの遅延時間と面積のスケーリング係数を最適化し、FSCとSSCの電圧、FSC閾値、FSC蛍光スケーリング、蛍光PMT電圧の設定を最適化、など
- FITCと側方散乱(SSC)の蛍光強度に基づいて、フローサイトメーター(FCM)のネガティブコントロールのサンプルを実行します。陰性対照には細胞はFITC - SSCの取得の表示により視覚化することができるようになるまで、FITCしきい値を増やします。
- 無添加のコントロールサンプルを実行し、FITCとSSCの買収に基づいて、10,000個の細胞の分布パターンを調べる。すべてのセルを囲むと、"低強度細胞"(LIS)としてそれを指定するためにゲートを定義します。
- DOC -修正サンプルを実行し、FITCとSSCに基づいて、10,000個の細胞の分布パターンを調べる。いくつかの細胞は、プリセットLIのゲートに表示されます。 LIはより高い蛍光強度を(HIS)を持つ細胞を囲むように別のゲートを定義します。ゲートセルの相対量を表示するには、統計情報を取得する。
- "1滴を浄化する"モードで500μlのPBSを含むコレクションチューブにソートHI細胞。 HISの数が500,000カウントに達したときにソートを終了する。
5。 16S rRNA遺伝子のフィルタPCR増幅
フィルタPCR手順はKirchmanらから変更されています。6
- 白、0.22μmの孔サイズ、25ミリ直径、ポリカーボネートメンブレンフィルター上にフィルターソートされた細胞。滅菌ブレードを使用していない細菌の細胞が含まれていないフィルターの端を切り落とす。 8等しい大きさにフィルターをスライス。
- 細胞がチューブの内側を向くように、PCR反応チューブに単一のフィルタピースを置きます。 PCRの反応チューブに45μlのPCRグレードの水を加えます。水に完全にフィルター水没。
- 2 ROCHE illustra PuReTaq読み取り- to - Goの、PCR反応チューブにPCRビーズを簡単にボルテックスを追加。 PCRの反応チューブのそれぞれに、2μlのよう27Fおよび1492R 7のような順方向と逆の16S rRNA遺伝子のプライマー(0.4μMの各プライマーの最終濃度)、のそれぞれを追加します。
- (オプション)吸着増幅阻害剤を助けるためにPCR反応液に1μlのウシ血清アルブミン溶液を(BSA、終濃度が30μg/100μL)を追加します。一BSAを追加しないことを選択した場合、水の代わりに、1μlを加える。
- サーマルサイクラーでのPCR増幅を行います。 PCRプログラムダウンタッチが推奨されて、これは順番に62から52に減少アニーリング温度を有し° Cを1 ° C 52のアニーリング温度で15サイクルが続く11サイクル、℃でのサイクルごとにすべてのサイクルは、(95℃)変性(62℃〜52℃)アニーリング、および拡張(72℃)50の持続時間のステップが含まれています。最初の3分の変性と最後の10分延長のステップはまた、PCRのプログラムに含まれています。
- エチジウムブロマイド染色した1%アガロースゲル電気泳動でPCR増幅を確認してください。 QIAGEN QIAquickゲル抽出キットでゲルとクリーンから消費税のPCRアンプリコンを。
- 各サンプルに2つの追加のPCR増幅を行う、するたびに新しいフィルタセクションを使用してください。 PCRゲルpurificati後で、一緒に同じサンプルのアンプリコンをプール。精製された16S rDNAのアンプリコンは、現在そのようなクローンのライブラリー構築と塩基配列決定などの分類学的同定を可能にする分子解析、多数のための準備が整いました。
6。代表的な結果:
代表的な結果は例としてプトレシン分解菌の研究を使用して記述されます。水試料は、グルジアの沿岸のサイトから収集し、上記の手順に従って処理した。 FACS分析は、プトレシン加えて、高BrdUの取り込み率(図1)を示す、高FITC蛍光強度を持つ細菌のグループの発展を誘導することを明らかにした。これらの細胞は、高のBrdU取り込み細胞(HIS)として指定され、主にプトレシン分解菌を含むことが期待された。彼は唯一のBrdUの取り込み(LIS)の低レベルを持つ細胞が含まれている無添加のコントロールに欠落していた。 LISは、主に追加さプトレシンを使用することができませんでしたbacterioplanktonを含むことが期待された。彼は、分離されたチューブに分類し、メンブレンフィルター上に採取された。 16S rRNA遺伝子のアンプリコンは、フィルタPCRを用いて、高HI細胞が得られた。
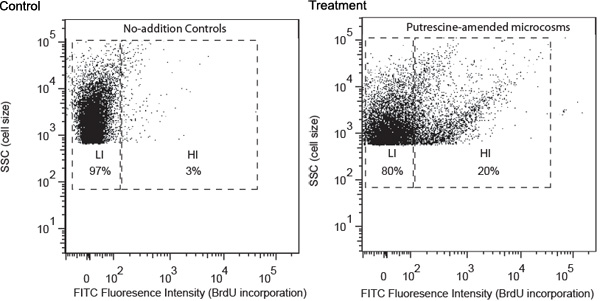
図1無添加制御とインキュベーションの24時間後に採取した(ここで例としてプトレシン)モデル化合物-改正されたサンプルのフローサイトメトリー分析。細胞の分布の分析は、積極的にBrdUの取り込みのレベルに関連してFITC標識(x軸)、の(1)蛍光強度に基づいていた、と(2)側方散乱(SSC、y軸)は、積極的に細胞に関連しているサイズ。ゲートの表記は、BrdUの取り込み、(; LI、低のBrdU取り込みHI、高のBrdU取り込み)のレベルに基づいています。 HIとLI細胞の相対的な割合は、対応するゲートに示されています。
ディスカッション
水生環境での個々のDOC成分を代謝するbacterioplanktonの種レベルの識別を可能にするために我々のアプローチのカップルBrdUの取り込み、FACSおよび16S rDNAの解析。のみ活性細菌の解析を可能にするため、休眠細胞が含まれていない代謝活動に基づいて、BrdU取り込みアッセイラベル細菌細胞、。我々のアプローチでは、BrdUの取り込みは、その後に可視化するBrdUの取り込みのレベルが異なる免?...
開示事項
利害の衝突は宣言されません。
謝辞
このプロジェクトの資金は、国立科学財団助成金OCE1029607(XMまで)とMCB0702125(MAMへ)とゴードンとベティムーア財団(MAMへ)によって提供されていました。
資料
| Name | Company | Catalog Number | Comments | |
| の名前 | タイプ | 会社 | カタログ番号 | コメント |
| BrdUを | 試薬 | シグマ | B5002 - 5G | |
| リゾチーム | 試薬 | シグマ | L6876 - 5G | |
| プロテイナーゼK | 試薬 | シグマ | P2308 - 25mgを | |
| In Situ細胞増殖キット、FLUOSで | キット | ロッシュ | 11810740001 | 抗BrdU - FLUOSと製造元が推奨する速度よりも反応あたりのインキュベーションバッファーをより多く消費する。 |
| フレームシールのインキュベーションチャンバー | 材料 | Bio - Rad社 | SLF - 1201 | |
| ポリカーボネート膜フィルター(142 - MM -直径、1.0μmの孔径) | 材料 | ミリポア | FALP14250 | |
| ポリカーボネート製メンブレンフィルター(25mmの直径、0.2μmの孔径) | 材料 | ミリポア | FGLP02500 | |
| illustra PuReTaqのReady - To - GoのPCRビーズ | キット | GEヘルスケア | 27-9559-01 | |
| QIAquickゲル抽出キット | キット | QIAGEN | 28704 | |
| FailSafeはPCRシステム | キット | EPICENTRE | FS99060 |
参考文献
- Pernthaler, A., Pernthaler, J., Schattenhofer, M., Amann, R. Identification of DNA-synthesizing bacterial cells in coastal North Sea plankton. Appl. Environ. Microbiol. 68, 5728-5728 (2002).
- Urbach, E., Vergin, K. L., Giovannoni, S. J. Immunochemical detection and isolation of DNA from metabolically active bacteria. Appl. Environ. Microbiol. 65, 1207-12 (1999).
- Mou, X. Z., Hodson, R. E., Moran, M. A. Bacterioplankton assemblages transforming dissolved organic compounds in coastal seawater. Environ. Microbiol. 9, 2025-2025 (2007).
- Hodson, R. E., Dustman, W. A., Garg, R. P., Moran, M. A. In situ PCR for visualization of microscale distribution of specific genes and gene products in prokaryotic communities. Appl. Environ. Microbiol. 61, 4074-4074 (1995).
- Dinjens, W. N. Bromodeoxyuridine (BrdU) immunocytochemistry by exonuclease III (Exo III) digestion. Histochemistry. 98, 199-199 (1992).
- Kirchman, D. L., Yu, L. Y., Fuchs, B. M., Amann, R. Structure of bacterial communities in aquatic systems as revealed by filter PCR. Aquat. Microb. Ecol. 26, 13-13 (2001).
- Delong, E. F., Wickham, G. S., Pace, N. R. Phylogenetic stains: ribosomal RNA-based probes for the identification of single cells. Science. 243, 1360-1360 (1989).
- Artursson, V., Jansson, J. K. Use of bromodeoxyuridine immunocapture to identify active bacteria associated with arbuscular mycorrhizal hyphae. Appl. Environ. Microbiol. 69, 6208-6208 (2003).
- Mou, X. Z. Bacterial carbon processing by generalist species in the coastal ocean. Nature. 451, 708-708 (2008).
- Mou, X. Flow-cytometric cell sorting and subsequent molecular analyses for culture-independent identification of bacterioplankton involved in dimethylsulfoniopropionate transformations. Appl. Environ. Microbiol. 71, 1405-1405 (2005).
- Dean, F. B. Comprehensive human genome amplification using multiple displacement amplification. Proc. Natl. Acad. Sci. U.S.A. 99, 5261-5261 (2002).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved