Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Bromodésoxyuridine (BrdU) l'étiquetage et ultérieures de tri cellulaire activé par fluorescence de la culture indépendante Identification de carbone organique dissous dégradants bactérioplancton
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Bactérioplancton environnement sont incubés avec un modèle de carbone organique dissous (DOC) composé et un réactif de marquage de l'ADN, bromodésoxyuridine (BrdU). Ensuite, DOC-dégradants cellules sont séparées de la communauté en vrac en fonction de leur incorporation de BrdU élevée en utilisant la fluorescence tri cellulaire (FACS). Ces cellules sont ensuite identifiés par des analyses moléculaires ultérieures.
Résumé
Les microbes sont les principaux agents de médiation de la dégradation de nombreuses carbone organique dissous (DOC) des substrats dans les milieux aquatiques. Toutefois, l'identification des taxons bactériens qui transforment les piscines spécifiques de COD dans la nature pose un défi technique.
Nous décrivons ici une approche qui couple bromodésoxyuridine (BrdU) l'incorporation, tri cellulaire par fluorescence (FACS), et ARNr 16S basée sur l'analyse moléculaire qui permet l'identification indépendante de la culture du bactérioplancton capables de dégrader un composé DOC spécifiques dans les milieux aquatiques. Microcosmes bactérioplancton en triple sont mis en place pour recevoir les deux BrdU et un composé modèle de DOC (DOC amendements), ou seulement BrdU (sans ajout de contrôle). Substituts BrdU les positions de la thymidine dans l'ADN bactérien nouvellement synthétisé et BrdU marqué l'ADN peut être facilement immunodétectée 1,2. Grâce à une incubation de 24 h, bactérioplancton qui sont capables d'utiliser le composé ajouté DOC devraient être activés sélectivement, et ont donc des niveaux plus élevés d'incorporation de BrdU (cellules HI) que les non-réactive les cellules dans les modifications du DOC et les cellules dans un no- Outre les contrôles (faible incorporation de BrdU cellules, les cellules LI). Après immunodétection de fluorescence, les cellules HI sont distingués et physiquement séparé des cellules LI par tri cellulaire activé par fluorescence (FACS) 3. Tri DOC-réactive (cellules HI) sont extraits de l'ADN et taxinomique identifiés par 16S ARNr ultérieure basée sur des analyses, y compris la PCR, la construction de bibliothèques clone et le séquençage.
Protocole
1. Traitement des échantillons d'eau
- Filtrer l'eau 10L de l'environnement grâce à une membrane filtrante à pores um de taille pour enlever les grosses particules et bacteriovores. Recueillir le filtrat de l'eau dans une bonbonne.
- Transfert de 36 ml de chaque filtrat dans 3 tubes Eppendorf stériles (50 ml) contenant 4 ml solution fraîchement préparée de paraformaldéhyde (PFA; 10%). Incuber pendant 2 heures à température ambiante pour préserver les cellules. Recueillir cellules sur 0,22 um de-la taille des pores des filtres à membrane blanche par filtration sous vide. Lavez le filtre en faisant passer 10 ml de tampon phosphate salin (PBS) à travers le filtre par filtration sous vide. Étiqueter les filtres de contrôles négatifs et de les stocker à -20 ° C.
Étapes 1.3 à 1.4 sont facultatives pour établir DOC-limitée conditions.
- Ajouter un mélange d'azote et de phosphore inorganiques (5 uM NH 4 Cl, 5 uM NaNO 3, et 1 uM NaH 2 PO 4 de concentration finale) dans la tourie.
- Incuber dans l'obscurité à température in situ pendant 48 heures avec agitation occasionnelle.
2. Microcosme création et l'incubation
- Remplissez chacune des six flacons de verre de 1 L avec 800 ml d'eau de l'échantillon de la bonbonne de l'étape 1.4 pour établir des microcosmes. Ajouter BrdU (10 uM, concentration finale) de chaque microcosme. Mélangez bien.
- Ajouter 1 ml de modèle composé solution DOC dans trois des microcosmes, ces servira modifications DOC. Ajouter 1 ml de PBS stérile dans les trois restantes microcosmes, ces servira sans plus de contrôle.
- Incuber tous les microcosmes dans un agitateur incubateur et incuber dans l'obscurité à température in situ tout en agitant à 100 rpm.
- Recueillir 36 ml d'échantillon d'eau de chaque microcosme et transférer dans des tubes de 50 ml Eppendorf stériles à des points de temps de 0, 8, 16 et 24 heures. Immédiatement ajouter 4 ml de frais PFA (10%) pour des tubes de prélèvement et incuber pendant 2 heures à température ambiante pour préserver les cellules.
- Filtre préservé cellules par 0,22 um-la taille des pores des filtres à membrane en polycarbonate. Laver les filtres avec 10 ml de PBS. Procéder immédiatement à l'étape suivante ou stocker les filtres à -20 ° C.
3. En immunodétection in situ pour l'incorporation de BrdU
- Décongeler les filtres (échantillons DOC modifiée, sans plus de contrôle et les contrôles négatifs) à température ambiante.
- Appliquer 1 ml de solution de lysozyme [10 mg / ml de lysozyme de blanc d'œuf dans 100 mM Tris, 50 nM EDTA (pH = 8)] 4 pour couvrir les cellules bactériennes sur le filtre. Incuber à température ambiante pendant 30 minutes. Lavez le filtre en faisant passer 10 ml de PBS à travers elle sous aspiration.
- Ajouter 1 ml de solution de protéinase K [2 mg / ml de protéinase K dans 100 mM Tris, 50 nM EDTA (pH = 8)] 4 pour couvrir les cellules bactériennes sur le filtre. Incuber à température ambiante pendant 30 minutes. Lavez le filtre en faisant passer 10 ml de PBS à travers elle sous aspiration.
- Ajouter une solution de exonucléase ml [exonucléase III (50 U / ml) dans 5 mM de MgCl2 et 50 mM Tris-HCl] 5 pour couvrir les cellules bactériennes sur le filtre. Incuber à 37 ° C pendant 30 minutes. Lavez le filtre en faisant passer 10 ml de PBS à travers elle sous aspiration.
- Assemblez chambres d'incubation cadre joint (Bio-Rad) selon les instructions du fabricant.
- Trancher le filtre en huitièmes à l'aide d'une lame stérile sur une consommation d'alcool nettoyé la surface sèche.
- En utilisant des pinces, placer une huitième section d'un filtre dans une chambre d'incubation assemblés cadre joint (huit cadres joint chambres sont nécessaires pour chaque échantillon de filtre). Le côté de la section de filtre qui contient des cellules doit faire face à la hausse. L'humidité à l'arrière du filtre permet le bâton de filtre à la diapositive. Si le filtre devient sèche, appliquer une petite goutte (2 pi) du dih 2 O dans le centre de la chambre avant de placer la section du filtre sur la diapositive.
Étapes 03.08 à 03.20 utilisent les réactifs de la ROCHE En Kit prolifération cellulaire in situ, fluos en suivant les procédures de modification des instructions du fabricant. Sauf pour PBS, tous les réactifs sont fournis dans le kit.
- Appliquer suffisamment de tampon d'incubation (0,5% d'albumine sérique bovine, 0,1% de Tween 20 dans du PBS), pour bien couvrir toute la section de filtre dans la chambre d'incubation sans causer de débordement fois le sceau est appliqué.
- Placez un couvercle en polyester cadre sur le cadre de la chambre d'incubation. Appuyez pour bien sceller la chambre d'incubation. Eviter les bulles d'air au-dessus du filtre. Incuber les chambres scellées pendant 10 min à température ambiante dans l'obscurité.
- Retirer le joint de polyester et d'ouvrir des chambres d'incubation. (Remarque: l'ouverture des chambres, parfois se tirer vers le haut le sceau cadre de la diapositive Dans ce cas, de préparer une nouvelle chambre pour les étapes suivantes..)
- Pipeter le tampon d'incubation d'un coin de la chambre. Eviter le contact avec le filtre.
- Lavez le filtre en douceur de pipetage 100 ul de PBS dans ethors de la chambre 3 fois.
- Préparer anti-BrdU-fluos solution de travail en suivant les étapes recommandées par le fabricant immédiatement avant utilisation.
- Pipeter 120 pi anti-BrdU-fluos solution de travail sur le filtre, en prenant soin de façon uniforme couvrir toute la surface.
- Refermer la chambre avec une housse de polyester neuf. Eviter les bulles d'air sur le dessus du filtre. Incuber la chambre dans l'obscurité à 37 ° C pendant 3 heures. Cette étape sera l'étiquette BrdU incorporé l'ADN in situ avec de l'isothiocyanate de fluorescéine (FITC).
- Retirez le couvercle en polyester et d'ouvrir la chambre d'incubation. Pipeter la solution anti-BrdU-fluos de travail. Lavez le filtre 3 fois avec du PBS.
- Transférer le filtre de la chambre d'incubation d'une surface stérile. Trancher la section filtre en petits morceaux à l'aide d'une lame stérile.
- Transférer les morceaux de filtre en 2 microtubes ml, contenant chacun 1 ml de PBS. Étroitement hermétiquement le tube et le joint avec du parafilm. Incuber à 37 ° C et 200 rpm pendant 10 min.
- Fixez les tubes sur un Vortexer et le vortex à la vitesse maximale pendant 5 minutes. Déposer le surnageant dans un tube stérile de 15 ml Eppendorf. Répétez les étapes d'incubation et de vortex pour 5 fois plus. Combinez le surnageant dans le même tube de 15 ml Eppendorf pour chaque échantillon. Typiquement, 80% de cellules peuvent être récupérés dans la suspension.
- Stocker le surnageant de cellules en suspension à 4 ° C. Trier dans les 2 jours.
4. Analyse FACS
Une procédure pour un cytomètre de flux BD FACSAria et du logiciel correspondant est décrit ici.
- Optimiser le réglage du débit étapes suivantes cytomètre recommandé par le fabricant. Cela implique: le réglage du contrôle d'amplification de définir les paramètres requis et breakoff le sweet spot, optimisation du délai laser et zone de facteurs d'échelle pour la pression de la gaine expérimenter et optimiser les réglages pour le FSC et SSC tensions, seuil FSC, FSC de fluorescence d'échelle, la fluorescence tensions PMT , etc
- Exécuter des échantillons de contrôle négatif sur le cytomètre en flux (FCM) en fonction de l'intensité de fluorescence du FITC et dispersent de côté (SSC). Augmenter le seuil FITC jusqu'au pas de cellules dans les contrôles négatifs peuvent être visualisés par l'affichage d'acquisition FITC-SSC.
- Exécuter des échantillons de contrôle sans plus et d'examiner le mode de distribution de 10 000 cellules en fonction de FITC et l'acquisition de la SSC. Définir une porte pour enfermer toutes les cellules et de les désigner comme des «cellules de basse intensité» (LIS).
- Exécuter DOC-amendé échantillons et d'examiner le modèle de distribution de 10.000 cellules basées sur FITC et SSC. Certaines cellules apparaissent dans la grille prédéfinie LI. Définir une autre porte pour enfermer les cellules qui ont une plus grande intensité de fluorescence (HIS) que Lis. Acquérir les statistiques pour voir l'abondance relative des cellules fermée.
- Trier les cellules dans des tubes HI collection contenant 500 ul de PBS à «purifier 1 goutte" mode. Terminez le tri lorsque le nombre de ses atteint 500 000 chefs d'accusation.
5. Filtre amplification par PCR des gènes ARNr 16S
Filtre les procédures de PCR sont modifiées de Kirchman et al. 6
- Filtre cellules triées sur un blanc, 0,22 um-pores de taille, 25 mm de diamètre, membrane en polycarbonate filtre. Coupez le bord du filtre qui ne contient pas de cellules bactériennes en utilisant une lame stérile. Trancher le filtre en 8 morceaux égaux de taille.
- Placez un seul morceau de filtre dans un tube de réaction PCR, avec les cellules vers l'intérieur du tube. Ajouter de l'eau 45 ul de grade PCR dans le tube de réaction PCR. Plonger le filtre entièrement dans l'eau.
- Ajouter 2 ROCHE illustra PuReTaq Read-To-Go Perles PCR dans le tube de réaction PCR, brièvement au vortex. Ajouter 2 ul de chacune des avant et arrière du gène ARNr 16S amorces (0,4 uM concentration finale pour chaque amorce), tels que 27F et 1492R 7, dans chacun des tubes de réaction PCR.
- (Facultatif) Ajoutez 1 microlitre bovins solution de sérum albumine (BSA, la concentration finale est 30μg/100 ul) au mélange réactionnel de PCR pour aider à l'amplification des inhibiteurs de s'adsorber. Si l'on choisit de ne pas ajouter de BSA, ajouter 1 microlitre d'eau à la place.
- Effectuer l'amplification par PCR sur un cycleur thermique. Une touche bas programme de PCR est recommandée, qui a la température de recuit séquentielle diminuant de 62 à 52 ° C par 1 ° C par cycle de 11 cycles, suivie par 15 cycles avec une température de recuit de 52 ° C. Tous les cycles comprennent dénaturant (à 95 ° C), le recuit (à 62 à 52 ° C), et l'extension (à 72 ° C) les étapes d'une durée de 50 ans. Un initiale de 3 min de dénaturation et dernière étape d'extension de 10 min est également inclus dans le programme de PCR.
- Confirmer l'amplification par PCR par électrophorèse sur un bromure d'éthidium teinté gel à 1% d'agarose. Accise amplicons PCR à partir du gel et de nettoyer avec le kit QIAGEN QIAquick gel extraction.
- Effectuer deux autres amplifications PCR pour chaque échantillon, chaque fois que l'utilisation d'une nouvelle section de filtre. Après un gel de PCR Purificatile, la piscine des amplicons du même échantillon ensemble. Purifié 16S ADNr amplicons sont maintenant prêtes pour un certain nombre d'analyses moléculaires qui permettent l'identification taxinomique, tels que la construction de bibliothèques clone et le séquençage.
6. Les résultats représentatifs:
Les résultats représentatifs sont décrits à l'aide d'une étude de putrescine bactéries dégradant comme un exemple. Des échantillons d'eau ont été recueillies à partir d'un site côtier de la Géorgie et traitées suivant les procédures décrites ci-dessus. Analyse FACS a révélé que plus de putrescine induit le développement d'un groupe de bactéries à haute intensité de fluorescence FITC, indiquant un taux élevé de l'incorporation de BrdU (Figure 1). Ces cellules ont été désignés comme étant à haut BrdU l'incorporation des cellules (HIS) et devaient contiennent principalement putrescine bactéries dégradant. HIS sont manquants dans les commandes sans plus, qui ne contenait que des cellules avec des niveaux inférieurs de l'incorporation de BrdU (LIS). Lis ont été s'attend à ce que contiennent principalement bactérioplancton ont été incapables d'utiliser la putrescine ajouté. HIS sont triés dans des tubes séparés et ensuite recueillis sur des filtres à membrane. Amplicons du gène 16S ARNr ont été obtenus pour les cellules en utilisant un filtre haute HI PCR.
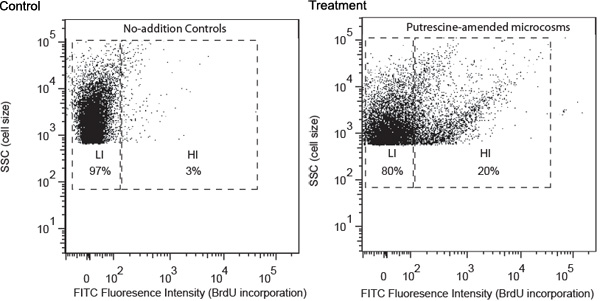
Figure 1. Cytométrie de flux sans plus de contrôle et de modèle composé modifiée (putrescine comme un exemple ici) échantillons prélevés après 24 h d'incubation. Analyse de la distribution cellulaire a été basée sur (1) l'intensité de fluorescence du FITC étiquetage (axe x), ce qui est positivement liée au niveau d'incorporation de BrdU, et (2) la diffusion latérale (SSC, axe des y), ce qui est positivement liée à la cellule taille. Notations Gate est basée sur le niveau d'incorporation de BrdU, (HI, haute-BrdU constitution; LI, faible BrdU constitution). Le pourcentage relatif de cellules HI et LI sont présentés dans les portes correspondantes.
Discussion
Notre approche de couples BrdU constitution, FACS et 16S ADNr pour permettre l'analyse des espèces au niveau d'identification du bactérioplancton qui métabolisent les composants individuels du DOC dans les milieux aquatiques. Le dosage de l'incorporation de BrdU étiquettes des cellules bactériennes en fonction des activités métaboliques, ce qui permet l'analyse uniquement sur les bactéries actives et ne comprennent donc pas des cellules dormantes. Dans notre approche, l'incorporation de BrdU ...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Le financement de ce projet a été fournie par la National Science Foundation accorde OCE1029607 (XM) et MCB0702125 (au MAM) et la Gordon and Betty Moore Foundation (au MAM).
matériels
| Name | Company | Catalog Number | Comments | |
| Nom | Tapez | Société | Le numéro de catalogue | Commentaires |
| BrdU | Réactifs | Sigma | B5002-5G | |
| Le lysozyme | Réactifs | Sigma | L6876-5G | |
| Protéinase K | Réactifs | Sigma | P2308-25MG | |
| In Situ prolifération cellulaire Kit, fluos | Kit | Roche | 11810740001 | Consommer plus de l'Anti-BrdU-fluos et le tampon d'incubation par réaction que suggéré par le fabricant. |
| Chambres d'incubation Cadre-Seal | Matériau | Bio-Rad | FSL-1201 | |
| Polycarbonate filtres à membranes (142 mm de diamètre, 1,0 um-la taille des pores) | Matériau | Millipore | FALP14250 | |
| Filtres à membrane en polycarbonate (25 mm de diamètre, de 0,2 um-la taille des pores) | Matériau | Millipore | FGLP02500 | |
| illustra PuReTaq Ready-To-Go PCR Perles | Kit | GE Healthcare | 27-9559-01 | |
| QIAquick gel extraction kit | Kit | QIAGEN | 28704 | |
| FailSafe PCR System | Kit | EPICENTRE | FS99060 |
Références
- Pernthaler, A., Pernthaler, J., Schattenhofer, M., Amann, R. Identification of DNA-synthesizing bacterial cells in coastal North Sea plankton. Appl. Environ. Microbiol. 68, 5728-5728 (2002).
- Urbach, E., Vergin, K. L., Giovannoni, S. J. Immunochemical detection and isolation of DNA from metabolically active bacteria. Appl. Environ. Microbiol. 65, 1207-12 (1999).
- Mou, X. Z., Hodson, R. E., Moran, M. A. Bacterioplankton assemblages transforming dissolved organic compounds in coastal seawater. Environ. Microbiol. 9, 2025-2025 (2007).
- Hodson, R. E., Dustman, W. A., Garg, R. P., Moran, M. A. In situ PCR for visualization of microscale distribution of specific genes and gene products in prokaryotic communities. Appl. Environ. Microbiol. 61, 4074-4074 (1995).
- Dinjens, W. N. Bromodeoxyuridine (BrdU) immunocytochemistry by exonuclease III (Exo III) digestion. Histochemistry. 98, 199-199 (1992).
- Kirchman, D. L., Yu, L. Y., Fuchs, B. M., Amann, R. Structure of bacterial communities in aquatic systems as revealed by filter PCR. Aquat. Microb. Ecol. 26, 13-13 (2001).
- Delong, E. F., Wickham, G. S., Pace, N. R. Phylogenetic stains: ribosomal RNA-based probes for the identification of single cells. Science. 243, 1360-1360 (1989).
- Artursson, V., Jansson, J. K. Use of bromodeoxyuridine immunocapture to identify active bacteria associated with arbuscular mycorrhizal hyphae. Appl. Environ. Microbiol. 69, 6208-6208 (2003).
- Mou, X. Z. Bacterial carbon processing by generalist species in the coastal ocean. Nature. 451, 708-708 (2008).
- Mou, X. Flow-cytometric cell sorting and subsequent molecular analyses for culture-independent identification of bacterioplankton involved in dimethylsulfoniopropionate transformations. Appl. Environ. Microbiol. 71, 1405-1405 (2005).
- Dean, F. B. Comprehensive human genome amplification using multiple displacement amplification. Proc. Natl. Acad. Sci. U.S.A. 99, 5261-5261 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon