Method Article
药理和功能的遗传分析处理涡虫的再生日本三角涡虫
摘要
在一个活的动物研究干细胞分化的一个有吸引力的模式是涡虫扁虫。再生是由简单的截肢手术,很容易在一个基本的实验室进行的实验研究和药理学和基因(适合在体内 RNAi)的操纵,在这篇文章中的协议详细。
摘要
自由生活涡虫扁虫有一个历史悠久的实验使用,因为他们非凡的再生能力 1 。从这些动物切除的小片断,改革原体计划,缺少的车身结构的再生。例如,如果一个“主干”的片段是从一个完整的蠕虫切割,一个新的“头”将再生前方一个“尾巴”后方将重新恢复了原有的“头部到尾部的车身结构的极性截肢前(图1A)。
涡虫被称为“neoblasts'〜30种不同类型的细胞分化,在正常的身体动态平衡和执行组织再生的干细胞,是由再生。这种再生过程是可靠和容易证明。由于几个创业实验室,许多工具和功能基因的方法的奉献精神,现在已经为这个模型系统进行了优化。因此,相当最近已经取得了进展,在理解和操作基础 2-9涡虫的发育可塑性的分子事件。
涡模型系统将会以一个科学家的广泛利益。神经学家,模型给予机会研究整个神经系统的再生,而不是简单的再生/修复单个神经细胞的过程中,通常是在许多已建立的模型研究的重点。涡虫表达的神经递质10过多,代表的一项重要制度,为研究中枢神经系统的11,12的演变,行为甄别潜在的13,14 。
再生的结果是经得起药理和遗传apparoaches操纵。例如,可以进行筛选药物对再生的影响,只需放置在身体片段的药物含截肢后在不同时间点的解决方案。使用击倒方法( 在体内的RNAi),这可以实现,通过显微注射周期或喂食细菌表达双链RNA的结构8,9,15,可以研究单个基因的作用。这两种方法都可以产生高外显率,例如,双极动物16-21再生的视觉冲击的表型。为了便于采用这种模式和实施这种方法,我们将展示使用涡虫日本三角涡虫的药理学和基因检测(在体内RNAi技术通过喂食)的视频在这条协议。
研究方案
1。中继片段再生实验
- 再生实验前至少5天停喂的蠕虫病毒的队列,以确保动物废物和摄取食物(30〜完整的蠕虫,每个蠕虫〜8-10毫米长的饥饿后)的。
- 在检测的一天,冲洗与水预先冷冻的平整的冰盘,并用保鲜膜覆盖平的冰面。使用镊子,一个滤纸上的保鲜膜。
- 几滴泉水浸湿的滤纸。转移到过滤器使用的移液管(<20%过滤蠕虫)涡虫。蠕虫过滤器,如果有必要可以重新定位,通过与更多的泉水repipetting。删除多余的液体。
- 使用手术刀,截肢动物的前尖,咽( 图1A)前结束之间约中途一个单切头。
- 使用手术刀,截去尾部区域,由一个单一的削减约中间动物的尾部和后咽( 图 1A)结束。删除多余的粘液,用手术刀切割后蘸用70%乙醇的纸巾。用手术刀多余的乙醇。
- 转移到培养皿中(100 x 25毫米深)通过含有泉水钳过滤器。等待〜3分钟(伤口缝合,观察一个捏和伤口变黑)。
- 冲洗干净,滤纸的Petri菜含有所需再生法和转移到恒温培养箱(24℃)介质的主干片段。
- 分数再生表型〜1周后,当再生结构识别( 图 1A) 。
2。吡喹酮诱导再生:两极药理操纵
- 准备溶药物在DMSO(62.4mg 1ML PZQ 200MM股票)吡喹酮(PZQ)的股票。当天使用。
- 设为50毫升含药溶液中缓冲Montjuch盐(PZQ 90微米)。涡〜1分钟,以确保分散。
- 执行主干片段[#1],露出的树干碎片队列的最后一步[1.7] PZQ协议的详细以上的再生法。
- 24-48小时后,交换PZQ含有温泉水的媒体,洗衣机蠕虫(> 3)次。
- 返回蠕虫孵化器。分数两极(两个头, 图1B)切割后的一周。
3。再生的基因操作:在体内RNAi的喂养协议
- 检测前,准备鸡肝匀浆。丢弃从foodstock脂肪,黄颜色的部分,菜泥其余肝。过滤菜泥通过滤网和存储等分悬浮液(1.5ml离心管,-20℃)。
- 检测之前,准备aliquoting到1ML样品供应,储存于-20 ° C牛的红血细胞(红细胞)
- 选择用于RNAi的蠕虫病毒。三个同伙:感兴趣的基因,阳性对照组(基因与已知的RNA干扰表型;图1C,D和 E)和负控制(无RNAi的表型的基因,也有用的评估击倒水平相比,实验队列)。对于每个队列,使用〜250的蠕虫(〜8-10毫米长至少5天的饥饿后)。存放在塑料桶泉水。
- 对于每个RNAi的队列中,大肠杆菌表达dsRNA的目标感兴趣的基因大肠杆菌解冻股票[8],随着鸡肝匀浆管[3.1]和红细胞管[3.2]。
- 佩莱细菌(13000克,1分钟)。移除上清液2xYT媒体(〜700μl)和重新悬浮颗粒。 Recentrifuge,在冰上,丢弃上清液,沉淀的地方。
- 创建大缸径P200的枪头,切断提示结束。在鸡肝匀浆(150μL)和红细胞(50μL)的混合物创建RNAi的喂养混合彻底重悬细菌沉淀。取出离心中的气泡。
- 觅食,删除包含蠕虫的塑料浴盆的水多数(离开〜1英寸深入)。涡流浴缸集中在这个容器中的中心的蠕虫,和吸管RNAi在一个网罗蠕虫圈饲养的组合。请使用移液管,仔细同轴电缆上的RNAi喂养混合逃出,没有其他食客的干扰!
- 喂奶后(约1小时),确定,美联储以及涡虫。这些蠕虫从摄入的红细胞深红色着色。丢弃他人。
- 小心地更换新鲜的泉水喂养的解决方案,最大限度地减少干扰,防止食物的排泄。要做到这一点,轻轻地本地化蠕虫使用移液管的管的一侧,倒入水混浊,并仔细地用清水取代倾盆而下的浴缸对面的墙上。长时间暴露在空气Resubmerge涡虫很快也会导致食物排泄。
- 松散SE铝容器的盖子,并返回到培养箱(24℃)
- 重复RNAi在多个周期中喂养协议(〜2-3天),穿插协议#1]屏幕RNAi的表型再生周期。许多基因的有效供料和再生的一个标准协议显示在图2〜1个月,总工期。
修改这个计划,针对不同的基因作为最佳的协议将取决于mRNA的稳定性,组织定位,蛋白质perdurance,或排除多个喂养周期后再生一个表型的发展等因素。评估只是表型的显性筛选(明显),或定量PCR方法,有针对性的mRNA水平与阴性对照队列击倒水平。
4。代表性的成果:
主干片段再生实验是健壮的所有蠕虫应重新正常前后(“头到尾”)极性。这可以切割后的5天,前眼点(asterixed图1A)的出现,促成尽快拿下。然而,一周后完成恢复失去结构的形态发生。药理操纵主干片段再生PZQ产量双头蠕虫( 图1B)也很强大(欧共体50 = 87(+ - )11%的双极片段,70μM为48 小时 20 PZQ) 。两极发生在一个单一的再生周期。下在这些实验的疗效可能与药物的溶解度和/或存储,或使用不同的扁虫种PZQ是无效的问题。对于外显率较 低的药物,通常用于anteriorization指数得分从 22完整的两极的中间表型搁置。表型从积极的控制结构的RNAi, 如图1所示:RNAi 的日本三角涡虫6 - 1(D J六个- 1)的结果,在无眼表型23( 图 1C),日本三角涡虫的PC2的RNAi(DJ - PC2)在损失厌恶的光响应24( 图1D)和RNAi的结果, 日本三角涡虫βcatenin- 1(βcateninDJ - 1)的结果在两个为首的表型16-19日,从主干片段( 图1E)21。这些表型,可以实现用下面这个简单的RNA干扰协议:DJ -六(FFxFx),DJ - PC2(FFFx),DJ -βcatenin- 1(FFxFx),其中F =饲养周期和x =分别再生周期。
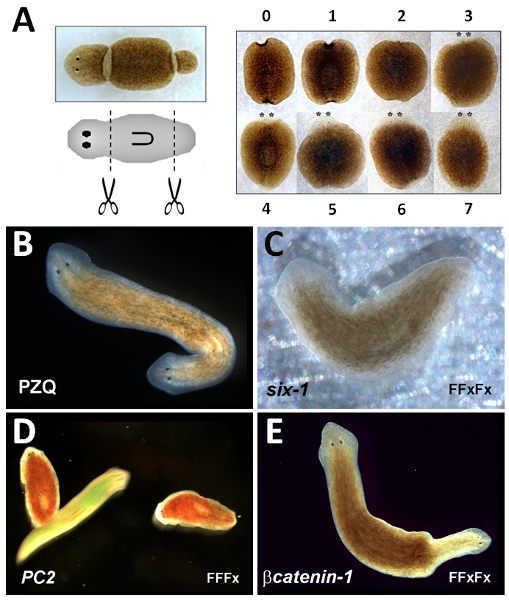
图1:主干片段再生检测涡虫(上)和原理图(中) 左 ,形象地展现切除主干片段的位置。右键,躯干片段再生timecourse显示外观再生片段在指定的时间(天)乙PZQ曝光产生。双头涡虫的图像C“无眼”DJ -六- 1 RNA干扰,D动的DJ - PC2的RNAi蠕虫(染成红色)轻厌恶反应,亏损相比,移动控制蠕虫(染色产生的蠕虫绿色),E双极型涡虫的DJ -βcatenin1的RNAi 。在Be,原来的RNAi蠕虫前到底是面向左侧。
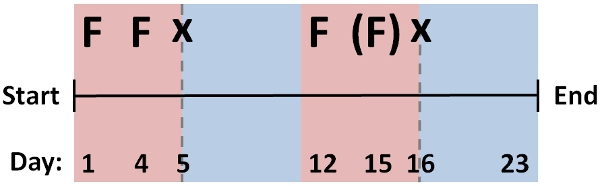
图2:典型RNAi的协议包括多个喂养(F)和再生周期(X)是有效的 D.许多基因敲除喂养和切割周期序列粳稻 。本议定书的个体基因可能需要修改,以产生最佳的结果,使用例如一个较少(括号)或更大数量的喂养和再生周期。
讨论
这里描述协议详细分析,研究和操纵涡虫三角涡虫再生的粳稻。他们很简单,不需要专用设备等,他们可以很容易地在实验室或教室进行。分析可以单独进行,或合并化学药物药效在体内的基因筛查,可以在候选基因水平上进行,或适应无偏见的,高通量筛选 8 。无论是耐人寻味的涡虫的生物学研究自己的权利,或在体内功能在哺乳动物的同源性研究组织再生的替代模型的评估,这些方法应推动多元化的研究人员的兴趣。
披露声明
没有利益冲突的声明。
致谢
在实验室的工作是由NSF(MCB0919933)和国立卫生研究院(GM088790)的支持。
材料
| Name | Company | Catalog Number | Comments |
| 试剂 | 供应商 | 目录编号 | 评论 |
|---|---|---|---|
| 泉水 | Kandiyohi。高级沃特公司明尼苏达州明尼阿波利斯 | N / A | 其他形式的泉水也很好。审判第一的可行性分析。 |
| 1 ×缓冲蒙特伊克(Montjuich)盐:氯化钠(1.6毫米),氯化钙(1MM),硫酸镁(1MM),氯化镁(0.1MM),氯化钾(0.1毫米),碳酸氢钠(1.2MM),HEPES(1.5)。 pH值7.4在24 ° C。 | 多个供应商 | N / A | 人工水再生实验期间的药物治疗,以确保pH缓冲。 5 / 8 Holtfreter的解决方案是一个另类。 |
| 2xYT肉汤 | Fisher Scientific则 | BP2467 - 500 | 媒体= 31 g / L的。高压灭菌。 |
| 培养皿(100x25mm) | Fisher Scientific则 | 08-757-11 | 在再生周期的房屋蠕虫 |
| 广场菜(100x100x15mm) | Fisher Scientific则 | 08 - 757 - 11A | 与水,冻结填补蠕虫切割面的冰盘 |
| 塑料桶:密保诺拉链式扭'N LOC(16盎司)。 | 各种零售商 | N / A | 便民水密封容器内的RNAi同伙 |
| 鸡肝 | 商业杂货店 | N / A | 偏见,对有机用品,由于避免抗生素。 |
| 手搅拌机 | 任何厨房供应商 | N / A | 使用鸡肝脏原浆 |
| 电线1毫米滤网 | 任何厨房供应商 | N / A | 使用使劲鸡肝脏原浆 |
| 牛红细胞 | Lampire生物实验室 | 7240807 | 冲到100%,并汇集了红细胞悬浮液 |
| 圆形滤纸 | 的Whatman#3 | 1003 055 | |
| 移液管 | Fisher Scientific则 | 13-711-41 | |
| 无菌,手术刀 | 多个供应商 | N / A | |
| ±吡喹酮 | 西格玛爱秩序 | P4668 | 在黑暗中商店粉末分装在4 ° C变干。 |
| DJ -六1 | GenBank中AJ557022.1 | RNAi的“眼”23表型阳性对照 | |
| DJ -βcatenin- 1 | GenBank中AB181913.1 | RNAi的阳性对照两个为首的表型 17 | |
| DJ - PC2 | RNAi的阳性对照光厌恶表型24损失 |
参考文献
- Morgan, T. H. Experimental studies of the regeneration of Planaria maculata. Arch Entwm Org. 7, 364-397 .
- Robb, S. M., Ross, E., Sánchez Alvarado, A. SmedGD: the Schmidtea mediterranea genome database. Nucleic Acids Res. 36, 599-606 (2008).
- Forsthoefel, D. J., Newmark, P. A. Emerging patterns in planarian regeneration. Curr Opin Genet Dev. 19, 412-4120 (2009).
- Adell, T., Cebrià, F., Saló, E. Gradients in Planarian Regeneration and Homeostasis. Cold Spring Harb Perspect Biol. 2, (2010).
- Agata, K., Watanabe, K. Molecular and cellular aspects of planarian regeneration. Semin Cell Dev Biol. 10, 377-383 (1999).
- Newmark, P. A. &. a. m. p. ;. a. m. p., Sánchez-Alvarado, A. Not your father's planarian: a classic model enters the era of functional genomics. Nat Rev Genet. 3, 210-219 (2002).
- Reddien, P. W., Sánchez-Alvarado, A. Fundamentals of planarian regeneration. Ann Rev Cell Dev Biol. 20, 725-757 (2004).
- Reddien, P. W., Bermange, A. L., Murfitt, K. J., Jennings, J. R., Sánchez-Alvarado, A. Identification of genes needed for regeneration, stem cell function, and tissue homeostasis by systematic gene perturbation in planaria. Dev Cell. 8, 635-649 (2005).
- Newmark, P. A., Reddien, P. W., Cebria, F. &. a. m. p. ;. a. m. p., Sánchez-Alvarado, A. Ingestion of bacterially expressed double-stranded RNA inhibits gene expression in planarians. Proc Natl Acad Sci USA. 100, 11861-11865 (2003).
- Collins, J. J. Genome-Wide Analyses Reveal a Role for Peptide Hormones in Planarian Germline Development. PLoS Biology. 8, e10000509-e10000509 (2010).
- Cebrià, F. Regenerating the central nervous system: how easy for planarians!. Dev Genes Evol. , 733-748 (2007).
- Mineta, K. Origin and evolutionary process of the CNS elucidated by comparative genomics analysis of planarian ESTs. Proc Natl Acad Sci USA. 100, 7666-7671 (2003).
- Oviedo, N. J., Nicolas, C. L., Adams, D. S., Levin, M. Emerging Model Organisms. A Laboratory Manual Chapter. 8, (2009).
- Buttarelli, F. R., Pellicano, C., Pontieri, F. E. Neuropharmacology and behavior in planarians: translations to mammals. Comp Biochem Physiol. 147, 399-408 (2008).
- Sánchez-Alvarado, A., Newmark, P. A. Double-stranded RNA specifically disrupts gene expression during planarian regeneration. Proc Natl Acad Sci U S A. 96, 5049-5054 (1999).
- Gurley, K. A., Rink, J. C., Sánchez-Alvarado, A. Beta-catenin defines head versus tail identity during planarian regeneration and homeostasis. Science. 319, 323-327 (2008).
- Yazawa, S., Umesono, Y., Hayashi, T., Tarui, H., Agata, K. Planarian Hedgehog/Patched establishes anterior-posterior polarity by regulating Wnt signaling. Proc Natl Acad Sci U S A. 106, 22329-22334 (2009).
- Petersen, C. P., Reddien, P. W. Smed-betacatenin-1 is required for anteroposterior blastema polarity in planarian regeneration. Science. 319, 327-330 (2008).
- Iglesias, M., Gomez-Skarmeta, J. L., Saló, E., Adell, T. Silencing of Smed-betacatenin1 generates radial-like hypercephalized planarians. Development. 135, 1215-1221 (2008).
- Nogi, T., Zhang, D., Chan, J. D., Marchant, J. S., S, J. A novel biological activity of praziquantel requiring voltage-operated Ca2+ channel beta subunits: subversion of flatworm regenerative polarity. PLoS Negl Trop Dis. 3, e464-e464 (2009).
- Oviedo, N. J. Long-range neural and gap junction protein-mediated cues control polarity during planarian regeneration. Dev Biol. 339, 188-199 (2010).
- Nogi, T., Levin, M. Characterization of innexin gene expression and functional roles of gap-junctional communication in planarian regeneration. Dev Biol. 287, 314-335 (2005).
- Mannini, L. Djeyes absent (Djeya) controls prototypic planarian eye regeneration by cooperating with the transcription factor Djsix-1. Dev Biol. 269, 346-359 (2004).
- Agata, K. Structure of the planarian central nervous system (CNS) revealed by neuronal cell markers. Zoolog Sci. 15, 433-440 (1998).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。