Method Article
I dosaggi farmacologici genetica e funzionale di manipolare Rigenerazione del Planaria Japonica Dugesia
In questo articolo
Riepilogo
Un modello interessante per studiare la differenziazione delle cellule staminali in un animale vivo è il verme piatto planaria. La rigenerazione è studiato da amputazione esperimenti semplici che sono facili da eseguire in un laboratorio di base e sono suscettibili di (farmacologica e genetica In vivo RNAi) manipolazione come descritto dai protocolli in questo articolo.
Abstract
A vita libera planaria vermi piatti hanno una lunga storia di causa dell'utilizzo sperimentale della loro notevoli capacità rigenerative 1. Piccoli frammenti prelevati da questi animali riformare il piano originale corpo dopo la rigenerazione delle strutture del corpo mancanti. Per esempio se frammento di un 'tronco' viene tagliato da un worm intatta, la 'testa' di un nuovo rigenererà anteriormente e una 'coda' rigenererà posteriormente ripristinare l'originaria polarità 'testa-coda' delle strutture del corpo prima amputazione (Figura 1A).
La rigenerazione è guidato da cellule staminali planaria, noto come 'neoblasts', che si differenziano in circa 30 diversi tipi di cellule normali del corpo durante l'omeostasi e la rigenerazione dei tessuti applicati. Questo processo rigenerativo è robusto e facile da dimostrare. Grazie alla dedizione di alcuni laboratori d'avanguardia, molti strumenti e metodi di genetica funzionale sono stati ottimizzati per questo sistema modello. Di conseguenza, notevoli recenti progressi sono stati fatti nella comprensione e manipolare gli eventi molecolari alla base planaria plasticità dello sviluppo 2-9.
Il sistema modello planaria sarà di interesse per una vasta gamma di scienziati. Per i neuroscienziati, il modello offre l'opportunità di studiare la rigenerazione di un intero sistema nervoso, piuttosto che semplicemente la ricrescita / riparazione del singolo processo delle cellule nervose che normalmente sono oggetto di studio in molti modelli consolidati. Planarie esprimere una pletora di neurotrasmettitori 10, rappresentano un importante sistema per lo studio dell'evoluzione del sistema nervoso centrale 11, 12 e hanno un potenziale di screening comportamentale 13, 14.
Risultati rigenerativa sono suscettibili di manipolazione da parte apparoaches farmacologiche e genetiche. Per esempio, i farmaci possono essere sottoposti a screening per gli effetti sulla rigenerazione semplicemente inserendo frammenti di corpo in soluzioni contenenti farmaci in diversi momenti dopo l'amputazione. Il ruolo dei singoli geni possono essere studiati utilizzando metodi knockdown (RNAi in vivo), che può essere realizzato attraverso cicli di microiniezione o alimentando batteri-espresso costruisce dsRNA 8, 9, 15. Entrambi gli approcci possono produrre fenotipi di grande impatto visivo ad alta penetranza, ad esempio, la rigenerazione degli animali bipolare 16-21. Per facilitare l'adozione di questo modello e implementazione di tali metodi, che in vetrina protocolli articolo il video per le analisi farmacologiche e genetiche (RNAi in vivo per l'alimentazione) utilizzando il Dugesia planaria japonica.
Protocollo
1. Tronco frammento saggio rigenerazione
- Smettere di alimentare una coorte di vermi per almeno 5 giorni prima del test rigenerativa per assicurare gli animali sono liberi di rifiuti e di cibo ingerito (~ 30 vermi intatti, ogni verme ~ 8-10 mm lungo post-fame).
- Il giorno del test, lavare un pre-congelato piatto ghiaccio livellato con acqua e coprire la superficie piana ghiacciata con pellicola trasparente. Uso di pinze, mettete una carta da filtro sulla pellicola di plastica.
- Bagnare la carta da filtro con qualche goccia di acqua di sorgente. Trasferimento planarie sul filtro (<20 vermi per filtro) con una pipetta di trasferimento. Worm può essere riposizionato sul filtro, se necessario, repipetting con acqua di sorgente più. Rimuovere il liquido in eccesso.
- Usando un bisturi, amputare la testa da un unico taglio fatto circa metà strada tra l'apice anteriore dell'animale e la fine anteriore della faringe (Figura 1A).
- Usando un bisturi, amputare la regione coda da un unico taglio fatto circa metà strada tra l'estremità della coda dell'animale e la fine posteriore della faringe (Figura 1A). Rimuovere il muco in eccesso sul bisturi con un fazzoletto di carta imbevuto di etanolo al 70% dopo il taglio. Pulire l'etanolo in eccesso dal bisturi.
- Trasferimento del filtro tramite pinze in una capsula di Petri (100 x 25mm profondità) contenente acqua di sorgente. Attendere ~ 3 minuti (per la chiusura della ferita, osservabili da un pizzicotti e oscuramento della ferita).
- Sciacquare frammenti di tronco da carta da filtro nella piastra di Petri contenente supporto desiderato per il dosaggio rigenerativa e trasferimento termostatati incubatore (24 ° C).
- Punteggio fenotipo rigenerativa dopo ~ 1 settimana, quando le strutture rigenerati sono identificabili (Figura 1A).
2. Manipolazione farmacologica della rigenerazione: bipolarismo indotto dal praziquantel
- Preparare praziquantel (PZQ) stock di farmaci solubilizzanti in DMSO (62.4mg PZQ in 1 ml di magazzino 200mm). Usa il giorno stesso.
- Fai 50mL di droga contenenti soluzione (90 mM PZQ) di sali Montjuch tamponata. Vortex per ~ 1 minuto per garantire la dispersione.
- Eseguire frammento saggio rigenerativa tronco come sopra [protocollo # 1], esponendo una coorte di frammenti di tronco di PZQ al passo finale [1.7].
- Dopo 24-48 ore, cambio PZQ contenenti supporti per acqua di sorgente, il lavaggio vermi diversi (> 3) volte.
- Ritorna worm per incubatrice. Punteggio bipolarità (a due teste, Figura 1B) una settimana dopo il taglio.
3. Manipolazione genetica di rigenerazione: nel protocollo vivo alimentazione RNAi
- Prima del dosaggio, la preparazione del fegato omogeneizzato di pollo. Eliminare grassi, le sezioni di colore giallo da foodstock, fegato e purea rimanente. Filtro purea attraverso mesh e sospensione aliquota per la conservazione (1,5 ml tubi, -20 ° C).
- Prima del dosaggio, preparare bovina globuli rossi (RBC) da aliquoting fornitura in campioni di 1 ml, conservati a -20 ° C.
- Seleziona worm per RNAi. Tre coorti sono utilizzati: gene di interesse, controllo positivo (gene noto con fenotipo RNAi; Figura 1C, D & E) e di controllo negativo (gene senza fenotipo RNAi, utile anche per la valutazione dei livelli knockdown rispetto alla coorte sperimentale). Per ciascuna coorte, utilizzare ~ 250 vermi (~ 8-10 mm molto tempo dopo muore di fame per almeno 5 giorni). Conservare in acqua di sorgente in vasca di plastica.
- Per ogni stock disgelo coorte RNAi, di E. coli che esprimono dsRNA gene targeting di interesse [8], insieme ad un tubo di omogenato di fegato di pollo [3.1] e un tubo di GR [3.2].
- Pellet di batteri (13.000 g, 1 minuto). Rimuovere il surnatante e risospendere pellet in 2xYT media (~ 700μl). Recentrifuge, scartare il surnatante, posto a pellet su ghiaccio.
- Creare un grande foro P200 punta della pipetta, tagliando la fine della punta. Accuratamente risospendere il pellet batterico in una miscela di fegato omogeneizzato di pollo (150 mL) e di globuli rossi (50 mL) per creare mix RNAi di alimentazione. Rimuovere le bolle d'aria per centrifugazione.
- Per l'alimentazione, rimuovere la maggior parte delle acque dalla vasca di plastica contenente vermi (lasciare profondità ~ 1 pollice). Vasca Swirl per concentrare i vermi al centro di questo contenitore, e pipetta mix di alimentazione RNAi in un cerchio corralling vermi. Utilizzare una pipetta di trasferimento di convincere attentamente fuggitivi di nuovo sul mix di alimentazione RNAi senza perturbare altri commensali!
- Dopo l'alimentazione (~ 1 ora), identificare planarie che alimentava bene. Questi worm mostrano una colorazione rosso intenso da globuli rossi ingerito. Scartare gli altri.
- Ricollocare accuratamente soluzione di alimentazione con acqua di sorgente fresca, riducendo al minimo disturbo per impedire egestion di cibo. Per fare questo, delicatamente localizzare i vermi su un lato della provetta con una pipetta di trasferimento, versare l'acqua torbida, e sostituire con acqua fresca versate nella parete opposta della vasca. Planarie Resubmerge velocemente come l'esposizione prolungata all'aria risultati anche in egestion cibo.
- Liberamente séal coperchio del contenitore e tornare alla incubatore (24 ° C)
- Ripetere l'alimentazione protocollo RNAi su più cicli (~ 2-3 giorni di distanza), intervallati da cicli di rigenerazione [protocollo # 1] per lo screening della fenotipi RNAi. Un protocollo standard per l'alimentazione e la rigenerazione efficace per molti geni è mostrato in figura 2, che si ~ 1 mese di durata totale.
Modificare questo schema per i geni diversi, come il protocollo ottimale dipenderà da fattori quali la stabilità di mRNA, localizzazione dei tessuti, perdurance proteine, o lo sviluppo di un fenotipo che preclude cicli di alimentazione multipla dopo la rigenerazione. Valutare i livelli di knockdown semplicemente screening per penetranza del fenotipo (se apparente), o da approcci qPCR per confrontare i livelli di mRNA bersaglio con coorte di controllo negativo.
4. Rappresentante dei risultati:
Il frammento saggio tronco rigenerazione è robusto in modo che tutti i worm dovrebbe rigenerarsi con le normali antero-posteriore ('testa alla coda') polarità. Questo può essere segnata appena cinque giorni dopo il taglio, facilitato dalla nascita del ocelli anteriore (asterixed, Figura 1A). Tuttavia, completo ripristino morfologico delle strutture perso si verifica dopo una settimana. Manipolazione farmacologica della rigenerazione tronco frammento da PZQ di cedere a due teste vermi (Figura 1B) è robusto (EC 50 = 87 (+ -) frammenti bipolare 11%, 70μM PZQ per 48 ore 20). Bipolarità avviene su un singolo ciclo rigenerativo. Minore efficacia in questi saggi si riferisce probabilmente a problemi con la solubilità della droga e / o stoccaggio o l'utilizzo di una specie di verme piatto diverso dove PZQ è inefficace. Per i farmaci con la più bassa penetranza, un indice anteriorization è spesso usato per punteggio fenotipi intermedi a parte da 22 bipolarità completa. Fenotipi che derivi dalla RNAi di costrutti di controllo positivo sono mostrati in figura 1: RNAi di Dugesia japonica sei-1 (D j-sei-1) si traduce in un fenotipo senza occhi 23 (Figura 1C), RNAi di Dugesia japonica PC2 (Dj-PC2) traduce in una perdita della risposta avversione luce 24 (Figura 1D) e RNAi di Dugesia japonica βcatenin-1 (Dj-βcatenin-1) si traduce in un due teste fenotipo 16-19, 21 da frammenti tronco (figura 1E). Questi fenotipi possono essere raggiunti utilizzando i seguenti protocolli di RNAi semplice: Dj-sei-1 (FFxFx), Dj-PC2 (FFFx), Dj-βcatenin-1 (FFxFx), dove F = ciclo di alimentazione e x = rispettivamente ciclo rigenerativo.
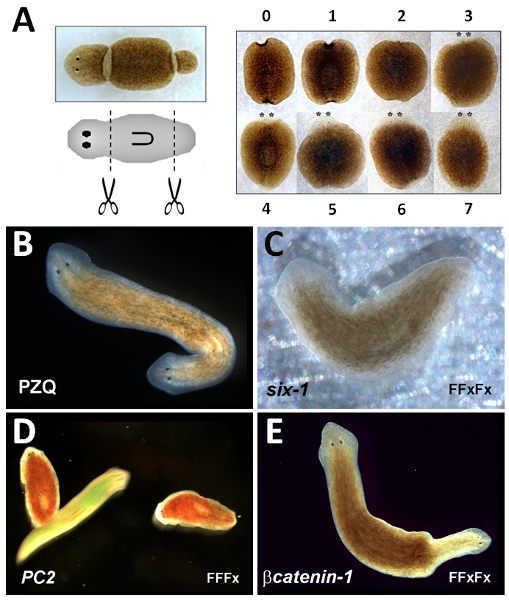
Figura 1:... Frammento saggio Tronco rigenerazione A sinistra, immagine di planaria (in alto) e schematica (al centro) per mostrare posizione del frammento asportato tronco diritto, timecourse di rigenerazione del tronco frammento che mostra l'aspetto del frammento rigenerante, a volte indicati (giorni) B immagine di due teste planaria prodotti dall'esposizione PZQ. C verme 'Eyeless' prodotta da Dj-sei-1 RNAi. D Perdita di risposta avversione luce in immobile Dj-PC2 RNAi vermi (macchiato rosso), rispetto al verme di controllo mobile (macchiata verde). E bipolare planaria prodotto da Dj-βcatenin-1 RNAi. In BE, la fine originale anteriore del worm RNAi è orientato a sinistra.
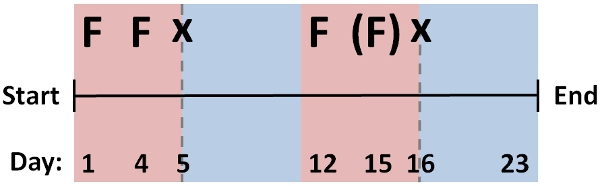
Figura 2: Sequenza di alimentazione e cicli di taglio tipico protocollo per RNAi composto da alimentazioni multiple (F), ed i cicli di rigenerazione (x) che è efficace per atterramento di molti geni in D. japonica. Modifica di questo protocollo per singoli geni possono essere necessari per ottenere risultati ottimali, per esempio utilizzando un numero inferiore (tra parentesi) o un numero maggiore di alimentazione e cicli di rigenerazione.
Discussione
I protocolli descritti dettagliatamente i test per lo studio e la manipolazione di rigenerazione della planaria Dugesia japonica. Sono semplici e non richiedono attrezzature specializzate tale da poter essere facilmente eseguito in laboratorio o in aula. Test può essere effettuato singolarmente o combinate (per lo screening genetico chimica dell'efficacia dei farmaci in vivo) e può essere eseguita a livello del gene candidato, o sono adattabili a imparziali, screening ad alto 8. Sia per lo studio della biologia intrigante di planarie in sé, o per la valutazione della funzione in vivo di omologhi mammiferi in un modello alternativo per studiare la rigenerazione dei tessuti, questi approcci dovrebbero catalizzare l'interesse di una vasta gamma di ricercatori.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Lavoro in laboratorio è supportato da NSF (MCB0919933) e NIH (GM088790).
Materiali
| Name | Company | Catalog Number | Comments |
| Reagente | Venditore | Numero di catalogo | Commenti |
|---|---|---|---|
| Sorgente d'acqua | Kandiyohi. Premio Waters Inc. Minneapolis, MN | n / a | Altre forme di acqua di sorgente funzionano bene anche. Prima prova nei test di vitalità. |
| 1 x tamponata Montjuïch sali: NaCl (1,6 mm), CaCl2 (1 mm), MgSO4 (1 mm), MgCl2 (0,1 mm), KCl (0,1 mm), NaHCO3 (1,2 mm), HEPES (1,5 mm). pH 7,4 a 24 ° C. | Diversi fornitori | n / a | Acqua artificiale per trattamenti farmacologici durante i test di rigenerazione per garantire buffer pH. Soluzione 5 / 8 Holtfreter è un'alternativa. |
| 2xYT brodo | Fisher Scientific | BP2467-500 | Media = 31 g / L. Sterilizzati in autoclave. |
| Petri Dish (100x25mm) | Fisher Scientific | 08-757-11 | Worm alloggio durante i cicli di rigenerazione |
| Piazza Dish (100x100x15mm) | Fisher Scientific | 08-757-11A | Riempire con acqua, congelare per il vassoio del ghiaccio utilizzato come superficie di taglio verme |
| Vaschetta di plastica: Ziploc Twist 'n Loc (16 once). | Vari rivenditori | n / a | Contenitori per l'acqua comoda stretto per coorti RNAi |
| Fegato di pollo | Commerciale di generi alimentari | n / a | Propensione fornisce organico, a causa di evitare gli antibiotici. |
| Mano Blender | Ogni cucina fornitore | n / a | Utilizzare per le purea di fegato di pollo |
| Filo 1 millimetro mesh | Ogni cucina fornitore | n / a | Utilizzare per filtrare purea di fegato di pollo |
| Bovina globuli rossi | Lampire Biological Laboratories | 7240807 | 100% lavato e raccolto sospensione RBC |
| Filtri circolari | Whatman # 3 | 1003 055 | |
| Trasferimento Pipettare | Fisher Scientific | 13-711-41 | |
| Sterili, lame chirurgiche | I fornitori multipli | n / a | |
| ± Praziquantel | Sigma Aldrich | P4668 | Aliquote polvere Conservare al buio a 4 ° C. Essiccare. |
| Dj-sei-1 | GenBank AJ557022.1 | RNAi controllo positivo per fenotipo "occhio-less" 23 | |
| Dj-βcatenin-1 | GenBank AB181913.1 | RNAi controllo positivo per due teste fenotipo 17 | |
| Dj-PC2 | RNAi controllo positivo per la perdita del fenotipo avversione leggeri 24 |
Riferimenti
- Morgan, T. H. Experimental studies of the regeneration of Planaria maculata. Arch Entwm Org. 7, 364-397 .
- Robb, S. M., Ross, E., Sánchez Alvarado, A. SmedGD: the Schmidtea mediterranea genome database. Nucleic Acids Res. 36, 599-606 (2008).
- Forsthoefel, D. J., Newmark, P. A. Emerging patterns in planarian regeneration. Curr Opin Genet Dev. 19, 412-4120 (2009).
- Adell, T., Cebrià, F., Saló, E. Gradients in Planarian Regeneration and Homeostasis. Cold Spring Harb Perspect Biol. 2, (2010).
- Agata, K., Watanabe, K. Molecular and cellular aspects of planarian regeneration. Semin Cell Dev Biol. 10, 377-383 (1999).
- Newmark, P. A. &. a. m. p. ;. a. m. p., Sánchez-Alvarado, A. Not your father's planarian: a classic model enters the era of functional genomics. Nat Rev Genet. 3, 210-219 (2002).
- Reddien, P. W., Sánchez-Alvarado, A. Fundamentals of planarian regeneration. Ann Rev Cell Dev Biol. 20, 725-757 (2004).
- Reddien, P. W., Bermange, A. L., Murfitt, K. J., Jennings, J. R., Sánchez-Alvarado, A. Identification of genes needed for regeneration, stem cell function, and tissue homeostasis by systematic gene perturbation in planaria. Dev Cell. 8, 635-649 (2005).
- Newmark, P. A., Reddien, P. W., Cebria, F. &. a. m. p. ;. a. m. p., Sánchez-Alvarado, A. Ingestion of bacterially expressed double-stranded RNA inhibits gene expression in planarians. Proc Natl Acad Sci USA. 100, 11861-11865 (2003).
- Collins, J. J. Genome-Wide Analyses Reveal a Role for Peptide Hormones in Planarian Germline Development. PLoS Biology. 8, e10000509-e10000509 (2010).
- Cebrià, F. Regenerating the central nervous system: how easy for planarians!. Dev Genes Evol. , 733-748 (2007).
- Mineta, K. Origin and evolutionary process of the CNS elucidated by comparative genomics analysis of planarian ESTs. Proc Natl Acad Sci USA. 100, 7666-7671 (2003).
- Oviedo, N. J., Nicolas, C. L., Adams, D. S., Levin, M. Emerging Model Organisms. A Laboratory Manual Chapter. 8, (2009).
- Buttarelli, F. R., Pellicano, C., Pontieri, F. E. Neuropharmacology and behavior in planarians: translations to mammals. Comp Biochem Physiol. 147, 399-408 (2008).
- Sánchez-Alvarado, A., Newmark, P. A. Double-stranded RNA specifically disrupts gene expression during planarian regeneration. Proc Natl Acad Sci U S A. 96, 5049-5054 (1999).
- Gurley, K. A., Rink, J. C., Sánchez-Alvarado, A. Beta-catenin defines head versus tail identity during planarian regeneration and homeostasis. Science. 319, 323-327 (2008).
- Yazawa, S., Umesono, Y., Hayashi, T., Tarui, H., Agata, K. Planarian Hedgehog/Patched establishes anterior-posterior polarity by regulating Wnt signaling. Proc Natl Acad Sci U S A. 106, 22329-22334 (2009).
- Petersen, C. P., Reddien, P. W. Smed-betacatenin-1 is required for anteroposterior blastema polarity in planarian regeneration. Science. 319, 327-330 (2008).
- Iglesias, M., Gomez-Skarmeta, J. L., Saló, E., Adell, T. Silencing of Smed-betacatenin1 generates radial-like hypercephalized planarians. Development. 135, 1215-1221 (2008).
- Nogi, T., Zhang, D., Chan, J. D., Marchant, J. S., S, J. A novel biological activity of praziquantel requiring voltage-operated Ca2+ channel beta subunits: subversion of flatworm regenerative polarity. PLoS Negl Trop Dis. 3, e464-e464 (2009).
- Oviedo, N. J. Long-range neural and gap junction protein-mediated cues control polarity during planarian regeneration. Dev Biol. 339, 188-199 (2010).
- Nogi, T., Levin, M. Characterization of innexin gene expression and functional roles of gap-junctional communication in planarian regeneration. Dev Biol. 287, 314-335 (2005).
- Mannini, L. Djeyes absent (Djeya) controls prototypic planarian eye regeneration by cooperating with the transcription factor Djsix-1. Dev Biol. 269, 346-359 (2004).
- Agata, K. Structure of the planarian central nervous system (CNS) revealed by neuronal cell markers. Zoolog Sci. 15, 433-440 (1998).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon