需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
利用电泳迁移分析(EMSA)和筛选功能的非编码基因变异DNA的亲和沉淀试验(DAPA)
* 这些作者具有相同的贡献
摘要
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
摘要
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
引言
测序和基因分型的基础研究,包括全基因组关联分析(GWAS),候选人基因研究和深测序研究,已经确定了在统计学上与疾病,性状或表型相关的许多基因变异。相反早期预测,大多数这些变体(85-93%)的分布在非编码区,并且不改变蛋白质1,2的氨基酸序列。解释这些非编码变体的功能和确定它们连接到相关的疾病的生物学机制,特征,或表型已被证明具有挑战性3-6。我们已开发,以确定链接变体的重要中间体的表型的分子机制的一般策略 - 基因表达。这条管线是专门设计来确定的TF由遗传变异体结合的调制。这一策略结合的目的是预测的计算方法和分子生物学技术在硅片的候选变体的生物学效应,并验证这些预测凭经验( 图1)。
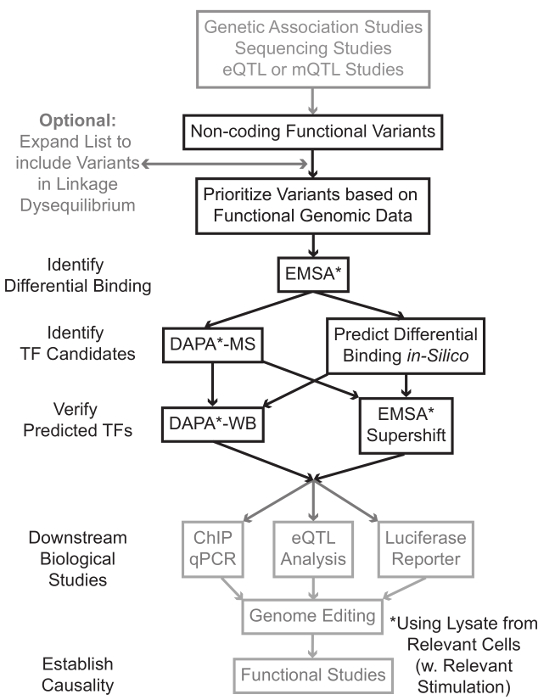
图1: 对于不包含在这个手稿以灰色阴影相关的详细协议的非编码基因变异步骤的分析战略方针 请点击此处查看该图的放大版本。
在许多情况下,通过扩大变体的列表以包括与每个统计学关联变体在高联动不平衡(LD)的所有那些开始是很重要的。 LD是在两个不同的染色体位置的等位基因的非随机关联,可以由R 2统计7被测量的量度。 R 2是林的措施卡格两个变量之间的不平衡,有两个变量之间的R 2 = 1表示完全连锁。在高LD等位基因被发现在整个人群祖先染色体上共分离。当前的基因分型阵列不包括在人类基因组中所有已知的变体。相反,他们利用了人类基因组中的LD和包括公知的变体作为代理为LD 8的一特定区域内的其它变体的一个子集。因此,在没有任何生物后果的变体可以与特定疾病,因为它是在与一个有意义的生物效应的因果变体的变体的LD相关联。在程序上,建议转换的1000基因组最新发布的项目9项变量调用文件(VCF)与PLINK 10,11,用于全基因组关联分析的开放源代码工具兼容的二进制文件。随后,LD R 2> 0.8所有其他遗传变异与每个输入遗传VAriant可确定为候选者。使用合适的参考人群该步进例如是很重要的,如果一个变种是在欧洲血统的科目,从类似的血统科目的数据应该用于LD扩展标识。
LD膨胀常常导致数十个候选变体,它是可能的,只有这些一小部分有助于疾病的机制。通常,它是不可行的实验逐一检查每个这些变体。因此,有用以利用数千公开可用功能基因组数据集作为过滤器的变体的优先次序。例如,编码财团12已经如DNA酶SEQ 13,ATAC进行数千描述转录因子的结合芯片起实验和辅因子,以及组蛋白标记在各种上下文,以及染色质辅助数据从技术-SEQ 14和FAIRE-SEQ 15。数据BASES和Web服务器如UCSC基因组浏览器16,路线图表观基因组17,蓝图表观基因组18,顺反组19,并重新映射20提供由这些和其他实验技术在广泛的细胞类型和条件产生的数据免费访问。当有太多的变体通过实验检验,这些数据可以被用来优先那些位于相关的细胞和组织类型的可能调控区之内。此外,在一个变型为特定蛋白的芯片起峰内的情况下,这些数据可以提供潜在线索作为对特定TF(S)或辅因子的结合可能影响。
接着,将所得的优先变体实验筛选,以验证预测的基因型依赖性蛋白使用EMSA 21,22结合。 EMSA测量的寡核苷酸上的非还原性TBE凝胶迁移的变化。荧光标记的寡核苷酸温育与核裂解物,以及核因子的结合将延缓在凝胶上的寡的运动。以这种方式,已经结合更多的核因子将呈现为对扫描更强的荧光信号寡。值得注意的是,EMSA不需要关于特定蛋白质的结合会受到影响的预测。
一旦变体被识别,它们位于预测的调节区之内,并且能够差动结合核因子中,采用计算的方法来预测具体TF(S),其结合它们可能会影响。我们优选使用CIS-BP 23,24,RegulomeDB 25,UniProbe 26和JASPAR 27。一旦候选人转录因子被确定,这些预测可以专门使用抗转录因子这些(EMSA-supershifts和DAPA-西部片)测试。一个EMSA-超迁移涉及增加了一个特定的TF-抗体与核裂解物和寡。阳性结果在EMSA-超迁移是再版esented如在EMSA频带的进一步转变,或带的损耗(在参考28中综述)。在互补DAPA,含有该变体和20个碱基对的侧翼核苷酸的5'-生物素化的寡核苷酸双链体与来自相关的细胞类型(多个)核裂解物温育以捕获任何核因子特异性结合的寡核苷酸。低聚双链核因子复合物是通过在一个磁列链霉微珠固定。结合的核因子直接通过洗脱29,48收集。结合预测可以然后通过使用特异于所述蛋白质的抗体的Western印迹来评估。在没有明显的预测,或过多的预测,从DAPA实验的变体拉起伏的洗脱可被发送到一个蛋白质组学芯使用质谱来识别候选转录因子的情况下,其可以随后使用验证这些先前描述的方法。
在该articl剩余即,提供了一种用于遗传变异体的EMSA和DAPA分析的详细协议。
研究方案
1.解决方案和试剂的制备
- 为了统一EMSA和DAPA使用定制的DNA寡核苷酸探针。
- 为了减少非特异性蛋白结合,设计短的寡核苷酸30,以及直接将感兴趣的变体在其17碱基对的内源性基因组序列侧接的中央(长度35-45碱基对(bp)之间)。对于EMSA寡核苷酸,加上5'荧光。对于DAPA寡核苷酸,加上5'生物素标记。
- 同时订购有义链及其反向互补链。另外,为了双工(退火前)寡核苷酸。当命名的寡核苷酸,立足于建立的参考基因组的术语。
注:"风险"和"非风险"指定可以是疾病和具体项目,而"参考"和"非参考"更普遍相关。 - 一旦寡核苷酸的到来,简要地在不含核酸酶的水含量和重悬降速至100μ的终浓度,M。储存于-20℃的悬浮股票。用铝箔包裹保护标记与光荧光团的寡核苷酸。
| 名称 | 序列 |
| rs76562819_REF_FOR | GTAATGCCTTAATGAGAGAG 一个 GTTAGTCATCTTCTCACTTC |
| rs76562819_REF_REV | GAAGTGAGAAGATGACTAACŧCTCTCTCATTAAGGCATTAC |
| rs76562819_NONREF_FOR | GTAATGCCTTAATGAGAGAG 摹 GTTAGTCATCTTCTCACTTC |
| rs76562819_NONREF_REV | GAAGTGAGAAGATGACTAACÇCTCTCTCATTAAGGCATTAC |
表1:实施例EMSA / DAPA寡核苷酸设计以测试SNP差分仓丁。"REF"代表参考等位基因,而"NONREF"代表非基准等位基因。 "FOR"表示正向链,而"REV"表示它的补。的SNP被认为是在红色。
- 制备胞质提取(CE)缓冲液有10mM HEPES(pH值7.9)的最终浓度,10毫KCl和0.1mM EDTA的在去离子水中。
- 制备核提取(NE)用20mM HEPES(pH值7.9)的最终浓度,0.4M NaCl和1mM EDTA中在去离子水中缓冲。
- 制备退火缓冲液的10mM Tris(pH值7.5-8.0)的终浓度,50mM的NaCl和1mM EDTA中在去离子水中。
从培养细胞核裂解液2.准备工作
注意:此实验方案是使用B淋巴母细胞系进行了优化,但在其他几个无关的贴/悬浮细胞系已经测试和工程普遍。
- 丘尔重新罗斯韦尔园区纪念研究所(RPMI)1640的B-类淋巴母细胞有2mM L-谷氨酰胺,10%胎牛血清,并含有100单位/ ml青霉素,100微克/毫升链霉素的1×抗生素 - 抗真菌剂,和250ng的/ ml两性霉素B的
- 种子在一系列200,000-500,000活细胞/ ml,并培育在37℃用5%二氧化碳培养瓶中与排出或松帽直立位置。
注意:当它们到达超过一百万个细胞/ ml的B-类淋巴母细胞的生长减缓。移液器向上和向下几次分手电池爆炸并返回细胞200,000-500,000细胞/ ml,以保持增长速度迅速。
- 种子在一系列200,000-500,000活细胞/ ml,并培育在37℃用5%二氧化碳培养瓶中与排出或松帽直立位置。
- 洗涤培养的细胞,用10ml冰冷的磷酸盐缓冲盐水(PBS)洗涤两次,降速在4℃,300×g离心5分钟,并通过抽吸除去PBS中。
- 计数每10 7个细胞使用血细胞计数器,重悬沉淀为1ml冷的PBS细胞。
注意:例如,如果溶解2×10 7 </ SUP>细胞悬浮在2毫升PBS。 - 等分试样1ml至1.5ml微量管中,使每个管含有10 7个细胞在PBS中。离心3300 XG为2分钟,4°C和吸掉PBS。
- 在使用之前,添加1mM的二硫苏糖醇(DTT),1×磷酸酶抑制剂,和1x蛋白酶抑制剂的CE缓冲器的工作原液。用400μl的CE缓冲器的重悬细胞沉淀,并在冰上孵育15分钟。
- 加入25微升10%的Nonidet P-40,并通过吹打混合。离心机在4℃,最高速度为3分钟。倒出并弃去上清液。
- 在使用前,加入1毫摩尔DTT,1×磷酸酶抑制剂,和1x蛋白酶抑制剂对NE缓冲器的工作原液。与NE缓冲的30微升悬浮细胞沉淀,并震荡混合。
- 在试管旋转器或在冰上10分钟温育在4℃。离心3300 XG 2分钟4℃。
- 在存放之前收集上清液(核裂解液)和分装-806℃至避免可能降解蛋白多冻融。发表10微升等分试样使用bichoninic酸测定法(BCA)31来测量蛋白质浓度。
3.电泳迁移分析(EMSA)
- 准备工作寡糖股票及EMSA凝胶。
- 如果寡核苷酸在双工排序,解冻100μM的库存和稀释1:在退火缓冲液2000实现了50nM的工作原液。
- 如果寡核苷酸定购单链的,解冻的100μM的股票和在退火缓冲液稀释1:10至达到100纳米工作的股票。在微量离心管中结合100微升彼此100nM的互补链溶液。
- 在一个热块放置在95℃下5分钟。关火块,并允许寡聚慢慢冷却到室温,使用前至少一小时。
- 预运行EMSA凝胶。
- 从预铸6%T取出滑动BE凝胶在去离子水冲洗数次,以便从井中去除任何缓冲。通过加入50毫升10×TBE的950毫升去离子水制备1升0.5×TBE缓冲液中。
- 组装凝胶电泳仪和填充0.5X TBE缓冲液内室检查是否有泄漏。如果没有缓冲泄漏到外室,填充外室的方式大致三分之二。
- 60分钟前在运行100伏的凝胶。
- 用200μl0.5×TBE缓冲液的冲洗每个孔中。
- 准备结合缓冲液母液。
- 制备10×100毫摩尔Tris,500mM的氯化钾,10毫摩尔DTT的终浓度结合缓冲液;在去离子水的pH值7.5。
- 在微量离心管中,创建一个主混合物由共同所有的反应(10微升10X结合缓冲液,10微升的DTT /聚山梨醇酯,5微升多聚d(IC)和2.5微升的鲑精DNA; 表2)的试剂。准备额外的10%考虑到由于移液体积损失。
| 试剂 | 终浓度。 | RXN#1 | RXN#2 | RXN#3 | RXN#4 |
| 超纯水 | 20微升体积。 | 13.5微升 | 11.98微升 | 13.5μl | 11.98微升 |
| 10X结合缓冲液 | 1X | 2微升 | 2微升 | 2微升 | 2微升 |
| DTT / TW-20 | 1X | 2微升 | 2微升 | 2微升 | 2微升 |
| 鲑鱼精子DNA | 500纳克/微升 | 0.5微升 | 0.5微升 | 0.5微升 | 0.5微升 |
| 1微克/微升多聚d(IC) | 1微克 | 1微升 | 1微升 | 1微升 | 1微升 |
| 核提取物(5.26微克/微升) | 8微克 | - | 1.52微升 | - | 1.52微升 |
| NE缓冲区 | 1.52微升 | - | 1.52微升 | - | |
| 参考等位基因寡 | 50飞摩尔 | 1微升 | 1微升 | - | - |
| 非参考等位基因寡 | 50飞摩尔 | - | - | 1微升 | 1微升 |
表2:实例EMSA反应SETU页表说明的例子EMSA测试的假设有基因型依赖性转录因子到特定的SNP的结合。
- 不含核酸酶的水添加到每个微量离心管中,使得在加入所有试剂的终体积为20微升。
- 主混合物的适当量(5.5微升)添加到每个离心管中。
- 添加8微克核裂解物到合适的离心管。包括含有低聚没有核提取物为阴性对照( 如 表2,RXN#1和#RXN 3)管。
注意:每反应裂解物的最佳量必须通过实验滴定来确定。通常,滴定范围2-10微克裂解物就足够了。 - 加入50 fmol的寡核苷酸到合适的离心管。轻弹混合和旋转简要内容到管的底部。孵育在室温下20分钟。
注意:如果attempti纳克一个超迁移,孵育用抗体的裂解物混合物在之前加入寡聚物室温20分钟。建议使用1微克的芯片级抗体以获得最佳效果。 - 加入2微升10X橙样染料到每个离心管中。移液器上下混合。
- 通过上下吹打混合,然后将每个样品排出到一个单独的井加载样本到运行前的6%TBE凝胶。在80 V上运行凝胶,直到橙色染料已迁移到2/3的一路下跌凝胶3/4。这大约需要60-75分钟。
- 由于撬它用凝胶刀开除去从塑料盒中的凝胶,然后将凝胶中的0.5%TBE缓冲液的容器中,以防止其干燥。
- 放置红外和化学发光成像系统的表面上的凝胶,作为确保消除,这将破坏图像的任何气泡或污染物。
- 使用扫描系统软件,点击"采集"标签然后选择"绘制新"周围绘制相应于该凝胶位于扫描器的表面上的区域中的框。
- 在"采集"选项卡中的"通道"部分中,选择相应的寡核苷酸的荧光标记的波长通道。在"扫描仪"部分中,单击"预览"来获得低质量的扫描预览。通过拖动周围所获取的预览图像向下到凝胶的部分蓝色框中调整扫描区域进行成像。
注:例如,如果使用标有700纳米荧光寡核苷酸,确保"700纳米"通道扫描之前选择。 - 在"扫描控件"部分,选择"84μM"分辨率选项和"中等"质量选项。设置聚焦偏移到凝胶的厚度的一半。注意:例如,1毫米的凝胶会使用0.5mm的聚焦偏移。
- 在"扫描仪"部分中,单击"开始"到begin扫描。
注意:在扫描过程中,亮度,对比度和颜色方案通常可以手动取决于扫描系统的制造商的调整。 - 扫描完成后,选择"图像"选项卡,然后点击"旋转或翻转"中的"创建"部分来纠正方向。通过点击"导出",在主菜单中保存图像文件,然后选择"单幅图像视图"。
4. DNA亲和纯化实验(DAPA)
- 5μM寡工作股票的制备。
- 如果寡核苷酸双工下令,解冻100微米的股票,并在退火缓冲液稀释1:20至达到5微米工作的股票。
- 如果寡核苷酸定购单链的,解冻的100μM的股票和在退火缓冲液稀释1:10至达到10微米的工作股票。结合10微升彼此10μM的互补链。在一个热块放置在95℃下5分钟。关火块,并允许寡聚慢慢冷却至在使用前室温。
- 之前开始,温暖的结合缓冲液,低严格洗涤缓冲液,高严格洗涤缓冲液和洗脱缓冲液至室温。
注:50毫微克的最终浓度/毫升多聚d(IC),可被加入到结合缓冲液,低严格洗涤缓冲液,并且高严格洗涤缓冲液,以降低蛋白质的寡聚物的潜在的非特异性结合。 - 准备对每个变量绑定的混合物。
- 混合1体积核裂解物与2倍体积的结合缓冲液。
注:需要裂解液量必须通过实验,由于不同的转录因子的丰度来确定。核裂解物的100-250微克之间使用每列是在大多数情况下,足够的。 - 添加1X磷酸酶抑制剂,1个蛋白酶抑制剂,和1x增强的结合(可选),轻弹管混匀数次。
注:100X结合增强包括750毫米氯化镁2毫米和300毫米氯化锌的。添加结合增强剂如果对TF的DNA的结合依赖于辅因子或还原剂。如果此信息不知道,添加结合增强剂。 - 加10μl5μM生物素标记的DNA捕获(50皮摩尔)的各有关结合混合物。孵育在室温下20分钟。
注意:孵育时间和温度可以根据对TF变化。的最优值必须通过实验确定。
- 混合1体积核裂解物与2倍体积的结合缓冲液。
- 加入100μl链亲和素微球。孵育在室温下10分钟。
- 对于所测试的每个寡探针,放置一个结合柱在磁分离器。直接放在离心管每个绑定列下,并应用100微升结合缓冲液冲洗柱子。
- 吸取各结合混合物成单独的列的内容,并允许液体通过该柱完全流入微量在继续之前管。确保与已在混合物中的异体寡核苷酸来标记列。标记流过的样品,并用新的微量离心管更换收集低严格洗涤。
- 应用100微升低严格洗涤缓冲液对柱;等到列水库是空的。重复洗4倍。标签低严格洗涤的样品,并与新的离心管代替收集高严格洗涤。
- 应用100微升高严格性洗涤缓冲液对柱;等到列水库是空的。重复洗4倍。标签的高严格洗涤样品,并与新的离心管代替收集预洗脱。
- 添加30微升天然洗脱缓冲液向柱,静置5分钟。标签洗脱前的样本及与新的离心管代替收集洗脱。
注意:此不洗脱结合蛋白;它洗剩余的高严格缓冲出列并用洗脱缓冲液代替它最大限度地洗脱的效率。 - 添加一个额外的50微升本地洗脱缓冲液来洗脱结合转录因子。对于较高的产量,但不太集中洗脱液,增加额外的50微升天然洗脱缓冲液并收集流通。
注意:通过质谱分析洗脱的样品,以确定结合的转录因子32的身份。此后,验证通过十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE),接着Western印迹33的蛋白质组的结果。如果质谱不可用,使用标准的技术,而不是一个Western印迹以确定表示基因型依赖性结合蛋白(S)的大小运行银染色。使用这些信息来缩小从介绍详细的计算方法预测课题组的列表。
结果
在这一节中,关于裂解液的质量,其特征在于执行EMSA或dapA和变异当提供所期待的代表性结果。例如,已经提出,冻融蛋白样品多次可能导致变性。为了探索在这些"冻融"循环的上下文EMSA分析的再现性,二35沸点寡聚在同一遗传变异体不同,用核裂解物的单一批次解冻并重新冷冻用于指定量孵育。次图2表明,冷冻和解冻这个特定B淋巴细胞核提取物达5倍,对蛋?...
讨论
虽然在测序和基因分型技术的进步大大提高了我们的能力,以找出与疾病相关的基因变异,我们了解这些变异影响的作用机制能力滞后。该问题的一个重要原因是,许多疾病相关变体位于在正对编码的基因组的区域,这有可能影响控制基因表达更难对预测的机制。在这里,我们提出了一种基于EMSA和DAPA技术,有价值的分子工具,用于识别基因型依赖TF绑定可能导致许多非编码变异体的功能活动...
披露声明
作者什么都没有透露。
致谢
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children's Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
材料
| Name | Company | Catalog Number | Comments |
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1 M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1x) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1 M) | Sigma | 646563 | Reducing agent |
| Protease Inhibitor Cocktail | Thermo Scientific | 87786 | Prevents catabolism of TFs |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10x) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/ml of penicillin, 10,000 µg/ml of streptomycin, and 25 µg/ml of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25 mM HEPES |
参考文献
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium--understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5'-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。