A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הקרנה עבור וריאנטים גנטיים ללא קידוד תפקודיים באמצעות ניידות Electrophoretic Shift Assay (Emsa) ו Assay משקע זיקת DNA (DAPA)
* These authors contributed equally
In This Article
Summary
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
Abstract
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
Introduction
סידור מחקרים המבוססים גנוטיפ, כוללים מחקרים אגודים הגנום כולו (GWAS), מחקרי מוקד המועמד, ומחקרי deep-sequencing, זיהו וריאנטים גנטיים רבים הקשורים סטטיסטי עם מחלה, תכונה, או הפנוטיפ. בניגוד לתחזיות המוקדמות, רוב גרסאות אלה (85-93%) נמצאים באזורים ללא קידוד ואל תשנה את רצף החומצות האמיניות של חלבונים 1,2. פירוש הפונקציה של הגירסות ללא קידוד אלה וקביעת המנגנונים הביולוגיים חיבורם המחלה הקשורה, תכונה, או הפנוטיפ הוכיח מאתגרת 3-6. פיתחנו אסטרטגיה כללית לזהות את המנגנונים המולקולריים המקשרים גרסאות כדי פנוטיפ ביניים חשוב - ביטוי גנים. צינור זה הוא תוכנן במיוחד כדי לזהות אפנון של TF המחייבת וריאנטים גנטיים. אסטרטגיה זו משלבת גישות חישוביות וטכניקות ביולוגיה מולקולרית שמטרתו לחזותהשפעות ביולוגיות של וריאנטים מועמד סיליקון, ולאמת תחזיות אלו באופן אמפירי (איור 1).
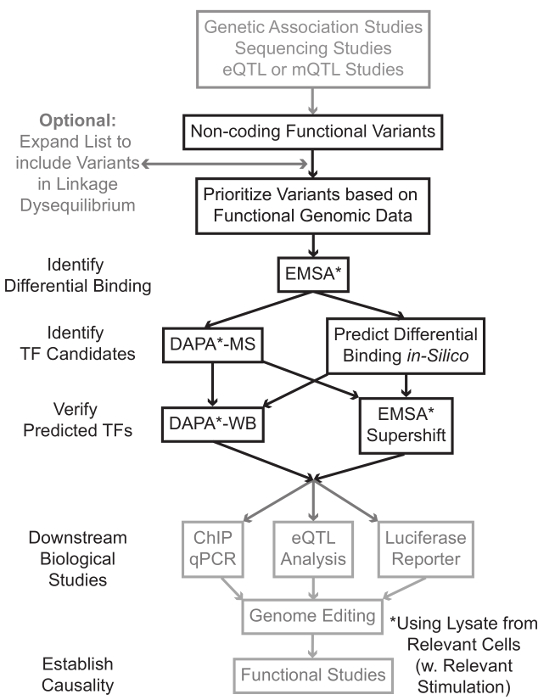
איור 1:.. גישה אסטרטגית לניתוח וריאנטים גנטיים ללא קידוד צעדים שאינם כלולים בפרוטוקול מפורט הקשורים כתב היד הזה מוצללות באפור אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
במקרים רבים, חשוב להתחיל על ידי הרחבת הרשימה של וריאנטים לכלול את כל אלה-איזון הצמדה גבוה (LD) עם כל גרסה קשורה מבחינה סטטיסטית. LD הוא מדד של העמותה הלא אקראית של אללים בשתי עמדות כרומוזומליות שונות, אשר ניתן למדוד על ידי נתון r 2 7. r 2 הוא מדד של ליןKage חוסר שיווי משקל בין שתי גרסאות, עם r 2 = 1 המציין הצמדה מושלמת בין שתי גרסאות. אללים ב LD הגבוהה נמצאים לשתף להפריד בכרומוזום פני אוכלוסיות אבות. מערכי genotyping נוכחיים אינם כוללים את כל הסוגים הידועים בגנום האנושי. במקום זאת, הם מנצלים את LD בתוך הגנום האנושי וכוללים משנה של הסוגים הידועים אשר פועלים פרוקסי עבור הגירסות אחרות בתוך אזור מסוים של LD 8. לפיכך, גרסה ללא כל תוצאה ביולוגית עשויה להיות קשורה למחלה מסוימת משום שהיא נמצאת LD עם וריאנט-סיבתית גרסה עם השפעה ביולוגית משמעותית. עניינים טכניים, מומלץ להמיר את המהדורה האחרונה של 1,000 הגנום להקרין 9 קבצים שיחת וריאנט (vcf) לתוך קבצים בינאריים תואמים עמד לירות 10,11, כלי קוד פתוח לניתוח עמותה הגנום כולו. בהמשך לכך, כל וריאנטים גנטיים אחרים עם LD r 2> 0.8 עם כל va קלט גנטיriant ניתן לזהות כמועמדים. חשוב להשתמש אוכלוסיית ההתייחסות המתאימה למשל החורג הזה, אם גרסה זוהתה במקצועות ממוצא אירופאי, נתונים הנבדקים ממוצא דומה אמורים לשמש להרחבת LD.
הרחבת LD לעתים קרובות תוצאות בעשרות גרסאות המועמד, וסביר להניח כי רק חלק קטן של אלה תורמים מנגנון המחלה. לעתים קרובות, זה מעשי לבחון כל הניסיון של הגירסות אלה בנפרד. לכן כדאי לנצל את אלף מערכי נתונים גנומי פונקציונליים בפומבי זמינים כמסנן לתעדף את הווריאציות. לדוגמה, הקונסורציום ENCODE 12 ביצע אלפי ניסויים שבב seq המתאר את עקדת TFS ושיתוף גורמים, וסימני היסטון במגוון רחב של הקשרים, יחד עם נתונים נגישות הכרומטין מטכנולוגיות כגון DNase-seq 13, ATAC -seq 14, ו -15 פייר-seq. Databases ושירת אינטרנט כגון דפדפן הגנום UCSC 16, מפת הדרכים epigenomics 17, Blueprint Epigenome 18, Cistrome 19, ולמפות 20 לספק גישה חופשית לנתונים המיוצרים על ידי אלה הניסיונות בטכניקות אחרות על פני מגוון רחב של סוגי תאים ותנאים. כאשר ישנן גרסות רבות מכדי לבחון באופן ניסיוני, נתונים אלה יכולים לשמש כדי לתעדף אלה הממוקמים בתוך אזורי רגולטוריים סבירים בסוגי תאים ורקמות רלוונטיים. יתר על כן, במקרים בהם קיימת גרסה נמצא במרחק שיא שבב seq עבור חלבון מסוים, נתונים אלה יכולים לספק מוביל פוטנציאל כמו ל TF ספציפי (ים) או שיתוף גורמים אשר מחייב העשוי להשפיע.
בשלב הבא, גרסות עדיפות וכתוצאה מוקרנות באופן ניסיוני כדי לאמת חלבון גנוטיפ תלוי חזה מחייב שימוש Emsa 21,22. Emsa מודד את שינוי ההגירה של אוליגו על ג'ל TBE הלא צמצום. אוליגו שכותרתו fluorescently הוא מודגרות עםlysate הגרעיני, ומחייב גורמים גרעיניים יהיה לעכב את התנועה של אוליגו על הג'ל. באופן זה, אוליגו אשר מחויב יותר גורמים גרעיניים יציגות כמו אות ניאון חזקה על סריקה. יש לציין, Emsa אינה דורשת תחזיות לגבי החלבונים הספציפיים אשר מחייבים יושפע.
ברגע הגירסות מזוהות הנמצאות בתוך אזורי רגולטוריים חזה ומסוגלים גורמים גרעיניים מחייבים באופן דיפרנציאלי, שיטות חישוביות מועסקות לחזות את TF (ים) הספציפי אשר מחייב הם עשויים להשפיע. אנחנו מעדיפים להשתמש CIS-BP 23,24, RegulomeDB 25, UniProbe 26, ו JASPAR 27. לאחר מועמד TFS מזוהים, תחזיות אלו ניתן לבדוק באופן ספציפי באמצעות נוגדנים נגד TFS אלה (Emsa-supershifts ו DAPA-מערבונים). Emsa-supershift תהיה כרוכה בתוספת של נוגדן TF-ספציפית lysate ו אוליגו גרעיני. תוצאה חיובית של גידול Emsa-supershift הוא represented כמעבר נוסף בלהקה Emsa, או הפסד של הלהקה (הנסקרת תוך התייחסות 28). בשנות ה DAPA משלימים, דופלקס אוליגו-biotinylated 5'המכיל את וריאנט ואת 20 בסיס-זוג איגוף נוקלאוטידים מודגרת עם lysate גרעיני מסוג תא הרלוונטיים (ים) כדי ללכוד כל הגורמים גרעיני כבילת oligos במיוחד. אוליגו המורכב גורם דופלקס-גרעיני הוא משותק על ידי streptavidin microbeads בטור מגנטי. הגורמים גרעיני כבול נאספים ישירות דרך elution 29,48. תחזיות כריכה ניתן להעריך מכן על ידי כתם מערבי באמצעות נוגדנים ספציפיים לחלבון. במקרים בם אין תחזיות ברורות, או תחזיות רבות מדי, elutions מ ומורד למשוך גרסה של ניסויי DAPA ניתן לשלוח גרעין פרוטאומיקה לזהות TFS המועמד באמצעות ספקטרומטר מסה, אשר לאחר מכן ניתן לאמת באמצעות שתואר לעיל אלה שיטות.
בשאר של articlדואר, הפרוטוקול המפורט לניתוח Emsa ו DAPA של וריאנטים גנטיים מסופק.
Protocol
1. הכנת פתרונות ריאגנטים
- להזמין בדיקות oligonucleotide DNA מותאמות אישית לשימוש Emsa ו DAPA.
- כדי להפחית חלבון הלא ספציפית מחייב, לעצב oligos קצר (בין 35-45 זוגות בסיסים (bp) אורך) 30, ומניחים את וריאנט של עניין ישירות במרכז מוקף רצף גנומי אנדוגני שלה 17 נ"ב. לקבלת oligos Emsa, להוסיף fluorophore 5 '. לקבלת oligos DAPA, הוסף תג ביוטין 5 ".
- להזמין הוא גדיל תחושת הגדיל המשלים ההפוך שלה. לחלופין, דופלקס סדר (טרום annealed) oligos. כאשר שיום oligos, לבסס את המינוח על הגנום נקבע.
הערה: "סיכון" ו "חסר סיכון" ייעוד יכול להיות מחלות פרויקט מסוים, תוך כדי "התייחסות" ו "השמטה" הן יותר אוניברסלית רלוונטי. - עם הגעתו של oligos, בקצרה לסובב את התוכן ו resuspend במים nuclease ללא לריכוז סופי של 100 μ;M. חנות resuspended המניה ב -20 ° C. הגן oligos מתויג עם fluorophore מן האור על ידי לפפה ברדיד אלומיניום.
| שֵׁם | סדר פעולות |
| rs76562819_REF_FOR | GTAATGCCTTAATGAGAGAG GTTAGTCATCTTCTCACTTC |
| rs76562819_REF_REV | GAAGTGAGAAGATGACTAAC T CTCTCTCATTAAGGCATTAC |
| rs76562819_NONREF_FOR | GTAATGCCTTAATGAGAGAG G GTTAGTCATCTTCTCACTTC |
| rs76562819_NONREF_REV | GAAGTGAGAAGATGACTAAC C CTCTCTCATTAAGGCATTAC |
טבלה 1: דוגמה Emsa / DAPA oligonucleotide עיצוב לבדוק SNP בבן ההפרשדינג. "REF" מייצג את אלל הפניה, תוך "NONREF" מייצג את אלל השמטה. "עבור" מייצג הגדיל קדימה, בעוד "REV" מציין המשלים שלו. ה- SNP נתפסת באדום.
- הכן מיצוי cytoplasmic (CE) חיץ עם ריכוז סופי של 10 מ"מ HEPES (pH 7.9), 10 מ"מ KCl, ו -0.1 מ"מ EDTA במים deionized.
- הכן מיצוי גרעיני (NE) חיץ עם ריכוז סופי של 20 מ"מ HEPES (pH 7.9), 0.4 M NaCl, ו 1 mM EDTA במים deionized.
- הכן חיץ חישול עם ריכוז סופי של 10 מ"מ טריס (pH 7.5-8.0), 50 מ"מ NaCl, ו 1 mM EDTA במים deionized.
2. הכנת גרעיני Lysate מ תאים בתרבית
הערה: פרוטוקול ניסוי זו הוטב באמצעות שורות תאי B-lymphoblastoid, אך נבדק בכמה שורות תאי מעטים אחרות חסידה / השעיה ועובד באופן אוניוורסלי.
- cultuמחדש תאי B-lymphoblastoid במכון הזיכרון רוזוול פארק (RPMI) 1640 עם 2 מ"מ L- גלוטמין, 10% בסרום שור העובר, וכן אנטיביוטיקה antimycotic 1x המכיל 100 יחידות / מ"ל פניצילין, 100 מיקרוגרם / מ"ל של סטרפטומיצין, ו -250 ng / מ"ל של B. amphotericin
- זרע בטווח של 200,000-500,000 תאים / מ"ל קיימא דגירה צלוחיות על 37 מעלות צלזיוס עם 5% פחמן דו חמצני בתנוחה זקופה עם כובעים פרקו או רופף.
הערה: הצמיחה של תאים lymphoblastoid B מאט כאשר הם מגיעים מעל 1,000,000 תאים / מ"ל. לשבור את תקיעות תא על ידי pipetting מעלה ומטה מספר פעמים ולחזור לתאי 200,000-500,000 תאים / מיליליטר כדי לשמור על קצב צמיחה מהיר.
- זרע בטווח של 200,000-500,000 תאים / מ"ל קיימא דגירה צלוחיות על 37 מעלות צלזיוס עם 5% פחמן דו חמצני בתנוחה זקופה עם כובעים פרקו או רופף.
- שטפו תאים בתרבית פעמיים עם 10 בופר פוספט קר מ"ל קרח (PBS), ספין למטה ב 4 ° C, 300 XG במשך 5 דקות ולהסיר PBS באמצעות שאיפה.
- ספירת תאים באמצעות גלולה hemocytometer ו resuspend כמו 1 מ"ל קרח קר PBS לכל 10 7 תאים.
הערה: לדוגמה, אם lysing 2 x 10 7 </ Sup> תאים, resuspend ב 2 מ"ל PBS. - Aliquot 1 מ"ל ל 1.5 מ"ל צינורות microcentrifuge כך שכל צינור המכיל 10 7 תאים PBS. צנטריפוגה ב 3,300 XG למשך 2 דקות 4 ° C ו לשאוב את PBS.
- לפני השימוש, להוסיף 1 מ"מ dithiothreitol (DTT), מעכב phosphatase 1x, ו פרוטאז 1x מעכב מניה עובד של חיץ לספירה. Resuspend תא גלול עם 400 μl של חיץ לספירת דגירה על קרח למשך 15 דקות.
- הוסף 25 μl של 10% Nonidet P-40 ומערבבים ידי pipetting. צנטריפוגה ב 4 ° C, ומהירות מרבית במשך 3 דקות. למזוג וזורקים supernatant.
- לפני השימוש, להוסיף 1 מ"מ DTT, מעכב phosphatase 1x, ו 1x פרוטאז מעכב מניה עובד של חיץ NE. Resuspend תא גלול עם 30 μl של חיץ NE ומערבב ידי vortexing.
- דגירה על 4 מעלות צלזיוס rotator צינור או על קרח למשך 10 דקות. צנטריפוגה 3,300 XG למשך 2 דקות 4 ° C.
- אסוף את supernatant הברור (lysate הגרעיני) ו aliquot לפני האחסון ב -806; C כדי למנוע להקפיא להפשיר מחזורים מרובים עלולים לבזות את החלבון. השאירו aliquot 10 μl למדוד ריכוז חלבון באמצעות assay חומצה bichoninic (BCA) 31.
3. Electrophoretic ניידות Shift Assay (Emsa)
- כן אוליגו עבודת סטוק Emsa ג'ל.
- אם oligos נצטוו בדופלקס, להפשיר את המניה 100 מיקרומטר לדלל 1: 2,000 במאגר חישול להשיג מניה עובד 50 ננומטר.
- אם oligos נצטווה חד גדילים, להפשיר את 100 מיקרומטר המניות לדלל 1:10 ב חיץ חישול להשיג 100 מניות עובדות ננומטר. מערבבים 100 μl של הפתרון המשלים גדיל 100 ננומטר אחד עם השני בתוך שפופרת microcentrifuge.
- מניחים בתוך גוש חום על 95 מעלות צלזיוס למשך 5 דקות. כבה את גוש חום ולאפשר oligos להתקרר לאט לטמפרטורת החדר במשך להשתמש לפני שעה אחת לפחות.
- טרום להפעיל את הג'ל Emsa.
- להסיר את השקופית מתוך% מראש יצוק 6 TBE ג'ל ולשטוף תחת מים ללא יונים מספר פעמים על מנת להסיר כל חיץ מהבארות. הכן 1 ליטר של חיץ 0.5x TBE על ידי הוספת 50 מ"ל של 10x TBE עד 950 מ"ל מים ללא יונים.
- להרכיב את מנגנון ג'ל אלקטרופורזה ולבדוק אם יש דליפות ידי מילוי החדר הפנימי עם חיץ TBE 0.5x. אם אין חיץ דליפות לתוך החדר החיצוני, למלא את החדר החיצוני כשני שליש הדרך.
- טרום להפעיל את הג'ל על 100 וולט במשך 60 דקות.
- לשטוף היטב עם כל 200 μl של חיץ 0.5x TBE.
- הכן חיץ מחייב מיקס מאסטר.
- הכן 10x חיץ מחייב עם ריכוז סופי של 100 מ"מ טריס, 500 מ"מ KCl, 10 מ"מ DTT; pH 7.5 במים deionized.
- בתוך צינור microcentrifuge, ליצור תמהיל מאסטר המורכב של חומרים כימיים משותפים לכל התגובות (10 μl 10x חיץ מחייב, 10 μl DTT / polysorbate, 5 μl פוליפוני ד (IC), ו -2.5 μl DNA זרע סלמון; טבלה 2). כן 10% נוספיםלתת דין וחשבון על אובדן נפח בשל pipetting.
| מֵגִיב | Conc הסופי. | Rxn # 1 | Rxn # 2 | Rxn # 3 | Rxn # 4 |
| מי ultrapure | עד 20 μl vol. | 13.5 μl | 11.98 μl | 13.5μl | 11.98 μl |
| 10x חיץ מחייב | 1x | 2 μl | 2 μl | 2 μl | 2 μl |
| DTT / TW-20 | 1x | 2 μl | 2 μl | 2 μl | 2 μl |
| DNA זרע סלמון | 500 ng / μl | 0.5 μl | 0.5 μl | 0.5 μl | 0.5 μl |
| 1μg / ד פוליפוני μl (IC) | 1 מיקרוגרם | 1 μl | 1 μl | 1 μl | 1 μl |
| תמצית גרעינית (5.26 מיקרוגרם / μl) | 8 מיקרוגרם | - | 1.52 μl | - | 1.52 μl |
| מאגר NE | 1.52 μl | - | 1.52 μl | - | |
| אוליגו אלל הפניה | 50 fmol | 1 μl | 1 μl | - | - |
| אוליגו אלל ללא הפניה | 50 fmol | - | - | 1 μl | 1 μl |
טבלה 2: Setu תגובת הדוגמא Emsap. הטבלה מדגימה דוגמא Emsa כדי לבדוק את ההשערה כי יש גנוטיפ תלוי עקדת TFS על SNP ספציפי.
- מוסיפים מים nuclease ללא כל צינור microcentrifuge כך נפח סופי בעקבות תוספת של כל ריאגנטים יהיה 20 μl.
- מוסיפים את הכמות המתאימה (5.5 μl) של תערובת אמן על צינור אחד microcentrifuge.
- הוסף 8 מיקרוגרם של lysate הגרעינית צינורות microcentrifuge המתאימים. כלול צינורות המכילים אוליגו ללא תמצית גרעינית כפי שולט שלילי (למשל טבלה 2, Rxn # 1 ו Rxn # 3).
הערה: הכמות האופטימלית של lysate לכל תגובה צריכה להיקבע באופן ניסיוני על ידי טיטרציה. באופן כללי, titrating מגוון של 2-10 מיקרוגרם של lysate מספיק. - הוסף 50 fmol של אוליגו אל צינורות microcentrifuge המתאימים. פליק כדי לערבב בקצרה לסובב את התוכן לחלק התחתון של הצינור. דגירה במשך 20 דקות בטמפרטורת החדר.
הערה: אם attempting supershift, דגירה תערובת lysate עם נוגדן עבור 20 דקות בטמפרטורת החדר לפני תוספת של oligos. מומלץ להשתמש 1 מיקרוגרם של נוגדן שבב כיתה לקבלת התוצאות הטובות ביותר. - הוסף 2 μl של Dye Loading 10x אורנג צינור אחד microcentrifuge. פיפטה מעלה ומטה כדי לערבב.
- טען את הדגימות לתוך הג'ל TBE 6% מראש המנוהל על ידי pipetting מעלה ומטה כדי לערבב ולאחר מכן גירוש כל דגימה לתוך באר נפרד. הפעל את הג'ל על 80 V עד לצבוע בכתום העביר 2/3 3/4 של הדרך למטה הג'ל. זה אמור לקחת בערך 60-75 דקות.
- הסר את הג'ל מתוך הקלטת פלסטיק ידי הוצאת אותו פתוח עם סכין ג'ל ולמקם את הג'ל בתוך מכולה עם חיץ 0.5% TBE לשמור עליו מפני התייבשות.
- מניחים את הג'ל על פני השטח של מערכת הדמיה אינפרא אדום chemiluminescence, להיות בטוח כדי לחסל כל בועות או מזהמים כי ישבש את התמונה.
- שימוש בתוכנת מערכת סריקה, לחץ על הכרטיסייה "לרכוש"ולאחר מכן בחר "צייר חדש" כדי ליצור תיבה סביב האזור המתאים שבו ג'ל ממוקם על פני השטח של הסורק.
- בקטע "ערוצים" של הכרטיסייה "לרכוש", בחרו בערוץ המתאים אורך הגל של תג fluorophore על אוליגו. בקטע "סורק", לחץ על "תצוגה מקדימה" כדי לקבל סריקה מקדימה באיכות נמוכה. התאם את אזור הסריקה על ידי גרירת התיבה הכחולה סביב תמונת התצוגה המקדימה רכשה עד החלק של ג'ל להיות צילמו.
הערה: לדוגמה, אם באמצעות oligos שכותרתו עם fluorophore 700 ננומטר, לוודא כי התחנה "700 ננומטר" נבחרה לפני הסריקה. - בקטע "בקרות סריקה", בחר ברזולוצית האפשרות "84 מיקרומטר" ואת אפשרות האיכות "הבינונית". הגדר את המיקוד כדי לקזז מחצית העובי של ג'ל. הערה: לדוגמה, ג'ל 1 מ"מ ישתמש מוקד 0.5 מ"מ לקזז.
- בקטע "סורק", לחץ על "התחל" כדי begin הסריקה.
הערה: במהלך הסריקה, בהירות, ניגודיות, ואת ערכת הצבעים לעתים קרובות יכולה להיות מותאמת באופן ידני בהתאם ליצרן של מערכת הסריקה. - לאחר הסריקה הסתיימה, בחר בכרטיסייה "תמונה" ולחץ על "לסובב או להפוך" בסעיף "צור" כדי לתקן את הכיוון. שמור את קובץ התמונה על ידי לחיצה על "ייצוא" בתפריט הראשי ולאחר מכן בחר "הצג תמונה אחת."
4. DNA זיקה Assay טיהור (DAPA)
- הכנת 5 מיקרומטר אוליגו עבודה Stock.
- אם oligos נצטווה בדופלקס, להפשיר את מניית 100 מיקרומטר לדלל 01:20 חיץ חישול להשיג מניה עובדת 5 מיקרומטר.
- אם oligos נצטווה חד גדילים, להפשיר את 100 מיקרומטר המניות לדלל 1:10 ב חיץ חישול להשיג 10 מיקרומטר עובדים מניות. שלב 10 μl של 10 מיקרומטר גדילים משלימים אחד עם השני. מניחים בתוך גוש חום על 95 מעלות צלזיוס למשך5 דקות. כבה את גוש חום ולאפשר oligos להתקרר לאט לטמפרטורת החדר לפני השימוש.
- לפני שמתחילים, לחמם את חיץ מחייב, חיץ לשטוף חומרא נמוך, חיץ לשטוף חומרה גבוהה, חיץ elution לטמפרטורת החדר.
הערה: ריכוז סופי של 50 ng / mL Poly ד (IC) ניתן להוסיף למאגר מחייב, חיץ לשטוף חומרא נמוך, חיץ לשטוף חומרה גבוהה להפחית מחייב הלא ספציפית פוטנציאל של חלבונים אל oligos. - מכינים את תערובת מחייב עבור כל חלופה.
- מערבבים 1 נפח של lysate גרעינית עם 2 כרכים של חיץ מחייב.
הערה: כמות lysate נדרש צריכה להיקבע באופן ניסיוני בשל משתנים שפע של TFS. שימוש בין 100-250 מיקרוגרם של lysate גרעינית לכל עמודה מספיקה ברוב המקרים. - הוסף מעכב phosphatase 1x, מעכבי פרוטאז 1x, משפר מחייב 1x (אופציונלי) ומערבבים ידי מצליף הצינור מספר פעמים.
הערה: 100x מחייבמשפר מורכב 750 מ"מ MgCl 2 ו 300 מ"מ ZnCl 2. להוסיף משפר מחייב אם המחייב של TF ל- DNA תלוי cofactors או סוכני צמצום. אם מידע זה אינו ידוע, מוסיף את המשפר המחייב. - הוסף 10 μl של ה- DNA לכידת biotinylated 5 מיקרומטר (50 pmol) לכל תערובת מחייב בהתאמה. דגירה במשך 20 דקות בטמפרטורת החדר.
הערה: דגירה זמן וטמפרטורה עשוי להשתנות תלוי TF. הערכים האופטימליים צריכים להיקבע באופן ניסיוני.
- מערבבים 1 נפח של lysate גרעינית עם 2 כרכים של חיץ מחייב.
- הוספת 100 μl של microbeads streptavidin. דגירה במשך 10 דקות בטמפרטורת החדר.
- עבור כל בדיקה אוליגו נבדקת, למקם טור כובל המפריד המגנטי. מניחים צינור microcentrifuge ישירות תחת כל עמודה מחייב ולהחיל 100 μl של חיץ מחייב לשטוף את העמודה.
- פיפטה את התוכן של כל תערובת מחייב לעמודות נפרדות ולאפשר לנוזל לזרום לחלוטין דרך העמודה לתוך microcentrifugeצינור לפני שתמשיך. הקפד לרשום את העמודות עם אוליגו החלופה ששמש את התערובת המחייבת. לייבל דגימות זרימה דרך ולהחליף עם צינורות microcentrifuge חדשים כדי לאסוף את השטיפות-חומרא הנמוך.
- החל 100 μl של חיץ לשטוף נמוכה חומרא לעמודה; לחכות עד מאגר הטור ריק. חזור על לשטוף 4x. לייבל לשטוף דגימות הנמוכה חומרא ולהחליף עם צינורות microcentrifuge חדשים לאסוף הכביסות הגבוהה חומרא.
- החל 100 μl של חיץ לשטוף גבוהה חומרא לעמודה; לחכות עד מאגר הטור ריק. חזור על לשטוף 4x. לייבל לשטוף דגימות הגבוהה חומרא ולהחליף עם צינורות microcentrifuge חדשים כדי לאסוף את-elution מראש.
- הוסף 30 μl של חיץ elution יליד לעמודה ולתת לעמוד במשך 5 דקות. לייבל הדגימות מראש elution ולהחליף עם צינורות microcentrifuge חדשים כדי לאסוף את elution.
הערה: זו אינה elute החלבון מחויב; שהוא שוטף ההיי-חומרא הנותרחיץ מהטור ומחליף אותו עם חיץ elution על מנת למקסם את היעילות של elution. - הוספת חיץ elution והמקורית נוסף 50 μl כדי elute את TFS כבול. לקבלת תשואה גבוהה יותר אך פחות eluate המרוכז, להוסיף 50 μl נוספים של חיץ elution ילידים לאסוף את הזרימה דרך.
הערה: ניתוח דגימות elution באמצעות ספקטרומטריית מסה כדי לקבוע את זהותו של כבול TFS 32. לאחר מכן, לאמת את התוצאות proteomic באמצעות ג'ל אלקטרופורזה polyacrylamide סולפיט dodecyl נתרן (SDS-PAGE) ואחריו כתם המערבי 33. אם ספקטרומטריית מסה אינה זמינה, לרוץ כתם כסף באמצעות טכניקה סטנדרטית במקום כתם מערבי כדי לקבוע את גודל החלבון (ים) מראה גנוטיפ תלוי מחייב. השתמש במידע זה כדי לצמצם את הרשימה של TFS לחזות על פי גישות חישוביות המפורטים במבוא.
תוצאות
בסעיף זה, תוצאות נציגים למה לצפות ניתנות בעת ביצוע Emsa או DAPA, ואת ההשתנות לגבי איכות lysate מאופיינת. לדוגמא, הוצע כי הקפאת דגימות חלבון הפשרה מספר פעמים עלולה לגרום denaturation. על מנת לחקור את שחזור של ניתוח Emsa בהקשר של אלה "להקפיא להפשיר" מחזורים, שני 35 ...
Discussion
למרות התקדמות בטכנולוגיות רצף גנוטיפ מאוד שפרה את היכולת שלנו לזהות וריאנטים גנטיים הקשורים למחלה, היכולת שלנו להבין את המנגנונים התפקודיים מושפעים הגירסות אלה מפגרת. מקור עיקרי של הבעיה הוא הרבה הגירסות הקשורים למחלות ממוקמות n על-קידוד האזורים בגנום, אשר סביר ?...
Disclosures
החוקרים אין לי מה לחשוף.
Acknowledgements
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children's Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
Materials
| Name | Company | Catalog Number | Comments |
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1 M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1x) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1 M) | Sigma | 646563 | Reducing agent |
| Protease Inhibitor Cocktail | Thermo Scientific | 87786 | Prevents catabolism of TFs |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10x) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/ml of penicillin, 10,000 µg/ml of streptomycin, and 25 µg/ml of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25 mM HEPES |
References
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium--understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5'-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved