Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Elektroforetik Hareketlilik Assay (EMSA) ve Shift kullanarak Fonksiyonel Olmayan kodlama Genetik Türevleri için tarama DNA afinite Yağış Deneyi (DAPA)
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
We present a strategic plan and protocol for identifying non-coding genetic variants affecting transcription factor (TF) DNA binding. A detailed experimental protocol is provided for electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genotype-dependent TF DNA binding.
Özet
Population and family-based genetic studies typically result in the identification of genetic variants that are statistically associated with a clinical disease or phenotype. For many diseases and traits, most variants are non-coding, and are thus likely to act by impacting subtle, comparatively hard to predict mechanisms controlling gene expression. Here, we describe a general strategic approach to prioritize non-coding variants, and screen them for their function. This approach involves computational prioritization using functional genomic databases followed by experimental analysis of differential binding of transcription factors (TFs) to risk and non-risk alleles. For both electrophoretic mobility shift assay (EMSA) and DNA affinity precipitation assay (DAPA) analysis of genetic variants, a synthetic DNA oligonucleotide (oligo) is used to identify factors in the nuclear lysate of disease or phenotype-relevant cells. For EMSA, the oligonucleotides with or without bound nuclear factors (often TFs) are analyzed by non-denaturing electrophoresis on a tris-borate-EDTA (TBE) polyacrylamide gel. For DAPA, the oligonucleotides are bound to a magnetic column and the nuclear factors that specifically bind the DNA sequence are eluted and analyzed through mass spectrometry or with a reducing sodium dodecyl sulfate polyacrylamide gel electrophoresis (SDS-PAGE) followed by Western blot analysis. This general approach can be widely used to study the function of non-coding genetic variants associated with any disease, trait, or phenotype.
Giriş
Sıralama ve genom çapında ilişkilendirme çalışmaları (GWAS), aday odağı çalışmaları da dahil olmak üzere genotiplendirme dayalı çalışmalar, ve derin sıralama çalışmaları, istatistiksel bir hastalık, özellik veya fenotip ile ilişkili birçok genetik varyantlar belirledik. Erken tahminler aksine, bu varyantlar (85-93%) en kodlayıcı olmayan bölgelerinde bulunur ve protein 1,2 amino asit dizisini değiştirmezler. Bu kodlayıcı olmayan varyantları işlevini yorumlama ve ilişkili hastalık onları bağlayan biyolojik mekanizmaları belirleyen özellik veya fenotip 3-6 zorlu kanıtlanmıştır. gen ifadesinin - Biz önemli bir ara fenotip için varyantları bağlantı moleküler mekanizmaları tanımlamak için genel bir strateji geliştirdik. Bu boru hattı, özellikle genetik varyantlar bağlama TF modülasyonu belirlemek için tasarlanmıştır. Bu strateji tahmin etmek amaçlanmıştır hesaplama yaklaşımları ve moleküler biyoloji teknikleri birleştirenBiyolojik siliko aday varyantları etkileri ve doğrulamak için şu tahminler ampirik (Şekil 1).
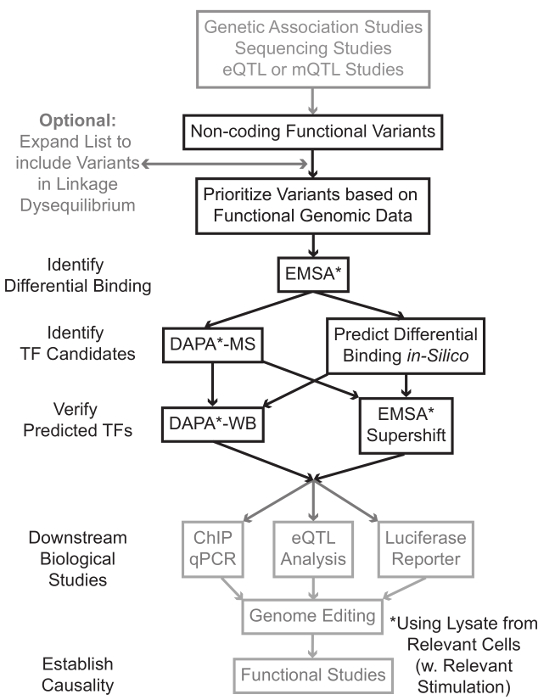
Şekil 1:.. Gri gölgeli bu yazının ilişkili detaylı protokolde yer almayan kodlayıcı olmayan genetik varyantları Adımlar analizi için stratejik yaklaşım bu rakamın daha büyük bir versiyonunu görmek için lütfen buraya tıklayınız.
Bir çok durumda, her bir istatistiksel bağlantılı varyantı ile, yüksek bir bağlantı-dengesizliği (LD) tüm bu dahil varyantların listesini genişleterek başlaması önemlidir. LD R2 istatistik 7 ile ölçülebilir iki farklı kromozom konumlarda allel rastgele olmayan federasyonu, bir ölçüsüdür. R2 lin bir ölçüsüdüriki modeli arasında bir r 2 = 1 belirten mükemmel bağlantı ile iki modeli arasındaki kage dengesizlik. yüksek LD alleller atalarının popülasyonlar arasında kromozom üzerindeki ortak ayırmak bulunmuştur. Güncel genotiplendirme dizileri insan genomu bilinen tüm varyasyonları dahil değildir. Bunun yerine, onlar insan genomunun içinde LD istismar ve LD 8 belirli bir bölgedeki diğer varyantları için temsilci olarak vekaleten oy bilinen çeşitleri bir alt kümesini içerir. anlamlı bir biyolojik etki ile nedensel varyantı-varyantı ile LD çünkü Böylece herhangi bir biyolojik sonucu olmayan bir varyant, belirli bir hastalık ile ilişkili olabilir. Prosedürel, 1,000 genomları son sürümü plink 10,11, tüm genom dernek analizi için bir açık kaynak aracı ile uyumlu ikili dosyaları içine 9 varyant çağrı dosyalarını (vcf) proje dönüştürmek için tavsiye edilir. Daha sonra, her giriş genetik va ile LD r 2> 0.8 ile diğer tüm genetik varyantlarRiant adayları olarak tespit edilebilir. Bir varyant Avrupa kökenli kişilerde, benzer kökenli konularda veri LD genişlemesi için kullanılması gerektiğini tespit edilmiştir eğer, bu üvey örneğin için uygun referans nüfusu kullanmak önemlidir.
LD genişletme genellikle aday türevleri onlarca neden olur ve sadece küçük bir fraksiyonu bir hastalık mekanizmasına katkıda bulunması olasıdır. Genellikle, deneysel bireysel bu varyantların her incelemek için olanaksız olduğunu. Varyantlar öncelik filtre olarak kamuya açık fonksiyonel genomik veri setleri binlerce kaldıraç nedenle faydalıdır. Örneğin, şifreleme konsorsiyum 12, örneğin, DNase seq 13 ATAC olarak teknolojilerden kromatin erişilebilirlik verileri ile birlikte, bağlamlarda çeşitli yonga-DİZ TFs bağlanmasını tarif eden deneyler ve ko-faktörler, ve histon işaretleri binlerce yürüttü -DİZ 14 ve FAIRE seq 15. databBöyle UCSC Genom Tarayıcı 16, Yol Haritası Epigenomics 17, Blueprint epigenom 18 Cistrome 19 ve eşleştirmek 20 olarak ases ve web sunucuları hücre tipleri ve koşullar geniş bir yelpazede bu ve diğer deneysel tekniklerle üretilen verilere ücretsiz erişim sağlar. Deneysel olarak incelemek için pek çok çeşidi vardır, bu veri, ilgili hücre ve doku tiplerinde olası düzenleyici bölgeler içinde yer alan bu öncelik için kullanılabilir. Bundan başka, bir varyantı belirli bir protein için bir çip DİZİ zirve içinde olduğu durumlarda, bu veriler, belirli bir TF (ler) olan ya da etki olabilir bağlama ko-faktör olarak potansiyel neden sağlayabilir.
Sonraki, öncelik ortaya çıkan varyantları EMSA 21,22 kullanarak bağlama tahmin genotip-bağımlı protein doğrulamak için deneysel taranmaktadır. EMSA bir indirgeyici olmayan TBE jeli üzerinde oligo geçiş değişimi ölçer. Floresan etiketli oligo kuluçkalanırNükleer lizat ve nükleer faktörlerin bağlayıcı jel üzerinde oligo hareketini geciktirir. Bu şekilde, tarama sırasında daha güçlü bir floresan sinyal olarak sunacak daha fazla nükleer faktörler bağlı olan oligo içinde. Özellikle, EMSA bağlayıcı etkilenir spesifik protein ilgili tahminlerde gerektirmez.
varyantlar tahmin düzenleyici bölgeler içinde yer alan ve farklı şekilde bağlayıcı nükleer faktörlerin yeteneğine sahip olduğu tespit edildikten sonra, hesaplama yöntemleri olan onlar etkileyebilecek bağlayıcı belirli TF (ler) tahmin etmek istihdam edilmektedir. Biz BDT-BP 23,24, RegulomeDB 25, UniProbe 26 ve Jaspar 27 kullanmayı tercih eder. Aday TF'ler belirlendikten sonra, bu öngörüler özellikle bu TFs (EMSA-Süper kaymalar ve DAPA-Batılıların) karşı antikorlar kullanılarak test edilebilir. Bir EMSA-süperkayma nükleer lizat ve oligo bir TF-özgü antikorun eklenmesini kapsamaktadır. Bir EMSA-süperkayma bir pozitif sonuç repr olduğunuEMSA bandında bir kayma ya da grubun bir kayıp olarak esented (referans 28'de incelenmiştir). Tamamlayıcı DAPA, varyant ve yan nükleotidlerini 20 baz çifti ihtiva eden bir 5'-biyotinlenmiş oligo dupleks spesifik oligolar bağlama nükleer faktörü yakalamak için uygun bir hücre tipinde (ler) den, nükleer lizatı ile inkübe edilir. Oligo dubleks nükleer faktör kompleksi manyetik sütunda mikroboncuklarm streptavidin tesbit edilir. Bağlı nükleer faktörler elüsyon 29,48 doğrudan tahsil edilir. Bağlanma tahminler daha sonra proteine özel antikorlar kullanılarak bir Western blot ile tespit edilebilir. sonradan kullanılarak valide edilebilir hiçbir belirgin tahminler, ya da çok fazla tahminler, kütle-spektrometresi kullanılarak aday TFs belirlemek için bir proteomik çekirdeğe gönderilebilir DAPA deneyleri varyant aşağı açılır menüden elüsyon olduğu durumlarda, bu daha önce açıklanan yöntemleri.
articl kalanındae, genetik varyantları EMSA ve DAPA analizi için detaylı bir protokol sağlanır.
Protokol
Çözümler ve Reaktiflerin hazırlanması 1.
- EMSA ve DAPA kullanılmak üzere özel DNA oligonükleotid probları sipariş edin.
- Kısa oligos tasarım spesifik olmayan bağlanma proteini azaltmak için 30, ve 17 bp'lik endojen genomik sekans ile çevrili merkezi doğrudan özel bir varyantı yer (35-45 baz uzunluğunda çifti (bp) arasında). EMSA oligo için, bir 5 'fluorofor ekleyin. DAPA oligos için, 5 'biotin etiketini ekleyin.
- duyu ipliği ve ters tamamlayıcı iplikçik hem sipariş edin. Alternatif olarak, sipariş dubleks (pre-tavlı) oligos. oligos adlandırırken, yerleşmiş bir referans genom üzerinde isimlendirme temel.
Not: "Risk" ve hastalık ve projeye özel olabilir "non-riski" atama "referans" ve "referans olmayan" daha evrensel alakalı ise. - oligo Girişte, kısa bir süre için 100 u arasında nihai bir konsantrasyona kadar nükleaz içermeyen su içeriği ve tekrar süspansiyon aşağı spinM. -20 ° C'de stoklarını yeniden süspanse edildi. alüminyum folyo ile sararak ışıktan bir fluorofor ile etiketlenmiş oligos koruyun.
| isim | Sıra |
| rs76562819_REF_FOR | GTAATGCCTTAATGAGAGAG bir GTTAGTCATCTTCTCACTTC |
| rs76562819_REF_REV | GAAGTGAGAAGATGACTAAC T CTCTCTCATTAAGGCATTAC |
| rs76562819_NONREF_FOR | GTAATGCCTTAATGAGAGAG G GTTAGTCATCTTCTCACTTC |
| rs76562819_NONREF_REV | GAAGTGAGAAGATGACTAAC Cı CTCTCTCATTAAGGCATTAC |
Tablo 1: Örnek EMSA / DAPA oligonükleotid tasarımı diferansiyel bin bir SNP test etmek"NONREF" referans dışı alel açılımı ise ding. "REF", referans alel anlamına gelir. "İÇİN" "REV" onun tamamlayıcısı gösterir iken, ileri iplikçik anlamına gelir. SNP kırmızı görülmektedir.
- bir son 10 mM HEPES (pH 7.9) 'ın konsantrasyonunun, 10 mM KCI ve deiyonize su içinde 0.1 mM EDTA ile sitoplazmik ekstraksiyon (CE) tamponu hazırlayın.
- Nükleer ekstraksiyon (NE) bir son 20 mM HEPES (pH 7.9) 'ın konsantrasyonunun, 0.4 M NaCI ve deiyonize su içinde 1 mM EDTA tampon hazırlayın.
- bir son 10 mM Tris (pH 7,5-8,0) konsantrasyonu, 50 mM NaCI ve deiyonize su içinde 1 mM EDTA ile tavlama tamponu hazırlayın.
Kültürlenmiş hücrelerin çekirdekleri, lizat 2. Hazırlık
Not: Bu deneysel protokol B limfoblastoid hücre çizgileri kullanılarak optimize edilmiştir, ancak birkaç diğer ilgisiz yapışkan / süspansiyon hücre hatları test edilmiş ve evrensel olarak çalışır.
- Cultu2 mM L-glutamin,% 10 fetal sığır serumu ve penisilin, 100 ünite / ml streptomisin, 100 ug / ml ihtiva eden 1x antibiyotik antimikotik ve 250 ng Roswell Park Memorial Institute (RPMI) 1640 B-lenfoblastoid hücreleri, yeniden amfoterisin B / ml
- 200,000-500,000 canlı hücre / ml 'lik bir mesafeden tohum ve delikli veya gevşek kapaklı dik olarak% 5 karbon dioksit ile 37 ° C'de şişeler inkübe edin.
Not: 1.000.000 hücre / ml ulaştığında B-lenfoblastoid hücrelerin büyümesi yavaşlar. yukarı pipetleme ve birkaç kez aşağı hücre patlamalar break up ve hızlı bir büyüme oranını korumak için 200,000-500,000 hücre / ml hücreleri dönün.
- 200,000-500,000 canlı hücre / ml 'lik bir mesafeden tohum ve delikli veya gevşek kapaklı dik olarak% 5 karbon dioksit ile 37 ° C'de şişeler inkübe edin.
- 5 dakika boyunca, 4 ° C'de, 300 xg durması ve aspirasyonla PBS kaldırmak, iki kez 10 ml buz soğukluğundaki fosfat tamponlu salin (PBS) ile kültürlenen hücreler yıkanır.
- 10 7 hücre başına soğuk PBS 1 ml buz gibi bir hemositometre ve tekrar süspansiyon pelet kullanarak hücreleri saymak.
Not: Örneğin, 2 x 10 7 lize halinde </ Sup> hücreler, 2 ml PBS içinde tekrar süspansiyon. - Kısım 1.5 ml mikrosantrifüj tüplerine 1 mi, her boru PBS içinde 10 7 hücre içerecek şekilde. PBS kapalı 2 dakika 4 ° C ve aspirat için 3300 xg'de santrifüj.
- Kullanmak CE tampon çalışma stok 1 mM ditiyotreitol (DTT), 1 x fosfataz inhibitörü ve 1 x proteaz inhibitörü ilave önce. Tekrar süspansiyon hücre CE tampon 400 ul pelet ve 15 dakika boyunca buz üzerinde inkübe edilir.
- % 10 Nonidet P-40 25 ul ekleyin ve pipetleme karıştırın. 4 ° C, 3 dakika boyunca maksimum hızda santrifüj. Durusu ve supernatant atın.
- Kullanmak NE tampon çalışma stok 1 mM DTT, 1 x fosfataz inhibitörü ve 1 x proteaz inhibitörü ilave önce. Tekrar süspansiyon hücre NE tamponu 30 ul pelet ve vorteks karıştırın.
- tüp rotator veya 10 dakika buz üzerinde 4 ° C'de inkübe edin. Santrifüj 2 dakika 4 ° C 3300 xg.
- En saklamadan önce berrak süpernatant (nükleer lizat) ve kısım toplayın -806 C proteini düşebilir birden donma-çözülme döngüleri önlemek için. Bichoninic asit deneyi (BCA) 31 kullanarak protein konsantrasyonunu ölçmek için bir 10 ul tablet bırakın.
3. Elektroforetik Devingenlik Değişimi Analizi (EMSA)
- Oligo Çalışma Stok ve EMSA Jel hazırlayın.
- oligos dubleks sipariş olsaydı, 100 mcM stok çözülme ve 1 sulandırmak: 2.000 tavlama tamponunda 50 nM çalışma stok elde etmek.
- oligo tek şeritli sipariş edilen ise, 100 uM stokları çözünmesi ve 100 nM çalışma stokları elde etmek için tavlama tamponu içinde 1:10 oranında seyreltin. mikrosantrifüj tüpü içinde birbirleri ile 100 nM tamamlayıcı zinciri çözeltisi 100 ul birleştirin.
- 5 dakika boyunca 95 ° C'de bir ısı bloğunda yerleştirin. Isı bloğu kapatın ve oligo yavaş yavaş kullanımdan önce en az bir saat boyunca oda sıcaklığına kadar soğumasını bekleyin.
- EMSA Gel Öncesi çalıştırın.
- bir ön döküm% 6 T slaytı kaldırmakjel BE ve kuyulardan herhangi bir tampon kaldırmak için deiyonize suyun altında birkaç kez durulayın. iyonu giderilmiş su, 950 ml 10x TBE, 50 ml ekleyerek 0.5x TBE tamponu 1 L hazırlayın.
- jel elektroforezi cihazı monte edin ve 0.5x TBE tamponu ile iç odasını doldurarak sızıntı olup olmadığını kontrol edin. tampon, dış odasına sızar ise, dış bölmesini şekilde kabaca üçte ikisi doldurun.
- 60 dakika boyunca 100 V'ta jel ön çalıştırın.
- 0.5x TBE tamponu 200 ul her iyi yıkayın.
- Tampon Master Mix Cilt hazırlayın.
- 100 mM Tris, 500 mM KCI, 10 mM DTT nihai bir konsantrasyona sahip bağlayıcı tampon 10x hazırlanması; iyonu giderilmiş su içinde, pH 7.5 olarak değiştirilmişti.
- Mikrosantrifüj tüpü içinde, bütün reaksiyonlar (Tablo 2 ise 10 ul 10x bağlanma tamponu, 10 ul DTT / polisorbat, 5 ul Poli D (IC) ve 2.5 ul somon sperm DNA) ortak olan maddeler arasında meydana gelen bir ana karışımı oluşturur. ek bir% 10 hazırlayınpipetleme nedeniyle hacim kaybı hesaba.
| reaktif | Final Kons. | 1. rxn | 2. rxn | 3. rxn | 4. rxn |
| Ultra Saf Su | 20 ul hacme. | 13.5 ul | 11.98 ul | 13.5μl | 11.98 ul |
| 10x Bağlama Tamponu | 1x | 2 ul | 2 ul | 2 ul | 2 ul |
| DTT / TW-20 | 1x | 2 ul | 2 ul | 2 ul | 2 ul |
| Salmon sperm DNA'sı | 500 ng / | il | 0.5 ul | 0.5 ul | 0.5 ul | 0.5 ul |
| 1 ug / ul Poli d (IC) | 1 ug | 1 ul | 1 ul | 1 ul | 1 ul |
| Nükleer Ekstrakt (5.26 ug / ul), | 8 ug | - | 1.52 ul | - | 1.52 ul |
| NE Tampon | 1.52 ul | - | 1.52 ul | - | |
| Referans alel oligo | 50 fmol | 1 ul | 1 ul | - | - |
| Sigara Referans alel oligo | 50 fmol | - | - | 1 ul | 1 ul |
Tablo 2: Örnek EMSA reaksiyon setus. tablo genotip-bağımlı belirli bir SNP için TFs bağlanmasının olduğunu hipotezini test etmek için bir örnek EMSA göstermektedir.
- gibi tüm reaktifler ilave edildikten sonra nihai hacim 20 ul olacak her mikrosantrifüj tüpüne nükleaz içermeyen su ilave edilir.
- Her mikrosantrifüj tüp ana karışımı uygun miktarda (5.5 ul) ekleyin.
- Uygun mikrosantrifüj tüpler nükleer lizat 8 mikrogram ekleyin. Nükleer negatif kontroller olarak ekstraktı (Tablo 2, Rxn 1. ve Rxn # 3) olmadan oligo içeren tüpler ekleyin.
Not: reaksiyon başına lizat optimal miktarı titrasyonu ile deneysel olarak tespit edilmelidir. Genel olarak, lizat 2-10 ug bir dizi titre yeterlidir. - Uygun mikrosantrifüj tüplerine oligo 50 fmol ekleyin. Flick karıştırın ve kısaca tüpün dibine içeriğini döndürmek için. Oda sıcaklığında 20 dakika boyunca inkübe edin.
Not: Eğer attemptive bir süper ng oligo ilavesinden önce oda sıcaklığında 20 dakika boyunca antikor ile lizat karışımı inkübe edin. En iyi sonuç için bir ChIP dereceli antikor 1 mikrogram kullanılması tavsiye edilir. - Her mikrosantrifüj tüpüne 10x Turuncu Yükleme Dye 2 ul ekleyin. yukarı pipet ve aşağı karıştırın.
- Ayrı bir kuyuya her örnek kovma sonra yukarı ve aşağı pipetleme karıştırmak ve öncesi işletilen% 6 TBE jel içine örnekleri yükleyin. turuncu boya jel yol aşağı 3/4 2/3 göç kadar 80 V jel çalıştırın. Bu, yaklaşık 60-75 dakika sürer.
- Bir jel bıçakla açık meraklı plastik kaset jel çıkarın ve kurumasını tutmak için% 0.5 TBE tamponu ile bir kapta jel yerleştirin.
- Görüntüyü bozacak herhangi bir kabarcıkları veya kirletici ortadan kaldırmak için emin olmak, kızılötesi ve chemilüminesan görüntüleme sistemi yüzeyinde jel yerleştirin.
- tarama sistemi yazılımı kullanarak, "Acquire" sekmesini tıklayınve daha sonra jel tarayıcının yüzeyinde bulunduğu karşılık gelen alanı çevresinde bir kutu çizmek için "Yeni Çiz" seçeneğini seçin.
- "Edinme" sekmesinin "Kanallar" bölümünde, oligo üzerinde florofor etiketinin dalga boyuna karşılık gelen kanalı seçin. "Tarayıcı" bölümünde, bir düşük kaliteli önizleme taraması almak için "önizleme" butonuna tıklayınız. jel kısmına aşağı edinilen Önizleme görüntüsünü çevreleyen mavi kutu sürükleyerek tarama alanını ayarlayın yansıması.
Not: Örneğin, 700 nm florofor ile işaretlenmiş oligos kullanıyorsanız, "700 nm" kanal taramadan önce seçili olduğundan emin olun. - "Tarama Kontroller" bölümünde, "84 uM" çözünürlük seçeneği ve "Orta" kalite seçeneği seçin. jel kalınlığının yarısına ofset odağı ayarlayın. Not: Örneğin, 1 mm'lik bir jel ofset 0.5 mm'lik bir odak kullanmak.
- "Tarayıcı" bölümünde, b "Başlat" a tıklayınTarama Egin.
Not: Tarama sırasında, parlaklık, kontrast ve renk düzeni genellikle tarama sisteminin üreticisine bağlı olarak manuel olarak ayarlanabilir. - Tarama bittikten sonra, "Resim" sekmesini seçin ve yönünü düzeltmek için "oluşturma" bölümünde "Döndür veya çevirin" tıklayın. Ana menüde "Export" tıklayarak görüntü dosyasını kaydedin ve ardından "Tek Resim Görünümü."
4. DNA çekimi saflaştırma deneyi (DAPA)
- 5 uM Oligo Çalışma Stokta hazırlanması.
- oligos dubleks sipariş olsaydı, 100 uM stok çözülme ve 5 uM çalışma stok elde etmek için tavlama tamponunda 1:20 sulandırmak.
- oligo tek iplikli sipariş olsaydı, 100 uM stokları çözülme ve stokları çalışan 10 mcM ulaşmak için tavlama tamponunda 1:10 sulandırmak. birbirleriyle 10 uM tamamlayıcı zincirlerinde 10 ul birleştirin. 95 ° C de bir ısı bloğunda yerleştirin5 dakika. Isı bloğu kapatın ve oligo yavaş yavaş kullanımdan önce oda sıcaklığına kadar soğumasını bekleyin.
- başlamadan önce, oda sıcaklığında bağlanma tamponu, düşük sıkılık yıkama tamponu, yüksek sertlikte yıkama tamponu ve elüsyon tamponu ısıtın.
Not: 50 ng nihai bir konsantrasyon / ml poli D (IC) 'oligo proteinlerin potansiyel spesifik olmayan bağlanmaların azaltmak için bağlama tamponu düşük sıkılık yıkama tamponu ve yüksek sertlikte yıkama tamponu ilave edilebilir. - Her varyant için bağlayıcı karışımlar hazırlayın.
- bağlama tamponu 2 hacim nükleer lizat 1 hacim karıştırın.
Not: lizat gerekli miktar bağlı TF'lerin bolluğu değişen deneysel olarak tespit edilmelidir. Sütun başına nükleer lizat ug 100-250 arasında kullanılması çoğu durumda yeterlidir. - 1x fosfataz inhibitörü, 1x proteaz inhibitörü ve 1x bağlayıcı arttırıcı (isteğe bağlı) ekleyin ve tüpü birkaç kez hafifçe vurarak karıştırın.
Not: 100x bağlayıcıgüçlendirici, 750 mM MgCl2 ve 300 mM ZnCI2 oluşur. DNA TF bağlayıcı kofaktör veya indirgeyici ajanlar bağlıdır eğer bağlayıcı geliştirici ekleyin. Bu bilgi bilinmiyorsa, bağlanma arttırıcı ekleyin. - her bir ilgili bağlama karışımına 5 uM biyotinlenmiş yakalama DNA (50 mmol) 10 ul ekle. Oda sıcaklığında 20 dakika boyunca inkübe edin.
Not: İnkübasyon süresi ve sıcaklık, TF bağlı olarak değişebilir. uygun değerler deneysel olarak tespit edilmesi gerekmektedir.
- bağlama tamponu 2 hacim nükleer lizat 1 hacim karıştırın.
- streptavidin mikro boncuklar 100 ul ekleyin. Oda sıcaklığında 10 dakika boyunca inkübe edin.
- Her oligo probu test edilen, manyetik ayırıcı bir bağlayıcı sütun yerleştirin. Her bağlayıcı sütununun altında doğrudan ependorf tüp yerleştirin ve sütun durulama bağlayıcı tampon 100 ul geçerlidir.
- ayrı sütunlar halinde her bağlayıcı karışımın içeriğini pipet ve sıvı mikrosantrifüj içine sütun aracılığıyla tamamen akmasına izinDevam etmeden önce tüp. bağlayıcı karışımında kullanılan varyant oligo sütunları etiket emin olun. akış örnekleri etiket ve düşük sertlikteki yıkar toplamak için yeni mikrosantrifüj tüpler ile değiştirin.
- sütununa düşük sertlikteki yıkama tamponu 100 ul uygulayın; Kolon rezervuar boşalana kadar bekleyin. yıkama 4x tekrarlayın. Düşük sıkılık yıkama örnekleri etiket ve yüksek sertlikte yıkar toplamak için yeni mikrosantrifüj tüpler ile değiştirin.
- sütununa yüksek sertlikteki yıkama tamponu 100 ul uygulayın; Kolon rezervuar boşalana kadar bekleyin. yıkama 4x tekrarlayın. yüksek sertlikteki yıkama örnekleri etiket ve ön elüsyon toplamak için yeni mikrosantrifüj tüpler ile değiştirin.
- sütun özgü elüsyon tamponu 30 ul ilave edin ve 5 dakika boyunca bekletin. Ön yıkama örnekleri Etiket ve elüsyon toplamak için yeni mikrosantrifüj tüpler ile değiştirin.
Not: Bu ilişkili protein elüt etmez; geri kalan, yüksek sertlikteki yıkamalarsütunun üzerinden tamponu ve elüsyon verimliliğini en üst düzeye çıkarmak için yıkama tamponu ile değiştirir. - bağlı TFs Zehir için ek 50 ul yerli elüsyon tamponu ekleyin. daha yüksek bir verim daha az konsantre edilmiş, ayrılmış malzeme için, ana elüsyon tamponu ek bir 50 ul ve akış-through toplar.
Not: bağlı TFs 32 kimliğini belirlemek için kütle spektrometresi ile elüsyon örnekleri analiz edin. Daha sonra, bir Western Blot 33 tarafından takip sodyum dodesil sülfit poliakrilamid jel elektroforezi (SDS-PAGE) ile proteomik sonuçları doğrulamak. kütle spektrometrisi mevcut değilse, genotip bağımlı bağlanma gösteren protein (ler) boyutunu belirlemek için yerine Western blot standart teknik kullanılarak gümüş leke çalıştırın. Girişte detaylı hesaplama yaklaşımları tahmin TFs listesini daraltmak için bu bilgileri kullanabilirsiniz.
Sonuçlar
lizat kalitesi özelliği açısından bir EMSA veya DAPA ve değişkenliği yaparken Bu bölümde, ne temsilcisi sonuçları verilmektedir. Örneğin, birden çok kez dondurma ve çözme, protein örnekleri denatürasyon neden olabilir öne sürülmüştür. Bu "dondurma-çözme" döngü bağlamında EMSA analizi tekrarlanabilirliğini incelemek için, bir gen varyant farklı iki adet 35 bp'lik oligo belirtilen miktarda çözülmüş ve yeniden dondurulmuştur nükleer liz...
Tartışmalar
sıralama ve genotiplendirme teknolojilerindeki gelişmeler büyük ölçüde hastalıkla ilgili genetik varyantları tanımlamak için kapasitemizi gelişmiş olmasına rağmen, bu varyantlar etkilenen fonksiyonel mekanizmaları anlamak için yeteneğimizi kalıyor. Sorunun ana kaynağı olasılıkla gen ekspresyonunu kontrol zor-arası tahmin mekanizmaları etkileyen ilgili kodlama genomunun bölgeler birçok hastalıkla ilişkili varyantları n yer olmasıdır. Burada, biz büyük olasılıkla çok kodlayıcı ...
Açıklamalar
Yazarlar ifşa hiçbir şey yok.
Teşekkürler
We thank Erin Zoller, Jessica Bene, and Lindsey Hays for input and direction in protocol development. MTW was supported in part by NIH R21 HG008186 and a Trustee Award grant from the Cincinnati Children's Hospital Research Foundation. ZHP was supported in part by T32 GM063483-13.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Custom DNA Oligonucleotides | Integrated DNA Technologies | http://www.idtdna.com/site/order/oligoentry | |
| Potassium Chloride | Fisher Scientific | BP366-500 | KCl, for CE buffer |
| HEPES (1 M) | Fisher Scientific | 15630-080 | For CE and NE buffer |
| EDTA (0.5M), pH 8.0 | Life Technologies | R1021 | For CE, NE, and annealing buffer |
| Sodium Chloride | Fisher Scientific | BP358-1 | NaCl, for NE buffer |
| Tris-HCl (1M), pH 8.0 | Invitrogen | BP1756-100 | For annealing buffer |
| Phosphate Buffered Saline (1x) | Fisher Scientific | MT21040CM | PBS, for cell wash |
| DL-Dithiothreitol solution (1 M) | Sigma | 646563 | Reducing agent |
| Protease Inhibitor Cocktail | Thermo Scientific | 87786 | Prevents catabolism of TFs |
| Phosphatase Inhibitor Cocktail | Thermo Scientific | 78420 | Prevents dephosphorylation of TFs |
| Nonidet P-40 Substitute | IBI Scientific | IB01140 | NP-40, for nuclear extraction |
| BCA Protein Assay Kit | Thermo Scientific | 23225 | For measuring protein concentration |
| Odyssey EMSA Buffer Kit | Licor | 829-07910 | Contains all necessary EMSA buffers |
| TBE Gels, 6%, 12 Wells | Invitrogen | EC6265BOX | For EMSA |
| TBE Buffer (10x) | Thermo Scientific | B52 | For EMSA |
| FactorFinder Starting Kit | Miltenyi Biotec | 130-092-318 | Contains all necessary DAPA buffers |
| Licor Odyssey CLx | Licor | Recommended scanner for DAPA/EMSA | |
| Antibiotic-Antimycotic | Gibco | 15240-062 | Contains 10,000 units/ml of penicillin, 10,000 µg/ml of streptomycin, and 25 µg/ml of Fungizone® Antimycotic |
| Fetal Bovine Serum | Gibco | 26140-079 | FBS, for culture media |
| RPMI 1640 Medium | Gibco | 22400-071 | Contains L-glutamine and 25 mM HEPES |
Referanslar
- Hindorff, L. A., et al. Potential etiologic and functional implications of genome-wide association loci for human diseases and traits. Proc Natl Acad Sci U S A. 106 (23), 9362-9367 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Ward, L. D., Kellis, M. Interpreting noncoding genetic variation in complex traits and human disease. Nat Biotechnol. 30 (11), 1095-1106 (2012).
- Paul, D. S., Soranzo, N., Beck, S. Functional interpretation of non-coding sequence variation: concepts and challenges. Bioessays. 36 (2), 191-199 (2014).
- Zhang, F., Lupski, J. R. Non-coding genetic variants in human disease. Hum Mol Genet. , (2015).
- Lee, T. I., Young, R. A. Transcriptional regulation and its misregulation in disease. Cell. 152 (6), 1237-1251 (2013).
- Slatkin, M. Linkage disequilibrium--understanding the evolutionary past and mapping the medical future. Nat Rev Genet. 9 (6), 477-485 (2008).
- Bush, W. S., Moore, J. H. Chapter 11: Genome-wide association studies. PLoS Comput Biol. 8 (12), e1002822 (2012).
- 1000 Genomes Project Consortium. An integrated map of genetic variation from 1,092 human genomes. Nature. 491 (7422), 56-65 (2012).
- Chang, C. C., et al. Second-generation PLINK: rising to the challenge of larger and richer datasets. Gigascience. 4, 7 (2015).
- Purcell, S., et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 81 (3), 559-575 (2007).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Crawford, G. E., et al. Genome-wide mapping of DNase hypersensitive sites using massively parallel signature sequencing (MPSS). Genome Res. 16 (1), 123-131 (2006).
- Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y., Greenleaf, W. J. Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nat Methods. 10 (12), 1213-1218 (2013).
- Giresi, P. G., Kim, J., McDaniell, R. M., Iyer, V. R., Lieb, J. D. FAIRE Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome Res. 17 (6), 877-885 (2007).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Roadmap Epigenomics Consortium. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Martens, J. H., Stunnenberg, H. G. BLUEPRINT: mapping human blood cell epigenomes. Haematologica. 98 (10), 1487-1489 (2013).
- Liu, T., et al. Cistrome: an integrative platform for transcriptional regulation studies. Genome Biol. 12 (8), R83 (2011).
- Griffon, A., et al. Integrative analysis of public ChIP-seq experiments reveals a complex multi-cell regulatory landscape. Nucleic Acids Res. 43 (4), e27 (2015).
- Staudt, L. M., et al. A lymphoid-specific protein binding to the octamer motif of immunoglobulin genes. Nature. 323 (6089), 640-643 (1986).
- Singh, H., Sen, R., Baltimore, D., Sharp, P. A. A nuclear factor that binds to a conserved sequence motif in transcriptional control elements of immunoglobulin genes. Nature. 319 (6049), 154-158 (1986).
- Weirauch, M. T., et al. Determination and inference of eukaryotic transcription factor sequence specificity. Cell. 158 (6), 1431-1443 (2014).
- Ward, L. D., Kellis, M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 40 (Database issue), D930-D934 (2012).
- Boyle, A. P., et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22 (9), 1790-1797 (2012).
- Hume, M. A., Barrera, L. A., Gisselbrecht, S. S., Bulyk, M. L. UniPROBE, update 2015: new tools and content for the online database of protein-binding microarray data on protein-DNA interactions. Nucleic Acids Res. 43 (Database issue), D117-D122 (2015).
- Mathelier, A., et al. JASPAR 2014: an extensively expanded and updated open-access database of transcription factor binding profiles. Nucleic Acids Res. 42 (Database issue), 142-147 (2014).
- Smith, M. F., Delbary-Gossart, S. Electrophoretic Mobility Shift Assay (EMSA). Methods Mol Med. 50, 249-257 (2001).
- Franza, B. R., Josephs, S. F., Gilman, M. Z., Ryan, W., Clarkson, B. Characterization of cellular proteins recognizing the HIV enhancer using a microscale DNA-affinity precipitation assay. Nature. 330 (6146), 391-395 (1987).
- . BCA Protein Assay Kit: User Guide Available from: https://tools.thermofisher.com/content/sfs/manuals/MAN0011430_Pierce_BCA_Protein_Asy_UG.pdf (2014)
- Wijeratne, A. B., et al. Phosphopeptide separation using radially aligned titania nanotubes on titanium wire. ACS Appl Mater Interfaces. 7 (21), 11155-11164 (2015).
- Silva, J. M., McMahon, M. The Fastest Western in Town: A Contemporary Twist on the Classic Western Blot Analysis. J. Vis. Exp. (84), (2014).
- Lu, X., et al. Lupus Risk Variant Increases pSTAT1 Binding and Decreases ETS1 Expression. Am J Hum Genet. 96 (5), 731-739 (2015).
- Ramana, C. V., Chatterjee-Kishore, M., Nguyen, H., Stark, G. R. Complex roles of Stat1 in regulating gene expression. Oncogene. 19 (21), 2619-2627 (2000).
- Fillebeen, C., Wilkinson, N., Pantopoulos, K. Electrophoretic Mobility Shift Assay (EMSA) for the Study of RNA-Protein Interactions: The IRE/IRP Example. J. Vis. Exp. (94), e52230 (2014).
- Heng, T. S., Painter, M. W. Immunological Genome Project, C. The Immunological Genome Project: networks of gene expression in immune cells. Nat Immunol. 9 (10), 1091-1094 (2008).
- Wu, C., et al. BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol. 10 (11), R130 (2009).
- Wu, C., Macleod, I., Su, A. I. BioGPS and MyGene.info: organizing online, gene-centric information. Nucleic Acids Res. 41 (Database issue), D561-D565 (2013).
- Wang, J., et al. Sequence features and chromatin structure around the genomic regions bound by 119 human transcription factors. Genome Res. 22 (9), 1798-1812 (2012).
- Holden, N. S., Tacon, C. E. Principles and problems of the electrophoretic mobility shift assay. J Pharmacol Toxicol Methods. 63 (1), 7-14 (2011).
- Xu, J., Liu, H., Park, J. S., Lan, Y., Jiang, R. Osr1 acts downstream of and interacts synergistically with Six2 to maintain nephron progenitor cells during kidney organogenesis. Development. 141 (7), 1442-1452 (2014).
- Yang, T. -. P., et al. Genevar: a database and Java application for the analysis and visualization of SNP-gene associations in eQTL studies. Bioinformatics. 26 (19), 2474-2476 (2010).
- Fort, A., et al. A liver enhancer in the fibrinogen gene cluster. Blood. 117 (1), 276-282 (2011).
- Solberg, N., Krauss, S. Luciferase assay to study the activity of a cloned promoter DNA fragment. Methods Mol Biol. 977, 65-78 (2013).
- Rahman, M., et al. A repressor element in the 5'-untranslated region of human Pax5 exon 1A. Gene. 263 (1-2), 59-66 (2001).
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır