需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
模拟信号蛋白的功能:走向人工信号转导疗法
摘要
We present guidelines for developing synthetic 'chemical transducers' that can induce communication between naturally unrelated proteins. In addition, detailed protocols are presented for synthesizing and testing a specific 'transducer' that enables a growth factor to activate a detoxifying enzyme and consequently, to regulate the cleavage of an anticancer prodrug.
摘要
Signal transduction pathways, which control the response of cells to various environmental signals, are mediated by the function of signaling proteins that interact with each other and activate one other with high specificity. Synthetic agents that mimic the function of these proteins might therefore be used to generate unnatural signal transduction steps and consequently, alter the cell's function. We present guidelines for designing 'chemical transducers' that can induce artificial communication between native proteins. In addition, we present detailed protocols for synthesizing and testing a specific 'transducer', which can induce communication between two unrelated proteins: platelet-derived growth-factor (PDGF) and glutathione-S-transferase (GST). The way by which this unnatural PDGF-GST communication could be used to control the cleavage of an anticancer prodrug is also presented, indicating the potential for using such systems in 'artificial signal transduction therapy'. This work is intended to facilitate developing additional 'transducers' of this class, which may be used to mediate intracellular protein-protein communication and consequently, to induce artificial cell signaling pathways.
引言
信号转导途径在几乎所有的细胞过程中发挥显著作用,并允许细胞迅速环境信号作出反应。1这些途径通常由结合的信号传导分子的触发以胞外受体,这导致细胞内酶的活化。扩增和细胞内的该信号的传播受信令形成蛋白质 - 蛋白质相互作用的网络,其中酶可逆地以高特异性活性的蛋白质的功能介导的。因为这些网络的失调经常导致癌症的发展,出现了在建立"的癌症的信号转导的治疗',2由此药物被设计为破坏恶性信号通路的兴趣。我们最近已提出了另一种方法的信号,依赖于药物来产生不自然的信号转导途径的能力转导的治疗。 3特别是,我们相信,通过设计模拟信号蛋白的功能的合成剂,这将有可能间接地调节细胞的功能。例如,这些人工网络可以使蛋白质生物标志物来激活切割前体药物的酶。或者,这些信号蛋白模拟物可能能够激活非天然细胞信号传导途径,导致治疗效果。
为了证明这种方法的可行性,我们最近创建的合成"化学换能器'4,使血小板衍生的生长因子(PDGF)通过激活谷胱甘肽-S-转移酶(GST),这是引发抗癌前药的切割不是其天然结合伴侣。这个'换能器'的结构由与二价抑制剂为GST改性的抗PDGF的DNA适体。因此,该合成剂属于家族分子与结合位点不同的蛋白质,二聚的5-7如化学诱导剂(的CID)8-10并且还基于寡核苷酸的合成分子偶联物的组蛋白粘合剂。11-21
这种系统的设计基础的一般原理,本文描述和提供了用于合成和测试该常规酶测定"换能器"的功能的详细协议。这项工作是为了便于开发这一类,其可以用于介导的细胞内蛋白质 - 蛋白质的通信,因此,以诱导人工细胞信号传导途径的额外的"换能器"。
图1示意性地描述了合成"化学传感器",可以调解不自然的蛋白质-蛋白质通信的工作原理。在该图中,"化学传感器",它集成了普罗特合成粘合剂EINS I和II(粘合剂I和II),使蛋白质二触发蛋白I的催化活性,这不是其天然结合伴侣。在不存在蛋白Ⅱ的,换能器结合的酶(蛋白质I)中的催化位点并抑制其活性( 图1,状态ⅱ)。该'换能器'的蛋白II的结合,然而,促进粘合剂I和蛋白II的表面( 图1,状态ⅲ),这降低了它的朝向蛋白I.亲和力结果,的有效浓度之间的相互作用的'在溶液中自由'换能器被缩小,这导致所述换能器蛋白的解离我复杂和蛋白I的活化( 图1,状态ⅳ)。两者合计,这些步骤突出的高效'换能器'的设计基础三个基本原则:(1)一个"换能器"应具有特定的粘合剂对于每个蛋白质靶的,(2)的相互作用betwe烯粘合剂II和蛋白II应该比粘合剂I和蛋白I,和(3)的粘合剂之间的相互作用我必须能够与蛋白Ⅱ的表面相互作用更强。这最后的原则并不一定要求粘合剂我单独将具有朝向蛋白质二高亲和力和选择性。相反,它是根据我们最近的研究这表明使合成分子接近的蛋白质是可能促进该分子与所述蛋白质的表面之间的相互作用。19,22,23
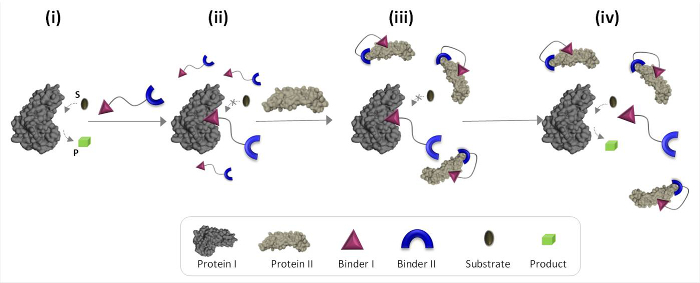
图1:的"化学换能器"的操作原则当"化学传感器"被添加到活性蛋白I(状态i),它通过粘结剂结合至其活性位点I和抑制其活性(状态II)。在蛋白Ⅱ的存在,然而,未结合的'化学吨ransducer'通过粘合剂二,这促进了粘合剂I和蛋白II的表面之间的相互作用与蛋白质II相互作用。这引起粘结剂I-II蛋白质相互作用降低了有效浓度粘结剂我的,这导致了"transducer'蛋白解离我复杂的蛋白质我复活(国IV)。 请点击此处查看该图的放大版本。
Access restricted. Please log in or start a trial to view this content.
研究方案
1."化学传感器"的合成
- 前期准备工作
- 通过混合278毫升三乙胺的应用114毫升乙酸和400ml的超纯水制备的2M乙酸三乙铵(TEAA)缓冲液。调节pH至7,并加水至1L的终体积保持它在黑暗的瓶子。
注意:此溶液是稳定的多年。 - 由18毫克抗坏血酸溶解在20毫升的超纯水制备的5mM抗坏血酸溶液。选用一个新鲜的解决方案;该溶液是稳定的一天。
- 通过在11个毫升二甲基亚砜(DMSO)中25毫克硫酸铜五水合物溶解于毫克TBTA 10毫升的超纯水和58制备的10mM铜(II)/三(benzyltriazolylmethyl)胺(TBTA)溶液。混合两种溶液。保持在室温下,保护它的光。
- 通过混合278毫升三乙胺的应用114毫升乙酸和400ml的超纯水制备的2M乙酸三乙铵(TEAA)缓冲液。调节pH至7,并加水至1L的终体积保持它在黑暗的瓶子。
- 偶联过程
- 在80微升溶解100纳摩尔修饰的寡核苷酸的核苷酸(ODN-1)新鲜的超纯水。加入20μl2M的TEAA,pH值= 7添加的抗坏血酸(5毫在水中)一个新鲜的溶液80微升。
- 溶解叠氮基修饰的利尿酸1.5微摩尔(574.5微克)在180微升DMSO中,并将其添加到该溶液中。脱气用氩为60秒,并迅速从铜(II)/ TBTA溶液(10mM在55%(体积/体积)的DMSO /水)加入40微升溶液。
- 再次用氩气清除,关闭严密,并搅拌过夜。
- 监测反应的进度和纯化通过RP-HPLC的缀合物(流动相:A)5%乙腈,5%TEAA,90%超纯水; B)65%乙腈,5%TEAA,30%超纯水)。24
2. PDGF控制GST活动
- 前期准备工作
- 通过混合33.9磷酸盐缓冲盐水(PBSx1)中的溶液用16.1超纯水的溶液制备将50ml测定缓冲液,以达到一个8毫米的最终磷酸盐浓度和广告ð23.8毫克氯化镁 ,实现一个5毫米的终浓度。
- 通过将蛋白质溶解于含有50mM Tris pH值7.5,50mM的NaCl的缓冲液制备的GST M1-1的贮备溶液,1mM的二硫苏糖醇(DTT)和5mM乙二胺四乙酸(EDTA),以30μM的终浓度。把这个解决方案成小分装和储存在-80℃。稀释新鲜,根据节2.1.5.1,在测定缓冲液,并保持在冰上。
注意:该解决方案将是稳定的约5小时,或直到有在酶的活性的降低。 - 根据以下说明制备的基板:
- 溶解10毫克在325微升的超纯水的原型谷胱甘肽(GSH)的至100mM的储备溶液的最终浓度。从这个原液在979微升分析缓冲液为2.1 mm工作液的终浓度稀释21微升。
- 在492微升电子商务溶解10毫克2,4-二硝基氯苯(CDNB)thanol至100mM的储备溶液的最终浓度。稀从956.8微升的分析缓冲液的原液43.2微升为4.32毫工作液的最终浓度。
- 在一个96孔板,把每个基片在一个单独的行(12个孔)。插入至少60微升每孔以使该溶液的快速和容易的撤出。覆盖铝板光保护板。
- 制备下列储备溶液中的测定缓冲液:
- 稀释的GST M1-1 50到的二聚体0.6μM的终浓度。
- 淡化"化学传感器'到30微米的原液。
- 稀释PDGF至40μm的终浓度。
- 稀释PDGF适体的250μM的最终浓度。
- 测量在"化学传感器"的存在和PDGF的GST活动。
- 搭建了一个实验的过程我N对于动力学测量板读数器。
- 创建一个新的实验,一个"标准的协议"。
- 在"程序"按打开程序设置窗口。
- 在窗口的上侧弹出列表,根据该板的制造商选择'384板"类型。
- 按'读'在左边的菜单上。
- 关于检测方法选择"吸收"。
- 关于读式选择"终点"。
- 写波长窗口340纳米。
- 对右上侧的"完全版"自下而上按,然后选择井来衡量。
- 按"确定"关闭"读取"窗口。
- 选择左侧菜单中的"启动动力"。
- 使运行时间10分钟。
- 选择最小间隔的选项。
- 按"确定"关闭窗口动能。
- 拖动"读取"我行n要动力学测量。
- 按"验证"按钮,然后单击"确定"按钮。
- 保存实验。
- 按"播放"按钮。一个对话框将会出现 - 只按"确定"按钮时,测量应启动。
- 为了执行三次重复实验,准备四个样品每片含3.25微升了"化学传感器"和3.25微升GST M1-1的。添加到每个样品0,1.2,2.4,或4.9微升PDGF和123.5,122.3,121.1的,或118.6微升测定缓冲液中,分别。
- 在室温下孵育该溶液10分钟。
- 在384透明孔板,插入40微升样品的每个孔中。插入只在奇数井或仅在同一行中的偶数孔中的样品,以允许基片除了使用多移液器的。
- 采用12通道多移液器,迅速从各t的加10微升他基材在96孔板(2.1.4节)进行预先准备的。轻轻混匀并迅速以避免气泡。插入板插入读卡器和启动动力学测量。由于GST动力学是相当快的,要尽量减少基底加法和动力学测量开始之间的时间。
- 搭建了一个实验的过程我N对于动力学测量板读数器。
- GST活化的"化学传感器"中介/抑制周期。
- 为了进行三次重复实验中,制备每片含84.5微升测定缓冲液中,3.25微升'化学传感器"的,和3.25微升的GST M1-1的5个样品。在室温下孵育3分钟。
- 添加3.65微升试验缓冲液的样品1和3.65微升PDGF与样品2-5。在室温下孵育3分钟。
- 添加3.12微升试验缓冲液的来样1-2和3.12微升PDGF适体样品3-5。在室温下孵育3分钟。
- 添加24.4微升试验缓冲液的来样1-3和24.4微升至PDGF样品4-5。在室温下孵育3分钟。
- 添加7.8微升测定缓冲液以样品1-4和7.8微升PDGF适体在室温下采样5.孵育5分钟。
- 在384透明孔板,插入40微升样品放入每个孔中。插入样本仅入奇数或仅将在同一行中的偶数孔中。
- 使用12通道多吸移管,迅速地从各底板添加10微升(在96孔板预先制备的)。轻轻混匀并迅速以避免气泡。插入板插入读卡器和启动动力学测量。
- 通过减去在在t 340毫微米=从外径0.5分钟,在t 340毫微米= 1.5分钟测量,以评估激活/抑制可回收测得的OD值计算每个条件下在V 0 [MOD /分钟]。25
- 评估"化学传感器"在环境变化的实时响应。
- PDGF除了实时生效
- 设置在板读数器用于动力学测定的实验程序。
- 重复步骤2.2.1.1-2.2.1.10。
- 使运行时间3.5分钟。
- 选择最小间隔的选项。
- 按"确定"关闭窗口动能。
- 在"读取"线拖到动力学测量。
- 选择盘开/在左边的菜单上。
- 选择选项"积垢(无对话)"。
- 选择左侧菜单中的"延迟"选项,并输入30秒。
- 选择盘开/在左边的菜单上。
- 选择选项"在(无对话)板"。
- 通过重复步骤2.2.1.4创建第二动力学测量 - 2.2.1.14但在节2.2.1.11设定动能为25分钟,而不是10分钟。
- 按"验证"按钮,然后单击"确定"按钮。
- 保存实验。
- 按"解放军Y'按钮。一个对话框将会出现 - 只按"确定"按钮时,测量应启动。
- 通过混合GST M1-1的1微升和1微升38微升分析缓冲的"化学传感器"的准备两个样品。插入样品到384透明孔板的两口井,留下两个井之间的空孔。
- 采用12通道多移液器,迅速加入10微升各底板,轻轻混匀并迅速以避免泡沫,将板插入阅读器并开始动力学的测量。
- 当板打开(后3.5分钟),迅速加入1.125微升PDGF向井之一 ,轻轻拌匀,并允许盘报收为剩余的动力学测量。
- 加入PDGF适体的实时效果
- 重复步骤2.4.1.1-2.4.1.15。
- 通过混合1微升GST M1-1的,1微升准备两个样本"化学传感器",并在36.9微升分析缓冲液1.125微升PDGF的。插入样品到384透明孔板的两口井,留下两个井之间的空孔。
- 采用12通道多移液器,迅速加入10微升各底板,轻轻混匀并迅速以避免泡沫,将板插入读卡器,并开始动力学的测量。
- 当板打开(后1.5分钟),迅速加入1.2微升的PDGF适体到孔中的一个 ,轻轻混合并允许在板收起来为剩余动力学测量。
- PDGF除了实时生效
- 测量的JS-K前体药活化由GST在'化学传感器'和PDGF的存在。
- 设置在板读数器用于动力学测定的实验程序。
- 创建一个新的实验,一个"标准的协议"。
- 在"程序"按打开程序设置窗口。
- 在窗口的上侧弹出列表,根据该板的制造商选择'384板"类型。
- 按'读'在左边的菜单上。
- 至于检测的方法选择了"吸收"。
- 关于读式选择"终点"。
- 写波长窗口305纳米。
- 对右上侧的"完全版"按钮,按下并选择好来衡量。
- 按"确定"关闭"读取"窗口。
- 选择左侧菜单中的"启动动力"。
- 使运行时间10分钟。
- 选择最小间隔的选项。
- 按"确定"关闭窗口动能。
- 在"读取"线拖到动力学测量。
- 按"验证"按钮,然后单击"确定"按钮。
- 保存实验。
- 按"播放"按钮。一个对话框博x将出现 - 只有按"确定"按钮时,测量应启动。
- 对于NO生产测量使用亚硝酸盐/硝酸盐热量套件。在一个96孔板插入50微升测定缓冲液为一列,70微升格里斯的我试剂到第二行,和70微升格里斯第二试剂进入第三行。
- 为了执行三次重复实验,准备四个样品每片含4.8微升了"化学传感器"的。为了样品1,增加155.2微升分析缓冲液;样品2,加GST-M1-1的3.2微升和152微升分析缓冲液;样品3,加入PDGF的9.6微升和145.6微升分析缓冲液;和样品4,加入3.2微升GST-M1-1的,9.6微升PDGF,以及142.4微升分析缓冲液。
- 在室温下孵育该溶液10分钟。
- 在384透明孔板,插入50微升样品放入每个孔中。插入样本仅在奇数或仅在即使在同一行的孔,以允许基片除了使用多移液器的。
- 添加的JS-K的0.54微升(5毫在DMSO中)到每个孔中。
- 采用12通道多移液器,快速添加从GSH溶液10微升(在96孔板事先准备好的),轻轻混匀并迅速以避免气泡。插入板插入读卡器和启动动力学测量。
- 动力学测定后,立即使用12通道多吸移管,采取从每个样品50μl的进测定缓冲行中的预制备的96孔板,并迅速添加到它的Griess 1试剂的50微升和50微升的格里斯试剂II。孵育而避光进行10分钟,在RT和在550nm处测量吸光度。
注:测定缓冲液和试剂体积依赖于试剂盒"的协议。
- 设置在板读数器用于动力学测定的实验程序。
Access restricted. Please log in or start a trial to view this content.
结果
设计,合成,以及"化学传感器"可诱导的PDGF和GST之间人工通信的作用机制在图2中呈现。在"换能器"的结构,集成了PDGF的DNA适体和双-依他尼酰胺(BEA ),其是分别为26纳米和144纳米,已知的GST抑制剂( 图2a)。19这些粘合剂使'换能器'既PDGF和GST具有不同的亲和力,即结合,以解离常数(K D'S) 4此外,根据本设计,?...
Access restricted. Please log in or start a trial to view this content.
讨论
We presented a method for designing and testing of a 'chemical transducer' that can induce artificial communication between two naturally unrelated proteins, GST and PDGF, without modifying the native proteins. The unnatural GST-PDGF communications could be detected in real time by using enzymatic assays that follow the changes in the activity of GST in the presence of the 'chemical transducer' and increasing the concentrations of PDGF. In addition to detecting the activation of GST by PDGF, these assays were used to fol...
Access restricted. Please log in or start a trial to view this content.
披露声明
The authors have nothing to disclose.
致谢
这项研究是由密涅瓦基金会,HFSP组织和欧洲研究理事会资助项目(开始格兰特338265)。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 1-chloro-2,4-dinitrobenzene | Sigma-Aldrich | 237329 | |
| Acetic acid | Bio Lab | 01070521 | |
| Acetnitrile | J.T.Baker | 9017-03 | |
| Ascorbic acid | Sigma-Aldrich | A4544 | |
| Copper(II) Sulfate pentahydrate | Merck-Millipore | 102790 | |
| Dimethyl sulfoxide | Merck-Millipore | 802912 | |
| Dulbecco's Phosphate Buffered Saline | Biological Industries | 02-023-5A | |
| Ethacrynic acid | Tokyo Chemical Industry Co. Ltd | E0526 | |
| Glutathione-s-transferase M1-1 | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| JS-K | Sigma-Aldrich | J4137 | |
| L-glutathione reduced | Sigma-Aldrich | G4251 | |
| Magnesium Chloride | J.T.Baker | 0162 | |
| nitrate/nitrite colorimetric assay kit | Cayman Chemical | 780001 | |
| Oligonucleotides | W. M. Keck Foundation Biotechnology at Yale University | custom order | |
| PDGF-BB | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| TBTA | Sigma-Aldrich | 678937 | |
| Triethylamine | Sigma-Aldrich | T0886 | |
| Desalting column | GE Healthcare | illustra MicroSpin G-25 Columns | |
| HPLC | Waters | 2695 separation module | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 4.6 mm × 50 mm) | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 10 mm × 50 mm) | |
| Plate reader | BioTek | synergy H4 hybrid |
参考文献
- Hunter, T. Signaling—2000 and Beyond. Cell. 100, 113-127 (2000).
- Levitzki, A., Klein, S. Signal transduction therapy of cancer. Mol Aspects Med. 31, 287-329 (2010).
- Peri-Naor, R., Motiei, L., Margulies, D. Artificial signal transduction therapy: a futuristic approach to disease treatment. Future Med. Chem. 7, 2091-2093 (2015).
- Peri-Naor, R., Ilani, T., Motiei, L., Margulies, D. Protein-Protein Communication and Enzyme Activation Mediated by a Synthetic Chemical Transducer. J. Am. Chem. Soc. 137, 9507-9510 (2015).
- Corson, T. W., Aberle, N., Crews, C. M. Design and Applications of Bifunctional Small Molecules: Why Two Heads Are Better Than One. ACS Chem. Biol. 3, 677-692 (2008).
- Rutkowska, A., Schultz, C. Protein Tango: The Toolbox to Capture Interacting Partners. Angew. Chem. Int. Ed. 51, 8166-8176 (2012).
- Meyer, C., Köhn, M. A Molecular Tête-à-Tête Arranged by a Designed Adaptor Protein. Angew. Chem. Int. Ed. 51, 8160-8162 (2012).
- Klemm, J. D., Schreiber, S. L., Crabtree, G. R. Dimerization as a Regulatory Mechanism in Signal Transduction. Annu. Rev. Immunol. 16, 569-592 (1998).
- DeRose, R., Miyamoto, T., Inoue, T. Manipulating signaling at will: chemically-inducible dimerization (CID) techniques resolve problems in cell biology. Pflugers Arch. 465, 409-417 (2013).
- Gestwicki, J. E., Marinec, P. S. Chemical control over protein-protein interactions: beyond inhibitors. Comb. Chem. High Throughput. Screen. 10, 667-675 (2007).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. 25, 848-862 (2013).
- Diezmann, F., Seitz, O. DNA-guided display of proteins and protein ligands for the interrogation of biology. Chem. Soc. Rev. 40, 5789-5801 (2011).
- Röglin, L., Ahmadian, M. R., Seitz, O. DNA-Controlled Reversible Switching of Peptide Conformation and Bioactivity. Angew. Chem. Int. Ed. 46, 2704-2707 (2007).
- Röglin, L., Altenbrunn, F., Seitz, O. DNA and RNA-Controlled Switching of Protein Kinase Activity. ChemBioChem. 10, 758-765 (2009).
- Harris, D. C., Chu, X., Jayawickramarajah, J. DNA-Small Molecule Chimera with Responsive Protein-Binding Ability. J. Am. Chem. Soc. 130, 14950-14951 (2008).
- Harris, D. C., Saks, B. R., Jayawickramarajah, J. Protein-Binding Molecular Switches via Host-Guest Stabilized DNA Hairpins. J. Am. Chem. Soc. 133, 7676-7679 (2011).
- Kim, Y., Cao, Z., Tan, W. Molecular assembly for high-performance bivalent nucleic acid inhibitor. Proc. Nat. Acad. Sci. U.S.A. 105, 5664-5669 (2008).
- Han, D., et al. A Logical Molecular Circuit for Programmable and Autonomous Regulation of Protein Activity Using DNA Aptamer-Protein Interactions. J. Am. Chem. Soc. 134, 20797-20804 (2012).
- Motiei, L., Pode, Z., Koganitsky, A., Margulies, D. Targeted Protein Surface Sensors as a Tool for Analyzing Small Populations of Proteins in Biological Mixtures. Angew. Chem. Int. Ed. 53, 9289-9293 (2014).
- Ranallo, S., Rossetti, M., Plaxco, K. W., Vallée-Bélisle, A., Ricci, F. A Modular, DNA-Based Beacon for Single-Step Fluorescence Detection of Antibodies and Other Proteins. Angew. Chem. Int. Ed. 54, 13214-13218 (2015).
- Franzini, R. M., et al. Identification of Structure-Activity Relationships from Screening a Structurally Compact DNA-Encoded Chemical Library. Angew. Chem. Int. Ed. 54, 3927-3931 (2015).
- Unger-Angel, L., et al. Protein recognition by bivalent, 'turn-on' fluorescent molecular probes. Chem. Sci. 6, 5419-5425 (2015).
- Nissinkorn, Y., et al. Sensing Protein Surfaces with Targeted Fluorescent Receptors. Chem. Eur. J. 21, 15981-15987 (2015).
- Huber, C. G., Oefner, P. J., Bonn, G. K. High-Resolution Liquid Chromatography of Oligonucleotides on Nonporous Alkylated Styrene-Divinylbenzene Copolymers. Anal. Biochem. 212, 351-358 (1993).
- Lyon, R. P., Hill, J. J., Atkins, W. M. Novel class of bivalent glutathione S-transferase inhibitors. Biochemistry. 42, 10418-10428 (2003).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. , 1-16 (2013).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。