このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
シグナル伝達タンパク質の機能を模倣:人工シグナル伝達療法に向けて
要約
We present guidelines for developing synthetic 'chemical transducers' that can induce communication between naturally unrelated proteins. In addition, detailed protocols are presented for synthesizing and testing a specific 'transducer' that enables a growth factor to activate a detoxifying enzyme and consequently, to regulate the cleavage of an anticancer prodrug.
要約
Signal transduction pathways, which control the response of cells to various environmental signals, are mediated by the function of signaling proteins that interact with each other and activate one other with high specificity. Synthetic agents that mimic the function of these proteins might therefore be used to generate unnatural signal transduction steps and consequently, alter the cell's function. We present guidelines for designing 'chemical transducers' that can induce artificial communication between native proteins. In addition, we present detailed protocols for synthesizing and testing a specific 'transducer', which can induce communication between two unrelated proteins: platelet-derived growth-factor (PDGF) and glutathione-S-transferase (GST). The way by which this unnatural PDGF-GST communication could be used to control the cleavage of an anticancer prodrug is also presented, indicating the potential for using such systems in 'artificial signal transduction therapy'. This work is intended to facilitate developing additional 'transducers' of this class, which may be used to mediate intracellular protein-protein communication and consequently, to induce artificial cell signaling pathways.
概要
シグナル伝達経路は、事実上すべての細胞プロセスにおいて重要な役割を果たし、細胞が急速に環境シグナルに応答することができます。1これらの経路は、多くの場合、細胞内酵素の活性化をもたらす細胞外受容体へのシグナル伝達分子の結合によってトリガされます。細胞内でこの信号の増幅及び伝播は酵素が可逆的に高い特異性で活性化されたタンパク質 - タンパク質相互作用のネットワークを形成するタンパク質のシグナル伝達機能によって媒介されます。これらのネットワークの調節不全は、しばしば癌の発生につながるので、薬は悪性のシグナル伝達経路を破壊するように設計されていることにより、2 '癌のシグナル伝達療法」を確立する上で多くの関心が集まっています。我々は最近、不自然なシグナル伝達経路を生成する薬物の能力に依存している形質導入療法をシグナリングするための代替的なアプローチを提案しています。 図3は、我々は、シグナル伝達タンパク質の機能を模倣する合成薬剤を設計することによって、間接的に、細胞の機能を調節することが可能であると考えています。例えば、これらの人工的なネットワークは、プロドラッグを切断する酵素を活性化するタンパク質バイオマーカーを使用可能にすることができます。あるいは、これらのシグナル伝達タンパク質の模倣物は、治療効果が得られ、不自然な細胞シグナル伝達経路を活性化することができるかもしれません。
このアプローチの実現可能性を実証するために、我々は最近であるグルタチオン-S-トランスフェラーゼ(GST)を、活性化することによって抗癌プロドラッグの切断を誘発する血小板由来増殖因子(PDGF)を可能にする合成「化学変換器4 ' を作成しましたない、その天然の結合パートナー。この「トランスデューサー」の構造は、GSTのための二価阻害剤で修飾されている抗PDGFのDNAアプタマーで構成されています。したがって、この合成剤は、への結合部位を有する分子のファミリーに属し異なるタンパク質、二量体化の5-7のような化学誘導物質(のCID)8-10と、オリゴヌクレオチド合成分子複合体に基づいてタンパク質-結合剤のグループに。11月21日
そのようなシステムの設計の基礎となる一般的な原理は、本明細書に記載されており、合成及び従来の酵素アッセイを使用してこの「変換器」の機能を試験するための詳細なプロトコルが提供されます。この作業は、細胞内のタンパク質 - タンパク質の通信を仲介し、その結果として、人工的な細胞シグナル伝達経路を誘導するために使用することができるこのクラスの追加の「変換器」を、開発を促進することを意図しています。
図1は、不自然なタンパク質-タンパク質の通信を媒介することができる合成「化学変換器」の動作原理を説明しています。 protのための合成バインダーを統合し、この図では、「化学変換器」では、アインスIおよびII(バインダーIおよびII)は、その天然の結合相手ではないタンパク質の触媒活性Iを、トリガするタンパク質IIを可能にします。タンパク質IIの非存在下では、トランスデューサは、酵素(タンパク質I)の触媒部位に結合した ( 図1、状態II)の活性を阻害します。タンパク質IIに「トランスデューサ」の結合が、その結果の有効濃度をタンパク質I.向かってその親和性を低下させるバインダーIおよびIIタンパク質の表面( 図1、状態III)の間の相互作用を促進します」溶液中の遊離」トランスデューサは、私は複雑で、タンパク質I( 図1、状態IV)の再活性化にトランスデューサタンパク質の解離につながる、減少します。 (1)「トランスデューサー」はタンパク質標的のそれぞれについて具体的なバインダーを持っている必要があり、(2)相互作用betwe:まとめると、これらの手順は、効率的な「変換器」の設計の基礎となる3つの基本的な原則を強調表示しましたバインダーIIおよびタンパク質II専用私はタンパク質IIの表面と相互作用することができなければならないIおよびタンパク質I、および(3)バインダーバインダーとの間の相互作用よりも強いでなければなりません。この最後の原則は必ずしもバインダーは、単独で私はタンパク質IIに対して高い親和性と選択性を有することは必要ありません。その代わりに、それは、タンパク質の近傍に合成分子をもたらすことは、この分子とタンパク質表面との間の相互作用を促進する可能性があることを示した我々の最近の研究に基づいている。19,22,23
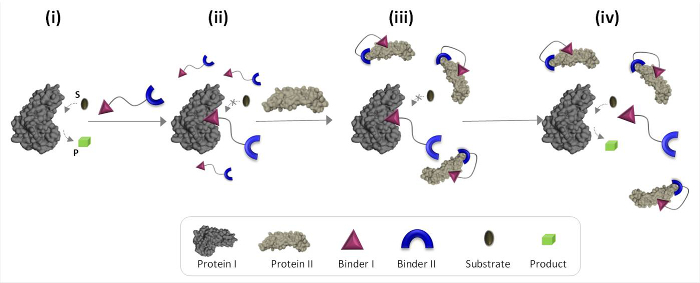
図1:「化学トランスデューサは'I(状態I)活性タンパク質に付加されている場合「化学変換器」の動作原理は 、それがバインダーIを介して、その活性部位に結合し、その活性(状態II)を阻害します。タンパク質II、但し、結合していない「化学Tの存在下でransducerは「バインダーI及びタンパク質IIの表面との間の相互作用を促進するバインダーIIを通じてタンパク質IIと相互作用します。この誘導された結合剤I-タンパク質IIの相互作用は、私は、複雑な「transducer'タンパク質の解離につながるバインダーの有効濃度Iを、減少させ、タンパク質に私が再活性化(状態IV)。 この図の拡大版をご覧になるにはこちらをクリックしてください。 。
Access restricted. Please log in or start a trial to view this content.
プロトコル
「化学トランスデューサー」の1合成
- 予備の準備
- 2 M酢酸トリエチルアンモニウム(TEAA)バッファは、超純水の酢酸の114ミリリットルと400ミリリットルとトリエチルアミン278ミリリットルを混合して準備します。暗いボトルに保管してpHを7に調整し、1 Lの最終体積に水を追加します。
注:このソリューションは、年間安定です。 - 超純水20ml中にアスコルビン酸の18ミリグラムを溶解することにより5mMのアスコルビン酸溶液を調製します。新鮮な溶液を使用してください。溶液を1日間安定です。
- 超純水の10ミリリットルと11ミリリットル中TBTA 58mgのジメチルスルホキシド(DMSO)で銅25mgの(II)五水和物を溶解することにより10mMの銅(II)/トリス(benzyltriazolylmethyl)アミン(TBTA)溶液を調製します。二つの溶液を混ぜます。室温でそれを維持し、光から保護します。
- 2 M酢酸トリエチルアンモニウム(TEAA)バッファは、超純水の酢酸の114ミリリットルと400ミリリットルとトリエチルアミン278ミリリットルを混合して準備します。暗いボトルに保管してpHを7に調整し、1 Lの最終体積に水を追加します。
- 結合手順
- 80μlの修飾オリゴヌクレオチドの100ナノモル(ODN-1)を溶解新鮮な超純水。 2 M TEAA20μlのはpH = 7は、アスコルビン酸の新たに作られた溶液(水中5 mM)の80μlのを追加。
- DMSOの180μlのアジド修飾されたエタクリン酸の1.5マイクロモル(574.5μgの)を溶解し、溶液に追加します。ドガ60秒間アルゴンを使用し、迅速にCu(II)/ TBTA溶液(55%中の10mM(v / v)のDMSO /水)から40μlを添加するソリューション。
- 近くにしっかりと、アルゴンで再びパージし、そして一晩攪拌します。
- 反応の進行状況を監視し、RP-HPLC(移動相:A)によって結合体を精製し、5%アセトニトリル、5%TEAA、90%の超純水; B)65%アセトニトリル、5%TEAA、30%超純水)24
2. PDGFによってGST活性を制御します
- 予備の準備
- 8mMの最終リン酸塩濃度および広告を達成するために、超純水16.1 mlのリン酸緩衝生理食塩水(PBSx1)33.9 mlと混合することによってアッセイ緩衝液を50mlの調製DのMgCl 2の23.8 mgを5mMの最終濃度を達成します。
- 50mMトリスpH7.5の、50mMのNaClを含む緩衝液中でタンパク質を溶解することによりGST M1-1のストック溶液を調製し、1mMジチオスレイトール(DTT)、30μMの最終濃度5mMのエチレンジアミン四酢酸(EDTA)。 -80℃で小分けし、ストアにこのソリューションを割ります。アッセイ緩衝液中で、セクション2.1.5.1によると、新鮮に希釈し、氷上に保ちます。
注:溶液を、約5時間、安定であるか、または酵素活性の減少があるまで。 - 以下の手順に従って基板を準備します。
- 100mMのストック溶液の最終濃度になるように超純水325μlの還元グルタチオン(GSH)10mgを溶解します。 2.1 mMのワーキング溶液の最終濃度のためにアッセイ緩衝液の979μlのこのストック溶液から21μLを希釈します。
- Eの492μlの2,4-ジニトロクロロベンゼン(CDNB)10mgを溶解させます100mMのストック溶液の最終濃度までthanol。 4.32 mMのワーキング溶液の最終濃度のためにアッセイ緩衝液の956.8μlの原液から43.2μLを希釈します。
- 96ウェルプレートでは、別の行(12ウエル)の各基板を置きます。ソリューションの迅速かつ簡単に撤退を可能にするために、各ウェルに少なくとも60μLを挿入します。光保護のためにアルミシートでプレートをカバーしています。
- アッセイ緩衝液中で、次のストック溶液を準備します。
- 二量体の0.6μMの最終濃度に50によってGST M1-1を希釈します。
- 30μMの原液に「化学変換器 'を希釈します。
- 40μMの最終濃度にPDGFを希釈します。
- 250μMの最終濃度にPDGFアプタマーを希釈します。
- 「化学変換器」とPDGFの存在下でのGST活性を測定します。
- 実験手順を設定する私動態測定のためにプレートリーダーをNです。
- 「標準プロトコル」などの新しい実験を作成します。
- 「手順」を押して、手順の設定ウィンドウを開きます。
- ウィンドウの上側のポップアップリストでは、プレートのメーカーによると「384プレート」タイプを選択します。
- 左側のメニューを押し「読みます」。
- 検出方法については「吸光度」を選択します。
- 読み取りタイプについて「エンドポイント」を選択します。
- 波長ウィンドウ上に340nmを書きます。
- 右上側の「フル板」底を押して、測定されるべき十分を選択します。
- 押して「OK」「読む」ウィンドウを閉じます。
- 選択し、左メニューの「運動を開始」。
- 実行時間10分を行います。
- 最小間隔オプションを選択します。
- プレス運動ウインドウを閉じるには「OK」。
- 私は、「読む」の行をドラッグします反応速度測定Ntoを。
- 「検証」ボタンを押して「OK」ボタンを押してください。
- 実験を保存します。
- 「プレイ」ボタンを押してください。ダイアログボックスが表示されます - 測定を開始しなければならない場合にのみ、「OK」ボタンを押してください。
- 三連の実験を行うためには、4つのサンプルに「化学変換器」とGST M1-1の3.25μlの各含む3.25μLを準備します。それぞれ、各サンプル0、1.2、2.4、またはPDGFおよび123.5、122.3、121.1の4.9μlの、またはアッセイ緩衝液の118.6μlに加えます。
- 10分間室温で溶液をインキュベートします。
- 384透明ウェルプレートで、各ウェルにサンプルの40μlのを挿入します。基質添加のためのマルチピペットの使用を可能にするためにのみ、奇数ウェル中や、同じ行にあっても、ウェル内のサンプルを挿入します。
- 12チャンネルマルチピペッターを使用して、すぐにTのそれぞれから10μLを加えます96ウェルプレート(セクション2.1.4)に予め用意した彼の基板。泡を避けるために、静かにかつ迅速に混ぜます。リーダーにプレートを挿入し、反応速度測定を開始します。 GST速度が非常に高速であるため、基質添加および反応速度測定の開始との間の時間を最小限に抑えるようにしてください。
- 実験手順を設定する私動態測定のためにプレートリーダーをNです。
- 「化学変換器」によって媒介されるGST活性化/阻害サイクル。
- 、三連の実験を行う5つのサンプルのアッセイ緩衝液84.5μlを、「化学変換器」の3.25マイクロリットル、およびGST M1-1の3.25μLを含むそれぞれを調製するために。 3分間、室温でインキュベートします。
- サンプル2-5にPDGFの1と3.65μLをサンプリングするアッセイ緩衝液の3.65μLを加えます。 3分間、室温でインキュベートします。
- サンプル3-5に、サンプル1-2とPDGFアプタマーの3.12μlにアッセイ緩衝液の3.12μLを加えます。 3分間、室温でインキュベートします。
- サンプルにアッセイ緩衝液の24.4μlを添加しますサンプル4-5のPDGFの1-3と24.4μlの。 3分間、室温でインキュベートします。
- 5分間、室温で5インキュベートをサンプリングするPDGFアプタマーのサンプル1-4と7.8μlにアッセイ緩衝液の7.8μlを添加します。
- 384透明ウェルプレートで、各ウェルにサンプルを40μlを挿入します。のみ奇数または、同じ行にあってもウェルににサンプルを挿入します。
- 12チャンネルマルチピペッターを使用して、すぐにそれぞれの基板からの10μLを加える(96ウェルプレート中に予め調製しました)。泡を避けるために、静かにかつ迅速に混ぜます。リーダーにプレートを挿入し、反応速度測定を開始します。
- tにおいて340nmで測定したODを差し引くことにより、各条件でV 0 [MOD /分]を算出= ODから0.5分、Tで340 nmで測定し= 1.5分活性化/阻害リサイクル性を評価すること。25
- 環境の変化に「化学変換器」のリアルタイム応答を評価します。
- PDGFの添加のリアルタイム効果
- 反応速度測定用のプレートリーダーでの実験手順を設定します。
- 繰り返しは2.2.1.1-2.2.1.10を繰り返します。
- 実行時間を3.5分にします。
- 最小間隔オプションを選択します。
- プレス運動ウインドウを閉じるには「OK」。
- 運動の測定に「読む」の行をドラッグします。
- 左側のメニュー/上にプレートアウトを選択します。
- オプション 'プレートアウト(ダイアログなし)」を選択します。
- 左メニューの「遅延」オプションを選択し、30秒を入力します。
- 左側のメニュー/上にプレートアウトを選択します。
- オプション '(なしダイアログ)でプレート]を選択します。
- 2.2.1.14をしかし、セクション2.2.1.11で25分の代わりに10分であることが運動を設定する - 手順2.2.1.4を繰り返すことにより、第2の運動の測定を作成します。
- 「検証」ボタンを押して「OK」ボタンを押してください。
- 実験を保存します。
- 「ナンプラーを押してくださいY 'ボタンをクリックします。ダイアログボックスが表示されます - 測定を開始しなければならない場合にのみ、「OK」ボタンを押してください。
- GST M1-1の1μlのとアッセイ緩衝液の38μlの中で「化学変換器」の1μLを混合することにより、二つのサンプルを準備します。これらの2つのウェル間の空井戸を残し、384透明ウェルプレートの2ウェルにサンプルを挿入します。
- すぐに、各基板から10μLを追加穏やかに混合し、すばやく泡を避けるために、リーダーにプレートを挿入し、反応速度測定を開始し、12チャンネルのマルチピペッターを使用しました。
- プレートは、(3.5分後)まで開いたら、すぐに、ウェルの1にPDGFの1.125μlを添加穏やかに混合し、そしてプレートは、残りの反応速度測定のためにクローズアップすることができます。
- PDGFアプタマーを追加のリアルタイム効果
- 繰り返しは2.4.1.1-2.4.1.15を繰り返します。
- GST M1-1の1μlを、1μLを混合することにより、二つのサンプルを準備します」化学変換器」、およびアッセイ緩衝液の36.9μlの中のPDGFの1.125μlの。これらの2つのウェル間の空井戸を残し、384透明ウェルプレートの2ウェルにサンプルを挿入します。
- 12チャンネルのマルチピペッターを使用して、迅速に、各基板から10μlを添加する気泡を避けるために、静かにかつ迅速に混ぜて、リーダーにプレートを挿入し、反応速度測定を開始します。
- プレートは、(1.5分後)まで開いたら、すぐに、ウェルの1にPDGFアプタマーの1.2μlを添加穏やかに混合し、プレートを残りの反応速度測定のためにクローズアップすることができます。
- PDGFの添加のリアルタイム効果
- 「化学変換器」とPDGFの存在下でGSTによってJS-Kプロドラッグ活性化を測定します。
- 反応速度測定用のプレートリーダーでの実験手順を設定します。
- 「標準プロトコル」などの新しい実験を作成します。
- 「手順」を押して、手順の設定ウィンドウを開きます。
- ウィンドウの上側のポップアップリストでプレートメーカーによると「384プレート」タイプを選択します。
- 左側のメニューを押し「読みます」。
- 検出方法については「吸光度」を選択しました。
- 読み取りタイプについて「エンドポイント」を選択します。
- 波長窓に305 nmのを書きます。
- 右上側の「フル板」ボタンを押して、測定されるべき十分を選択します。
- 押して「OK」「読む」ウィンドウを閉じます。
- 選択し、左メニューの「運動を開始」。
- 実行時間10分を行います。
- 最小間隔オプションを選択します。
- プレス運動ウインドウを閉じるには「OK」。
- 運動の測定に「読む」の行をドラッグします。
- 「検証」ボタンを押して「OK」ボタンを押してください。
- 実験を保存します。
- 「プレイ」ボタンを押してください。ダイアログBOXが表示されます - 測定を開始しなければならない場合にのみ、「OK」ボタンを押してください。
- NO産生の測定のために亜硝酸/硝酸熱量測定キットを使用します。 96ウェルプレートでは、1行に私は3番目の行に2行目に試薬、およびグリースII試薬70μlのグリースの70μLをアッセイ緩衝液50μlを挿入します。
- 三連の実験を行うためには、4つのサンプルに「化学変換器」のそれぞれを含む4.8μLを準備します。 1をサンプリングするために、アッセイ緩衝液の155.2μlを添加します。 2をサンプリングするために、GST-M1-1およびアッセイ緩衝液の152μlに3.2μlを添加します。 3をサンプリングするために、PDGFの9.6μlのとアッセイ緩衝液の145.6μlを添加します。そして、4をサンプリングするために、GST-M1-1の3.2μlを、PDGFの9.6μlを、およびアッセイ緩衝液の142.4μlを添加します。
- 10分間室温で溶液をインキュベートします。
- 384透明ウェルプレートで、各ウェルに50μlのサンプルを挿入します。のみ奇数またはだけででサンプルを挿入同じ行であってもウェルは、基質添加のためのマルチピペットの使用を可能にします。
- 各ウェルにJS-Kの0.54μlの(DMSO中の5mM)を追加します。
- すぐに気泡を避けるために迅速に(96ウェルプレート中に予め調製した)を、穏やかに混合GSH溶液10μLを加えると、12チャンネルマルチピペッターを使用して。リーダーにプレートを挿入し、反応速度測定を開始します。
- すぐに運動測定後、12チャンネルのマルチピペッターを使用して、事前に用意した96ウェルプレートでアッセイバッファー行に各試料から50μLを取り、すぐにそれにグリースI試薬を50μl及び50μlのを追加グリースII試薬。 RTで10分間、光から保護しながらインキュベートし、550 nmの吸光度を測定します。
注意:アッセイ緩衝液および試薬のボリュームがキット」プロトコルに依存しています。
- 反応速度測定用のプレートリーダーでの実験手順を設定します。
Access restricted. Please log in or start a trial to view this content.
結果
設計、合成、およびPDGFとGSTとの間に人工的な通信を誘導することができる「化学変換器」の作用の機構は、図2に示されている。「変換器」の構造は、PDGF DNAアプタマー及びビスエタクリンアミドを統合(BEA )、これは既知のGST阻害剤( 図2A)である。19これらのバインダーは、それぞれ、26 nmおよび144 nMでの解離定数(K dの?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
We presented a method for designing and testing of a 'chemical transducer' that can induce artificial communication between two naturally unrelated proteins, GST and PDGF, without modifying the native proteins. The unnatural GST-PDGF communications could be detected in real time by using enzymatic assays that follow the changes in the activity of GST in the presence of the 'chemical transducer' and increasing the concentrations of PDGF. In addition to detecting the activation of GST by PDGF, these assays were used to fol...
Access restricted. Please log in or start a trial to view this content.
開示事項
The authors have nothing to disclose.
謝辞
この研究はミネルバ財団、HFSP組織、および欧州研究評議会助成金(グラント338265を開始する)によってサポートされていました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 1-chloro-2,4-dinitrobenzene | Sigma-Aldrich | 237329 | |
| Acetic acid | Bio Lab | 01070521 | |
| Acetnitrile | J.T.Baker | 9017-03 | |
| Ascorbic acid | Sigma-Aldrich | A4544 | |
| Copper(II) Sulfate pentahydrate | Merck-Millipore | 102790 | |
| Dimethyl sulfoxide | Merck-Millipore | 802912 | |
| Dulbecco's Phosphate Buffered Saline | Biological Industries | 02-023-5A | |
| Ethacrynic acid | Tokyo Chemical Industry Co. Ltd | E0526 | |
| Glutathione-s-transferase M1-1 | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| JS-K | Sigma-Aldrich | J4137 | |
| L-glutathione reduced | Sigma-Aldrich | G4251 | |
| Magnesium Chloride | J.T.Baker | 0162 | |
| nitrate/nitrite colorimetric assay kit | Cayman Chemical | 780001 | |
| Oligonucleotides | W. M. Keck Foundation Biotechnology at Yale University | custom order | |
| PDGF-BB | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| TBTA | Sigma-Aldrich | 678937 | |
| Triethylamine | Sigma-Aldrich | T0886 | |
| Desalting column | GE Healthcare | illustra MicroSpin G-25 Columns | |
| HPLC | Waters | 2695 separation module | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 4.6 mm × 50 mm) | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 10 mm × 50 mm) | |
| Plate reader | BioTek | synergy H4 hybrid |
参考文献
- Hunter, T. Signaling—2000 and Beyond. Cell. 100, 113-127 (2000).
- Levitzki, A., Klein, S. Signal transduction therapy of cancer. Mol Aspects Med. 31, 287-329 (2010).
- Peri-Naor, R., Motiei, L., Margulies, D. Artificial signal transduction therapy: a futuristic approach to disease treatment. Future Med. Chem. 7, 2091-2093 (2015).
- Peri-Naor, R., Ilani, T., Motiei, L., Margulies, D. Protein-Protein Communication and Enzyme Activation Mediated by a Synthetic Chemical Transducer. J. Am. Chem. Soc. 137, 9507-9510 (2015).
- Corson, T. W., Aberle, N., Crews, C. M. Design and Applications of Bifunctional Small Molecules: Why Two Heads Are Better Than One. ACS Chem. Biol. 3, 677-692 (2008).
- Rutkowska, A., Schultz, C. Protein Tango: The Toolbox to Capture Interacting Partners. Angew. Chem. Int. Ed. 51, 8166-8176 (2012).
- Meyer, C., Köhn, M. A Molecular Tête-à-Tête Arranged by a Designed Adaptor Protein. Angew. Chem. Int. Ed. 51, 8160-8162 (2012).
- Klemm, J. D., Schreiber, S. L., Crabtree, G. R. Dimerization as a Regulatory Mechanism in Signal Transduction. Annu. Rev. Immunol. 16, 569-592 (1998).
- DeRose, R., Miyamoto, T., Inoue, T. Manipulating signaling at will: chemically-inducible dimerization (CID) techniques resolve problems in cell biology. Pflugers Arch. 465, 409-417 (2013).
- Gestwicki, J. E., Marinec, P. S. Chemical control over protein-protein interactions: beyond inhibitors. Comb. Chem. High Throughput. Screen. 10, 667-675 (2007).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. 25, 848-862 (2013).
- Diezmann, F., Seitz, O. DNA-guided display of proteins and protein ligands for the interrogation of biology. Chem. Soc. Rev. 40, 5789-5801 (2011).
- Röglin, L., Ahmadian, M. R., Seitz, O. DNA-Controlled Reversible Switching of Peptide Conformation and Bioactivity. Angew. Chem. Int. Ed. 46, 2704-2707 (2007).
- Röglin, L., Altenbrunn, F., Seitz, O. DNA and RNA-Controlled Switching of Protein Kinase Activity. ChemBioChem. 10, 758-765 (2009).
- Harris, D. C., Chu, X., Jayawickramarajah, J. DNA-Small Molecule Chimera with Responsive Protein-Binding Ability. J. Am. Chem. Soc. 130, 14950-14951 (2008).
- Harris, D. C., Saks, B. R., Jayawickramarajah, J. Protein-Binding Molecular Switches via Host-Guest Stabilized DNA Hairpins. J. Am. Chem. Soc. 133, 7676-7679 (2011).
- Kim, Y., Cao, Z., Tan, W. Molecular assembly for high-performance bivalent nucleic acid inhibitor. Proc. Nat. Acad. Sci. U.S.A. 105, 5664-5669 (2008).
- Han, D., et al. A Logical Molecular Circuit for Programmable and Autonomous Regulation of Protein Activity Using DNA Aptamer-Protein Interactions. J. Am. Chem. Soc. 134, 20797-20804 (2012).
- Motiei, L., Pode, Z., Koganitsky, A., Margulies, D. Targeted Protein Surface Sensors as a Tool for Analyzing Small Populations of Proteins in Biological Mixtures. Angew. Chem. Int. Ed. 53, 9289-9293 (2014).
- Ranallo, S., Rossetti, M., Plaxco, K. W., Vallée-Bélisle, A., Ricci, F. A Modular, DNA-Based Beacon for Single-Step Fluorescence Detection of Antibodies and Other Proteins. Angew. Chem. Int. Ed. 54, 13214-13218 (2015).
- Franzini, R. M., et al. Identification of Structure-Activity Relationships from Screening a Structurally Compact DNA-Encoded Chemical Library. Angew. Chem. Int. Ed. 54, 3927-3931 (2015).
- Unger-Angel, L., et al. Protein recognition by bivalent, 'turn-on' fluorescent molecular probes. Chem. Sci. 6, 5419-5425 (2015).
- Nissinkorn, Y., et al. Sensing Protein Surfaces with Targeted Fluorescent Receptors. Chem. Eur. J. 21, 15981-15987 (2015).
- Huber, C. G., Oefner, P. J., Bonn, G. K. High-Resolution Liquid Chromatography of Oligonucleotides on Nonporous Alkylated Styrene-Divinylbenzene Copolymers. Anal. Biochem. 212, 351-358 (1993).
- Lyon, R. P., Hill, J. J., Atkins, W. M. Novel class of bivalent glutathione S-transferase inhibitors. Biochemistry. 42, 10418-10428 (2003).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. , 1-16 (2013).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved