JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
신호 전달 단백질의 기능을 흉내 낸 : 인공 신호 전달 요법을 향해
요약
We present guidelines for developing synthetic 'chemical transducers' that can induce communication between naturally unrelated proteins. In addition, detailed protocols are presented for synthesizing and testing a specific 'transducer' that enables a growth factor to activate a detoxifying enzyme and consequently, to regulate the cleavage of an anticancer prodrug.
초록
Signal transduction pathways, which control the response of cells to various environmental signals, are mediated by the function of signaling proteins that interact with each other and activate one other with high specificity. Synthetic agents that mimic the function of these proteins might therefore be used to generate unnatural signal transduction steps and consequently, alter the cell's function. We present guidelines for designing 'chemical transducers' that can induce artificial communication between native proteins. In addition, we present detailed protocols for synthesizing and testing a specific 'transducer', which can induce communication between two unrelated proteins: platelet-derived growth-factor (PDGF) and glutathione-S-transferase (GST). The way by which this unnatural PDGF-GST communication could be used to control the cleavage of an anticancer prodrug is also presented, indicating the potential for using such systems in 'artificial signal transduction therapy'. This work is intended to facilitate developing additional 'transducers' of this class, which may be used to mediate intracellular protein-protein communication and consequently, to induce artificial cell signaling pathways.
서문
신호 전달 경로는 거의 모든 세포 과정에 중요한 역할을 담당하고, 셀이 급속히 환경 신호에 응답 할 수있다. (1)이 경로는 종종 세포 내 효소의 활성 결과 세포 외 수용체에 시그널링 분자의 결합에 의해 유발된다. 증폭 및 세포 내 신호 전달이 효소는 높은 특이성을 가역적으로 활성화되는 단백질 - 단백질 상호 작용의 네트워크를 형성하는 신호 전달 단백질의 작용에 의해 매개된다. 이러한 네트워크의 조절 곤란 자주 암 발생을 유도하기 때문에, 약물 악성 신호 전달 경로를 방해하도록 설계된다 '암 신호 전달 요법'이 확립에 많은 관심이 있었다. 최근 부자연 신호 전달 경로를 생성하는 약물의 능력에 의존하여 전달 요법을 알리기 위해 다른 방법을 제안 하였다. 3, 우리는 신호 전달 단백질의 작용을 모방 합성 제제를 설계함으로써, 간접적으로 세포의 기능을 조절하는 것이 가능하다고 판단된다. 예를 들어, 이들 네트워크는 인공 드럭을 절단하는 효소를 활성화하는 생체 단백질을 가능하게 할 수있다. 또한, 이러한 신호 전달 단백질 모방 체는 치료 효과의 결과로, 자연스러운 세포 신호 전달 경로를 활성화 할 수 있습니다.
이러한 접근의 가능성을 증명하기 위해, 최근 인 글루타치온 에스 - 트랜스퍼 라제 (GST)를 활성화시켜 항암 전구 약물의 분해를 유발 혈소판 유래 성장 인자 (PDGF)를 가능하게하는 합성 '화학 변환기'4 작성한 하지 자연 바인딩 파트너. 이 '변환기'의 구조에 대한 GST 가의 억제제 수정 항 PDGF의 DNA 앱 타머 구성된다. 따라서,이 합성 에이전트에 결합 부위와 분자의 가족에 속하는다른 단백질, 이량의 5-7와 같은 화학 유도 (식별자) 8 ~ 10도 올리고 뉴클레오티드 합성 분자 복합체를 기반으로 단백질 바인더의 그룹. 11-21
이러한 시스템의 설계의 기초가되는 일반적인 원리는 본 명세서에 기술되고 합성 종래 효소 분석법이 '변환기'의 기능을 테스트하기위한 상세한 프로토콜이 제공된다. 이 작업은 세포 단백질 간의 통신을 매개하고, 따라서, 인공 세포 신호 전달 경로를 유도하는 데 사용될 수있다,이 클래스의 추가적인 '센서'를 개발 촉진하기위한 것이다.
도 1은 비 천연 단백질 간의 통신을 매개 할 수있는 합성 '화학 센서'의 동작 원리를 설명한다. 제자를위한 합성 바인더 통합이 그림은 '화학 센서'에서고용주 식별 번호 (EIN) I 및 II (바인더 I 및 II)는 자연 바인딩 파트너가 아닌 단백질의 촉매 활성 I를 트리거하는 단백질 II 수 있습니다. II 단백질의 부재에서, 변환기는 효소 (단백질 I)의 촉매 사이트를 결합하고 그 활성 (도 1의 상태 II)를 억제한다. 단백질 II의 '변환기'결합, 단, 그 결과의 유효 농도를 단백질 I. 향해 친화력을 감소 바인더 I 단백질 II의 표면 (도 1의 상태 ⅲ) 사이의 상호 작용을 촉진 ' 솔루션 무료 '트랜스 듀서는 트랜스 듀서 단백질의 해리 리드하는 감소 I 복잡하고 단백질 I의 재 활성화에 (그림 1, 상태 ⅳ). (1) '변환기', 단백질 목표의 각각에 대해 (2) 상호 작용 betwe을 특정 바인더가 있어야합니다 함께 다음 단계는 효율적인 '변환기'의 디자인을 기본 세 가지 기본 원칙을 강조바인더 II 단백질 엉 II 내가 단백질 II의 표면과 상호 작용 할 수 있어야 바인더 I 단백질 I, (3) 바인더 사이의 상호 작용보다 더 강한해야합니다. 이 마지막 원칙은 반드시 단백질 II 향해 높은 친화력 및 선택성을해야 내가 혼자 바인더를 필요로하지 않습니다. 대신, 단백질에 근접 합성 분자를 가져 오는 것은,이 단백질 분자의 표면 사이의 상호 작용을 촉진 할 가능성이 있음을 보여 우리의 최근의 연구에 기초한다. 19,22,23
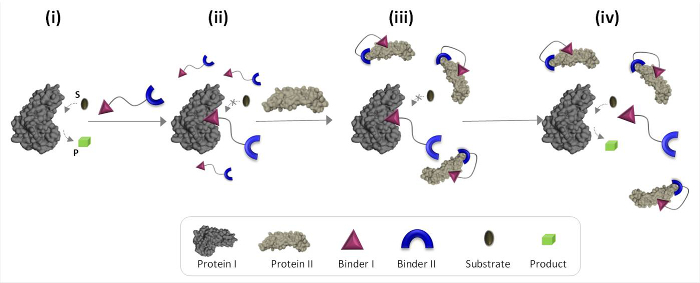
그림 1 :. '화학 트랜스 듀서는'활성 단백질 I (상태 I)에 추가 될 때 '화학 센서'의 작동 원리, 그것은 바인더를 통해 활성 부위에 I를 결합하여 그 활성 (주 II)를 억제한다. II 단백질의 존재하지만, 결합되지 않은 "화학 transducer '는 바인더 I 단백질 II의 표면 사이의 상호 작용을 촉진 바인더 II를 통해 단백질 II와 상호 작용합니다. 이 유도 바인더 I-단백질 II 상호 작용은 내가 복잡하고 단백질에 내가 재 활성화 (상태 IV). 'transducer' 단백질의 해리 리드 바인더 I의 유효 농도를 감소 여기를 클릭하십시오이 그림의 더 큰 버전을 볼 수 .
Access restricted. Please log in or start a trial to view this content.
프로토콜
'화학 트랜스 듀서'의 1. 합성
- 사전 준비
- 초순수 114 ml의 아세트산 및 400 mL의 트리 에틸 아민 278 mL를 혼합하여 2 M 트리 에틸 암모늄 아세테이트 (TEAA) 버퍼를 준비한다. 어두운 병에 보관 7 pH를 조정하고 1 L.의 최종 볼륨에 물을 추가합니다.
참고 :이 솔루션은 수년간 안정적입니다. - 초순수 20ml에 아스코르브 산 18 mg을 용해시켜 5 mM의 아스코르브 산 용액을 제조 하였다. 새로운 솔루션을 사용; 용액을 하루 동안 안정하다.
- 11 ml의 디메틸 설폭 사이드 (DMSO)에 TBTA 10 초순수 ml의 물과 58 mg의 구리 (II) 설페이트 수화물 25 mg을 용해시켜 10mM의 구리 (II) / 트리스 (benzyltriazolylmethyl) 아민 (TBTA) 용액을 제조 하였다. 두 용액을 혼합한다. 실온에서 보관하고 빛으로부터 보호.
- 초순수 114 ml의 아세트산 및 400 mL의 트리 에틸 아민 278 mL를 혼합하여 2 M 트리 에틸 암모늄 아세테이트 (TEAA) 버퍼를 준비한다. 어두운 병에 보관 7 pH를 조정하고 1 L.의 최종 볼륨에 물을 추가합니다.
- 활용 절차
- 80 μL에서 수정 된 올리고 뉴클레오티드의 100 nmol의 (ODN-1) 용해신선한 초순수. pH가 = 7. 아스코르브 산 (물에 5 밀리미터)의 갓 만든 용액 80 μl를 추가, 2 M의 TEAA의 20 μl를 추가합니다.
- DMSO 180 μL에 아지 수정 에타 크린 산의 1.5 μmol (574.5 μg의)를 용해 용액에 추가합니다. 구리 (II) / TBTA 용액 (55 %에서 10 밀리미터 (v / v)의 DMSO / 물)에서 40 μl를 추가 신속하게 60 초 동안 아르곤을 사용하여 솔루션을 드가.
- 가까운 단단히, 아르곤 다시 제거하고, 밤새 교반.
- (이동상 : A) 반응의 진행을 모니터링하고 RP-HPLC에 의해 복합체를 정제 5 % 아세토 니트릴, 5 % TEAA, 90 % 초순수; B) 65 % 아세토 니트릴, 5 % TEAA, 30 % 초순수). 24
2. PDGF에 의해 GST의 활동을 제어
- 사전 준비
- 8 mM의 최종 농도로 인산 및 광고를 달성하기 초순수 16.1 ㎖의 인산염 완충 식염수 (PBSx1) 33.9 mL를 혼합하여 분석 완충액 50 ㎖를 준비D의 MgCl 2 23.8 mg을 5 mM의 최종 농도를 달성한다.
- 50 mM 트리스 (pH 7.5), 50 mM의 NaCl을 함유하는 완충액으로 단백질을 용해시켜 GST M1-1의 원액을 제조, 1 mM 디티 오 트레이 톨 (DTT) 및 30 μM의 최종 농도 5 mM의 에틸렌 디아민 테트라 아세트산 (EDTA). -80 ℃에서이 작은 분량 씩에 솔루션 및 저장소를 나눈다. 정량법 버퍼에, 섹션 2.1.5.1에 따르면, 갓 희석하고 얼음에 보관하십시오.
주 : 효소 활성의 감소가있을 때까지 상기 용액을 약 5 시간 동안 안정하거나한다. - 다음 지침에 따라 기판을 준비 :
- 100 mM의 스톡 용액의 최종 농도로 초순수 325 μL의 환원 글루타티온 (GSH) 10 ㎎을 녹인다. 2.1 mM의 작업 용액의 최종 농도에 대한 분석 버퍼의 979 μl의이 원액에서 21 μl를 희석.
- (E)의 492 μL에 2,4- 디 니트로 클로로 벤젠 10 ㎎ (CDNB)을 녹이고100 mM의 스톡 용액의 최종 농도 thanol. 4.32 mM의 작업 용액의 최종 농도에 대한 분석 완충액 956.8 μL의 스톡 용액으로부터 43.2 μL 희석.
- 96 웰 플레이트에서 별도의 라인 (12 웰)의 각 기판을 넣어. 솔루션의 빠르고 쉽게 철회 할 수 있도록 각 웰에 최소 60 μl를 삽입합니다. 빛 보호를위한 알루미늄 시트와 함께 접시를 커버.
- 상기 분석 버퍼는 다음 원액을 준비한다 :
- 이량 체 0.6 μM의 최종 농도로 50 GST M1-1 희석.
- 30 μM 원액에 '화학 물질 변환기를'희석.
- 40 μM의 최종 농도로 희석 PDGF.
- 250 μM의 최종 농도 PDGF 앱 타머를 희석.
- 상기 '화학 센서'의 존재와 PDGF의 GST의 활동을 측정합니다.
- 실험 절차의 난을 설정동역학 측정을 위해 플레이트 판독기 n 개의.
- 는 '표준 프로토콜'과 같은 새로운 실험을 만듭니다.
- '절차'를 눌러 절차 설정 창을 엽니 다.
- 가 상단에 팝업 목록에서 플레이트 제조 업체에 따라 '384 판'유형을 선택합니다.
- 왼쪽 메뉴를 눌러 '읽기'.
- 상기 검출 방법은 '흡광도'선택에.
- 읽기 유형에 대해서는 '종점'을 선택합니다.
- 파장 창에서 340 nm의 쓰기.
- 상단 오른쪽에있는 '전체 판'바닥에 눌러 잘 선택을 측정한다.
- 를 눌러 '확인'을 닫습니다 '읽기'창.
- 선택 왼쪽 메뉴에 '운동 시작'.
- 실행 시간 10 분을합니다.
- 최소 간격 옵션을 선택합니다.
- 를 눌러 '확인'운동 창을 닫습니다.
- '읽기'라인 전을 드래그운동 측정 NTO.
- '검증'버튼을 누른 다음 '확인'버튼을 누릅니다.
- 실험을 저장합니다.
- '재생'버튼을 누릅니다. 대화 상자가 나타납니다 - 측정이 시작되어야 할 때 단지 '확인'버튼을 누릅니다.
- 상기 삼중 실험을 수행하는 네 개의 샘플링 '화학 센서'및 GST M1-1 3.25 μL를 각각 함유하는 3.25 μl를 준비하기 위해. 각각, 각 샘플 0, 1.2, 2.4, 또는 PDGF 및 123.5, 122.3, 121.1의 4.9 μL, 또는 분석 버퍼의 118.6 μL에 추가합니다.
- 10 분 동안 실온에서 용액을 인큐베이션.
- 384 투명 웰 플레이트에 각 웰에 샘플의 40 μl를 삽입합니다. 기판 첨가 멀티 피펫의 사용을 허용하는 경우에만, 홀수 또는 웰은 동일한 행에 짝수 웰에 샘플을 삽입한다.
- 12 채널 다중 피펫을 사용하여, 신속 t 각 10 μL를 추가96 웰 플레이트 (섹션 2.1.4)에 미리 준비 하였다 그는 기판. 거품을 방지하기 위해 신속하고 부드럽게 혼합한다. 독자에 플레이트를 삽입하고 운동 측정을 시작합니다. GST 동력학이 매우 빠르게되기 때문에, 기판을 첨가하고 반응 속도 측정의 시작 사이의 시간을 최소화하려고.
- 실험 절차의 난을 설정동역학 측정을 위해 플레이트 판독기 n 개의.
- GST 활성화 '화학 변환기'에 의해 중재 / 억제 사이클.
- 상기 삼중 실험을 수행 5 샘플 분석 완충액 84.5 μL, '화학 센서'의 3.25 μL 및 GST M1-1 3.25 μl를 함유하는 각각을 준비하기 위해. 3 분 동안 실온에서 인큐베이션.
- 분석 버퍼의 3.65 μL 샘플 2-5에 PDGF의 1과 3.65 μl를 샘플 추가합니다. 3 분 동안 실온에서 인큐베이션.
- 샘플 1-2 분석 버퍼의 3.12 μL 샘플 3-5에 PDGF 앱 타머의 3.12 μl를 추가합니다. 3 분 동안 실온에서 인큐베이션.
- 샘플 분석 버퍼의 24.4 μl를 추가샘플 4-5에 PDGF의 1-3 24.4 μL. 3 분 동안 실온에서 인큐베이션.
- 5 분 동안 실온에서 5 품어을 샘플링 샘플 1 ~ 4 및 PDGF 앱 타머의 7.8 μL에 대한 분석 버퍼의 7.8 μl를 추가합니다.
- 384 투명 웰 플레이트에 각 웰에 시료 40 μl를 삽입합니다. 만 홀수로 또는 만 같은 줄에서조차 우물에 삽입 샘플.
- 12 채널 다중 피펫을 사용하여 신속하게 각 기판 10 μL를 추가 (96- 웰 플레이트에 미리 제조 됨). 거품을 방지하기 위해 신속하고 부드럽게 혼합한다. 독자에 플레이트를 삽입하고 운동 측정을 시작합니다.
- t에서 340 나노 = 활성화 / 금지 재활용 성을 평가하기 위해 t에서 340 내지 = 1.5 분에서 측정 된 OD 0.5 분으로 측정 한 OD를 감산하여 각 조건 하에서 V 0 [MOD / 분]를 계산한다. 25
- 환경의 변화에 '화학 센서'의 실시간 응답을 평가합니다.
- <리> PDGF 첨가 실시간 효과
- 운동 측정 용 플레이트 리더의 실험 절차를 설정합니다.
- 반복 2.2.1.1-2.2.1.10 단계를 반복합니다.
- 실행 시간 3.5 분합니다.
- 최소 간격 옵션을 선택합니다.
- 를 눌러 '확인'운동 창을 닫습니다.
- 운동 측정에 '읽기'라인을 끕니다.
- 왼쪽 메뉴에 /에서 아웃 플레이트를 선택합니다.
- 옵션 '아웃 플레이트 (아무 대화 상자)'을 선택합니다.
- 왼쪽 메뉴의 '지연'옵션을 선택하고 30 초를 입력합니다.
- 왼쪽 메뉴에 /에서 아웃 플레이트를 선택합니다.
- 옵션 '(어떤 대화)의 판'을 선택합니다.
- 2.2.1.14을하지만 섹션 2.2.1.11에서 25 분 대신 10 분으로 운동을 설정 - 단계 2.2.1.4를 반복하여 두 번째 운동 측정을 만듭니다.
- '검증'버튼을 누른 다음 '확인'버튼을 누릅니다.
- 실험을 저장합니다.
- '괞 찮아을 눌러Y '버튼을 클릭합니다. 대화 상자가 나타납니다 - 측정이 시작되어야 할 때 단지 '확인'버튼을 누릅니다.
- GST M1-1의 1 μL 및 분석 버퍼 38 μL의 '화학 센서'의 1 μl를 혼합하여 두 개의 샘플을 준비합니다. 이 두 우물 사이에 빈 우물을 떠나, 384 투명 잘 판의 두 우물에 샘플을 삽입합니다.
- 신속하게 각 기판 10 μL를 추가 부드럽게 혼합하고 빠르게 거품을 방지하기 위해, 리더로 플레이트를 삽입 운동 측정을 시작하는 12 채널 다중 피펫 사용.
- 플레이트는 (3.5 분 후)를 열 때, 신속하게, 우물 중 하나에 PDGF의 1.125 μl를 추가 부드럽게 믹스, 플레이트 나머지 운동 측정을 가까이 할 수 있습니다.
- PDGF 앱 타머를 추가 실시간 효과
- 반복 2.4.1.1-2.4.1.15 단계를 반복합니다.
- GST M1-1의 1 μL, 1 μl를 혼합하여 두 개의 샘플을 준비합니다 '화학 센서 '및 분석 버퍼의 36.9 μL에서 PDGF의 1.125 μL. 이 두 우물 사이에 빈 우물을 떠나, 384 투명 잘 판의 두 우물에 샘플을 삽입합니다.
- 거품을 방지 리더에 플레이트를 삽입하고 운동 측정을 시작하기 위해 빨리 빨리, 각각의 기판에서 10 μl를 추가 부드럽게 혼합하고, 12 채널 멀티 피펫을 사용하여.
- 플레이트는 (1.5 분 후)를 열 때, 신속하게, 우물 중 하나에 PDGF 앱 타머의 1.2 μl를 추가 부드럽게 믹스와 플레이트 나머지 운동 측정을 가까이 할 수 있습니다.
- '화학 센서'와 PDGF의 존재에 GST에 의해 JS-K 전구 약물 활성화를 측정한다.
- 운동 측정 용 플레이트 리더의 실험 절차를 설정합니다.
- 는 '표준 프로토콜'과 같은 새로운 실험을 만듭니다.
- '절차'를 눌러 절차 설정 창을 엽니 다.
- 가 상단에 팝업 목록에서 플레이트 제조 업체에 따라 '384 판'유형을 선택합니다.
- 왼쪽 메뉴를 눌러 '읽기'.
- 상기 검출 방법은 '흡광도'선택에.
- 읽기 유형에 대해서는 '종점'을 선택합니다.
- 파장 창에서 305 nm의 쓰기.
- 상단 오른쪽에있는 '전체 판'버튼을 누르면 잘 선택을 측정한다.
- 를 눌러 '확인'을 닫습니다 '읽기'창.
- 선택 왼쪽 메뉴에 '운동 시작'.
- 실행 시간 10 분을합니다.
- 최소 간격 옵션을 선택합니다.
- 를 눌러 '확인'운동 창을 닫습니다.
- 운동 측정에 '읽기'라인을 끕니다.
- '검증'버튼을 누른 다음 '확인'버튼을 누릅니다.
- 실험을 저장합니다.
- '재생'버튼을 누릅니다. 대화 보X 표시됩니다 - 측정이 시작되어야 할 때 단지 '확인'버튼을 누릅니다.
- NO 생산을위한 측정은 아질산염 / 질산염 열량 키트를 사용합니다. 96 웰 플레이트에서 세 번째 행으로, 한 행에 내가 두 번째 행에 시약은 Griess 70 μL, 및은 Griess II 시약의 70 μl를 분석 버퍼 50 μl를 삽입합니다.
- 상기 삼중 실험을 수행하는 네 개의 샘플링 '화학 센서'각각 함유하는 4.8 μL를 준비하기 위해. 분석 완충액 155.2 μL를 추가 샘플 1; GST-M1-1의 3.2 μL 및 분석 버퍼의 152 μl를 추가, 2를 샘플링하는 단계; PDGF의 9.6 μL 및 분석 버퍼의 145.6 μl를 추가, 3을 샘플링하는 단계; 와, 4 샘플 GST-M1-1의 3.2 μL, PDGF의 9.6 μL 및 분석 버퍼의 142.4 μl를 추가합니다.
- 10 분 동안 실온에서 용액을 인큐베이션.
- 384 투명 웰 플레이트에 각 웰에 시료 50 μl를 삽입합니다. 만 홀수 또는 단지를에 삽입 샘플동일한 라인에도 웰 기질 첨가 멀티 피펫의 사용을 허용한다.
- 각 웰에 JS-K의 0.54 μL (DMSO 5 mm)를 추가합니다.
- 빠르게 거품을 방지하기 위해 신속하게 (96 웰 플레이트에 미리 제조 됨)을 조심스럽게 믹스 GSH 용액 10 μL를 추가하고, 12 채널 다중 피펫 사용. 독자에 플레이트를 삽입하고 운동 측정을 시작합니다.
- Griess의 I 시약 50 μL와 50 μL 즉시 운동 측정 한 후, 12 채널 다중 피펫을 사용하여, 미리 준비된 96- 웰 플레이트에서 분석 완충액 행에 각 시료 50 μL를 가지고 신속하게 추가 은 Griess II 시약. RT에서 10 분 동안 광으로부터 보호하면서 인큐베이션하고 550 nm에서 흡광도를 측정한다.
참고 : 분석 완충액 및 시약의 볼륨이 키트 '프로토콜에 따라 달라집니다.
- 운동 측정 용 플레이트 리더의 실험 절차를 설정합니다.
Access restricted. Please log in or start a trial to view this content.
결과
설계, 합성 및 PDGF와 GST 사이에 인공 커뮤니케이션을 유도 할 수있는 '화학 센서'의 작용 메커니즘은 그림 2에 제시되어있다. '변환기'의 구조는 PDGF DNA 앱 타머 및 비스 - 에타 크린 아미드를 통합 (BEA ), 각각 26 nm의 144 nm의, 알려진 GST 억제제 (그림 2a). (19)이 바인더 (해리 상수, K d 개를 '변환기', 즉 다른 친 화성을 ...
Access restricted. Please log in or start a trial to view this content.
토론
We presented a method for designing and testing of a 'chemical transducer' that can induce artificial communication between two naturally unrelated proteins, GST and PDGF, without modifying the native proteins. The unnatural GST-PDGF communications could be detected in real time by using enzymatic assays that follow the changes in the activity of GST in the presence of the 'chemical transducer' and increasing the concentrations of PDGF. In addition to detecting the activation of GST by PDGF, these assays were used to fol...
Access restricted. Please log in or start a trial to view this content.
공개
The authors have nothing to disclose.
감사의 말
이 연구는 미네르바 재단, HFSP 조직 및 유럽 연구위원회 그랜트에 의해 지원되었다 (부여 338265 시작).
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 1-chloro-2,4-dinitrobenzene | Sigma-Aldrich | 237329 | |
| Acetic acid | Bio Lab | 01070521 | |
| Acetnitrile | J.T.Baker | 9017-03 | |
| Ascorbic acid | Sigma-Aldrich | A4544 | |
| Copper(II) Sulfate pentahydrate | Merck-Millipore | 102790 | |
| Dimethyl sulfoxide | Merck-Millipore | 802912 | |
| Dulbecco's Phosphate Buffered Saline | Biological Industries | 02-023-5A | |
| Ethacrynic acid | Tokyo Chemical Industry Co. Ltd | E0526 | |
| Glutathione-s-transferase M1-1 | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| JS-K | Sigma-Aldrich | J4137 | |
| L-glutathione reduced | Sigma-Aldrich | G4251 | |
| Magnesium Chloride | J.T.Baker | 0162 | |
| nitrate/nitrite colorimetric assay kit | Cayman Chemical | 780001 | |
| Oligonucleotides | W. M. Keck Foundation Biotechnology at Yale University | custom order | |
| PDGF-BB | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| TBTA | Sigma-Aldrich | 678937 | |
| Triethylamine | Sigma-Aldrich | T0886 | |
| Desalting column | GE Healthcare | illustra MicroSpin G-25 Columns | |
| HPLC | Waters | 2695 separation module | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 4.6 mm × 50 mm) | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 10 mm × 50 mm) | |
| Plate reader | BioTek | synergy H4 hybrid |
참고문헌
- Hunter, T. Signaling—2000 and Beyond. Cell. 100, 113-127 (2000).
- Levitzki, A., Klein, S. Signal transduction therapy of cancer. Mol Aspects Med. 31, 287-329 (2010).
- Peri-Naor, R., Motiei, L., Margulies, D. Artificial signal transduction therapy: a futuristic approach to disease treatment. Future Med. Chem. 7, 2091-2093 (2015).
- Peri-Naor, R., Ilani, T., Motiei, L., Margulies, D. Protein-Protein Communication and Enzyme Activation Mediated by a Synthetic Chemical Transducer. J. Am. Chem. Soc. 137, 9507-9510 (2015).
- Corson, T. W., Aberle, N., Crews, C. M. Design and Applications of Bifunctional Small Molecules: Why Two Heads Are Better Than One. ACS Chem. Biol. 3, 677-692 (2008).
- Rutkowska, A., Schultz, C. Protein Tango: The Toolbox to Capture Interacting Partners. Angew. Chem. Int. Ed. 51, 8166-8176 (2012).
- Meyer, C., Köhn, M. A Molecular Tête-à-Tête Arranged by a Designed Adaptor Protein. Angew. Chem. Int. Ed. 51, 8160-8162 (2012).
- Klemm, J. D., Schreiber, S. L., Crabtree, G. R. Dimerization as a Regulatory Mechanism in Signal Transduction. Annu. Rev. Immunol. 16, 569-592 (1998).
- DeRose, R., Miyamoto, T., Inoue, T. Manipulating signaling at will: chemically-inducible dimerization (CID) techniques resolve problems in cell biology. Pflugers Arch. 465, 409-417 (2013).
- Gestwicki, J. E., Marinec, P. S. Chemical control over protein-protein interactions: beyond inhibitors. Comb. Chem. High Throughput. Screen. 10, 667-675 (2007).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. 25, 848-862 (2013).
- Diezmann, F., Seitz, O. DNA-guided display of proteins and protein ligands for the interrogation of biology. Chem. Soc. Rev. 40, 5789-5801 (2011).
- Röglin, L., Ahmadian, M. R., Seitz, O. DNA-Controlled Reversible Switching of Peptide Conformation and Bioactivity. Angew. Chem. Int. Ed. 46, 2704-2707 (2007).
- Röglin, L., Altenbrunn, F., Seitz, O. DNA and RNA-Controlled Switching of Protein Kinase Activity. ChemBioChem. 10, 758-765 (2009).
- Harris, D. C., Chu, X., Jayawickramarajah, J. DNA-Small Molecule Chimera with Responsive Protein-Binding Ability. J. Am. Chem. Soc. 130, 14950-14951 (2008).
- Harris, D. C., Saks, B. R., Jayawickramarajah, J. Protein-Binding Molecular Switches via Host-Guest Stabilized DNA Hairpins. J. Am. Chem. Soc. 133, 7676-7679 (2011).
- Kim, Y., Cao, Z., Tan, W. Molecular assembly for high-performance bivalent nucleic acid inhibitor. Proc. Nat. Acad. Sci. U.S.A. 105, 5664-5669 (2008).
- Han, D., et al. A Logical Molecular Circuit for Programmable and Autonomous Regulation of Protein Activity Using DNA Aptamer-Protein Interactions. J. Am. Chem. Soc. 134, 20797-20804 (2012).
- Motiei, L., Pode, Z., Koganitsky, A., Margulies, D. Targeted Protein Surface Sensors as a Tool for Analyzing Small Populations of Proteins in Biological Mixtures. Angew. Chem. Int. Ed. 53, 9289-9293 (2014).
- Ranallo, S., Rossetti, M., Plaxco, K. W., Vallée-Bélisle, A., Ricci, F. A Modular, DNA-Based Beacon for Single-Step Fluorescence Detection of Antibodies and Other Proteins. Angew. Chem. Int. Ed. 54, 13214-13218 (2015).
- Franzini, R. M., et al. Identification of Structure-Activity Relationships from Screening a Structurally Compact DNA-Encoded Chemical Library. Angew. Chem. Int. Ed. 54, 3927-3931 (2015).
- Unger-Angel, L., et al. Protein recognition by bivalent, 'turn-on' fluorescent molecular probes. Chem. Sci. 6, 5419-5425 (2015).
- Nissinkorn, Y., et al. Sensing Protein Surfaces with Targeted Fluorescent Receptors. Chem. Eur. J. 21, 15981-15987 (2015).
- Huber, C. G., Oefner, P. J., Bonn, G. K. High-Resolution Liquid Chromatography of Oligonucleotides on Nonporous Alkylated Styrene-Divinylbenzene Copolymers. Anal. Biochem. 212, 351-358 (1993).
- Lyon, R. P., Hill, J. J., Atkins, W. M. Novel class of bivalent glutathione S-transferase inhibitors. Biochemistry. 42, 10418-10428 (2003).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. , 1-16 (2013).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유