需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
细菌生长的精确、高通量分析
摘要
细菌生长的定量评价是理解微生物生理学作为系统级现象的关键。本文介绍了一种实验操作和分析方法的协议, 允许对细菌生长进行精确、高通量的分析, 这是系统生物学研究的一个重要课题。
摘要
细菌生长是现代微生物生理学发展的一个中心概念, 也是在系统层面上对细胞动力学的研究。最近的研究报告了细菌生长和全基因组事件之间的相关性, 如基因组减少和转录重组。正确分析细菌生长对理解生长依赖的基因功能和细胞成分的协调至关重要。因此, 需要以高通量的方式对细菌生长进行精确的定量评价。新兴的技术发展提供了新的实验工具, 允许更新用于研究细菌生长的方法。这里介绍的协议雇用了一个微的读者与一个高度优化的实验程序, 对细菌生长的可再生和精确的评估。此协议用于评估以前描述的几种大肠杆菌菌株的生长情况。该议定书的主要步骤如下: 在小瓶中制备大量的细胞储存, 用于重复试验, 具有重现性的结果, 使用 96-井板进行高通量增长评价, 并手工计算两个主要表示增长动态的参数 (即、最大增长率和人口密度)。与传统的菌落形成单元 (CFU) 测定法相比, 它对琼脂板上的玻璃管中培养的细胞进行了计数, 目前的方法更加有效, 提供了更详细的生长变化的时间记录, 但具有更严格低人口密度的检测限制。总之, 所描述的方法有利于精确和可重复的高通量分析细菌生长, 可用于绘制概念性结论或作出理论观察。
引言
微生物研究经常从细菌细胞的培养和细菌生长曲线的评估开始, 这代表细菌生理学的一个基本现象1,2,3。基本文化原则在已出版的研究文献和教科书中得到广泛应用, 因为细菌培养是一种基本的方法。在替补席上, 传统上一直关注于优化生长培养基和培养条件, 但控制生长速度, 这可能会提供更大的理解微生物生理学, 没有广泛研究了4。对于指数生长的细菌来说, 细胞状态的一个关键参数是生长速率, 据报告, 它与基因组、转录和蛋白质群相协调5,6,7,8.因此, 对细菌生长的定量评价是了解微生物生理学的关键。
为了评价细菌的生长, 用于估计生物量的实验方法已经建立了9,10 , 并基于检测生化、物理或生物参数, 如光学浊度。此外, 用于捕获生长变化动态特性的分析方法通常基于建立的非线性模型11,12,13, 例如, 逻辑方程。生长动力学通常通过测量光学浊度或进行菌落形成单元 (CFU) 测定来获得细胞生长的定时取样。这些培养和检测方法的局限性在于, 数据点不是人口动态的真实反映, 因为测量间隔通常是小时, 因为培养条件 (例如, 温度的变化和曝气) 在取样时受到干扰。必须利用技术和理解方面的最新发展来更新文化和分析技术。微读者的最新进展使 real-time 观察细菌生长, 并大大降低劳动力成本。利用这些先进的设备, 最新的细菌生长研究报告了高通量测量的分析方法14,15。
本议定书的目的是以高通量的方式评估精确的增长动态, 这对于最终解决增长率如何确定以及影响增长率的问题的定量研究将是有价值的。该议定书处理的所有因素, 应考虑到可重复和精确的定量细菌生长。本文主要对实验方法和分析进行了详细的介绍。这种方法允许以高通量的方式精确和重现地分析细菌的生长。微生物学可以利用这个协议从他们的实验证据中获得额外的定量结果。该协议也可用于系统生物学的研究, 试图得出概念性结论或实现对增长的理论概述。
研究方案
1. 准备生长培养基
注意: 最小介质 M63 的化学成分如下:62 mm K 2 HPO 4 , 39 mm, 2 PO 4 , 15 mm (NH 4 ) 2 因此 4 、1.8 和 #181; m FeSO 4 、15和 #181; m 硫胺素-HCl、0.2 mm MgSO 4 和 22 mm 葡萄糖。M63 是由混合三库存解决方案: 五 X 解决方案, 20% 葡萄糖和 MgSO 4 硫胺素溶液。将所有解决方案存储在4和 #176; C.
- 准备五 X 解决方案
- 以准备 FeSO 4 解决方案, 请使用电子吸管和一次性血清吸管将 ddH 2 O 添加到50毫升离心管中。使用 P-200 添加0.06 毫升 hcl, 以获得0.01 米 hcl. 添加36毫克 FeSO 4 -7H 2 O 并混合好.
- 测量 160 ml ddH 2 O 在计量缸中, 并将其添加到500毫升的烧杯中。按以下顺序添加下列内容: 10.72 g K 2 HPO 4 , 5.24 g, 2 PO 4 , 2.0 g (NH 4 ) 2 所以 4 , 和 0.5 mL FeSO 4 解决方案 (在步骤1.1.1 中准备).
- 使用精密 ph 计, 将 ph 值调整到 7.0, 增加2米。将解决方案倒入测量缸中, 并将 ddH 2 O 添加到总体积为200毫升。使用250毫升过滤装置 (PVDF、0.22 和 #181; M) 对解决方案进行消毒.
- 在测量缸中准备20% 葡萄糖溶液
- 测量 200 ml ddH 2 O, 并将其倒入一个500毫升的烧杯中。在混合使用磁力搅拌器的同时, 加入50克葡萄糖。稀释溶液在一个测量缸到总体积为250毫升。按照步骤1.1.3 中的说明对解决方案进行灭菌.
- 准备 MgSO 4 硫胺素解决方案
- 向500毫升的烧杯添加 150 ml ddH 2 O。在与磁力搅拌器混合时, 加入1.0 克硫胺-HCl 和10克 MgSO 4 -7H 2 o. 将测量缸中的溶液稀释到总容积200毫升。按照步骤1.1.3 中的说明对解决方案进行灭菌.
- 准备最小介质 M63
- 放置五 X 解决方案、20% 葡萄糖解决方案、MgSO 4 硫胺素解决方案、电子吸管、P-200 吸管和 200-#181; 在干净的长凳上的吸管提示.
- 测量 155.8 ml ddH 2 O 在计量缸中, 并将其倒入500毫升的烧杯中.
- 使用电子吸管和一次性血清吸管, 将40毫升的五 X 溶液和4毫升20% 葡萄糖溶液添加到测量的 ddH 2 O。然后, 添加0.2 毫升 MgSO 4 硫胺素溶液与 P-200。按照步骤1.1.3 中的说明对解决方案进行灭菌.
2。准备甘油库存
- 单元格区域性
注意: 细菌菌株 (如 、W3110 及其减少的基因组) 可从应变库组织中获得。菌株通常以琼脂板上的菌落的形式获得。- 将五消毒玻璃管与硅橡胶塞子、电子吸管、P-1,000、1000-#181; l 吸管提示, P-200, 200 和 #181; l 吸管提示, 和目标菌株 (板块上的殖民地) 在一个干净的长凳上.
- 在打开硅胶瓶塞之前, 将玻璃管的口暴露在本生燃烧器上。在管子打开后, 将硅橡胶塞子暴露在火焰中, 然后轻轻地将瓶盖放回玻璃管上.
- 使用电子吸管和一次性血清吸管将5毫升 M63 添加到其中一个玻璃管和4.5 毫升 M63 到其他四管.
- 使用 P-200 提示选择一个菌落, 并将其接种在含有5毫升 M63 的玻璃管中.
- 涡流管使其悬浮。然后, 通过将本解决方案的 0.5 ml 转换为包含 4.5 ml M63 的四管之一, 将溶液稀释10倍.
- 重复步骤2.1.5 中描述的剩余管的过程。五种不同浓度的稀释系列 (稀释1、10、100、1000和 1万) 现在已经准备就绪.
- 消毒玻璃管和硅橡胶塞子的嘴, 如步骤2.1.2 所述。用塞子把管子盖上。避免不起皱的硅橡胶塞子的污染.
- 将五根管子放在5ml 摇动孵化箱中, 在37和 #176; C, 在 200 rpm 处晃动。在夜间或10至30小时孵育养殖.
- 选择甘油库存的区域性
- 将5ml 室温 M63 介质、P-1,000、1000和 #181; L 吸管提示和一次性试管在干净的长凳上.
- 添加1000和 #181; 我 M63 一个 P-1,000 的一次性试管。将一次性试管置于分光光度计中, 以 600 nm 的固定波长启动程序, 并测量空白.
- 将五玻璃管从摇动孵化器移到干净的长凳上.
- 丢弃 M63 从一次性试管, 并添加1000和 #181; L 文化到相同的一次性试管与 P-1,000。测量单元区域性的光学浊度 (OD 600 ), 如步骤2.2.2 中所述.
注意: 为避免任何污染, 并实现精确测量, 将玻璃管和塞子暴露在火焰中, 并在取样前对其进行涡流。为了确保可靠的结果, 建议重复测量, 特别是当细胞密度较低. - 在五单元格区域性中, 选择一个位于早期的指数增长阶段 (OD 600 = 0.01-0.05), 用于甘油库存.
注意: 如果多个区域性在最佳范围内有密度, 则通常选择最接近0.05 的一个.
- 为重复测试进行甘油储存.
注: 这是为准备十股票的描述。根据实验要求, 可以做较大或更小的量。- 将灭菌的60% 甘油溶液、十消毒1.5 毫升管内、P-1,000 和 P-200、1000-和 200-和 #181; L 吸管提示, 微站在干净的长凳上.
- 添加250和 #181; 我用60% 的甘油溶液和750和 #181 消毒; 选择的细胞培养微1.5 毫升的移.
注意: 库存量是可变的, 但始终保持1:3 的比率60% 甘油的细胞培养;这导致最终浓度为15% 甘油. - 将剩余的九管内放在微支架上, 并分配100和 #181; 在步骤2.3.2 中准备的混合物的 L。现在有十相同的甘油库存供将来使用.
- 将库存存储在-80 和 #176 的深度冷冻库中; C.
3。获取增长曲线
- 设置微读取器.
注: 用引号显示的术语显示了在这里使用的平板阅读器的具体语法 (参见材料表)。- 打开该软件。开放和 #34;P rotocols 和 #34; #34; 任务管理器和 #34; 选择和 #34; 创建新的 #34;。选择和 #34; 标准协议和 #34;.
- 打开和 #34;P 程序和 #34;, 并调整设置。打开和 #34; 设置温度和 #34; 选择和 #34; 孵化器和 #34;设置和 #34; 温度和 #34; 37 和 #176; c 和 #34; 渐变和 #34; 到0和 #176; #34;P 再热 #34; 在继续下一步之前。打开和 #34; 启动动力学和 #34; 设置和 #34; 运行时间和 #34; 24:00:00 或 48:00:00, 和 #34; 间隔和 #34; 00:30:00 或 01:00:00.
注: 大约需要1分钟才能阅读整个96井板. - 打开和 #34; 抖动和 #34; 设置和 #34; 抖动模式和 #34; #34; 线性和 #34;检查和 #34; Constitution 震动和 #34; 设置和 #34; 频率和 #34; 在 567 cpm。打开和 #34; 读取和 #34; 检查和 #34; 吸光度和 #34;, #34; 终点/动能和 #34; 和 #34; 单色 #34; 设置和 #34; 波长和 #34; 到 600.
- 单击和 #34; 验证和 #34; 确认程序正确。单击并 #34; 保存和 #34; 将其保存为新程序以备将来使用.
- 实时增长记录
- 绘制一个96井板象形 (8 和 #215; 12 表), 以指示在96井板上接种的培养样品的位置。打印出表并将其用作实验的参考.
注: 位于微边缘的井只因蒸发而含有空白介质. - 放置一个灭菌的96平底微, 盖子、P-1000、1000和 #181; 吸管提示, P-200, 200 和 #181; l 吸管提示, 数1.5 毫升管内, 8 多通道吸管, 灭菌试剂库, 室温 M63, 甘油库存 (在步骤2.3 中准备) 在一个干净的长凳上.
- 在试剂库中添加大约25毫升 M63。将此库存量用于以下所有步骤.
- 添加900和 #181; 我 M63 管内准备制作串口稀释.
- 在室温下解冻甘油库存。添加900和 #181; 我 M63 到解冻的甘油库存和涡流。这导致了10倍的稀释原甘油库存.
- 转移100和 #181; 将10倍稀释的 l 转到另一微, 其中包含900和 #181; l M63 和涡旋。这导致100倍的稀释.
- 重复步骤3.2.6 直到达到所需的稀释数.
- 用200和 #181 填充微边缘的井; M63 使用8通道吸管 (P-200).
注: 这些井可以用作空白. - 加载200和 #181; 根据参考表 (步骤 3.2.1), 在步骤 3.2.4 3.2.7 对微井进行的每个稀释样品的 L。在板上不同位置的多个井中加载和装载相同样品之前, 先将稀释过的样品涡旋.
- 将96井微放在平板读卡器上.
- 打开和 #34; 现在阅读和 #34; #34; 任务管理器和 #34; 选择程序 (3.1 节)。单击并 #34; 确定 #34; 开始测量。将录制保存为数据分析的新实验文件 (4 节).
- 绘制一个96井板象形 (8 和 #215; 12 表), 以指示在96井板上接种的培养样品的位置。打印出表并将其用作实验的参考.
4。数据分析
- 将 real-time 增长速率数据 (第3.2 节) 的结果导出到 USB 内存棒作为文本文件.
注:96 个格式化的结果 (曲线) 将显示在 real-time 的平板阅读器上。每小时记录 (OD 值) 可以导出为表;行和列分别表示井号 ( 例如 、A1、B1) 和测量时间 ( 例如 , 00:00:59). - 使用电子表格软件打开文本文件.
- 按小时 OD 600 将96井微的读数复制到新工作表中以进行进一步分析.
- 从每个样本的每小时读取中减去背景读取时间为零.
注: 为方便起见, 含 M63 (步 3.2.8) 的空井均值可作为背景值. - 计算五连续 OD 的平均值 600 读取以估计最大人口密度.
注意: 五连续 od 600 读取的最大平均值定义为相应增长曲线的最大 od 600 。 - 计算增长率, 和 #181; (h -1 ), 通过对 OD 600 的所有对连续的值应用下列公式:
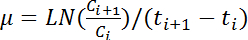
注意: 在这个等式中, c i 和 c i+1 表示任意两个连续时间点 (t i 和 t i+1 的 OD 600 值,分别)。LN 表示自然对数. - 根据每小时 OD 600 按照步骤4.6 进行读取, 计算增长速率随时间的变化。计算五连续增长率的平均值和标准差, 以估计最大增长率.
注意: 最大的平均值与最小的标准偏差被定义为相应增长曲线的最大增长率.
5。确认96井读数 (可选) 的全局偏差
注意: 板读写器和可消耗的 96-井板都可能导致偏差测量。为了获得高精度和可重复性的定量结果, 强烈建议确认96井板的全球偏差.
- 为偏压测试准备96井板和平板读卡器, 如第3.2.2 节所述.
- 添加20毫升 M63 到一个灭菌50毫升离心管。将甘油库存添加到相同的离心管和涡流中。将悬浮溶液转移到灭菌试剂库.
- 使用8通道吸管转移200和 #181; L 的悬浮解决方案的 96-井板.
- 将 96-井板放在平板读卡器上并开始测量.
- 记录每小时 OD 600 读取和分析, 如4节所述。比较各井的最大生长速率和人口密度, 以确定96井读数的位置偏差.
结果
所描述的方法提供了一种手段, 以连续的, 高通量的方式捕捉动态细菌生长, 利用96井格式的阅读器, 在不同的时间间隔采取多种光学密度测量 (从分钟到小时到天)。在一个单一的实验中, 可以精确地获得表达各种基因组的大肠杆菌菌株的生长曲线 (图 1A)。与所描述的方法相比, 传统方法 (CFU 分析) 通常需要更长的采样时间间隔 (图 ...
讨论
协议中的关键步骤包括准备一股指数增长的细胞的普通股, 以及在微的不同位置的多个井中复制相同的样品。以前, 微生物学从一夜培养开始了文化。虽然这种方法可以减少细菌生长的滞后时间, 但很难获得可再生的生长曲线。如图 2所示, 使用普通甘油库存的独立测量导致几乎相同的生长曲线, 从而提供了重现性和精确的结果。如图 3所示, 不同位置的水...
披露声明
我们感谢平土提供 CFU 化验的例子。这项工作得到了部分财政支助, 其经费来自日本教育、文化、体育、科学和技术部 (补助金) 26506003 号 (BWY) 的科学研究。
致谢
作者没有什么可透露的。
材料
| Name | Company | Catalog Number | Comments |
| K2HPO4 | Wako | 164-04295 | |
| KH2PO4 | Wako | 166-04255 | |
| (NH4)2SO4 | Wako | 019-03435 | |
| MgSO4-7H2O | Wako | 138-00415 | |
| Thiamine-HCl | Wako | 201-00852 | |
| glucose | Wako | 049-31165 | |
| HCl | Wako | 080-01066 | |
| Iron (II) sulfate heptahydrate (FeSO4-7H2O) | Wako | 094-01082 | |
| KOH | Wako | 168-21815 | |
| Glycerol | Wako | 075-00611 | |
| Centrifuge tube (50 mL, sterilized) | WATSON | 1342-050S | |
| Pipette Tips, 200 µL | WATSON | 110-705Y | |
| Pipette Tips, 1,000 µL | WATSON | 110-8040 | |
| Microtube (1.5 mL) | WATSON | 131-715C | |
| 8 multichannel-pipette | WATSON | NT-8200 | |
| PASORINA STIRRER | AS ONE | 2-4990-02 | |
| Glass cylinder (200 mL) | AS ONE | 1-8562-07 | |
| Precision pH mater | AS ONE | AS800 / 1-054-01 | |
| Pipetman P-200 | GILSON | 1-6855-05 | |
| Pipetman P-1000 | GILSON | 1-6855-06 | |
| Disposable Serolocical Pipettes (10 mL) | SANPLATEC | SAN27014 | |
| Disposable Serolocical Pipettes (25 mL) | SANPLATEC | SAN27015 | |
| Microtube stand | BM Bio | 801-02Y | |
| Vortex | BM Bio | BM-V1 | |
| Corning Costar 96-well microplate with lid (Flat bottom, Clear) | Sigma-Aldrich | Corning, 3370 | |
| Corning Costar reagent reservoir (50 mL) | Sigma-Aldrich | Corning, 4870 | |
| Stericup GV PVDF (250 mL, 0.22 µM) | Merck Millipore | SCGVU02RE | |
| Pipet-Aid XP | DRUMMOND | 4-000-101 | |
| Bioshaker (BR-23UM MR) | TAITEC | 0053778-000 | |
| Disposal cell (1.5 mL) | Kartell | 1938 / 2-478-02 | |
| DU 730 Life Science UV/Vis Spectrophotometer | Beckman Coulter | A23616 | |
| EPOCH2 | BioTek | 2014-EP2-002 / EPOCH2T | |
| Beaker (500 mL) | IWAKI | 82-0008 | |
| BIO clean bench | Panasonic | MCV-B131F | |
| Glass tubes | NICHIDEN RIKA GLASS | P-10M~P-30 /101019 | |
| Silicone rubber stoppers | ShinEtsu Polymer | T-19 | |
| Bacterial strains | Strain bank organization; National Bio Resource Project (NBRP) in Japan |
参考文献
- Kovarova-Kovar, K., Egli, T. Growth kinetics of suspended microbial cells: from single-substrate-controlled growth to mixed-substrate kinetics. Microbiol Mol Biol Rev. 62 (3), 646-666 (1998).
- Soupene, E., et al. Physiological studies of Escherichia coli strain MG1655: growth defects and apparent cross-regulation of gene expression. J Bacteriol. 185 (18), 5611-5626 (2003).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Egli, T. Microbial growth and physiology: a call for better craftsmanship. Front Microbiol. 6, 287 (2015).
- Kurokawa, M., Seno, S., Matsuda, H., Ying, B. W. Correlation between genome reduction and bacterial growth. DNA Res. 23 (6), 517-525 (2016).
- Matsumoto, Y., Murakami, Y., Tsuru, S., Ying, B. W., Yomo, T. Growth rate-coordinated transcriptome reorganization in bacteria. BMC Genomics. 14, 808 (2013).
- Nahku, R., et al. Specific growth rate dependent transcriptome profiling of Escherichia coli K12 MG1655 in accelerostat cultures. J Biotechnol. 145 (1), 60-65 (2010).
- Dai, X., et al. Reduction of translating ribosomes enables Escherichia coli to maintain elongation rates during slow growth. Nat Microbiol. 2, 16231 (2016).
- Madrid, R. E., Felice, C. J. Microbial biomass estimation. Crit Rev Biotechnol. 25 (3), 97-112 (2005).
- Harris, C. M., Kell, D. B. The estimation of microbial biomass. Biosensors. 1 (1), 17-84 (1985).
- Yates, G. T., Smotzer, T. On the lag phase and initial decline of microbial growth curves. J Theor Biol. 244 (3), 511-517 (2007).
- Kargi, F. Re-interpretation of the logistic equation for batch microbial growth in relation to Monod kinetics. Lett Appl Microbiol. 48 (4), 398-401 (2009).
- Peleg, M., Corradini, M. G. Microbial growth curves: what the models tell us and what they cannot. Crit Rev Food Sci Nutr. 51 (10), 917-945 (2011).
- Sprouffske, K., Wagner, A. Growthcurver: an R package for obtaining interpretable metrics from microbial growth curves. BMC Bioinformatics. 17, 172 (2016).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Mol Biol Evol. 31 (1), 232-238 (2014).
- Hermsen, R., Okano, H., You, C., Werner, N., Hwa, T. A growth-rate composition formula for the growth of E.coli on co-utilized carbon substrates. Mol Syst Biol. 11 (4), 801 (2015).
- Engen, S., Saether, B. E. r- and K-selection in fluctuating populations is determined by the evolutionary trade-off between two fitness measures: Growth rate and lifetime reproductive success. Evolution. 71 (1), 167-173 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。