Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Präzise, Hochdurchsatz-Analyse des Bakterienwachstums
In diesem Artikel
Zusammenfassung
Quantitative Bewertung des Bakterienwachstums unbedingt mikrobielle Physiologie als Systemebene Phänomen zu verstehen. Ein Protokoll für experimentelle Manipulation und ein analytischer Ansatz eingeführt, ermöglicht präzise, Hochdurchsatz-Analyse der bakteriellen Wachstum, das ist ein zentrales Thema des Interesses in der Systembiologie.
Zusammenfassung
Bakterielles Wachstum ist ein zentrales Konzept in der Entwicklung der modernen mikrobielle Physiologie, sowie bei der Untersuchung der zelluläre Dynamik auf der Systemebene. Jüngste Studien haben Zusammenhänge zwischen Bakterienwachstum und genomweite Ereignisse, z. B. Genom Reduktion und Transkriptom Reorganisation berichtet. Richtig analysieren Bakterienwachstum ist entscheidend für das Verständnis der Wachstum-abhängige Koordination von Genfunktionen und zellulären Komponenten. Dementsprechend ist die präzise quantitative Auswertung des Bakterienwachstums in gewissem Sinne Hochdurchsatz-erforderlich. Neue technologische Entwicklungen bieten neue experimentelle Tools, die Updates über die Methoden zur Untersuchung von Bakterienwachstum zu ermöglichen. Die hier vorgestellte Protokoll beschäftigt einen Mikrotestplatte Leser mit einem hochoptimierten experimentelle Verfahren für die präzise und reproduzierbare Auswertung des Bakterienwachstums. Dieses Protokoll wurde verwendet, um das Wachstum von mehreren Werten zuvor beschriebenen Escherichia-coli -Stämme. Die wichtigsten Schritte des Protokolls sind wie folgt: die Vorbereitung einer großen Anzahl der Zelle Bestände in kleinen Fläschchen für wiederholte Prüfungen mit reproduzierbaren Ergebnissen, die Verwendung von 96-Well Platten für Hochdurchsatz-Wachstum-Bewertung und die manuelle Berechnung von zwei großen Parameter (d.h. maximale Wachstum Rate und Bevölkerung Dichte) repräsentieren die Wachstumsdynamik. Im Vergleich zu den traditionellen koloniebildenden Einheit (KBE) Assay, der die Zellen, die in Glasröhren im Laufe der Zeit auf Agarplatten kultiviert werden zählt, die vorliegende Methode ist effizienter und bietet detailliertere zeitliche Aufzeichnungen über Wachstum Veränderungen, aber hat eine strengere Nachweisgrenze bei niedrigen Bevölkerungsdichte. Zusammenfassend ist das beschriebene Verfahren vorteilhaft für die präzise und reproduzierbare Hochdurchsatz-Analyse des bakteriellen Wachstums, der konzeptionellen Schlussfolgerungen ziehen oder theoretische Bemerkungen verwendet werden kann.
Einleitung
Mikrobiologische Untersuchungen beginnen oft mit der Kultur der Bakterienzellen und der Bewertung der Bakterienwachstum Kurven, die ein grundlegendes Phänomen der bakteriellen Physiologie1,2,3darstellen. Grundlegende Kultur Prinzipien sind weit verbreitet in der veröffentlichten Literatur und Lehrbücher, weil Bakterienkultur eine grundlegende Methodik ist. Die Ebene der Bank erhebliche Aufmerksamkeit hat traditionell auf die Optimierung Wachstumsmedien und Kultivierung Bedingungen, sondern steuern die Wachstumsrate, die wahrscheinlich noch größeres Verständnis für mikrobielle Physiologie bieten würde wurde nicht ausgiebig studiert4. Für exponentiell wachsenden Bakterien, ist ein wichtiger Parameter der zellulären Staat die Wachstumsrate, die berichtet wurde, das Genom, Transkriptom und Proteom5,6,7,8 abgestimmt sein . So ist die quantitative Bewertung des Bakterienwachstums entscheidend für das Verständnis der mikrobielle Physiologie.
Um bakterielles Wachstum zu bewerten, die experimentellen Methoden zur Schätzung der Biomasse sind gut etablierte9,10 und basieren auf dem Nachweis von biochemischen, physikalische oder biologische Parameter, z. B. optische Trübung. Darüber hinaus basieren die analytischen Methoden verwendet, um die dynamischen Eigenschaften des Wachstums Änderungen erfassen häufig auf etablierte nichtlineare Modelle11,12,13, z. B. logistische Gleichungen. Wachstumsdynamik sind in der Regel durch zeitgesteuerte Sampling des Zellwachstums in der Kultur durch optische Trübungsmessung oder Durchführung von koloniebildenden Einheit (KBE) Assays erworben. Die Einschränkung dieser Kultivierung und Erkennung Methoden ist, dass die Datenpunkte kein getreues Spiegelbild der Populationsdynamik sind, da die Messintervalle oft in Stunden sind und die Kultur-Bedingung (z. B.Veränderungen in Temperatur und Belüftung) ist zum Zeitpunkt der Probenahme gestört. Kultur und Analysetechniken müssen aktualisiert werden, mit den letzten Entwicklungen in Technologie und Verständnis. Jüngste Fortschritte in der Mikrotestplatte Leser erlauben die Echtzeit-Beobachtung des Bakterienwachstums und Arbeitskosten deutlich zu verringern. Mit diesen fortschrittlichen Geräten, haben die neuesten Studien über Bakterienwachstum Analysemethoden für Hochdurchsatz-Messungen14,15berichtet.
Dieses Protokoll soll die genaue Wachstumsdynamik in gewissem Hochdurchsatz-bewerten, die quantitative Studien nützlich sein werden, die letztlich die Fragen wie die Wachstumsrate bestimmt wird und welche Faktoren beeinflussen die Wachstumsrate. Das Protokoll befasst sich mit allen Faktoren, die für die reproduzierbare und genaue Quantifizierung des Bakterienwachstums berücksichtigt werden sollten. Die experimentelle Methode und Analyse sind detailliert im Haupttext beschrieben. Diese Methode erlaubt die präzise und reproduzierbare Analyse des Bakterienwachstums in gewissem Sinne Hochdurchsatz. Mikrobiologen können dieses Protokoll verwenden, um zusätzliche quantitative Ergebnisse aus ihrer experimentellen Beweise ableiten. Dieses Protokoll kann auch für Studien in der Systembiologie verwendet werden, die versuchen, konzeptionellen Schlussfolgerungen zu ziehen oder einen theoretischen Überblick über Wachstum zu erreichen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Vorbereitung das Wachstumsmedium
Hinweis: die chemische Zusammensetzung der minimale mittlere M63 lautet wie folgt: 62 mM K 2 HPO 4, 39 mM KH 2 PO 4, 15 mM (NH 4) 2 SO 4 1,8 µM FeSO 4, 15 µM Thiamin-HCl, 0,2 mM MgSO 4 und 22 mM Glukose. M63 erfolgt durch das Mischen von drei Stammlösungen: 5 X Lösung, 20 % Glukose und MgSO 4 Thiamin-Lösung. Speichern Sie alle Lösungen bei 4 ° c
- Herstellung der fünf X-Lösung
- , FeSO 4 Lösung vorzubereiten benutzen eine elektrische Pipette und ein Einweg-serologische Pipette eine 50 mL Zentrifugenröhrchen DdH 2 O hinzu. Verwenden Sie eine P-200 hinzufügen 0,06 mL HCl zu erhalten 0,01 M HCl. Add 36 mg FeSO 4-7 H 2 O und gut verrühren.
- 160 mL DdH 2 O in einem Messzylinder messen und einen 500-mL-Becherglas hinzufügen. Fügen Sie die folgenden in dieser Reihenfolge: 10,72 g K 2 HPO 4, 5,24 g KH 2 PO 4, 2, 0 g (NH 4) 2 SO 4 und 0,5 mL FeSO 4-Lösung (vorbereitet in Schritt 1.1.1).
- Mit dem Präzisions-pH-Meter, den pH-Wert auf 7,0 durch Zugabe von 2 M KOH einstellen. Gießen Sie die Lösung in einen Glaszylinder und einem Gesamtvolumen von 200 mL fügen Sie DdH 2 O hinzu. Sterilisieren Sie die Lösung mit einem 250-mL-Filteranlage (PVDF, 0,22 µM).
- Herstellung der 20 % Glukoselösung
- 200 mL DdH 2 O in einem Messzylinder zu messen und um einen 500-mL-Becherglas gießen. Fügen Sie beim Mischen mit einem Magnetrührer, 50 g Glukose. Verdünnen Sie die Lösung in einem Messzylinder auf ein Gesamtvolumen von 250 mL. Die Lösung zu sterilisieren, wie unter Schritt 1.1.3.
- Herstellung der MgSO 4 Thiamin Lösung
- einen 500-mL-Becherglas 150 mL DdH 2 O hinzufügen. Fügen Sie beim Mischen mit einem Magnetrührer ein Gesamtvolumen von 200 mL 1,0 g Thiamin-HCl und 10 g MgSO 4-7 H 2 O. verdünnt die Lösung in einem Messzylinder hinzu. Die Lösung zu sterilisieren, wie unter Schritt 1.1.3.
- Vorbereitung minimale mittlere M63
- legen die fünf X-Lösung, die 20 % Glukoselösung MgSO 4 Thiamin Lösung, eine elektronische Pipette, einer P-200-Pipette und 200 µL Pipettenspitzen auf einer sauberen Werkbank.
- Maßnahme 155,8 mL DdH 2 O in einem Messzylinder und gießen Sie sie in einen 500-mL-Becherglas.
- Mit dem elektronischen Pipette und Einweg-serologische Pipette, Hinzufügen der gemessenen DdH 2 O. 40 mL 5 X und 4 mL 20 % Glucoselösung Fügen Sie dann 0,2 mL MgSO 4 Thiamin-Lösung mit der P-200. Die Lösung zu sterilisieren, wie unter Schritt 1.1.3.
2. Vorbereitung der Glycerin-Lager
- Zelle Kultur
Hinweis: die Bakterienstämme (z.B., W3110 und seine reduzierte Genome) gibt es von Belastung Bank Organisationen. Die Stämme werden in der Regel in Form von Kolonien auf Agarplatten erhalten.- Platz fünf sterilisierte Glasröhren mit Silikon Gummistopper, eine elektronische Pipette, P-1,000, 1.000 µL Pipettenspitzen, P-200, 200-µL-Pipettenspitzen und die Ziel-Stämme (Kolonien auf Tellern) auf einer sauberen Werkbank.
- Aussetzen die Mündung des Glasrohrs auf dem Bunsenbrenner vor dem Öffnen des Silikon-Gummistopfens. Setzen Sie den Silikon-Gummistopfen in die Flamme nach dem Öffnen der Tube und leicht setzen Sie die Kappe wieder auf die Glasröhre.
- Verwenden die elektronische Pipette und Einweg-serologische Pipette eines der Glasröhren und 4,5 mL M63, die anderen vier Röhrchen 5 mL M63 hinzu.
- P-200 Tipp verwenden, um eine Kolonie zu wählen und in das Glasrohr mit 5 mL M63 impfen.
- Vortex das Rohr zu einer Suspension. Verdünnen Sie die Lösung dann 10-fach durch Übertragung von 0,5 mL dieser Lösung auf eine der vier Röhren mit 4,5 mL M63.
- Wiederholen Sie den Vorgang im Schritt 2.1.5 für die übrigen Röhren beschrieben. Eine Verdünnungsreihe mit fünf verschiedenen Konzentrationen (Verdünnungen von 1, 10, 100, 1000 und 10.000) ist jetzt bereit.
- Sterilisieren die Mündern von Glasröhren und Silikon-Gummistopfen wie unter Punkt 2.1.2 beschrieben. Verschließen Sie die Rohre mit dem Stopfen. Kontamination vermeiden, indem Sie nicht knittern die Silikon-Gummistopper.
- Legen die fünf Röhren in einem vorgewärmten schütteln Inkubator bei 37 ° C und bei 200 u/min schütteln. Inkubation die Kultur über Nacht oder für 10 bis 30 h
- Auswahl der Kultur für das Glycerin Lager
- legen das vorgewärmte Raumtemperatur M63 Medium, P-1,000, 1.000 µL Pipettenspitzen und ein Einweg-Küvette auf einer sauberen Werkbank.
- Hinzufügen 1000 µL M63, eine Einweg-Küvette mit einem p-1, 000. Legen Sie die Einweg-Küvette in einem Spektrophotometer, starten Sie das Programm bei einer festen Wellenlänge von 600 nm und Maßnahme des Rohlings.
- Ans der sauberen Bank die fünf Glasröhren aus dem Schütteln Inkubator.
- Verwerfen M63 von Einweg-Küvette und die gleichen Einweg-Küvette mit einem p-1, 000 1.000 µL Kultur hinzufügen. Die optische Trübung der Zellkultur (OD 600) zu messen, wie in Schritt 2.2.2 beschrieben.
Hinweis: Um eine Kontamination zu vermeiden und eine präzise Messung zu erreichen, setzen Sie die Glasröhren und die Stopper der Flamme wie beschrieben und Wirbel der Kultur vor der Probenahme. Um zuverlässige Ergebnisse zu gewährleisten, sind wiederholte Messungen empfohlen, besonders wenn die Zelldichte niedrig ist. - Der fünf Zellkulturen, wählen Sie in der frühen Phase des exponentiellen Wachstums (OD 600 = 0,01 - 0,05) für den Glycerin bestand.
Hinweis: Wenn mehrere Kulturen dichten im optimalen Bereich, die am nähsten zu 0,05 ist allgemein ausgewählt.
- Machen die Glycerin-Bestände für wiederholte Prüfungen.
Hinweis: Dies ist für die Zubereitung von zehn Aktien beschrieben. Größere oder kleinere Mengen können je nach den experimentellen Anforderungen erfolgen.- Legen Sie die sterilisierten 60 % Glycerin Lösung, zehn sterilisiert 1,5 mL Mikroröhrchen, P-1,000 und P-200, 1.000 und 200 µL Pipettenspitzen und eine Reaktionscup auf einer sauberen Werkbank stehen.
- Fügen Sie 250 µL 60 % Glycerin Lösung und 750 µL der ausgewählten Zellkultur 1,5 mL Reaktionscup und Mischung von Pipettieren sterilisiert.
Hinweis: Das Lager Volumen ist variabel, aber behalten Sie immer eine 1:3-Verhältnis von 60 % Glycerin, Zellkultur; Dadurch wird eine Endkonzentration von 15 % Glycerin. - Stellen die restlichen neun Mikroröhrchen in die Reaktionscup Ständer und 100 µL der Mischung in 2.3.2 Schritt für jedes Rohr vorbereitet zu verzichten. Mittlerweile gibt es zehn identische Glycerin-Beständen für die zukünftige Verwendung.
- Speichern die Bestände in einer Tiefkühltruhe bei-80 ° c
3. Erwerb der Wachstumskurven
- der Mikrotestplatte Leser einrichten.
Hinweis: Die Begriffe in Anführungszeichen zeigen die spezifische Formulierung auf der Platte-Leser verwendet hier verwendet (siehe Tabelle Materialien).- Offene Software. Offene " Protokolle " in " Task-Manager " und wählen Sie " neu erstellen ". Wählen Sie " Standardprotokoll ".
- Open " Verfahren ", und passen Sie die Einstellungen. Offene " Set Temperatur ", und wählen Sie " Inkubator auf ". Set " Temperatur " auf 37 ° C und " Gradient " auf 0 ° C. Check " vorheizen " bevor Sie mit dem nächsten Schritt fortfahren. Offene " starten kinetische ", Set " Laufzeit " für 24:00:00 oder 48:00:00, und " Intervall " 00:30:00 oder 01:00.
Hinweis: Es dauert ca. 1 min zu lesen, eine gesamte 96 gut Platte. - Open " schütteln, " und " Shake Modus " als " Linear ". Überprüfen Sie " CoNstitution Shake " und " Frequenz " auf 567 cpm. Offene " lesen ", überprüfen " Absorption ", " Endpunkt/kinetische ", und " Monochromatoren. " gesetzt " Wellenlänge " bis 600.
- Klicken Sie " Validate " zu bestätigen, dass das Verfahren korrekt ist. Klicken Sie " speichern Sie " als ein neues Programm für die spätere Verwendung zu speichern.
- Echtzeit-Aufnahme des Wachstums
- zeichnen einen 96-Well-Platte-Piktogramm (8 × 12 Tabelle) um die Positionen der beimpften Kultur Proben auf der 96-Well-Platte anzugeben. Drucken Sie die Tabelle und verwenden Sie es als Referenz für das Experiment.
Hinweis: Der Brunnen befindet sich am Rande der Mikrotestplatte sollte nur leere Medium wegen Verdunstung enthalten. - Ort eine sterilisierte 96-Well-flache Unterseite Mikrotestplatte mit Deckel, P-1000, 1000 µL Pipettenspitzen, P-200, 200-µL-Pipettenspitzen, mehrere 1,5 mL Mikroröhrchen, ein 8-Mehrkanal-Pipette, sterilisierte Reagenz Reservoir, Raumtemperatur M63 und Glycerin Aktien (vorbereitet in Schritt 2.3) auf einer sauberen Werkbank.
- Ca. 25 mL M63 hinzufügen das Reagenz-Reservoir. Dieses Reservoir-Lager für die folgenden Schritte verwenden.
- Fügen Sie 900 µL M63, Mikroröhrchen in Vorbereitung für die Herstellung von Verdünnungsreihen.
- Lassen die Glycerin-Aktie bei Raumtemperatur auftauen. Fügen Sie 900 µL M63 aufgetauten Glycerin Lager und Wirbel. Dies führt zu einer 10-divisibel Verwässerung der ursprünglichen Glycerin Aktie.
- Transfer 100 µL der 10-divisibel Verdünnung zu einer anderen Reaktionscup mit 900 µL M63 und Wirbel. Dies führt zu einer 100-fold Verdünnung.
- Wiederholen Sie Schritt 3.2.6 bis die gewünschte Anzahl von Verdünnungen erreicht werden.
- Füllen die Brunnen am Rande der Mikrotestplatte mit 200 µL M63 mit einer 8-Kanal-Pipette (P-200).
Hinweis: Diese Brunnen können als Rohling verwendet werden. - Last 200 µL der einzelnen verdünnt Probe vorbereitet in Schritten 3.2.4 - 3.2.7 um die Mikrotestplatte Brunnen nach der Referenztabelle (Schritt 3.2.1). Wirbel der verdünnten Proben vor dem Laden und laden die gleiche Probe in mehreren Wells in vielfältig Standorte auf der Platte.
- 96-Well Mikrotestplatte auf der Platte-Leser legen.
- Open " Read Now " in " Task-Manager " und wählen Sie das Programm (Abschnitt 3.1). Klicken Sie auf " OK " Messung zu beginnen. Speichern Sie die Aufnahme als neue experimentelle Datei für die Datenanalyse (Abschnitt 4).
- zeichnen einen 96-Well-Platte-Piktogramm (8 × 12 Tabelle) um die Positionen der beimpften Kultur Proben auf der 96-Well-Platte anzugeben. Drucken Sie die Tabelle und verwenden Sie es als Referenz für das Experiment.
4. Analyse der Daten
- Export die Ergebnisse der Daten in Echtzeit Wachstumsrate (Abschnitt 3.2) auf einen USB-Speicher-stick als eine Text-Datei.
Hinweis: Eine 96-Well formatierte Ergebnisse (Kurven) werden auf der Platte Leser in Echtzeit angezeigt. Die Stundenaufzeichnungen (OD-Werte) können als Tabelle exportiert werden; die Zeilen und Spalten darstellen, die gut Zahlen (z.B. A1, B1) und die Messzeit (z.B. 00:00:59), beziehungsweise. - Öffnen Sie die Textdatei mit einem Tabellenkalkulations-Software.
- Kopieren Sie die stündlichen OD 600 lautet der 96-Well Mikrotestplatte in ein neues Arbeitsblatt zur weiteren Analyse.
- Subtrahieren Hintergrund liest vor Zeit Null von der stündlichen liest jeder Probe gut.
Hinweis: der Einfachheit halber der Mittelwert des Rohlings Brunnen enthaltenden M63 (Schritt 3.2.8) kann verwendet werden, als den Hintergrundwert. - Berechnen Sie den Mittelwert aus fünf aufeinander folgenden OD 600 liest die maximale Bevölkerungsdichte abzuschätzen.
Hinweis: Der größte Mittelwert der fünf aufeinander folgenden OD 600 lautet ist definiert als die maximale OD 600 entsprechende Wachstumskurve. - Berechnung die Wachstumsrate µ (h -1), durch die Anwendung der folgenden Gleichung für alle Paare aufeinander folgender Werte OD 600:
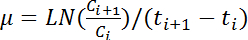
Hinweis: In dieser Gleichung C ich und C i + 1 repräsentieren die OD 600 Werte zwei aufeinander folgenden Zeitpunkten (t ich und t i + 1, beziehungsweise). LN gibt natürlichen Logarithmus. - Berechnen die Veränderungen bei der Zunahme im Laufe der Zeit auf Stundenbasis OD 600 basierend liest laut Schritt 4.6. Berechnen Sie den Mittelwert und die Standardabweichung der fünf aufeinander folgenden Wachstumsraten, die maximale Wachstumsrate zu schätzen.
Hinweis: Der größte Mittelwert mit der kleinsten Standardabweichung ist definiert als die maximale Wachstumsrate der entsprechenden Wachstumskurve.
5. Bestätigt die globale Vorspannung des 96-Well lautet (Optional)
Hinweis: sowohl die Platte Leser und die Verbrauchsmaterialien 96-Well-Platte können dazu führen, dass voreingenommene Messungen. Um sehr präzise und reproduzierbar quantitative Ergebnisse zu erzielen, bestätigt die globale Vorspannung der 96-Well-Platte ist sehr zu empfehlen.
- 96-Well-Platte und der Platte-Leser auf den Bias Test vorbereiten, wie in Abschnitt 3.2.2 beschrieben.
- Fügen Sie 20 mL M63, eine sterilisierte 50 mL Zentrifugenröhrchen. Die Brühe Glycerin auf die gleichen Zentrifugenröhrchen und Wirbel. Übertragen die abgehängte Lösung zu einem sterilisierten Reagenz Reservoir.
- Verwenden Sie einen 8-Kanal-Pipette, um 200 µL der suspendierten Lösung auf die 96-Well-Platte zu übertragen.
- 96-Well-Platte auf den Teller-Leser und starten Sie die Messung.
- Datensatz die stündliche OD 600 liest und analysiert, wie in Abschnitt 4 beschrieben. Vergleichen Sie die berechnete maximale Wachstum Rate und Bevölkerung Dichte von jedem gut zu bestimmen, standortbezogene Voreingenommenheit des 96-Well lautet.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Das beschriebene Verfahren bietet die Möglichkeit, dynamische Wachstum von Bakterien in einer kontinuierlichen, Hochdurchsatz-Weise durch den Einsatz eines 96-Well-Format-Reader, der mehrere optische Dichtemessungen in verschiedenen Zeitabständen (von Minuten bis Stunden bis Tage) nimmt zu erfassen. Die Wachstumskurven aus einer Zusammenstellung von E. Coli -Stämme, die mit dem Ausdruck verschiedener Genome können genau in einem einzigen Experiment (Abbil...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Wichtige Schritte im Protokoll sind die Vorbereitung einer Stammaktie der exponentiell wachsenden Zellen und die Replikation der gleichen Proben in mehreren Brunnen an verschiedenen Positionen auf die Mikrotestplatte. Zuvor, Mikrobiologen die Kultur angefangen eine Übernachtung Vorkultur. Während diese Methode die Verzögerungszeit des Bakterienwachstums verringern kann, ist es schwierig, reproduzierbare Wachstumskurven zu erreichen. Wie in Abbildung 2dargestellt, führte die unabhängigen...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Wir danken für die Bereitstellung der KBE-Assay-Beispiel Kohei Tsuchiya. Diese Arbeit wurde teilweise finanziell durch eine Beihilfe für wissenschaftliche Forschung (C) Nr. 26506003 (zu BWY) vom Ministerium für Bildung, Kultur, Sport, Wissenschaft und Technologie, Japan unterstützt.
Danksagungen
Die Autoren haben nichts preisgeben.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| K2HPO4 | Wako | 164-04295 | |
| KH2PO4 | Wako | 166-04255 | |
| (NH4)2SO4 | Wako | 019-03435 | |
| MgSO4-7H2O | Wako | 138-00415 | |
| Thiamine-HCl | Wako | 201-00852 | |
| glucose | Wako | 049-31165 | |
| HCl | Wako | 080-01066 | |
| Iron (II) sulfate heptahydrate (FeSO4-7H2O) | Wako | 094-01082 | |
| KOH | Wako | 168-21815 | |
| Glycerol | Wako | 075-00611 | |
| Centrifuge tube (50 mL, sterilized) | WATSON | 1342-050S | |
| Pipette Tips, 200 µL | WATSON | 110-705Y | |
| Pipette Tips, 1,000 µL | WATSON | 110-8040 | |
| Microtube (1.5 mL) | WATSON | 131-715C | |
| 8 multichannel-pipette | WATSON | NT-8200 | |
| PASORINA STIRRER | AS ONE | 2-4990-02 | |
| Glass cylinder (200 mL) | AS ONE | 1-8562-07 | |
| Precision pH mater | AS ONE | AS800 / 1-054-01 | |
| Pipetman P-200 | GILSON | 1-6855-05 | |
| Pipetman P-1000 | GILSON | 1-6855-06 | |
| Disposable Serolocical Pipettes (10 mL) | SANPLATEC | SAN27014 | |
| Disposable Serolocical Pipettes (25 mL) | SANPLATEC | SAN27015 | |
| Microtube stand | BM Bio | 801-02Y | |
| Vortex | BM Bio | BM-V1 | |
| Corning Costar 96-well microplate with lid (Flat bottom, Clear) | Sigma-Aldrich | Corning, 3370 | |
| Corning Costar reagent reservoir (50 mL) | Sigma-Aldrich | Corning, 4870 | |
| Stericup GV PVDF (250 mL, 0.22 µM) | Merck Millipore | SCGVU02RE | |
| Pipet-Aid XP | DRUMMOND | 4-000-101 | |
| Bioshaker (BR-23UM MR) | TAITEC | 0053778-000 | |
| Disposal cell (1.5 mL) | Kartell | 1938 / 2-478-02 | |
| DU 730 Life Science UV/Vis Spectrophotometer | Beckman Coulter | A23616 | |
| EPOCH2 | BioTek | 2014-EP2-002 / EPOCH2T | |
| Beaker (500 mL) | IWAKI | 82-0008 | |
| BIO clean bench | Panasonic | MCV-B131F | |
| Glass tubes | NICHIDEN RIKA GLASS | P-10M~P-30 /101019 | |
| Silicone rubber stoppers | ShinEtsu Polymer | T-19 | |
| Bacterial strains | Strain bank organization; National Bio Resource Project (NBRP) in Japan |
Referenzen
- Kovarova-Kovar, K., Egli, T. Growth kinetics of suspended microbial cells: from single-substrate-controlled growth to mixed-substrate kinetics. Microbiol Mol Biol Rev. 62 (3), 646-666 (1998).
- Soupene, E., et al. Physiological studies of Escherichia coli strain MG1655: growth defects and apparent cross-regulation of gene expression. J Bacteriol. 185 (18), 5611-5626 (2003).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Egli, T. Microbial growth and physiology: a call for better craftsmanship. Front Microbiol. 6, 287(2015).
- Kurokawa, M., Seno, S., Matsuda, H., Ying, B. W. Correlation between genome reduction and bacterial growth. DNA Res. 23 (6), 517-525 (2016).
- Matsumoto, Y., Murakami, Y., Tsuru, S., Ying, B. W., Yomo, T. Growth rate-coordinated transcriptome reorganization in bacteria. BMC Genomics. 14, 808(2013).
- Nahku, R., et al. Specific growth rate dependent transcriptome profiling of Escherichia coli K12 MG1655 in accelerostat cultures. J Biotechnol. 145 (1), 60-65 (2010).
- Dai, X., et al. Reduction of translating ribosomes enables Escherichia coli to maintain elongation rates during slow growth. Nat Microbiol. 2, 16231(2016).
- Madrid, R. E., Felice, C. J. Microbial biomass estimation. Crit Rev Biotechnol. 25 (3), 97-112 (2005).
- Harris, C. M., Kell, D. B. The estimation of microbial biomass. Biosensors. 1 (1), 17-84 (1985).
- Yates, G. T., Smotzer, T. On the lag phase and initial decline of microbial growth curves. J Theor Biol. 244 (3), 511-517 (2007).
- Kargi, F. Re-interpretation of the logistic equation for batch microbial growth in relation to Monod kinetics. Lett Appl Microbiol. 48 (4), 398-401 (2009).
- Peleg, M., Corradini, M. G. Microbial growth curves: what the models tell us and what they cannot. Crit Rev Food Sci Nutr. 51 (10), 917-945 (2011).
- Sprouffske, K., Wagner, A. Growthcurver: an R package for obtaining interpretable metrics from microbial growth curves. BMC Bioinformatics. 17, 172(2016).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Mol Biol Evol. 31 (1), 232-238 (2014).
- Hermsen, R., Okano, H., You, C., Werner, N., Hwa, T. A growth-rate composition formula for the growth of E.coli on co-utilized carbon substrates. Mol Syst Biol. 11 (4), 801(2015).
- Engen, S., Saether, B. E. r- and K-selection in fluctuating populations is determined by the evolutionary trade-off between two fitness measures: Growth rate and lifetime reproductive success. Evolution. 71 (1), 167-173 (2017).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten