Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Análisis precisos, alto rendimiento de crecimiento bacteriano
En este artículo
Resumen
Evaluación cuantitativa del crecimiento bacteriano es fundamental para entender la fisiología microbiana como un fenómeno de nivel de sistemas. Se introduce un protocolo de manipulación experimental y un enfoque analítico, permitiendo análisis precisos, alto rendimiento de crecimiento bacteriano, que es un tema clave de interés en biología de sistemas.
Resumen
Crecimiento bacteriano es un concepto central en el desarrollo de la moderna fisiología microbiana, así como en la investigación de la dinámica celular a nivel de sistemas. Estudios recientes han reportado correlaciones entre el crecimiento bacteriano y eventos de todo el genoma, como reorganización de reducción y transcriptoma de genoma. Analizar correctamente el crecimiento bacteriano es fundamental para entender la coordinación dependiente del crecimiento de las funciones de genes y componentes celulares. Por consiguiente, se requiere la evaluación cuantitativa precisa del crecimiento bacteriano en forma de alto rendimiento. Desarrollos tecnológicos emergentes ofrecen nuevas herramientas experimentales que permiten la actualización de los métodos utilizados para el estudio de crecimiento bacteriano. El protocolo introducido el emplea a un lector de microplacas con un procedimiento experimental altamente optimizado para la evaluación precisa y reproducible del crecimiento bacteriano. Este protocolo fue utilizado para evaluar el crecimiento de varias cepas de Escherichia coli ha descrito anteriormente. Los pasos principales del protocolo son las siguientes: la elaboración de un gran número de poblaciones de células en pequeños viales de repetidas pruebas con la reproducibilidad, la utilización de placas de 96 pocillos para la evaluación del crecimiento de alto rendimiento y el cálculo manual de dos grandes parámetros (es decir, densidad de población y tasa de crecimiento máxima) que representa la dinámica de crecimiento. En comparación con el tradicional análisis de unidad (UFC) formadoras de colonias, que cuenta con las células que se cultivan en tubos de vidrio con el tiempo en placas de agar, el presente método es más eficiente y proporciona más registros temporales de los cambios de crecimiento, pero tiene una más estricta límite de detección a densidades de población bajas. En Resumen, el método descrito es ventajoso para el análisis de alto rendimiento precisa y reproducible del crecimiento bacteriano, que puede utilizarse para sacar conclusiones conceptuales o hacer observaciones teóricas.
Introducción
Estudios microbiológicos a menudo comienzan con el cultivo de las células bacterianas y la evaluación de las curvas de crecimiento bacteriano, que representan un fenómeno fundamental de la fisiología bacteriana1,2,3. Principios básicos de la cultura están ampliamente disponibles en la literatura de los estudios publicados y los libros de texto porque la cultura bacteriana es una metodología fundamental. A nivel Banco, considerable atención se ha centrado tradicionalmente en optimización de medios de cultivo y condiciones de cultivo, pero controla la tasa de crecimiento, que probablemente daría aún mayor comprensión de la fisiología microbiana, no ha sido ampliamente estudiados4. Para el crecimiento exponencial de bacterias, un parámetro clave del estado celular es la tasa de crecimiento, que se ha divulgado para ser coordinado con el genoma, transcriptoma y proteoma5,6,7,8 . Así, la evaluación cuantitativa del crecimiento bacteriano es crucial para la comprensión de fisiología microbiana.
Para evaluar el crecimiento bacteriano, los métodos experimentales utilizados para estimar la biomasa están bien establecidos9,10 y se basan en la detección de parámetros bioquímicos, físicos o biológicos, como la turbidez óptica. Además, los métodos analíticos utilizados para capturar las propiedades dinámicas de los cambios de crecimiento se basan comúnmente en modelos no lineales establecidos11,12,13, por ejemplo, las ecuaciones logísticas. Dinámica de crecimiento se adquiere generalmente por tiempo muestreo del crecimiento celular en cultivo por medición de turbidez óptica o realizar pruebas de unidad (UFC) formadoras de colonias. La limitación de estos métodos de cultivo y detección es que los puntos no son un fiel reflejo de la dinámica de la población porque los intervalos de medición son a menudo en horas y la condición de la cultura (p. ej., cambios de temperatura y aireación) se altera en el momento de muestreo. Técnicas de cultivo y análisis deben ser actualizadas con los acontecimientos recientes en la tecnología y la comprensión. Los avances recientes en los lectores de microplacas permiten la observación en tiempo real del crecimiento bacteriano y disminuyen significativamente los costos laborales. Usando estos dispositivos avanzados, los estudios más recientes sobre crecimiento bacteriano han publicado métodos analíticos para mediciones de alto rendimiento14,15.
El propósito de este protocolo es evaluar la dinámica de crecimiento precisa de una manera de alto rendimiento, que será de gran valor para estudios cuantitativos que en última instancia, responder las preguntas de cómo se determina la tasa de crecimiento y los factores que afectan la tasa de crecimiento. El protocolo aborda todos los factores que deben tenerse en cuenta para la cuantificación precisa y repetible de crecimiento bacteriano. El método experimental y el análisis se describen en detalle en el texto principal. Este método permite el análisis preciso y reproducible de crecimiento bacteriano en forma de alto rendimiento. Microbiólogos pueden utilizar este protocolo para obtener resultados cuantitativos adicionales de su evidencia experimental. Este protocolo también puede utilizarse para estudios en biología de sistemas que tratan de sacar conclusiones conceptuales o para lograr un resumen teórico de crecimiento.
Protocolo
1. preparar el medio de crecimiento
Nota: la composición química de M63 medio mínimo es el siguiente: 62 m m K 2 HPO 4, 39 m m KH 2 PO 4, 15 mM (NH 4) 2 SO 4 1,8 μm FeSO 4, 15 μm tiamina-HCl, 0,2 mM MgSO 4 y 22 mM de glucosa. M63 se hace mezclando tres soluciones: solución X 5, 20% de glucosa y solución de MgSO 4 tiamina. Almacene todas las soluciones a 4 ° C.
- Preparación de la solución de X 5
- para preparar solución de FeSO 4, utilice una pipeta eléctrica y una pipeta serológica desechable para agregar ddH 2 O a un tubo de centrífuga de 50 mL. Utilizar un P-200 Añadir 0,06 mL de HCl para obtener 36 mg FeSO 4 0.01 M HCl. Añadir-7 H 2 O y mezcle bien.
- Medir 160 mL ddH 2 O en una probeta y añadir un vaso de precipitados de 500 mL. Agregue lo siguiente en este orden: 10,72 g K 2 HPO 4, 5,24 g KH 2 PO 4, 2.0 g de (NH 4) 2 SO 4 y 0,5 mL de solución de FeSO 4 (preparado en el paso 1.1.1).
- Usando un medidor de pH de precisión, ajustar el pH a 7.0 agregando 2 M KOH. Vierta la solución en una probeta y añadir ddH 2 O a un volumen total de 200 mL. Esterilizar la solución utilizando una unidad de filtración de 250 mL (PVDF, 0,22 μm).
- Preparación de la solución de glucosa 20%
- medir 200 mL ddH 2 O en una probeta y verter un vaso de precipitados de 500 mL. Mientras se mezcla con un agitador magnético, agregar 50 g de glucosa. Diluir la solución en una probeta hasta un volumen total de 250 mL. Esterilizar la solución como se describe en el paso 1.1.3.
- Preparación de la solución de MgSO 4 tiamina
- añadir 150 mL ddH 2 O un vaso de precipitados de 500 mL. Mientras se mezcla con un agitador magnético, agregar 1,0 g de tiamina-HCl y 10 g de MgSO 4-7 H 2 O. diluir la solución en una probeta hasta un volumen total de 200 mL. Esterilizar la solución como se describe en el paso 1.1.3.
- Preparación mínima M63 medio
- Coloque la solución de X 5, la solución de glucosa 20%, la solución de MgSO 4 tiamina, una pipeta electrónica, una pipeta P-200 y puntas de pipeta de 200 μL en un banco limpio.
- ML 155,8 ddH 2 O en una probeta y verter en un vaso de precipitados de 500 mL.
- Usando la pipeta electrónica y pipeta serológica desechable, agregar 40 mL de solución de 5 X y solución glucosa al de 20% 4 mL con el ddH medido 2 O. Luego, agregar 0,2 mL de solución de MgSO 4 tiamina con la P-200. Esterilizar la solución como se describe en el paso 1.1.3.
2. Preparar el caldo glicerol
cultura- celular
Nota: las cepas bacterianas (p. ej., W3110 y sus genomas reducidos) están disponibles de organizaciones Banco de tensión. Las cepas se obtienen generalmente en forma de colonias en placas de agar.- Colocar cinco tubos de cristal esterilizado con tapones de goma de silicona, una pipeta electrónica, P-1,000, puntas de pipeta de 1.000 μl, P-200, puntas de pipeta de 200 μl y las cepas target (colonias en las placas) en un banco limpio.
- Exponga la boca del tubo de vidrio con un mechero de Bunsen antes de abrir el tapón de goma de silicona. Exponer el tapón de goma de silicona para la llama después de que se abre el tubo y luego ligeramente Coloque la tapa en el tubo de cristal.
- Utilice la pipeta electrónica y pipeta serológica desechable para añadir a uno de los tubos de vidrio y 4,5 mL M63 a los otros cuatro tubos 5 mL M63.
- Utilice la punta del P-200 una colonia y inocular en el tubo de vidrio que contiene 5 mL M63.
- El tubo de vórtice para hacer una suspensión. Entonces, la solución diluida 10 veces al transferir 0,5 mL de esta solución a uno de los cuatro tubos conteniendo 4,5 mL M63.
- Repetir el proceso descrito en el paso 2.1.5 para los tubos restantes. Una serie de diluciones con cinco concentraciones diferentes (diluciones de 1, 10, 100, 1000 y 10.000) ya está listo.
- Esterilizar las bocas de los tubos de vidrio y los tapones de goma de silicona como se describe en el paso 2.1.2. Los tubos con los tapones de la tapa. Evitar la contaminación por no arrugar los tapones de goma de silicona.
- Colocar cinco tubos en una incubadora con agitación a 37 ° C y se agita a 200 rpm. Incubar el cultivo durante la noche o para h. de 10 a 30
- Selección de la cultura para el stock de glicerol
- Coloque el medio precalentado temperatura M63, P-1,000, puntas de pipeta de 1.000 μl y una cubeta desechable en un banco limpio.
- Añadir 1000 μl M63 a una cubeta desechable con un P-1, 000. Coloque la cubeta desechable en un espectrofotómetro, iniciar el programa en una longitud de onda fija de 600 nm y medir el espacio en blanco.
- Hacia los tubos de vidrio de cinco de la incubadora sacude el Banco limpio.
- M63 descartar de la cubeta desechable y añadir 1.000 cultura μl a la misma cubeta desechable con un P-1, 000. Medir la turbidez óptica de la cultura de célula (OD 600) como se describe en el paso 2.2.2.
Nota: Para evitar cualquier contaminación y lograr una medición precisa, exponga los tubos de vidrio y los tapones de la llama como se describe y el vórtice de la cultura antes de muestreo. Para asegurar resultados confiables, se recomiendan mediciones repetidas, sobre todo cuando la densidad celular es baja. - De los cinco cultivos celulares, elegir una que se encuentra en la fase temprana de crecimiento exponencial (OD 600 = 0.01 - 0.05) para el caldo glicerol.
Nota: Si varias culturas tienen densidades dentro de la gama óptima, el uno más cercano a 0,05 es comúnmente seleccionado.
- Hacer las poblaciones de glicerol por repetidas pruebas.
Nota: Esto se describe para la preparación de las diez poblaciones. Cantidades más grandes o más pequeños se pueden hacer según los requisitos experimentales.- Coloque la solución de glicerina 60% esterilizada, diez esterilizar microtubos de 1,5 mL, P-1,000 y P-200, puntas de pipeta de 1.000 y 200 μl y un microtubo en un banco limpio.
- Añadir 250 μl esterilizada 60% solución de glicerol y 750 μl de la cultura de la celda seleccionada a los microtubos de 1,5 mL y mezclar mediante pipeteo.
Nota: El volumen de acciones es variable, pero siempre mantener una relación de 1:3 de 60% de glicerol para cultivo celular; Esto resulta en una concentración final de glicerol al 15%. - Colocar los nueve microtubos restantes en el stand de microtubos y dispensar 100 μl de la mezcla preparada en el paso 2.3.2 a cada tubo. Ahora hay diez poblaciones de glicerol idénticos para su uso futuro.
- Almacenar las existencias en un congelador a -80 ° C.
3. Adquisición de las curvas de crecimiento
- configurar el lector de microplacas.
Nota: Los términos que se muestra en las comillas muestran el fraseo específico utilizado en el lector de placa utilizado aquí (véase la tabla de materiales).- Abrir el software. Abierto " protocolos de " en " el administrador de tareas " y elija " crear nuevo ". Elegir " protocolo estándar ".
- Abierta " procedimiento " y ajustar la configuración. Abierto " temperatura " y seleccione " en la incubadora ". Set " temperatura " a 37 ° C y " gradiente " a 0 ° C. Revise " precalentamiento " antes de continuar con el siguiente paso. Abierto " empezar cinética ", establecer " tiempo de ejecución " para 24:00:00 o 48:00:00, y " intervalo de " de 00:30:00 o 1:00.
Nota: Tarda aproximadamente 1 minuto para leer un entero 96 placa bien. - Abierta " Shake " y " modo de agitar " como " lineal ". Compruebe " Constitution Shake " y " frecuencia " en 567 cpm. Abierto " leer ", compruebe " absorbancia ", " punto final/cinética ", y " monocromadores. " Set " longitud de onda " 600.
- Clic " validar " para confirmar que el procedimiento es correcto. Haga clic " guardar " para guardar como un nuevo programa para el uso futuro.
- En tiempo real de grabación de crecimiento
- dibujar un pictograma de la placa de 96 pozos (8 × 12 tabla) para indicar las posiciones de las muestras de cultivo inoculados en la placa de 96 pocillos. Imprimir la tabla y utilizarla como referencia para el experimento de.
Nota: Los pozos situados en los bordes de la microplaca sólo deben contener medio en blanco debido a la evaporación.
Microplacas de fondo - lugar un piso de 96 pocillos estéril con tapa P-1000, puntas de pipeta de 1000 μl, P-200, puntas de pipeta de 200 μL, varios microtubos de 1,5 mL, una pipeta multicanal de 8, un reservorio de reactivo esterilizada, temperatura M63 y el glicerol acciones (preparadas en el paso 2.3) en un banco limpio.
- Añadir aproximadamente 25 mL M63 para el depósito de reactivo. Uso de este stock de reserva de todos los pasos siguientes.
- Agregar 900 μl M63 en los microtubos en preparación para hacer diluciones seriadas.
- Descongelar el stock de glicerol a temperatura ambiente. Agregar 900 μl M63 stock de glicerol descongeladas y vortex. Esto resulta en una dilución de 10 veces del original stock de glicerol de.
- Transferir 100 μl de la dilución de 10 veces a otro microtubo que contiene 900 μl de M63 y vortex. Esto resulta en una 100-fold dilución.
- Repetir paso 3.2.6 hasta conseguir el número de diluciones deseado es.
- Llenar los pozos en el borde de la microplaca con 200 μL M63 utilizando una pipeta de 8 canales (P-200).
Nota: Estos pozos se pueden utilizar como el espacio en blanco. - Carga 200 μL de cada muestra en pasos 3.2.4 - 3.2.7 en los pocillos de la microplaca según la tabla de referencia (paso 3.2.1) diluida. Vórtice el antes las muestras diluidas de carga y carga la misma muestra en pozos múltiples en variados lugares en la placa de.
- Coloque la microplaca de 96 pocillos en el lector de placas de.
- Abierta " leer ahora " en " el administrador de tareas " y elegir el programa (sección 3.1). Haga clic en " OK " para comenzar a medir. Guardar la grabación como un nuevo archivo experimental para análisis de datos (sección 4).
- dibujar un pictograma de la placa de 96 pozos (8 × 12 tabla) para indicar las posiciones de las muestras de cultivo inoculados en la placa de 96 pocillos. Imprimir la tabla y utilizarla como referencia para el experimento de.
4. Análisis de datos
- exportar los resultados de los datos en tiempo real tasa de crecimiento (sección 3.2) a una memoria USB stick como un archivo de texto.
Nota: Un resultados con formato de 96 pocillos (curvas) se mostrarán en el lector de la placa en tiempo real. Los registros por hora (valores de OD) pueden ser exportados como una tabla; las filas y las columnas representan los números bien (por ejemplo, A1, B1) y el tiempo de medición (por ejemplo, 00:00:59), respectivamente. - Abrir el archivo de texto con un software de hoja de cálculo.
- Copiar las lecturas cada hora de 600 OD de la microplaca de 96 pozos en una nueva hoja de cálculo para su posterior análisis.
- Restar el fondo lee en el tiempo cero de las lecturas cada hora de cada muestra bien.
Nota: Para mayor comodidad, el valor medio del espacio en blanco pozos que contienen M63 (paso 3.2.8) puede ser utilizado como el valor de fondo. - Calcular la media de cinco consecutivos OD 600 lecturas para estimar la densidad de población máxima.
Nota: El mayor valor promedio de OD 600 cinco Lee consecutivo se define como el máximo de OD 600 de la curva de crecimiento correspondiente. - Calcular la tasa de crecimiento, μ (h -1), aplicando la siguiente ecuación para todos los pares de valores consecutivos de OD 600:
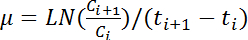
Nota: en esta ecuación, C y C i + 1 representan los valores de OD 600 de cualquier dos puntos de tiempo consecutivos (t y t i + 1, ««««respectivamente). LN indica logaritmo natural. - Calcular los cambios en la tasa de crecimiento en el tiempo basados en la hora OD 600 Lee según el paso 4.6. Calcular la media y la desviación estándar de cinco crecimientos consecutivos para estimar la tasa de crecimiento máxima.
Nota: El promedio mayor con la menor desviación estándar se define como la tasa de crecimiento máxima de la curva de crecimiento correspondiente.
5. Confirmando la tendencia Global de las lecturas de 96 pocillos (opcional)
Nota: tanto la placa del lector y la placa de 96 pocillos consumible pueden causar mediciones sesgadas. Para lograr resultados cuantitativos muy precisos y reproducibles, confirmando la tendencia global de la placa de 96 pocillos está altamente-recomendado.
- Preparar la placa de 96 pocillos y el lector de la placa para la prueba de sesgo, como se describe en la sección 3.2.2.
- Añadir 20 mL M63 a un tubo de centrífuga de 50 mL esterilizado. Añadir el caldo glicerol al mismo tubo de centrífuga y vortex. Transferir la solución suspendida a un reservorio de reactivo esterilizada.
- Use una pipeta de 8 canales para transferir 200 μL de la solución de suspensión a la placa de 96 pozos.
- Coloque la placa de 96 pocillos en el lector de placas y comenzar la medición.
- Registro hora OD 600 Lee y analiza como se describe en la sección 4. Comparar la densidad de población y tasa de crecimiento máxima calculada de cada pozo para determinar sesgo localización de 96 pocillos Lee.
Resultados
El método descrito proporciona un medio para capturar la dinámico crecimiento bacteriano de manera continua, de alto rendimiento utilizando un lector de formato de 96 pozos que tiene varias mediciones de densidad óptica a diferentes intervalos de tiempo (desde minutos a horas o días). Las curvas de crecimiento de una variedad de cepas de e. coli expresan diversos genomas pueden adquirirse precisamente en un solo experimento (figura 1A). En compar...
Discusión
Pasos críticos en el protocolo incluyen la preparación de una acción común de crecimiento exponencial las células y la replicación de las mismas muestras en pozos múltiples en diversas posiciones en la microplaca. Previamente, microbiólogos comenzaron la cultura de una cultura de la noche. Mientras que este método puede reducir el tiempo de retraso del crecimiento bacteriano, es difícil de conseguir las curvas de crecimiento reproductivo. Como se muestra en la figura 2, las medicio...
Divulgaciones
Agradecemos a Kohei Tsuchiya para dar el ejemplo de ensayo de UFC. Este trabajo fue parcialmente apoyado por una subvenciones para la investigación científica (C) no. 26506003 (a BWY) desde el Ministerio de educación, cultura, deportes, ciencia y tecnología, Japón.
Agradecimientos
Los autores no tienen nada que revelar.
Materiales
| Name | Company | Catalog Number | Comments |
| K2HPO4 | Wako | 164-04295 | |
| KH2PO4 | Wako | 166-04255 | |
| (NH4)2SO4 | Wako | 019-03435 | |
| MgSO4-7H2O | Wako | 138-00415 | |
| Thiamine-HCl | Wako | 201-00852 | |
| glucose | Wako | 049-31165 | |
| HCl | Wako | 080-01066 | |
| Iron (II) sulfate heptahydrate (FeSO4-7H2O) | Wako | 094-01082 | |
| KOH | Wako | 168-21815 | |
| Glycerol | Wako | 075-00611 | |
| Centrifuge tube (50 mL, sterilized) | WATSON | 1342-050S | |
| Pipette Tips, 200 µL | WATSON | 110-705Y | |
| Pipette Tips, 1,000 µL | WATSON | 110-8040 | |
| Microtube (1.5 mL) | WATSON | 131-715C | |
| 8 multichannel-pipette | WATSON | NT-8200 | |
| PASORINA STIRRER | AS ONE | 2-4990-02 | |
| Glass cylinder (200 mL) | AS ONE | 1-8562-07 | |
| Precision pH mater | AS ONE | AS800 / 1-054-01 | |
| Pipetman P-200 | GILSON | 1-6855-05 | |
| Pipetman P-1000 | GILSON | 1-6855-06 | |
| Disposable Serolocical Pipettes (10 mL) | SANPLATEC | SAN27014 | |
| Disposable Serolocical Pipettes (25 mL) | SANPLATEC | SAN27015 | |
| Microtube stand | BM Bio | 801-02Y | |
| Vortex | BM Bio | BM-V1 | |
| Corning Costar 96-well microplate with lid (Flat bottom, Clear) | Sigma-Aldrich | Corning, 3370 | |
| Corning Costar reagent reservoir (50 mL) | Sigma-Aldrich | Corning, 4870 | |
| Stericup GV PVDF (250 mL, 0.22 µM) | Merck Millipore | SCGVU02RE | |
| Pipet-Aid XP | DRUMMOND | 4-000-101 | |
| Bioshaker (BR-23UM MR) | TAITEC | 0053778-000 | |
| Disposal cell (1.5 mL) | Kartell | 1938 / 2-478-02 | |
| DU 730 Life Science UV/Vis Spectrophotometer | Beckman Coulter | A23616 | |
| EPOCH2 | BioTek | 2014-EP2-002 / EPOCH2T | |
| Beaker (500 mL) | IWAKI | 82-0008 | |
| BIO clean bench | Panasonic | MCV-B131F | |
| Glass tubes | NICHIDEN RIKA GLASS | P-10M~P-30 /101019 | |
| Silicone rubber stoppers | ShinEtsu Polymer | T-19 | |
| Bacterial strains | Strain bank organization; National Bio Resource Project (NBRP) in Japan |
Referencias
- Kovarova-Kovar, K., Egli, T. Growth kinetics of suspended microbial cells: from single-substrate-controlled growth to mixed-substrate kinetics. Microbiol Mol Biol Rev. 62 (3), 646-666 (1998).
- Soupene, E., et al. Physiological studies of Escherichia coli strain MG1655: growth defects and apparent cross-regulation of gene expression. J Bacteriol. 185 (18), 5611-5626 (2003).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Egli, T. Microbial growth and physiology: a call for better craftsmanship. Front Microbiol. 6, 287 (2015).
- Kurokawa, M., Seno, S., Matsuda, H., Ying, B. W. Correlation between genome reduction and bacterial growth. DNA Res. 23 (6), 517-525 (2016).
- Matsumoto, Y., Murakami, Y., Tsuru, S., Ying, B. W., Yomo, T. Growth rate-coordinated transcriptome reorganization in bacteria. BMC Genomics. 14, 808 (2013).
- Nahku, R., et al. Specific growth rate dependent transcriptome profiling of Escherichia coli K12 MG1655 in accelerostat cultures. J Biotechnol. 145 (1), 60-65 (2010).
- Dai, X., et al. Reduction of translating ribosomes enables Escherichia coli to maintain elongation rates during slow growth. Nat Microbiol. 2, 16231 (2016).
- Madrid, R. E., Felice, C. J. Microbial biomass estimation. Crit Rev Biotechnol. 25 (3), 97-112 (2005).
- Harris, C. M., Kell, D. B. The estimation of microbial biomass. Biosensors. 1 (1), 17-84 (1985).
- Yates, G. T., Smotzer, T. On the lag phase and initial decline of microbial growth curves. J Theor Biol. 244 (3), 511-517 (2007).
- Kargi, F. Re-interpretation of the logistic equation for batch microbial growth in relation to Monod kinetics. Lett Appl Microbiol. 48 (4), 398-401 (2009).
- Peleg, M., Corradini, M. G. Microbial growth curves: what the models tell us and what they cannot. Crit Rev Food Sci Nutr. 51 (10), 917-945 (2011).
- Sprouffske, K., Wagner, A. Growthcurver: an R package for obtaining interpretable metrics from microbial growth curves. BMC Bioinformatics. 17, 172 (2016).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Mol Biol Evol. 31 (1), 232-238 (2014).
- Hermsen, R., Okano, H., You, C., Werner, N., Hwa, T. A growth-rate composition formula for the growth of E.coli on co-utilized carbon substrates. Mol Syst Biol. 11 (4), 801 (2015).
- Engen, S., Saether, B. E. r- and K-selection in fluctuating populations is determined by the evolutionary trade-off between two fitness measures: Growth rate and lifetime reproductive success. Evolution. 71 (1), 167-173 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados