需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
脂肪酸13C Isotopologue 分析提供了对无脊椎动物消费者营养碳转移和脂质代谢的洞察力
摘要
脂肪酸营养标记方法,即,将脂肪酸同化为整个分子, 并转移到没有或轻微修饰的消费者组织中, 这是由于小土壤无脊椎动物脂肪酸代谢的知识缺口所阻碍的。Isotopologue 分析作为一种宝贵的工具来解开营养的相互作用。
摘要
脂肪酸 (FAs) 是食品网络生态学中有用的生物标志物, 因为它们通常被同化为一个完整的分子, 并转移到使用小的或没有修改的消费者组织中, 从而允许不同营养水平之间的膳食路由。然而, 由于对土壤动物脂质代谢的知识有限, 足总营养标记方法仍受到阻碍。本研究使用全标签棕榈酸 (13C16:0, 99 atom%) 作为示踪剂在脂肪酸代谢通路的两个广泛的土壤跳虫, Protaphorura fimata和Heteromurus 大足。为了研究这种前驱体的命运和代谢修饰, 提出了一种利用单离子监测的质谱法进行 isotopologue 分析的方法。此外, 还介绍了上游实验室饲养实验, 以及显性脂质组分 (中性脂质、磷脂) 的提取和甲基化, 以及相关的配方和计算。Isotopologue 分析不仅会产生从13c 标记为前体的脂肪酸中的总的13c 富集, 而且还产生超过母体离子质量的 isotopologues 的模式 (即, FA 分子离子m+) 每个标记为 FA 的一个或多个质量单位 (m+1, m+2, m+3,等等)。这一知识允许对完全消耗的 FA 的膳食路径与从头生物合成的比值进行结论。isotopologue 分析被认为是评价土壤动物脂肪酸代谢的有用工具, 以解开营养的相互作用。
引言
在一个神秘的栖息地如土壤, 营养关系是难以解决, 并进一步限制小规模的动物。在过去的十年中, 生物化学生态学取得了进展, 特别是在使用脂肪酸作为生物标志物, 以确定田间条件下土壤动物的饲养策略1,2,3。这是基于一个事实, 即从资源中脂肪酸可以被纳入到消费组织作为整个分子, 一个过程称为膳食路由4。从真菌到线虫到跳虫 5, 已经报告了三多种营养水平的脂肪酸转移。最近, 捕食动物区系被认为是6,7和第一次关于脂肪酸的评论作为土壤食物网的营养标记被发布了8,9。
通过脂肪酸稳定同位素探针 (FA-SIP) 获得了更详细的营养相互作用信息。在饮食和消费者中脂肪酸的13c/12c 比值的确定可以归因于二进制链接和估计伴生的碳流动, 并且被使用了在地球、淡水和海洋食物网10,11 ,12,13。基本的假设是, 膳食路由脂肪酸不受酶过程的影响;因此, 它们的13C 信号,即 13c/12c 比脂肪酸, 在消费者中类似于饮食1。然而, 在水生系统中报告了食物链的13C 签名逐渐枯竭, 从而阻碍了在营养研究中广泛应用 FA SIP14,15,16。此外, 陆地食物网中大多数无脊椎动物的脂代谢知识仍然有限。
了解消费者的脂质代谢通路, 对于利用营养标记脂肪酸作为测定食品网络生态学中定量碳流量的手段是至关重要的。考虑到这一点, 13C-isotopologue 分析, 原则上可以应用于对任何生物系统的碳代谢的研究17, 这是一个有希望的方法。在引入13c 标记的碳基板之后, 在新陈代谢网络中的13C 的分布是可追踪的, 因为在消费者中产生的代谢产物显示了特定的 isotopologue 分布。这可以通过定量的核代谢共振光谱学18,19或质谱20,21来评估, 后者在生物样品中偏爱低生物量由于其较高的灵敏度。
虽然 isotopologue 分析已成功应用于氨基酸, 并提供了对细菌病原体的体内碳代谢的洞察力17,22,23, 它的实施在脂肪酸已经落后了。第一次详细分析了稳定同位素标签前体脂肪酸的命运, 其膳食路由或降解通过β氧化, 在土壤无脊椎动物的消费者, 最近进行了门泽尔et 等。24. 在这里, 给出了与13C 标记脂肪酸结合实验的方法学基础, 并对常见的土壤无脊椎动物 (跳虫) 中的关键子代进行了 isotopologue 分析。这些 microarthropods 是一个很好的模型组, 因为它们构成了土壤食物网的重要组成部分, 并对其营养标记脂肪酸8,25进行了良好的调查。
了解消费者的脂质代谢通路, 对于利用营养标记脂肪酸作为测定食品网络生态学中定量碳流量的手段是至关重要的。本协议给出了实验室饲养实验的设计和建立, 以及从跳虫中提取和甲基化显性脂质分数 (中性脂质、磷脂) 的生物化学程序。通过质谱分析, 说明了脂肪酸的 isotopologue 组成, 并描述了相关的公式和计算。此过程的结果是: (i) isotopologues 超过父离子质量的比率 (即,脂肪酸分子离子 M+) 由一个或多个质量单位 (m+1, m+2, M+3,等) 和 (二) 总体13c. 从13C 标记的前驱体中提炼出的脂肪酸。虽然使用跳虫, 这种方法一般可以适用于任何其他捕食者-猎物互动的前提下, 这些是可培养在足够数量的控制条件下, 以确保成功的标签摄取和随后验证。
研究方案
被描述的协议不属于动物道德的能力。然而, 当人们将所描述的协议与更高的动物相适应时, 请注意动物伦理委员会批准了《牲畜处理议定书》。
1. 动物的耕种
注意: 所有解释的实验步骤都基于已建立良好的协议26,27,28。Biotests 在实验室需要持续供应容易可培养有机体。在这里, 使用了跳虫物种Protaphorura fimata (Gisin、1952) 和Hetermurus 大足(坦普尔顿, 1835)。这两种动物都是简单的维持作为生产的实验室文化喂养贝克的酵母。
- 在塑料缩影与紧的贴合盖子 (直径 7 cm, 高度 4.5 cm), 增加活性炭的混合物, 巴黎石膏和蒸馏水提供高度潮湿的养殖基体 (图 1A)。
- 当准备缩影混合足够的基板时,例如,在批处理中为10缩影。将巴黎 (225 克) 和1部干活性炭 (25 克) 的石膏9个部分混合在一个石膏锅中, 小心地添加大约10个蒸馏水 (250 毫升), 并允许在室温下不搅拌的情况下坐5分钟。
- 用实验勺子以顺时针方向搅拌, 避免气泡, 直到达到厚糊状的一致性。立即倒入缩影的高度约1厘米。
- 平滑的石膏, 轻轻敲击板凳和旋转。注意, 孔 (气泡的随机产品) 和沟 (积极添加无菌铲) 可能会鼓励肥沃的跳虫产卵。这项研究避免了孔和沟有利于具有相同的可再生条件。但是, 为了演示目的,图 1B显示了一些漏洞。
- 允许在室温下干燥 1-2 天;孵育在60°c 可能减少那时间到 1-2 h。
- 在使用前先滋润缩影, 再用吸管加入自来水, 直到基体稍潮湿。保持微观湿润, 定期添加蒸馏水, 因为跳虫有一个柔软的角质层和容易脱水。
- 使用简单的吸入管将跳虫容易地转移到石膏底座上,即长的硅管, 长约25厘米, 带有一个小网格的吸管尖端, 以防止动物吸入管内。或者, 通过允许它们附着在小刷子上的刚毛来转移动物。
- 转移 (参见步骤 1.2) 30 新鲜地孵化了跳虫入新的缩影并且提供粒状干面包师的酵母作为食物 (关于刀子小费) (图 1B);每周至少更新两次。计划三独立复制每个取样日;在这个研究天 0-7 和14。在黑暗中孵化15摄氏度。保持恒温是必不可少的, 因为脂肪酸是由动物新陈代谢改变, 以满足膜流动性的要求。
- 饲料跳虫与贝克的酵母四周前开始的暴露实验, 以获得一个均匀的13c/12c 信号和模式的脂肪酸。使用吸入管或刷子去除所有的鸡蛋 (图 1C), 粪便颗粒, 并定期蜕皮动物可能饲料, 从而改变他们的血脂剖面。

图 1: 跳虫的种植.(A) 填充有繁殖基质的缩影, 一种由巴黎石膏、活性炭和蒸馏水混合而成的干燥混合物。(B) 和 (C) Protaphorura fimata区域性的代表性标本;注意干燥面包师的小掘金作为食物来源并且作为孔在繁殖基体 (黑箭头) (B) 并且二个蛋 (白色箭头) (C)。请单击此处查看此图的较大版本.
2. 标签饮食、收获和样品处理
- 标签
- 四星期在建立跳虫文化之后, 安置30个人入新的缩影和孵化在15°c 在黑暗中。
- 通过将13C 标记的棕榈酸与贝克的酵母混合在一个比 0.5: 1,例如, 5 g, 给他们喂食面包师酵母包含完全13C 标记的棕榈酸 (13C16:0, 99 atom%), 引入脉冲标签13C16:0 和10克干面包师的酵母。在每个缩影上放置一个刀尖。
- 6小时后, 用完全未标记的面包酵母取代这个标签食品。
-
进一步耕种
- 在实验过程中, 每三天更新一次酵母的饮食, 增加的数量比跳虫在那个时期内消耗的要高。最重要的是, 使用吸入管或刷子清除 Collembolan 鸡蛋, 粪便颗粒和蜕皮定期, 以确保动物专属喂养的提供的食物。
- 收获
- 样品缩影破坏性每日直到天7。然后在14天, 收集三独立复制在每个取样时间;不同的取样时间是可能的。
- 准备10毫升玻璃管, 配备聚四氟乙烯涂层螺丝帽, 每个样品一个。事先清洗这些管子在玻璃器皿洗涤器和漂洗两次与去离子水之后。最后, 通过添加2毫升氯仿 (HPLC 级)、涡流大致和废弃溶剂, 去除疏水性污染物的痕迹。
- 对于控制样本 (0 天), 采取 3 30 非暴露跳虫从前文化作为天0样品。
- 对于暴露的样品 (1 天和以后), 采取每日样品在每个案件 3 30 暴露的跳虫从文化 intermediately 哺养与13C 标记棕榈酸和贝克酵母的混合物。
- 用超天平记录跳虫的新鲜重量。使用排或刷子将动物转移到适当的刻度盘上。为确保在秤盘中称量时容易处理跳虫, 震凉动物之前 (-80 °c 为2小时)。或者, CO2流为10分钟可以安全地电击动物。
- 直接称量后把动物从鳞片锅里仔细地放入10毫升玻璃试管中。用1毫升甲醇 (HPLC 级) 填充管, 并贮存在-20 摄氏度, 直到分析。
注: 从这一步骤开始, 避免与塑料设备的样品处理, 因为有机溶剂涉及;改用适合于溶剂和玻璃容器的分配器和吸管。
3. 动物组织和 Methanolysis 的脂质提取
- 准备三玻璃试管 (配备聚四氟乙烯涂层螺钉帽), 每批仅含1毫升甲醇作为空白值。重要的是, 添加或转让本协议中使用的任何溶剂仅由玻璃吸管或氯仿/甲醇冲洗耐溶剂的分配器。
- 在油脂提取过程开始时, 通过蒸发降低用于贮存 (或冲裁) 的甲醇;推荐一种配备真空泵和冷阱的紧凑型台式旋转真空集中器 (RVC)。将开管转入 RVC, 蒸发至干燥50摄氏度, 真空压力为 200 hPa, 20 分钟。
- 加入5毫升的单相萃取溶剂 (氯仿/甲醇/0.05 米磷酸盐缓冲 1:2: 0.8, pH 7.4) 到每个样品 (包括空白) 和提取 Collembolan 脂在室温下通过摇晃过夜 (~ 200 rpm)。
- 将溶剂转移到新的管中, 再用2.5 毫升的萃取溶剂, 通过晃动3小时来重新提取样品。之后, 结合两个步骤的提取物;建议使用玻璃巴斯德吸管。添加0.8 毫升氯仿和0.8 毫升蒸馏水, 然后混合和离心机在2000克在20°c 为5分钟。最后, 允许样品为水和氯仿相分离提供5分钟的支持。
- 对于脂肪酸模式分析, 将跳虫的总细胞脂分为中性脂质、糖脂宁和磷脂组分。
- 为每个样品, 准备一个硅酸专栏 (商业专栏与 0.5 g 硅酸, 网格大小100-200 µm, 参见材料目录) 通过增加1毫升氯仿 (预处理)。为了加快这一过程, 在真空块上安装柱, 通常用于固相萃取色谱。不要使用尼龙针;用不锈钢管。
- 在用于预处理的氯仿通过柱转移后, 将每个样品的氯仿的完全较低的一步转化为单个的柱。为了简化这一过程, 上部水相可以提前删除。使用吸管的玻璃, 但是, 注意列不干燥。
- 先后洗脱5毫升氯仿 (包括中性脂脂肪酸, NLFAs), 10 毫升丙酮 (糖脂 cell-不分析在这个项目) 和5毫升甲醇 (包括磷脂脂肪酸, PLFAs)。收集各个玻璃容器中的每一个分数。
- 在萃取结束时, 在 RVC 中通过蒸发减少氯仿 (NLFAs) 和甲醇 (PLFAs)。将开管转移到 RVC, 蒸发至干燥, 90 分钟60摄氏度, 真空度为 24 hPa。
- 开始从韦尔奇 (1991)29中添加1毫升的氢氧化钠-甲醇溶液 (45 克氢氧化钠, 150 毫升甲醇和150毫升蒸馏水) 后的 NLFA 和 PLFA 馏分的脂肪酸皂化, 并孵化100在水浴中°c 30 分钟。在冰冷的水中冷却样品2分钟, 然后把样品放回长凳上, 继续在室温下工作。
- 将内部标准添加到每个示例中, 包括空白。选择在实验生物体中不常见的脂肪酸;还可以使用饱和脂肪酸, 通过裂解和选择具有中间链长度的分子来最小化损失。对于许多用途, 奇数编号的 nonadecanoic 酸 (19:0) 工作良好。因此, 在异辛烷中添加30µL 的0.74 毫米溶液。确切的数量是非常重要的-一定要事先检查你的吸管的精确度与天平。
- 加入2毫升盐酸-甲醇 (混合325毫升 6.0 N 盐酸与275毫升甲醇), 孵化在80°c 为10分钟在水浴和冷却迅速在冰为2分钟。这一步是时间和温度敏感;使用 80, 1 °c 和 10 @ 1 分钟检查多少样品可以进入水浴一次, 以保持80°c。
注: 本程序的结果是脂肪酸甲酯 (FAMEs),即,脂肪酸分析仪稳定的汽化气相色谱。 - 最后, 加入1.25 毫升的己烷/甲基叔丁基醚 (1:1) 和岩石轻轻地10分钟, 然后离心机在2000克为5分钟. 移除底相, 保持顶相由 FAMEs 组成;使用玻璃巴斯德吸管。添加3毫升的水氢氧化钠 (10.8 克的氢氧化钠溶解在900毫升蒸馏水), 为洗涤步骤。
- 岩石和离心机。
- 使用玻璃巴斯德吸管完成上含脂的整个阶段, 并转移到装有聚四氟乙烯隔膜的气相色谱样品瓶中。避免包括少量的水相, 因为这将导致 GC 测量的问题。封装瓶和贮存在-20 °c, 直到分析。
4. 用气相色谱法定量测定脂肪酸
- 用气相色谱法对跳虫 (动物) 脂的 NFLA 和 PLFA 馏分中的 FAMEs 进行鉴别和定量。气相色谱仪 (GC) 装有火焰电离检测器 (FID) 是一个已建立和证实的设备30。
- 对于 FAMEs 的识别, 请将样本中峰值的保留时间与名望标准的比较。这些是质量或定量标准混合物的关键 FAMEs 覆盖各种食品和有机体。
- 使用著名的标准混合物, 包括代表 FAs 的调查生物组和实验性饮食。在跳虫中, 这些都是真核生物的脂肪酸特征,例如, 长链多不饱和脂肪酸, 如花生四烯酸 (20:4ω6)。当使用酵母作为饮食这些真菌标记, 如亚油酸 (18:2ω6)。一个好的选择是所谓的名声混合, 包括37种不同脂肪酸频繁的动物, 真菌和植物材料, 和细菌酸甲酯 (BAME) 混合 (见材料表)。
- 要在 GC 上运行示例, 请使用该仪器的相应软件设置一个序列。有关说明, 请参阅制造商手册。该序列从外部标准混合物 (例如名望和 BAME 混合) 开始, 后跟示例。请注意, 有一个保留时间移位,也就是说,在运行示例时, 每次运行时, 从 GC 列中的脂肪酸的洗脱时间略有延迟!无论是在示例序列中运行的每 10th或使用保留时间锁定棕榈酸 (16:0) 的标准。
- 适应与仪器相关的 GC 设置。以下程序建议为高性能 (HP) 毛细管柱 (25 m x 0.2 毫米身份证, 膜厚度0.33 µm. 将注入量设置为1µL 分流进样模式, 并以氢气为载体气体。使用温度程序开始在50°c (举行1分钟) 和增加25°c 最小-1到175°c 后跟3°c 最小-1到230°c (举行为5.7 分钟)。
- 计算 nmol 脂肪酸每克新鲜 (干) 重的有机体使用的反应为每个名望应用已知的数额为各自脂肪酸使用以下惯例:
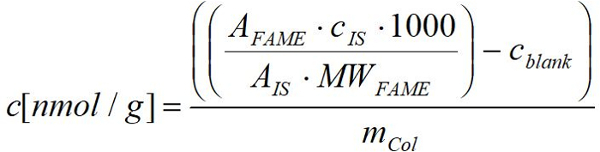
名望: 样品的各自名望的高峰区域
兆瓦FM: 各自名望的分子重量在µg 或µmol
C是: µg 内部标准的集中度
是: 内部标准的峰值区域
m 列: 在 g 中各自跳虫样品的新鲜 (干) 重量
1000: 转换因素从µmol 到 nmol
c生物武器: nmol 中相应空白值的平均值中各自名望的集中度
5. 13C 通过 Isotopologue 剖析分析
- 使用一个 GC 系统耦合到一个质量选择检测器 (MS) 提供一个电子电离 (EI) 来源的 isotopologue 测定。
- 使用极性毛细管柱 (例如, DB 23, CP-88), 因为这进一步允许分离不饱和脂肪酸, 即使有相同数量的双键。GC 列的选择对结果至关重要, 因为它决定了分子离子在脂肪酸中的良好表现。
- 对于 DB 23 列 (60 m x 0.25 毫米的 id, 膜厚度0.15 µm), 开始烤箱温度在130°c 和增加6.5 °c/分钟到170°c。跟随以增量3°c/分钟到203°c 并且举行1.9 分钟跟随以40°c/分钟的增量到230°c 和举行8.3 分钟. 设置传输线温度为280摄氏度。再次, 调整 GC 方法的仪器。
- 使用包括已知量的 FAMEs 的量化标准, 用于为13C 公司的所有脂肪酸进行调查。将这些标准放在每个示例序列运行的开始和结束处。从这些标准中取出感兴趣的脂肪酸的保留时间。
- 测量样品从实验总是从未标记的样品开始, 并且标记的探针尔后。应用适合于样本浓度的拆分比率,例如1:12.5. 如果可用于仪器, 在每个样品运行后应用一个反冲, 以清除从剩余的分析仪中的柱。
- 将保留时间锁应用于 GC-MS 方法, 使 SIM 采集不受移位, 且有可重现的分析物保留时间。
- 使用该仪器的选定离子监测 (SIM) 模式, 确定13C 在脂肪酸的分子离子中加入。在 SIM 模式下的操作可以检测出与完全扫描模式相关的灵敏度增加的特定的方法。
- 首先运行一个初始扫描, 看看有什么存在, 然后运行 SIM 在适当的离子。通过选择包含各自脂肪酸的色谱峰值时间的 m-/z 扫描窗口 (SIM 组), 获取分子质量感兴趣的数据。通常, 每个分析和时间窗口监控两到四个离子。
- 为了提高灵敏度, 调整质量扫描速率和停留时间 (花在每个肿块上的时间)。最好的质量数据是以尽可能低的速度获得的, 而 SIM 中的一般规则是对分析物峰值的8到12扫描。仪表设置的代理是每质量9毫秒的平均停留时间, 周期为6秒, 扫描时间为 175 ms 周期-1。
- 检测各自脂肪酸及其所有 isotopologues (m+1、m+2等) 的分子离子 (m+)。有关示例, 请参见具有代表性的结果。
- 记录每个离子片段 (isotopologue) 的丰度。注意到分子离子的丰度和 isotopologues 的数量相对较低, 定量的质量很大程度上取决于 MS 系统的性能。在开始一个大样本序列 (实验) 之前运行一首曲子, 并在必要时清洁离子源。
注意: 首先, 这些数据通过消耗的前体 (这里 16:0) 产生每个脂肪酸的总的13C 充实。 - 使用标记脂肪酸的同位素组成与未标记的对应物的比例来分配营养碳通量。使用步骤6.1 中的 atom% 公式计算标记碳 (原子百分比, atom%) 在各自脂肪酸中的百分比。将13c 中的跳虫脂肪酸的百分比与标签 (1 天和更高版本) 和未标记动物 (0 天) 的比例进行比较, 作为13C 从饮食到消费者流动的相对指示。
- 将13C 的位置分配到脂肪酸链中。根据 isotopologues 的分布, 将整个标记示踪脂肪酸 (这里 16:0) 的膳食路由从链伸长的脂质代谢中解开。虽然整个标记分子的同化增加了最远的 isotopologues 的丰度到父离子 (M+),例如, m+15, m+16, 与链伸长由使用13C 标记 C2片段 (13C 乙酰 CoA) isotopologues 接近分子离子变得更加频繁。
6. 13C 富集的计算
- 根据 isotopologues 的分布情况, 使用以下关系计算标记碳 (atom%) 在各自脂肪酸中的总百分比: atom% = (比率13C isotopologue 合并) x (频率各自 isotopologue)
这是在 Kuppardt et之后计算的。31为: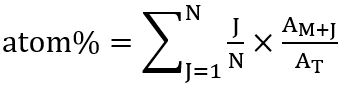
其中N是脂肪酸中的碳原子数, J是13C 同位素的数量, M + J 是各自 isotopologue 的丰度, T 所有 isotopologues 的总丰度。 - 计算时, 用 SIM-MS 分析法检测到各自 FA 和所有 isotopologues (m+1、m+2等) 的分子离子 (m+) 的峰值面积值, 并将其设置为100% 相对丰度。按照三的规则, 计算每个检测到的 isotopologue 的部分很容易。
- 从实验值中减去非标记的天0控制值 (自然13c 背景), 以获取只追溯到执行的外部13C 标记的最终数据。
结果
跳虫的鲜重和脂质含量
在所述实验过程中, NLFAs 和 PLFAs 的含量在一段时间内没有显著变化, 而试样的新鲜重量略有增加, 但不显著24。两个参数都表明跳虫标本的身体素质良好。注意调查跳虫的新鲜体重和脂质含量在整个实验中对应于取样日的脂肪酸和同位素分析。请注意, 在实验期间, 体重的减少和/或脂质含量的降低表明测试机体的适应?...
讨论
Isotopologue 分析
对 FAs 中13C 分布的数量方面的详细分析需要尖端技术来分配食物网中的碳分割。目前的工作使用 isotopologue 分析来评估热带相互作用的共同 FA 生物标志物的13c/12c 比值。采用液相色谱法 (LC) 对氨基酸进行分析, 并应用于致病细菌的碳代谢研究17,23。直到最近, isotopologue 的分析进一步...
致谢
门泽尔和 l. Ruess 的财政支持由德意志 Forschungsgemeinschaft (汝 RU780/11-1) 感激地承认。r. Nehring 由汝 780/10-1 资助。最后, 我们非常感谢榛 Ruvimbo Maboreke 博士校对我们的手稿。
材料
| Name | Company | Catalog Number | Comments |
| neoLab-Round jars | neoLab | 2-1506 | 69 x 40 mm, 10 pacs/pack |
| Charcoal activated | Carl Roth | X865.1 | p.a., powder, CAS No. 7440-44-0 |
| Alabaster Dental | RÖHRICH-GIPSE | --- | http://www.roehrich-gipse.de/dentalgipse.php |
| Chloroform | Carl Roth | 7331.1 | HPLC ≥ 99,9 % |
| Methanol | Carl Roth | P717.1 | HPLC ≥ 99,9 % |
| Hexan | Carl Roth | 7339.1 | HPLC ≥ 98 % |
| tert-Butyl methyl ether (MTBE) | Carl Roth | T175.1 | HPLC ≥ 99,5 % |
| Aceton | Carl Roth | 7328.2 | HPLC ≥ 99,9 % |
| NaOH | Carl Roth | 6771.1 | p.a. ≥99 %, in pellets |
| di-Natriumhydrogenphosphat | Carl Roth | P030.1 | p.a. ≥99 % , water free |
| Na-dihydrogenphosphat Dihydrat | Carl Roth | T879.1 | p.a. ≥99 % |
| Hypochloric acid (6 N) | VWR International | 26,115,000 | AVS TITRINORM vol. solution |
| Bond Elut (Columns) | Agilent Tech. | 14102037 | HF Bond Elut-SI, 500 mg, 3 mL, 50/PK |
| Präparatengläser Duran | Glasgerätebau Ochs | 135215 | Ø 16 x 100 mm, plus screw cap with handy knurl and integrated PTFE/silicone gasket |
| Supelco 37 Component FAME Mix | Sigma-Aldrich | 47885-U Supelco | 10 mg/mL in methylene chloride, analytical standard |
| FlowMesh | Carl Roth | 2796.1 | Polypropylene mesh, approximately 0.3 mm thick, with 1 mm strand spacing |
| Bacterial Acid Methyl Ester (BAME) Mix | Sigma-Aldrich | 47080-U Supelco | 10 mg/mL in methyl caproate, analytical standard |
| Methyl nonadecanoate | Sigma-Aldrich | 74208 | analytical standard ≥ 98.0 % |
| Hexadecanoic acid-1-13C (Palmitic) | Larodan Fine Chemicals | 78-1600 | GC ≥ 98.0 % (13C: 99.0 %) |
| RVC 2-25 CDplus | Martin Christ Gefrier-trocknungsanlagen | Compact benchtop midi concentrator | |
| Alpha 2-4 LDplus | Martin Christ Gefrier-trocknungsanlagen | Drying manifold | |
| MZ 2C NT | Vacuubrand GMBH | Vacuum pump | |
| Roto-Shake Genie | Scientific Industries | Combined rocking and rotating device | |
| XP64 Micro Comparator | Mettler Toledo | Super high precision balance | |
| GC-System 7890A | Agilent Tech. | Gas chromatograph | |
| 7000 GC/MS Triple Quad | Agilent Tech. | Triple Quad mass spectrometer | |
| 7683B Series Injector | Agilent Tech. | Sample injector | |
| Heraeus Multifuge 3SR+ | Thermo Scientific | Centrifuge with 10 ml tube rotor |
参考文献
- Ruess, L., et al. Application of lipid analysis to understand trophic interactions in soil. Ecology. 86 (8), 2075-2082 (2005).
- Ruess, L., et al. Lipid composition of Collembola and their food resources in deciduous forest stands - Implications for feeding strategies. Soil Biology and Biochemistry. 39 (8), 1990-2000 (1990).
- Chamberlain, P. M., Bull, I. D., Black, H. I. J., Ineson, P., Evershed, R. P. Fatty acid composition and change in Collembola fed differing diets: identification of trophic biomarkers. Soil Biology and Biochemistry. 37 (9), 1608-1624 (2005).
- Stott, A. W., Davies, E., Evershed, R. P., Tuross, N. Monitoring the routing of dietary and biosynthesised lipids through compound-specific stable isotope (delta C-13) measurements at natural abundance. Naturwissenschaften. 84 (2), 82-86 (1997).
- Ruess, L., Haggblom, M. M., Langel, R., Scheu, S. Nitrogen isotope ratios and fatty acid composition as indicators of animal diets in belowground systems. Oecologia. 139 (3), 336-346 (2004).
- Pollierer, M. M., Scheu, S., Haubert, D. Taking it to the next level: Trophic transfer of marker fatty acids from basal resource to predators. Soil Biology and Biochemistry. 42 (6), 919-925 (2010).
- Ferlian, O., Scheu, S., Pollierer, M. M. Trophic interactions in centipedes (Chilopoda, Myriapoda) as indicated by fatty acid patterns: Variations with life stage, forest age and season. Soil Biology and Biochemistry. 52, 33-42 (2012).
- Ruess, L., Chamberlain, P. M. The fat that matters: Soil food web analysis using fatty acids and their carbon stable isotope signature. Soil Biology and Biochemistry. 42 (11), 1898-1910 (2010).
- Traugott, M., Kamenova, S., Ruess, L., Seeber, J., Plantegenest, M. Empirically characterising trophic networks: What emerging DNA-based methods, stable isotope and fatty acid analyses can offer. Adv Ecol Res. 49, 177-224 (2013).
- Hammer, B. T., Fogel, M. L., Hoering, T. C. Stable carbon isotope ratios of fatty acids in seagrass and redhead ducks. Chemical Geology. 152 (1-2), 29-41 (1998).
- Budge, S. M., Iverson, S. J., Koopman, H. N. Studying trophic ecology in marine ecosystems using fatty acids: A primer on analysis and interpretation. Marine Mammal Science. 22 (4), 759-801 (2006).
- Haubert, D., et al. Trophic structure and major trophic links in conventional versus organic farming systems as indicated by carbon stable isotope ratios of fatty acids. Oikos. 118 (10), 1579-1589 (2009).
- Ngosong, C., Raupp, J., Richnow, H. H., Ruess, L. Tracking Collembola feeding strategies by the natural 13C signal of fatty acids in an arable soil with different fertilizer regimes. Pedobiologia. 54 (4), 225-233 (2011).
- Bec, A., et al. Assessing the reliability of fatty acid-specific stable isotope analysis for trophic studies. Methods in Ecology and Evolution. 2 (6), 651-659 (2011).
- Gladyshev, M. I., Makhutova, O. N., Kravchuk, E. S., Anishchenko, O. V., Sushchik, N. N. Stable isotope fractionation of fatty acids of Daphnia fed laboratory cultures of microalgae. Limnologica. 56 (Supplement C. 56 (Supplement C), 23-29 (2016).
- Gladyshev, M. I., Sushchik, N. N., Kalachova, G. S., Makhutova, O. N. Stable isotope composition of fatty acids in organisms of different trophic levels in the Yenisei river. PLoS One. 7 (3), e34059 (2012).
- Eisenreich, W., Dandekar, T., Heesemann, J., Goebel, W. Carbon metabolism of intracellular bacterial pathogens and possible links to virulence. Nature Reviews Microbiology. 8 (6), 401-412 (2010).
- Eylert, E., Bacher, A., Eisenreich, W. NMR-based isotopologue profiling of microbial carotenoids. Methods Mol Biol. 892, 315-333 (2012).
- Garton, N. J., O'Hare, H. M. Tuberculosis: feeding the enemy. Chemical Biology. 20 (8), 971-972 (2013).
- Rosenblatt, J., Chinkes, D., Wolfe, M., Wolfe, R. R. Stable isotope tracer analysis by GC-MS, including quantification of isotopomer effects. Am J Physiol. 263 (3), E584-E596 (1992).
- Fernandez, C. A., Des Rosiers, C., Previs, S. F., David, F., Brunengraber, H. Correction of 13C mass isotopomer distributions for natural stable isotope abundance. J Mass Spectrom. 31 (3), 255-262 (1996).
- Heuner, K., Eisenreich, W. The intracellular metabolism of legionella by isotopologue profiling. Methods Mol Biol. 954, 163-181 (2013).
- Willenborg, J., et al. Characterization of the pivotal carbon metabolism of Streptococcus suis serotype 2 under ex vivo and chemically defined in vitro conditions by isotopologue profiling. J Biol Chem. 290 (9), 5840-5854 (2015).
- Menzel, R., Ngosong, C., Ruess, L. Isotopologue profiling enables insights into dietary routing and metabolism of trophic biomarker fatty acids. Chemoecology. 27 (3), 101-114 (2017).
- Buse, T., Ruess, L., Filser, J. New trophic biomarkers for Collembola reared on algal diets. Pedobiologia. 56 (3), 153-159 (2013).
- Hutson, B. R. Effects of variations of the plaster-charcoal culture method on a Collembolan, Folsomia candida. Pedobiologia. 18, 138-144 (1978).
- Fountain, M. T., Hopkin, S. P. Folsomia candida (Collembola): a "standard" soil arthropod. Annu Rev Entomol. 50, 201-222 (2005).
- ISO, I. O. f. S. . Soil Quality-Inhibition of reproduction of Collembola (Folsomia candida) by soil pollutants. , (1999).
- Welch, D. F. Applications of cellular fatty acid analysis. Clin Microbiol Rev. 4 (4), 422-438 (1991).
- Dodds, E. D., McCoy, M. R., Rea, L. D., Kennish, J. M. Gas chromatographic quantification of fatty acid methyl esters: flame ionization detection vs. electron impact mass spectrometry. Lipids. 40 (4), 419-428 (2005).
- Kuppardt, S., Chatzinotas, A., Kastner, M. Development of a fatty acid and RNA stable isotope probing-based method for tracking protist grazing on bacteria in wastewater. Appl Environ Microbiol. 76 (24), 8222-8230 (2010).
- Zhang, X., He, H., Amelung, W. A GC/MS method for the assessment of 15N and 13C incorporation into soil amino acid enantiomers. Soil Biology and Biochemistry. 39 (11), 2785-2796 (2007).
- Vetter, W., Thurnhofer, S. Analysis of fatty acids by mass spectrometry in the selected ion monitoring mode. Lipid Technol. 19 (8), 184-186 (2007).
- Thurnhofer, S., Vetter, W. A gas chromatography/electron ionization-mass spectrometry-selected ion monitoring method for determining the fatty acid pattern in food after formation of fatty acid methyl esters. J Agric Food Chem. 53 (23), 8896-8903 (2005).
- Haubert, D., Haggblom, M. M., Scheu, S., Ruess, L. Effects of fungal food quality and starvation on the fatty acid composition of Protaphorura fimata (Collembola). Comparative Biochemistry and Physiology B-Biochemistry & Molecular Biology. 138 (1), 41-52 (2004).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。