このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
脂肪酸13C Isotopologue プロファイリング栄養炭素転送および無脊椎動物消費者の脂質代謝への洞察力を提供します。
要約
すなわち、分子全体消費者組織への伝達と脂肪酸の同化脂肪酸の栄養マーカー アプローチ変更が小さい土壌無脊椎動物の脂肪酸代謝の知識のギャップによって妨げられて無またはマイナー。Isotopologue ・ プロファイリングは、栄養相互作用を分離するための貴重なツールとして提供されます。
要約
通常完全な分子として同化、マイナーまたはなしの変更は、異なる栄養段階の間食事ルーティングできるように消費者組織に転送されるため、脂肪酸 (FAs)、食物網の生態学的に有用なバイオ マーカーです。しかし、FA 栄養マーカー アプローチはまだ土壌動物相の脂質代謝の限られた知識によって妨げられます。この調査は、脂肪酸代謝経路の 2 つの広範な土壌トビムシ、 Protaphorura fimataとHeteromurus 光沢苔のトレーサーとして完全ラベル パルミチン酸 (13C16:0、99 の atom %) を使用しました。運命、この前駆体の新陳代謝の変更を調査するために isotopologue のプロファイリングの手法を提案する単一イオンのモニタリングを用いた質量分析法によって行います。さらに、上流の研究室実験を供給、抽出および支配的な脂質画分 (中性脂質、リン脂質) と関連数式および計算のメチル化して説明します。Isotopologue プロファイリング13C 標識前駆体由来の脂肪酸で全体の13C 濃縮収量のみではなく、また親イオン (すなわちFA の分子イオンの質量を超える isotopologues のパターンを生成M+) それぞれのラベルの 1 つまたは複数の質量単位(M+1、+2M、M+3等)で FA。この知識は、デノボ合成と比較して完全に消費された FA の栄養経路の比率に結論をことができます。Isotopologue のプロファイリングは、栄養相互作用を分離する土壌動物における脂肪酸代謝の評価のための便利なツールとして使用することをお勧めします。
概要
土壌など不可解な生息地で栄養関係解決困難な動物のサイズが小さいによってさらに制限されます。最後の十年は、フィールド条件1,2,3の下の土壌動物相の供給戦略を定義するためのバイオ マーカーとしての脂肪酸の使用で特に、生化学的な生態学の進歩を見ています。これは全体の分子として消費者組織のリソースから脂肪酸を組み込むことができるという事実に基づく、食事ルーティング4と呼ばれる、プロセス。脂肪酸の転送は、以上 3 つ栄養のレベル、すなわち、菌類から粘管目5線虫に報告されています。最近では、捕食動物は、6、7を考慮され、土壌食物網における栄養マーカーとして脂肪酸の最初のレビューが公開された8,9をされています。
栄養相互作用の詳細については、脂肪酸の安定同位体プロービング (FA SIP) によって達成されます。13C の定量/ダイエットと消費することができます脂肪酸12C 比バイナリ リンクのせいと関連付けられている炭素フローを見積もるし、地上波で採用されている、淡水、海洋の食物網10,11 ,12,13。基本的な前提は、ルーティングされた脂肪酸が酵素プロセスの対象とはしたがっての13C 信号、すなわち、 13C/12C 消費者の脂肪酸の比はダイエット1に類似しています。ただし、食物連鎖を13C 署名の段階的な減少は栄養研究14,,1516FA SIP の広範なアプリケーションをそれにより妨げる水生システムで報告されています。さらに、地上の食物網におけるほとんどの無脊椎動物の脂質代謝の知識はまだ限られています。
栄養マーカー脂肪酸食物網生態学における定量的炭素フローの決定のための手段としての使用のための消費者における脂質代謝経路の理解が欠かせません。念頭、 13C isotopologue のプロファイリングは、原理的にはすべての生物学的システムの17、炭素代謝の調査のため適用できる有望な方法であります。13C 標識炭素基板、 13C 代謝ネットワーク内の分布の導入は消費者を特定の isotopologue 分布の生成された代謝産物からトレースできます。これは定量核代謝共鳴分光法18,19または質量20,は21、その高いため低バイオマス後者の支持された生体試料によって評価されることができます。感度。
ただし、isotopologue プロファイルがされて正常にアミノ酸に適用し、細菌性病原体17,22,23, 脂肪酸の実装の体内の炭素代謝に洞察力を提供酸が遅れています。安定同位体標識前駆体脂肪酸、その食物ルーティングや劣化、土壌無脊椎動物の消費者に β 酸化を介しての運命の最初の詳細な分析、最近メンツェルらによって実行されました。24. 13C 標識脂肪酸 isotopologue による頻繁な土壌無脊椎動物、トビムシ、キーの子孫の後で導入実験の方法論的基礎を提供するここでは。これらの植栽は、土壌食物網とがよく、栄養マーカー脂肪酸8,25の調査の重要なコンポーネントは、フォームの良いモデル グループです。
栄養マーカー脂肪酸食物網生態学における定量的炭素フローの決定のための手段としての使用のための消費者における脂質代謝経路の理解が欠かせません。現在のプロトコルは、トビムシから実験、および抽出および支配的な脂質画分 (中性脂質、リン脂質) のメチル化のための生化学的なプロシージャを供給所のデザインと設定を与えます。質量分析法による脂肪酸の isotopologue 組成を分析する方法について説明し、関連の数式と計算について説明します。この手順の結果: (i) 1 つ以上によって (すなわち、脂肪酸分子イオン M+) 親イオンの質量を超える isotopologues の比質量単位 (M+1、+2M、M+3、等) および (ii) 全体1313C 標識前駆体由来の脂肪酸で C 濃縮。このアプローチは、これらは、成功したラベルの吸収量を確保するための制御された条件下で十分な量の培養とその後を前提に他の捕食者-被食者相互作用に適用できる一般にトビムシに使用、検証します。
プロトコル
記述されていたプロトコルは、動物倫理の能力には該当しません。しかし、人々 は、高等動物の記述されていたプロトコルを適応し場合、により、機関の動物倫理委員会承認動物処理するためのプロトコルであること注意します。
1. 動物の栽培
注: すべては確立プロトコル26,27,28に基づいて実験の手順を説明します。バイオテスト研究所では、簡単に培養生物の連続的な供給を必要があります。ここでは、 Protaphorura fimata (Gisin、1952) とHetermurus 光沢苔(テンプルトン、1835) トビムシ種が使用されています。両種は、生産的な検査室で培養の酵母にうんざりとして維持するために簡単です。
- タイト フィットの蓋とプラスチック マイクロコズム (径 7 cm、高さ 4.5 cm) 活性炭、パリの石膏の混合物を追加し、非常に湿った繁殖基板 (図 1 a) を提供するために蒸留水します。
- ときに十分な基板、例えばバッチで 10 小宇宙の小宇宙ミックスを準備しています。パリのプラスター (225 g) のミックス 9 パーツとドライ活性炭 (25 g) 一緒に石膏の鍋の 1部蒸留水 (250 mL) の慎重に約 10 の部品を追加し、室温で攪拌せず 5 分間座ってできるように。
- 厚酒に酔って一貫性を達成するまでに空気の泡を避けるために時計回り方向に適度な速度でラボのスプーンでかき混ぜます。約 1 cm の高さに小宇宙にすぐに注ぐ。
- ベンチにタップして旋回を穏やかな石膏を滑らかに。(空気泡のランダムな製品) の穴と溝 (滅菌スパチュラ積極的に追加) は肥沃なトビムシが産卵を促すことがありますに注意してください。本研究は、穴と同じ再現性を持っていることを支持して溝を避けます。但し、デモンストレーション用図 1 bは、いくつかの穴を示します。
- 部屋の温度で約 1-2 日の乾燥させる60 ° c の孵化は 1-2 時間にその時間を短縮可能性があります。
- 基板は少し湿気があるまでピペットで水道水を追加して使用する前に小宇宙を湿らせます。トビムシ柔らかい表皮と乾燥に敏感である定期的に蒸留水を追加することによって小宇宙を湿った保ちます。
- シンプルな吸引チューブ、すなわち、約 25 cm の長いシリコン チューブを使用して石膏ベースにトビムシを簡単に転送ピペット チップと長いチューブに動物の吸引を防ぐために小さなメッシュを装備。また、小さなブラシの毛に付着することにより動物を譲渡します。
- (手順 1.2 参照) を転送 30 新鮮な新しい小宇宙にトビムシを孵化し、食品 (約ナイフ先端) として粒状乾燥パン酵母を提供する (図 1 b)。週に少なくとも 2 回を更新します。プラン 3 つの独立したサンプリング日あたり; 複製します。この研究の日 0 - 7 と 14。暗闇の中では、15 ° C で孵化させなさい。本質的な一定の温度を維持する脂肪酸として、膜流動性の要件を満たすために動物の新陳代謝によって変更されます。
- トビムシをパン酵母と同種13C を取得する屋外暴露実験を開始する前に 4 週間のフィード/12C 信号と脂肪酸のパターン。全卵 (図 1)、糞便のペレットを削除吸引チューブやブラシを使用し、ぬけがら定期的に動物として可能性がありますそれらのフィードそれらの脂質プロファイルを変更すること。

図 1: トビムシの栽培します。(A) 小宇宙基板、パリのプラスター、活性炭と蒸留水の乾燥混合物を繁殖でいっぱい。(B) と (C) Protaphorura fimata文化の代表的な標本乾燥パン酵母の食料源として、また繁殖基板 (黒矢印) の穴として使用される小さなナゲットに注意してください (B) 2 つの卵 (白い矢印) だけでなく、(C)。この図の拡大版を表示するのにはここをクリックしてください。
2. ラベルのダイエット、収穫、および検体の取扱い
- ラベリング
- トビムシの文化を確立した後 4 週間は、新しい小宇宙に 30 人を置き、暗闇の中で, 15 ° C で。
- 餌付け比脂肪、例えば、5 g でヘラでパン酵母と13C 標識パルミチン酸を混合することによって完全13C 標識パルミチン酸 (13C16:0、99 の atom %) を含有パン酵母、パルスのラベルをご紹介します。13C16:0 と 10 g パンの酵母を乾燥します。についてそれぞれの小宇宙にナイフの先端を配置します。
- 6 時間後に、完全にラベルのない酵母食品のラベルこれを置き換えます。
-
さらに栽培
- 実験の過程で酵母ダイエット 3 日ごとの更新、トビムシがその期間内に消費するものよりも高い金額を追加します。最も重要なは、トビムシの卵、糞、提供された食品に動物によって排他的な供給を確保するために定期的にぬけがらを削除するのに吸引チューブやブラシを使用します。
- 収穫
- 7 日目まで毎日破壊的小宇宙をサンプルします。14 日に、各サンプリング時 3 つの独立した複製を収集します。異なるサンプリング時間可能です。
- 10 mL ガラス管テフロン加工スクリュー キャップ、各サンプルを 1 つ装備を準備します。あらかじめガラス洗濯機にこれらの管をきれいにし、脱イオン水で 2 回リンス、その後。最後に、疎水性のすべてのトレースを削除する汚染物質をクロロホルム (HPLC 級) 2 mL を追加で 2 回洗う渦約溶媒を破棄します。
- コントロールのサンプル (日 0)、0 日サンプルとして前の文化から 3 30 非曝露トビムシを取る。
- 露出サンプル (1 日目以降)、取る文化からの各場合 3 の 30 の毎日のサンプル公開のトビムシ中間の13C 標識パルミチン酸と酵母のミックスを供給します。
- 超振動によってトビムシの新鮮重を記録します。排気装置または適切なスケール鍋にブラシを使用して動物を譲渡します。スケール鍋、(2 時間-80 ° C) の前に衝撃のクールな動物で計量中にトビムシを簡単に処理できるように。また、10 分の CO2ストリームすることができます安全に動物を気絶させます。
- 直接後計量動物を入れてスケール鍋から慎重に 10 mL のガラス試験管に。1 ml のメタノール (HPLC 級)、-20 ° C でストアの分析まで、管を入力します。
注: このステップからサンプル処理プラスチック製の装置で有機溶剤が関与している; を避ける代わりにディスペンサーとガラス器と同様に、溶剤に適しているピペットを使用します。
3. 動物組織メタノリシスから脂質の抽出
- 3 ガラス試験管 (テフロン加工スクリュー キャップ付け) 空白値としてのみ 1 mL のメタノールが含まれているバッチあたりを準備します。重要なは、追加またはガラス ピペットまたはクロロホルム/メタノール洗浄溶剤用ディスペンサーによってのみこのプロトコルで使用溶剤を転送します。
- 脂質抽出プロセスの初めに、減らすメタノール蒸発; によってストレージ (またはブランク) の適用コンパクトなベンチトップ型回転真空コンセントレーター (RVC) 真空ポンプとコールド トラップ装備をお勧めします。RVC に開いてチューブを転送し、20 分間 200 hPa の 50 ° C と真空圧下で乾燥するまで蒸発します。
- (空白を含む) 各サンプルに単相抽出溶媒 (chloroform/methanol/0.05 M リン酸バッファー 1:2:0.8、pH 7.4) 5 mL を追加し、一晩を揺することによって室温でトビムシ脂質を抽出 (~ 200 rpm)。
- 新しいチューブに溶媒を転送、再抽出溶媒の 2.5 mL の追加で 3 h に振ることによってサンプルを抽出します。その後、両方のステップからの抽出物を組み合わせるパスツール ピペット ガラスの使用をお勧めします。クロロホルムと 0.8 mL の蒸留水、0.8 mL を追加し、ミックス 5 分 20 ° C で 2,000 g の遠心分離機します。最後に、水溶液の分離のための 5 分間放置し、クロロホルムにサンプルを許可する段階。
- 脂肪酸パターン解析のためには、中性脂質、糖脂質、リン脂質画分にトビムシの総細胞脂質を分割します。
- 各サンプルのため準備するシリカの酸列 (0.5 g ケイ酸酸、メッシュ商業列 100-200 μ m のサイズ、材料の表を参照してください) (前処理) クロロホルム 1 mL を追加することによって。このプロセスを高速化、一般固相抽出クロマトグラフィー用真空ブロックの列をマウントします。ナイロン針; を使用しないでください。管の上部のステンレス鋼を使用します。
- 前処理用クロロホルムの後を通過した列転送各サンプルの完全な低いクロロホルム段階個々 の列に。この手順を簡素化するには、上層の水相を事前に削除することができます。ただし、ガラスのパスツール ピペットを使用して、列が乾燥しないでください注意してください。
- 順次溶出脂質画分 (含む中性脂質脂肪酸、NLFAs)、クロロホルム 5 mL と 10 mL のアセトン (糖脂質 - このプロジェクトでは分析されません)、メタノール (リン脂質脂肪酸組成、PLFAs を含む) の 5 mL。個々 のガラス容器の各分画を収集します。
- 抽出の最後に、クロロホルム (NLFAs) と、RVC の蒸発によってメタノール (PLFAs) を削減します。RVC に開いてチューブを転送し、乾燥、蒸発 〜 60 ° C および 24 hPa の真空で 90 分。
- 脂質 (NLFA と PLFA の分画) ・ ウェルチ (1991)29水酸化ナトリウム ・ メタノール溶液 (水酸化ナトリウム 45 g、メタノール、150 mL と 150 mL の蒸留水) 1 mL の添加からプロトコルを次の鹸化を開始し、, 100 で° C の水浴中で 30 分間。2 分、冷たい水でサンプルを冷却し、ベンチに戻るサンプルを入れて室温で作業を続行します。
- 内部標準をブランクを含む各サンプルに追加します。実験生物でない一般的な脂肪酸を選択します。また胸の谷間で損失を最小限に抑え、中間のチェーン長さをもつ分子を選択飽和脂肪酸を使用します。奇数 nonadecanoic 酸 (19:0) うまく、多くの目的のため。だから、イソオクタンで 0.74 mM ソリューションの 30 μ L を追加します。事前に、天秤と、ピペットの精度をチェックして必ず正確な量が非常に重要です - です。
- 塩酸 - メタノール (ミックス 275 ml のメタノールの 6.0 N 塩酸 325 mL) 2 mL を加えて、水浴とクールな 2 分の氷で急速に 10 分で 80 ° C で孵化させなさい。この手順は、時間と温度に敏感。80 ± 1 ° C と 10 ± 1 分チェックで使用水浴に行くことができますどのように多くのサンプルを一度に 80 ° c. を維持するには
注: この手順の結果脂肪酸メチルエステル (FAMEs)、すなわち、脂肪酸分析ガスクロマトグラフィーにおける蒸発現象の安定化します。 - 最後に、ヘキサン/第三ブチル エーテル (1:1) の 1.25 mL を追加、静かに 10 分後 2,000 g で 5 分間遠心ロックを削除下相維持上のフェーズから成る FAMEs;パスツール ピペット ガラスを使用します。洗浄工程の水性水酸化ナトリウム (NaOH 900 mL の蒸留水に溶解の 10.8 g) の 3 mL を追加します。
- ロックと遠心分離機をもう一度。
- 完全な上部脂質を含む段階をガス ・ クロマトグラフィーのサンプルバイアル テフロン隔壁を装備するガラス パスツール ピペットや転送を使用してください。これ GC 測定で問題が発生、水様段階の少量を含むを避けるため。バイアルをカプセル化し、解析まで-20 ° C で保存します。
4. ガスクロマト グラフによる脂肪酸の定量
- ガス ・ クロマトグラフィーを使用して識別し、トビムシ (動物) 脂質の NFLA と PLFA の分画で骨組を定量化します。水素炎イオン化検出器 (FID) 付ガスクロマトグラフィー (GC) は、確立された実績のある機器30です。
- FAMEs の識別のためには、名声標準でサンプルのピークの保持時間を比較します。これらはキー FAMEs 食料品及び生物の様々 なカバーの質的または量的の標準的な混合物です。
- 調査生物グループおよび実験的ダイエットの代表 FAs を含む名声標準混合物を採用してください。これらは、トビムシ、真核生物、例えば長鎖多価不飽和脂肪酸アラキドン酸 (20:4ω6) などの脂肪酸特性です。ダイエットとして酵母を採用する場合これら、リノール酸 (18:2ω6) などの真菌のマーカーです。良い選択は 37 の異なる脂肪酸動物、真菌に頻繁にで構成される、いわゆる名声ミックス、植物材料と細菌酸メチル (バメ) ミックス (材料の表を参照してください)。
- ガスクロマト グラフのサンプルを実行するには、楽器のそれぞれのソフトでシーケンスを設定します。手順については、製造元のマニュアルを参照してください。シーケンスは、外部標準混合物 (例えば名声とバメ ミックス)、サンプルの順に始まります。保持時間シフト、すなわち、わずかな遅延にある、サンプルの実行中に GC カラムから脂肪酸の溶出時間に注意してください!どちらかすべての 10thサンプル シーケンスで実行あるいはパルミチン酸 (16:0) のロック保持時間を使用標準が含まれます。
- GC 設定器に依存を適応します。次のプログラムが示唆された高性能 (HP) 毛細管カラム (25 m × 0.2 mm 内径、フィルム厚み 0.33 μ m 設定注入量スプリットレス モードで 1 μ L に、キャリアガスとして水素を使用します。50 ° c (1 分間開催) 温度プログラム開始と 230 ° C (5.7 分開催) に 3 ° C 分-1に続く 175 ° c の 25 ° C 分-1増を採用してください。
- 次の数式を使用してそれぞれの脂肪酸の知られていた量を適用する各名声の FID によって得られた応答を用いた生物のグラム新鮮な (ドライ) 重量あたり nmol 脂肪酸を計算します。
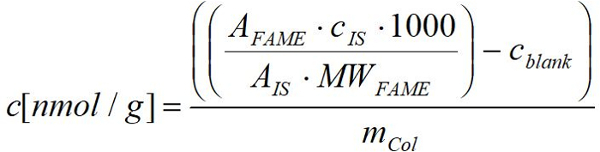
名声: サンプルのそれぞれの名声のピーク面積
MWFM: μ g/µmol でそれぞれの名声の分子量
Cは: μ g の内部標準物質の濃度
IS: 内部標準物質のピーク面積
mコル: g でそれぞれのトビムシ サンプル量の新鮮な (ドライ)
1000: nmol へ µmol からの換算係数
cBW: nmol で対応するブランク値の平均値でそれぞれの名声の濃度
5. 13C Isotopologue プロファイリングによる解析
- 結合する、質量選択検出器 (MS) isotopologue 定量電子イオン化 (EI) ソースに付属している GC システムを使用します。
- これはさらに二重結合数が同じでも不飽和脂肪酸の分離により、極毛細管カラム (例えば、 DB 23 CP Sil 88) を使用します。GC カラムの結果の重要な脂肪酸の分子イオンの良い表現が決定されるためです。
- DB 23 列の (60 m × 0.25 mm i. d.、フィルム厚さ 0.15 μ m) 130 ° c のオーブンの温度を起動し、170 ° c 6.5 ° C/分増203 ° C に 3 ° C/分の増加に従って、1.9 分 230 ° C に 40 ° C/分の増加とフォローと 8.3 分設定の転送ライン温度 280 ° C に保持計測器 GC メソッドを再度調整します。
- 13C の混入のために調査するすべての脂肪酸の FAMEs の知られていた量を含む定量的な基準を使用します。先頭と各サンプル シーケンスの実行の末尾にこれらの標準を置きます。これらの基準からの興味の脂肪酸の保持時間を取る。
- その後ラベルのサンプル、および標識プローブで始まる常に実験からのサンプルを測定します。サンプル濃度、例えば1:12.5.If、楽器使用可能な適切な分割比率を適用、残りの検体から列をオフに実行各サンプルの後ヘリウムでバック フラッシュを適用します。
- GC-MS 法にロック SIM 取得にシフト苦しむされず、再現可能な検体保存期間保存期間を適用します。
- 選択したイオン監視 (SIM) 計測器のモードを使用して GC/英-MS による脂肪酸の分子イオンの13C の定款を決定します。SIM モードでの操作は、フルスキャン モードの相対感度の向上と特定検体を検出することができます。
- 何が存在する最初の初期スキャンを実行し、適切なイオンの SIM。興味の分子の固まりでデータを取得するには、それぞれの脂肪酸のクロマト グラフのピーク時間を取囲む m/z スキャン ウィンドウ (SIM グループ) を選択します。通常、2 ~ 4 時間となり, ウィンドウごとイオンをモニターします。
- 、感度を高めるために大量のスキャン レートを調整し、(時間ごとの質量を見て) 回住みます。最低の可能な速度で最高品質のデータが得られたと、SIM 内のルールが試料ピーク 8 に 12 のスキャンを一般的に。機器の設定のプロキシは 9 ms の質量あたり 6 のサイクル タイムの平均滞留時間と 175 ms cyle-1のスキャン時間の s。
- 分子イオン (M+) のそれぞれの脂肪酸組成とそのすべての isotopologues (M+1、+2 M など) を検出します。例については、代表の結果を参照してください。
- 各イオン フラグメント (isotopologue) の豊かさを記録します。分子イオンとその isotopologues の豊かさは比較的低いと定量化の品質は MS システムの性能に大きく依存します。大規模なサンプル シーケンス (実験) を開始する前に曲を実行し、必要に応じてイオン源をクリーニングします。
注意: まず、これらのデータになる全体的な13C 強化消費の前駆体 (ここ 16:0) による各脂肪酸の。 - 標識脂肪酸回答マークされていない相手からの同位体組成の割合を使用して、食物連鎖の炭素フラックスを割り当てます。それぞれの脂肪酸の標識炭素 (atom %、atom %) の割合を計算するのにステップ 6.1 で atom % 数式を使用します。13C 標識 (日 1 およびそれ以降) とラベルのない動物 (0 日) トビムシの脂肪酸の割合を比較すると、消費者に食事から13C の流れの相対的な徴候として。
- 13C 混入脂肪酸鎖の位置を割り当てます。分布に基づく、isotopologues 収拾全体のマークされたトレーサー脂肪酸 (ここ 16:0) の食物ルーティング鎖伸長から脂質代謝によって。全体のマーカー分子の同化増加親イオン (+M)、例えばに最も遠い isotopologues の豊富な M+15、M+16、 13C の使用によって鎖の伸長とラベル C2フラグメント (13C アセチル coa) 分子イオンに近い isotopologues をより頻繁に得る。
6. 13C 濃縮の計算
- Isotopologues 分布によると次の関係を使用してそれぞれの脂肪酸 (atom %) でラベル付けされた炭素の全体的な割合を計算: 原子 % = (組み込まれる比13C isotopologue) × (周波数それぞれ isotopologue)
これは Kuppardtら後に計算されます。31として: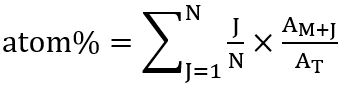
Nは、脂肪酸の炭素原子の数Jは13C 同位体の数、 M + Jはそれぞれ isotopologue とTすべて isotopologues の合計豊富な豊かさ。 - 計算、合計それぞれ FA とすべて isotopologues (M+1、+2、M など)、分子イオン (M+) のピーク面積値 SIM MS 分析によって検出および相対的な豊富を 100% に設定します。各検出された isotopologue の部分は簡単に次の 3 つのルールを計算します。
- 実行される外部13C 標識にだけさかのぼる最後のデータを取得する実験値から非標識日 0 コントロール値 (背景自然13C) を差し引きます。
結果
トビムシの新鮮な重量と脂質含量
記述されている実験の過程で NLFAs および PLFAs の内容変わりませんでした大幅時間をかけて大幅24が、供試体の重量がわずかに増加したに対し。両方のパラメーターは、トビムシ標本の体力の良いレベルを示します。脂肪酸組成と同位体分析のためのサンプリング日に対応する実験中のトビムシの新鮮...
ディスカッション
Isotopologue のプロファイル
FAs 13C 分布の定量的側面の詳細な分析では、炭素の食物網でのパーティション分割を割り当てるには最先端の技術を必要があります。現在の仕事は isotopologue 13C を評価するためにプロファイリングを採用/熱帯の相互作用のための共通の FA バイオ マーカー12C 比。このメソッドでは、液体クロマトグラフィー (...
謝辞
R ・ メンツェルとドイツ研究振興協会 (RU RU780/11-1) によって L. ・ ルッスの財政支援は、感謝します。R. Nehring RU 780/10-1 によって資金を供給されました。最後に、原稿の校正のため博士ハシバミ Ruvimbo Maboreke に非常に感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| neoLab-Round jars | neoLab | 2-1506 | 69 x 40 mm, 10 pacs/pack |
| Charcoal activated | Carl Roth | X865.1 | p.a., powder, CAS No. 7440-44-0 |
| Alabaster Dental | RÖHRICH-GIPSE | --- | http://www.roehrich-gipse.de/dentalgipse.php |
| Chloroform | Carl Roth | 7331.1 | HPLC ≥ 99,9 % |
| Methanol | Carl Roth | P717.1 | HPLC ≥ 99,9 % |
| Hexan | Carl Roth | 7339.1 | HPLC ≥ 98 % |
| tert-Butyl methyl ether (MTBE) | Carl Roth | T175.1 | HPLC ≥ 99,5 % |
| Aceton | Carl Roth | 7328.2 | HPLC ≥ 99,9 % |
| NaOH | Carl Roth | 6771.1 | p.a. ≥99 %, in pellets |
| di-Natriumhydrogenphosphat | Carl Roth | P030.1 | p.a. ≥99 % , water free |
| Na-dihydrogenphosphat Dihydrat | Carl Roth | T879.1 | p.a. ≥99 % |
| Hypochloric acid (6 N) | VWR International | 26,115,000 | AVS TITRINORM vol. solution |
| Bond Elut (Columns) | Agilent Tech. | 14102037 | HF Bond Elut-SI, 500 mg, 3 mL, 50/PK |
| Präparatengläser Duran | Glasgerätebau Ochs | 135215 | Ø 16 x 100 mm, plus screw cap with handy knurl and integrated PTFE/silicone gasket |
| Supelco 37 Component FAME Mix | Sigma-Aldrich | 47885-U Supelco | 10 mg/mL in methylene chloride, analytical standard |
| FlowMesh | Carl Roth | 2796.1 | Polypropylene mesh, approximately 0.3 mm thick, with 1 mm strand spacing |
| Bacterial Acid Methyl Ester (BAME) Mix | Sigma-Aldrich | 47080-U Supelco | 10 mg/mL in methyl caproate, analytical standard |
| Methyl nonadecanoate | Sigma-Aldrich | 74208 | analytical standard ≥ 98.0 % |
| Hexadecanoic acid-1-13C (Palmitic) | Larodan Fine Chemicals | 78-1600 | GC ≥ 98.0 % (13C: 99.0 %) |
| RVC 2-25 CDplus | Martin Christ Gefrier-trocknungsanlagen | Compact benchtop midi concentrator | |
| Alpha 2-4 LDplus | Martin Christ Gefrier-trocknungsanlagen | Drying manifold | |
| MZ 2C NT | Vacuubrand GMBH | Vacuum pump | |
| Roto-Shake Genie | Scientific Industries | Combined rocking and rotating device | |
| XP64 Micro Comparator | Mettler Toledo | Super high precision balance | |
| GC-System 7890A | Agilent Tech. | Gas chromatograph | |
| 7000 GC/MS Triple Quad | Agilent Tech. | Triple Quad mass spectrometer | |
| 7683B Series Injector | Agilent Tech. | Sample injector | |
| Heraeus Multifuge 3SR+ | Thermo Scientific | Centrifuge with 10 ml tube rotor |
参考文献
- Ruess, L., et al. Application of lipid analysis to understand trophic interactions in soil. Ecology. 86 (8), 2075-2082 (2005).
- Ruess, L., et al. Lipid composition of Collembola and their food resources in deciduous forest stands - Implications for feeding strategies. Soil Biology and Biochemistry. 39 (8), 1990-2000 (1990).
- Chamberlain, P. M., Bull, I. D., Black, H. I. J., Ineson, P., Evershed, R. P. Fatty acid composition and change in Collembola fed differing diets: identification of trophic biomarkers. Soil Biology and Biochemistry. 37 (9), 1608-1624 (2005).
- Stott, A. W., Davies, E., Evershed, R. P., Tuross, N. Monitoring the routing of dietary and biosynthesised lipids through compound-specific stable isotope (delta C-13) measurements at natural abundance. Naturwissenschaften. 84 (2), 82-86 (1997).
- Ruess, L., Haggblom, M. M., Langel, R., Scheu, S. Nitrogen isotope ratios and fatty acid composition as indicators of animal diets in belowground systems. Oecologia. 139 (3), 336-346 (2004).
- Pollierer, M. M., Scheu, S., Haubert, D. Taking it to the next level: Trophic transfer of marker fatty acids from basal resource to predators. Soil Biology and Biochemistry. 42 (6), 919-925 (2010).
- Ferlian, O., Scheu, S., Pollierer, M. M. Trophic interactions in centipedes (Chilopoda, Myriapoda) as indicated by fatty acid patterns: Variations with life stage, forest age and season. Soil Biology and Biochemistry. 52, 33-42 (2012).
- Ruess, L., Chamberlain, P. M. The fat that matters: Soil food web analysis using fatty acids and their carbon stable isotope signature. Soil Biology and Biochemistry. 42 (11), 1898-1910 (2010).
- Traugott, M., Kamenova, S., Ruess, L., Seeber, J., Plantegenest, M. Empirically characterising trophic networks: What emerging DNA-based methods, stable isotope and fatty acid analyses can offer. Adv Ecol Res. 49, 177-224 (2013).
- Hammer, B. T., Fogel, M. L., Hoering, T. C. Stable carbon isotope ratios of fatty acids in seagrass and redhead ducks. Chemical Geology. 152 (1-2), 29-41 (1998).
- Budge, S. M., Iverson, S. J., Koopman, H. N. Studying trophic ecology in marine ecosystems using fatty acids: A primer on analysis and interpretation. Marine Mammal Science. 22 (4), 759-801 (2006).
- Haubert, D., et al. Trophic structure and major trophic links in conventional versus organic farming systems as indicated by carbon stable isotope ratios of fatty acids. Oikos. 118 (10), 1579-1589 (2009).
- Ngosong, C., Raupp, J., Richnow, H. H., Ruess, L. Tracking Collembola feeding strategies by the natural 13C signal of fatty acids in an arable soil with different fertilizer regimes. Pedobiologia. 54 (4), 225-233 (2011).
- Bec, A., et al. Assessing the reliability of fatty acid-specific stable isotope analysis for trophic studies. Methods in Ecology and Evolution. 2 (6), 651-659 (2011).
- Gladyshev, M. I., Makhutova, O. N., Kravchuk, E. S., Anishchenko, O. V., Sushchik, N. N. Stable isotope fractionation of fatty acids of Daphnia fed laboratory cultures of microalgae. Limnologica. 56 (Supplement C. 56 (Supplement C), 23-29 (2016).
- Gladyshev, M. I., Sushchik, N. N., Kalachova, G. S., Makhutova, O. N. Stable isotope composition of fatty acids in organisms of different trophic levels in the Yenisei river. PLoS One. 7 (3), e34059 (2012).
- Eisenreich, W., Dandekar, T., Heesemann, J., Goebel, W. Carbon metabolism of intracellular bacterial pathogens and possible links to virulence. Nature Reviews Microbiology. 8 (6), 401-412 (2010).
- Eylert, E., Bacher, A., Eisenreich, W. NMR-based isotopologue profiling of microbial carotenoids. Methods Mol Biol. 892, 315-333 (2012).
- Garton, N. J., O'Hare, H. M. Tuberculosis: feeding the enemy. Chemical Biology. 20 (8), 971-972 (2013).
- Rosenblatt, J., Chinkes, D., Wolfe, M., Wolfe, R. R. Stable isotope tracer analysis by GC-MS, including quantification of isotopomer effects. Am J Physiol. 263 (3), E584-E596 (1992).
- Fernandez, C. A., Des Rosiers, C., Previs, S. F., David, F., Brunengraber, H. Correction of 13C mass isotopomer distributions for natural stable isotope abundance. J Mass Spectrom. 31 (3), 255-262 (1996).
- Heuner, K., Eisenreich, W. The intracellular metabolism of legionella by isotopologue profiling. Methods Mol Biol. 954, 163-181 (2013).
- Willenborg, J., et al. Characterization of the pivotal carbon metabolism of Streptococcus suis serotype 2 under ex vivo and chemically defined in vitro conditions by isotopologue profiling. J Biol Chem. 290 (9), 5840-5854 (2015).
- Menzel, R., Ngosong, C., Ruess, L. Isotopologue profiling enables insights into dietary routing and metabolism of trophic biomarker fatty acids. Chemoecology. 27 (3), 101-114 (2017).
- Buse, T., Ruess, L., Filser, J. New trophic biomarkers for Collembola reared on algal diets. Pedobiologia. 56 (3), 153-159 (2013).
- Hutson, B. R. Effects of variations of the plaster-charcoal culture method on a Collembolan, Folsomia candida. Pedobiologia. 18, 138-144 (1978).
- Fountain, M. T., Hopkin, S. P. Folsomia candida (Collembola): a "standard" soil arthropod. Annu Rev Entomol. 50, 201-222 (2005).
- ISO, I. O. f. S. . Soil Quality-Inhibition of reproduction of Collembola (Folsomia candida) by soil pollutants. , (1999).
- Welch, D. F. Applications of cellular fatty acid analysis. Clin Microbiol Rev. 4 (4), 422-438 (1991).
- Dodds, E. D., McCoy, M. R., Rea, L. D., Kennish, J. M. Gas chromatographic quantification of fatty acid methyl esters: flame ionization detection vs. electron impact mass spectrometry. Lipids. 40 (4), 419-428 (2005).
- Kuppardt, S., Chatzinotas, A., Kastner, M. Development of a fatty acid and RNA stable isotope probing-based method for tracking protist grazing on bacteria in wastewater. Appl Environ Microbiol. 76 (24), 8222-8230 (2010).
- Zhang, X., He, H., Amelung, W. A GC/MS method for the assessment of 15N and 13C incorporation into soil amino acid enantiomers. Soil Biology and Biochemistry. 39 (11), 2785-2796 (2007).
- Vetter, W., Thurnhofer, S. Analysis of fatty acids by mass spectrometry in the selected ion monitoring mode. Lipid Technol. 19 (8), 184-186 (2007).
- Thurnhofer, S., Vetter, W. A gas chromatography/electron ionization-mass spectrometry-selected ion monitoring method for determining the fatty acid pattern in food after formation of fatty acid methyl esters. J Agric Food Chem. 53 (23), 8896-8903 (2005).
- Haubert, D., Haggblom, M. M., Scheu, S., Ruess, L. Effects of fungal food quality and starvation on the fatty acid composition of Protaphorura fimata (Collembola). Comparative Biochemistry and Physiology B-Biochemistry & Molecular Biology. 138 (1), 41-52 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved