Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Fettsäure- 13C Isotopologue Profilierung gibt Einblick in trophische Carbon Transfer und Fettstoffwechsel der Wirbellosen Verbraucher
In diesem Artikel
Zusammenfassung
Die Fettsäure trophische Marker Ansatz, d. h. die Aufnahme von Fettsäuren als gesamte Molekül und Transfer in das Gewebe der Verbraucher ohne oder geringfügige Änderung, durch Wissenslücken im Fettsäure-Stoffwechsel der kleinen Boden Wirbellosen behindert wird. Isotopologue Profilierung dient als ein wertvolles Instrument zur trophische Interaktionen zu entwirren.
Zusammenfassung
Fettsäuren (FAs) sind nützliche Biomarker im Nahrungsnetz Ökologie, denn sie sind in der Regel als vollständige Molekül assimiliert und in Verbraucher-Gewebe mit Minderjährigen oder keine Änderung, so dass die diätetische routing zwischen verschiedenen trophischen Ebenen übertragen. Der FA-trophische Marker-Ansatz ist allerdings immer noch durch die begrenzte Kenntnisse in Lipidstoffwechsel von der Bodenfauna behindert. Diese Studie verwendete völlig gekennzeichneten Palmitinsäure (13C16:0, 99 % der Atom) als Tracer in Fettsäure-Stoffwechsel Bahnen von zwei weit verbreitete Boden Collembolen, Protaphorura Fimata und Heteromurus Redwood. Um das Schicksal und die metabolischen Veränderungen dieser Vorstufe zu untersuchen, ist eine Methode des Isotopologue profiling vorgestellt, durch Massenspektrometrie mit dem einzelnen Ion monitoring durchgeführt. Darüber hinaus wird die vorgelagerten Labor Fütterungsversuch sowie die Extraktion und die Methylierung von dominanten Lipid Brüche (neutralen Lipiden, Phospholipiden) und die zugehörige Formel und Berechnungen beschrieben. Nicht nur Ausbeute 13C beschriftet Vorläufer die insgesamt 13C-Anreicherung in Fettsäuren abgeleitet sondern produziert auch das Muster der Isotopologues überschreiten die Masse des Ions (d. h. das FA molekularen Ion Elternteil Isotopologue Profilierung M+) jeweils mit der Bezeichnung FA durch eine oder mehrere Masseneinheiten (M+ 1M+ 2M+ 3, etc.). Dieses Wissen erlaubt Rückschlüsse auf das Verhältnis der diätetischen routing von einem völlig verbrauchte FA im Vergleich zu de-Novo -Biosynthese. Die Isotopologue Profilierung empfiehlt sich als ein nützliches Instrument zur Bewertung der Fettsäure-Stoffwechsel in Bodentiere trophische Interaktionen zu entwirren.
Einleitung
In einem kryptischen Lebensraum wie Boden trophische Beziehungen sind schwer zu Adresse und sind durch die geringe Größe der Fauna weiter beschränkt. Im letzte Jahrzehnt hat Fortschritte in biochemische Ökologie, insbesondere bei der Verwendung von Fettsäuren als Biomarker für die Definition von Fütter Strategien der Bodenfauna unter Feld Bedingungen1,2,3gesehen. Dies ist, dass Fettsäuren aus Ressourcen in Verbraucher-Gewebe als gesamte Moleküle eingebaut werden können, bezeichnet ein Prozess diätetische routing4. Übertragung von Fettsäuren ist über drei trophischen Ebenen, d. h. aus Pilzen, Nematoden, Collembolen5berichtet worden. Vor kurzem, die räuberischen Fauna galt6,7 und die ersten Reviews auf Fettsäuren als trophische Markierungen im Boden Nahrungsnetze wurden veröffentlicht8,9.
Nähere Informationen zu trophischen Interaktionen wird durch Fettsäure-stabiler Isotope probing (FA-SIP) erreicht. Die Bestimmung von 13C /12C-Verhältnisse in Fettsäuren in Diäten und Verbraucher können zuschreiben binäre Verknüpfungen und Abschätzung den damit verbundenen CO2-Fluss und beschäftigt in terrestrischen, Süßwasser und marinen Nahrungsnetze10,11 ,12,13. Die Grundannahme ist, dass weitergeleitete diätetischen Fettsäuren nicht enzymatischen Prozessen unterworfen sind; daher ihre 13C signal, d. h. 13C /12C Verhältnis der Fettsäure in der Konsumgüterindustrie ist ähnlich wie in der Diät-1. Jedoch wurde eine allmähliche Entleerung der 13C Signatur oben in der Nahrungskette in aquatischen Systemen, dadurch behindern breite Anwendung des FA-SIP trophische Studien14,15,16berichtet. Kenntnisse in den Lipidstoffwechsel in den meisten Wirbellosen in terrestrischen Nahrungsketten ist übrigens nach wie vor begrenzt.
Das Verständnis der Lipid Metabolismus Bahnen bei den Verbrauchern ist Voraussetzung für die Nutzung der trophischen Marker Fettsäuren als Mittel zur Bestimmung der quantitativen Kohlenstoff Strömung im Nahrungsnetz Ökologie. Mit diesem im Verstand, 13C-Isotopologue Profilierung, welche im Prinzip für die Untersuchung von Kohlenstoff-Stoffwechsel jedes biologische System17, angewendet werden kann ist eine vielversprechende Methode. Nach der Einführung des 13C gekennzeichnet Carbon Substrat, die Verteilung der 13C in der metabolischen Netzwerkes ist seit der erzeugten Stoffwechselprodukte in der Consumer-Show eine spezifische Isotopologue Verteilung nachvollziehbar. Dies kann durch quantitative nukleare metabolische Resonanz Spektroskopie18,19 oder Massenspektrometrie20,21, mit der letzteren bevorzugten in biologischen Proben mit niedrigen Biomasse aufgrund seiner höheren beurteilt werden Empfindlichkeit.
Obwohl Isotopologue Profilierung wurde erfolgreich angewendet, Aminosäuren und bot Einblick in den in-Vivo Kohlenstoff-Stoffwechsel der bakteriellen Krankheitserregern17,22,23, deren Umsetzung in Fettsäuren Säuren ist zurückgeblieben. Die erste detaillierte Analyse über das Schicksal von einem stabilen isotopenmarkierter Vorläufer Fettsäure, diätetische routing oder Abbau durch β-Oxidation, Boden Wirbellosen Verbraucher, wurde vor kurzem von Menzel Et Al. durchgeführt. 24. hier werden die methodischen Grundlagen für Einbau Experimente mit 13C gekennzeichnet, gefolgt von Isotopologue Analyse der wichtigsten Nachkommen in häufigen Boden Wirbellosen, die Collembolen Fettsäuren zur Verfügung gestellt. Diese Microarthropods sind ein gutes Modell-Gruppe, da sie wichtige Bestandteile des Bodens Nahrungsnetz und sind gut für ihre trophische Marker Fettsäuren8,25untersucht bilden.
Das Verständnis der Lipid Metabolismus Bahnen bei den Verbrauchern ist Voraussetzung für die Nutzung der trophischen Marker Fettsäuren als Mittel zur Bestimmung der quantitativen Kohlenstoff Strömung im Nahrungsnetz Ökologie. Dieses Protokoll bietet das Design und die Einrichtung für ein Labor Ernährung Experiment und die biochemische Verfahren zur Extraktion und Methylierung von dominanten Lipide Brüche (neutralen Lipiden, Phospholipiden) von Collembolen. Es zeigt, wie die Isotopologue Zusammensetzung der Fettsäuren durch Massenspektrometrie analysiert wird und beschreibt die zugehörige Formel und Berechnungen. Dieses Verfahren führt zu: (i) die Verhältnisse der Isotopologues überschreiten die Masse des Ions (d. h. die Fettsäure Molekulare Ionen M+) Eltern von einem oder mehreren mass-Einheiten (M+ 1M+ 2M+ 3, etc.) und (Ii) die gesamte 13 C-Anreicherung in Fettsäuren abgeleitet aus den 13C gekennzeichneten Vorläufer. Obwohl für Collembolen verwendet, kann dieser Ansatz in der Regel auf andere Räuber-Beute-Interaktion auf der Prämisse angewendet werden, dass diese sind in ausreichender Menge unter kontrollierten Bedingungen zu eine erfolgreichen Label Akzeptanz zu gewährleisten und anschließende Überprüfung.
Protokoll
Das beschriebene Protokoll fällt nicht unter die Zuständigkeit der Tier-Ethik. Jedoch wenn Leute die beschriebenen Protokolle zu höheren Tieren anzupassen, achten Sie darauf, dass institutionelle Tier-Ethik-Kommission das Protokoll für den Umgang mit Tieren genehmigt.
1. Anbau von Tieren
Hinweis: Alle erläutert die experimentellen Schritte basieren auf etablierte Protokolle26,27,28. Biotests im Labor benötigen eine kontinuierliche Versorgung mit leicht kultivierbaren Organismen. Hier wurden die Collembolen Arten Protaphorura Fimata (Gisin, 1952) und Hetermurus Redwood (Templeton, 1835) verwendet. Beide Arten sind einfach zu pflegen als produktives Labor Kulturen gefüttert mit Bäckerhefe.
- In Kunststoff Mikrokosmen mit dicht schließendem Deckel (Durchmesser 7 cm, Höhe 4,5 cm), fügen Sie eine Mischung aus Aktivkohle, Gips, und destilliertes Wasser zu einem sehr feuchten Zucht Substrat (Abbildung 1A).
- Wenn Sie Mikrokosmen Mischung genug Substrat, z. B. für 10 Mikrokosmen in einem Batch vorbereiten. Mix 9 Teile der Gips (225 g) und 1 Teil des trockenen Aktivkohle (25 g) zusammen in einem Topf Gips vorsichtig ca. 10 Teile destilliertes Wasser (250 mL) hinzufügen und für 5 Minuten ohne rühren bei Raumtemperatur stehen lassen.
- Rühren Sie mit einem Labor-Löffel mit mäßiger Geschwindigkeit im Uhrzeigersinn, um Luftblasen zu vermeiden, bis eine Dicke suppenartige Konsistenz erreicht ist. Gießen Sie sofort in Mikrokosmen bis zu einer Höhe von ca. 1 cm.
- Glätten Sie den Putz durch sanftes klopfen auf der Bank und wirbeln. Beachten Sie, dass die Löcher (zufällige Produkte von Luftblasen) und Furchen (aktiv mit einem sterilen Spatel hinzugefügt) fruchtbaren Collembolen zur Eiablage es fördern können. Diese Studie vermieden Löcher und Furchen zu Gunsten der mit den gleichen reproduzierbaren Bedingungen. Abbildung 1 b stellt jedoch zu Demonstrationszwecken einige Löcher.
- Lassen Sie für ca. 1-2 Tage bei Zimmertemperatur trocknen; Inkubation bei 60 ° C kann dieser Zeit auf ca. 1-2 h reduzieren.
- Befeuchten Sie Mikrokosmen vor der Verwendung von Leitungswasser mit einer Pipette hinzu, bis das Substrat leicht feucht ist. Halten Sie Mikrokosmos feucht, indem Sie regelmäßig destilliertes Wasser als Collembolen haben eine weiche Nagelhaut und Austrocknung anfällig ist.
- Übertragen Sie Collembolen leicht auf den Putzgrund mit einer einfachen Saugrohr, d.h., eine lange Silikonschlauch ca. 25 cm lang mit einer PIPETTENSPITZE ausgestattet mit einem kleinen Netz zum Ansaugen von Tieren in das Rohr zu verhindern. Alternativ übertragen Sie Tiere, indem sie sich auf die Borsten einer Bürste haften.
- Übertragen (siehe Schritt 1.2) 30 frisch geschlüpften Collembolen in neue Mikrokosmen und bieten granulierter trocken Backhefe als Nahrung (etwa eine Messerspitze) (Abbildung 1 b); mindestens zweimal pro Woche zu erneuern. Plan drei unabhängige Wiederholungen pro Tag der Probenahme; in dieser Studie Tagen 0 - 7 und 14. Bei 15 ° C im Dunkeln inkubieren. Die Aufrechterhaltung eine Konstante Temperatur notwendig ist, werden als Fettsäuren von tierischen Stoffwechsel der membranfluidität Anforderungen verändert.
- Feed Collembolen mit Bäckerhefe für vier Wochen vor Beginn der Exposition Experimente zu einem homogenen 13C /12C Signal und Muster in den Fettsäuren. Ein Saugrohr oder einem Pinsel verwenden, um alle Eizellen (Abbildung 1), fäkale Pellets zu entfernen und Exuviae regelmäßig als Tiere können fressen sie damit eine Veränderung ihrer Lipid-Profil.

Abbildung 1: Anbau von Collembolen. (A) Mikrokosmos mit Zucht Substrat, eine getrocknete Mischung aus Gips, Aktivkohle und destilliertem Wasser gefüllt. (B) und (C) repräsentative Probe einer Protaphorura Fimata Kultur; Beachten Sie die kleinen Nuggets von trockenen Bäckerhefe verwendet als die Nahrungsquelle sowie auch Löcher in die Zucht-Substrat (schwarzer Pfeil) (B) sowie zwei Eiern (weißer Pfeil) (C). Bitte klicken Sie hier für eine größere Version dieser Figur.
(2) Kennzeichnung, Ernährung, Ernte und Probenbehandlung
- Kennzeichnung
- Vier Wochen nach der Gründung der Collembolen Kulturen, 30 Personen in neuen Mikrokosmen und bei 15 ° C im Dunkeln inkubieren.
- Einführen der Puls-Label durch Fütterung mit Bäckerhefe, enthält eine völlig 13C beschriftet Palmitinsäure (13C16:0, 99 % der Atom) durch das Mischen von 13C beschriftet Palmitinsäure mit Bäckerhefe mit einem Spatel in einem Verhältnis 0.5:1, z. B.5 g 13 C16:0 und 10 g Trockenhefe des Bäckers. Legen Sie über eine Messerspitze auf jeden Mikrokosmos.
- Ersetzen Sie nach 6 h das Essen mit vollkommen unbeschrifteten Bäckerhefe beschriftet.
-
Weitere Kultivierung
- Im Verlauf des Experiments erneuern Sie die Hefe Diät alle drei Tage zu, und fügen Sie Beträge höher als das, was innerhalb dieses Zeitraums Collembolen verbrauchen wird. Verwenden Sie am wichtigsten ist, ein Saugrohr oder Bürste, um Collembolan Eiern, fäkale Pellets und Exuviae regelmäßig, um sicherzustellen, ausschließliche Fütterung von den Tieren auf dem bereitgestellten Lebensmittel zu entfernen.
- Ernte
- Probieren Sie Mikrokosmen destruktiv täglich bis Tag 7. Sammeln Sie dann am 14. Tag, drei unabhängige Wiederholungen zu jedem Zeitpunkt der Probenahme; verschiedenen Abtastzeiten sind möglich.
- Bereiten Sie 10 mL Glasröhrchen mit Teflon beschichtet Schraubverschlüsse, eine für jede Probe ausgestattet. Vorher reinigen Sie diese Rohre in einer Glas-Scheibe und anschließend zweimal mit entionisiertem Wasser spülen. Schließlich, alle Spuren von hydrophoben entfernen Schadstoffe waschen zweimal durch Zugabe von 2 mL Chloroform (HPLC-Klasse), Wirbel etwa und das Lösungsmittel zu verwerfen.
- Nehmen Sie für Kontrollproben (Tag 0) 3-30 nicht exponierten Collembolen aus der Pre-Kulturen als Tag 0 Proben.
- Für exponierte Beispiele (Tag 1 und ab), nehmen Sie für die tägliche Proben jeweils 3 30 Collembolen aus den Kulturen Klosterturm ausgesetzt gefüttert mit der Mischung aus 13C beschriftet Palmitinsäure und Bäckerhefe.
- Rekord Collembolen Frischgewicht durch eine Ultra-Mikrowaage. Tiere mit einer Absaugung oder Pinsel, um entsprechende Skala-Pfannen zu übertragen. Einfache Handhabung der Collembolen beim Wiegen in der Skala-Pfannen, Schock-Cool Tiere vor (-80 ° C 2 h) zu gewährleisten. Alternativ kann ein CO-2 -Stream für 10 min sicher Tiere betäuben.
- Direkt nach dem wiegen setzen Tiere von den Skala-Pfannen sorgfältig in 10 mL Glas Reagenzgläser. Füllen Sie die Röhrchen mit 1 mL Methanol (HPLC-Klasse) und Store bei-20 ° C bis zur Analyse.
Hinweis: Von diesem Schritt ab, vermeiden Sie Probe Umgang mit Kunststoff Ausrüstung wie organische Lösungsmitteln beteiligt sind; Stattdessen verwenden Sie Spender und Pipetten, die Lösungsmittel sowie Glasgefäße geeignet sind.
(3) Lipid-Extraktion aus tierischem Gewebe und Methanolysis
- Bereiten Sie drei Test-Glasröhren (ausgestattet mit Teflon beschichtet Schraubverschlüsse) pro Charge mit nur 1 mL Methanol als leere Werte. Wichtig ist, hinzufügen oder alle Lösungsmittel nur von glaspipetten oder Chloroform/Methanol gespült lösemittelbeständig Spender in diesem Protokoll verwendet.
- Reduzieren Sie zu Beginn der Lipid-Extraktion das Methanol beantragt Speicher (oder ausblenden) durch Verdampfung; ein kompaktes Benchtop Rotations Vakuum Konzentrator (RVC) ausgestattet mit einer Vakuum-Pumpe und eine Kühlfalle wird empfohlen. Übertragen Sie die offenen Rohre in der RVC und Verdampfen bei 50 ° C und Vakuum Druck von 200 hPa für 20 min trocknen.
- Jede Probe (einschließlich Leerzeichen) 5 mL des einphasigen Extraktion Lösungsmittel (chloroform/methanol/0.05 M Phosphat-Puffer 1:2:0.8, pH 7,4) hinzu und Collembolan Fette bei Raumtemperatur durch Schütteln über Nacht zu extrahieren (~ 200 u/min).
- Das Lösungsmittel auf neue Röhren übertragen und erneut Proben durch Schütteln für 3 h mit einer zusätzlichen 2,5 mL Lösungsmittel Extraktion zu extrahieren. Danach verbinden Sie Auszüge aus beiden Schritten; die Verwendung von Glas, die Pasteur Pipetten wird empfohlen. 0,8 mL Chloroform und 0,8 mL destilliertem Wasser, dann mischen und bei 2.000 g bei 20 ° C für 5 min zentrifugieren. Abschließend Proben stehen für 5 min für die Trennung von wässrigen und chloroform Phasen.
- Für die Fettsäure-Muster Analyse Teilen der gesamten zellulären Lipide der Collembolen in neutral Lipid, Glycolipid und Phospholipid-Brüche.
- Für jede Probe vorbereiten eine Kieselsäure sauren Spalte (kommerzielle Spalte mit 0,5 g Kieselsäure, Mesh Größe 100-200 µm, siehe Tabelle der Materialien) durch Zugabe von 1 mL Chloroform (Vorkonditionierung). Um diesen Vorgang zu beschleunigen, montieren Sie die Spalten auf einen Vakuum-Block wie gewöhnlich in Chromatographie für Festphasenextraktion. Verwenden Sie keine Nylon Nadeln; Edelstahl über die Rohre zu verwenden.
- Nach dem Chloroform zur Vorkonditionierung hat die komplette untere Chloroform Phase jeder Probe durch die Spalte Übertragung einer einzelnen Spalte übergeben. Um dieses Verfahren zu vereinfachen, kann die oberen wässrige Phase vorher entfernt werden. Pasteur Pipetten aus Glas, aber benutzen, achten Sie darauf, dass die Spalten nicht austrocknen.
- Sukzessive eluieren Sie Lipid Brüche mit 5 mL Chloroform (einschließlich neutral Lipid Fettsäuren, NLFAs), 10 mL Aceton (Glycolipide - nicht in dieses Projekt analysiert) und 5 mL Methanol (einschließlich Phospholipid-Fettsäuren, PLFAs). Sammeln Sie jede Fraktion in einzelnen Glasgefäße.
- Am Ende der Extraktion reduzieren Sie Chloroform (NLFAs) und Methanol (PLFAs) über Verdunstung in eine RVC. Die offene Rohre auf der RVC übertragen und Verdampfen bis trocken, ~ 90 min bei 60 ° C und ein Vakuum von 24 hPa.
- Starten Sie Verseifung von Lipiden (NEAT und PLFA Fraktionen) nach dem Protokoll von Welch (1991)29 mit Zugabe von 1 mL Natriumhydroxid-Methanol-Lösung (45 g Natriumhydroxid, 150 mL Methanol und 150 mL destilliertem Wasser) zu und inkubieren Sie bei 100 ° C für 30 min in einem Wasserbad. Abkühlen der Probe im eisigen Wasser für 2 min, dann setzen Sie die Proben wieder auf der Bank und bei Raumtemperatur weiterarbeiten.
- Jede Probe, einschließlich der Leerzeichen fügen Sie des internen Standards hinzu. Wählen Sie eine Fettsäure in der experimentellen Organismen nicht üblich; auch eine gesättigte Fettsäure Verlust zu minimieren, durch Spaltung, und wählen ein Molekül mit mittlerer Kettenlänge. Für viele Zwecke, die ungeraden Nonadecanoic Säure (19:0) funktioniert gut. So fügen Sie 30 µL einer Lösung von 0,74 mM in isooktan. Achten Sie die genaue Menge ist sehr wichtig - darauf, die Genauigkeit Ihrer Pipette mit einer Mikrowaage vorab eingecheckt haben.
- Fügen Sie 2 mL Salzsäure-Methanol (Mix 325 mL 6,0 N Salzsäure mit 275 mL Methanol), bei 80 ° C für 10 Minuten in ein Wasserbad und kühlen schnell auf Eis für 2 min inkubieren. Dieser Schritt ist Zeit und temperaturempfindlich. Verwenden Sie 80 ± 1 ° C und 10 ± 1 min. Check mit wie viele Proben in das Wasserbad gehen können gleichzeitig um die 80 ° c zu halten
Hinweis: Dieses Verfahren führt zu Fettsäuren Methylester (Kamera), d. h. Fettsäure Analyten für Verdampfung Gaschromatographie stabilisiert. - Zu guter Letzt fügen Sie 1,25 mL Hexan/METHYLTErTiäBUTYLäTHEr (1:1) und Rock für 10 min, dann Zentrifuge bei 2.000 g für 5 min. sanft zu entfernen, die untere Phase und halten Sie die obere Phase von FAMEs enthalten; Verwenden Sie Glas, die Pasteur Pipetten. Hinzugeben Sie 3 mL wässriger Natriumhydroxid (10,8 g NaOH in 900 mL destilliertem Wasser aufgelöst) für ein Waschschritt.
- Rock und Zentrifuge wieder.
- Nehmen Sie die obere Lipid-haltigen Phase beendet mit einem Glas Pasteurpipette und Transfer zu einem Gaschromatographie Probe-Fläschchen mit einem Teflon-Septum ausgestattet. Vermeiden Sie auch kleine Mengen von der wässrigen Phase, da dies zu Problemen mit der GC-Messung wird. Fläschchen zu kapseln und bis zur Analyse bei-20 ° C lagern.
(4) Quantifizierung der Fettsäuren durch GC-FID
- Verwenden Sie Gaschromatographie identifizieren und quantifizieren die Berühmtheiten in den NFLA und PLFA Fraktionen von Lipiden Collembolen (Tier). Ein Gaschromatograph (GC) ausgestattet mit Ionisation Flammenwächter (FID) ist eine etablierte und bewährte Ausrüstung30.
- Vergleichen Sie für die Identifikation von FAMEs Retentionszeit von Gipfeln in der Probe, die in einem Standard-Ruhm. Das sind qualitative oder quantitative Standardmischung Key Kamera für eine Vielzahl von Lebensmitteln und Organismen.
- Beschäftigen Sie FAME standard Mischungen bestehend aus Vertreter FAs des untersuchten Organismus Konzerns und experimentelle Ernährung. In Collembolen handelt es sich um Fettsäuren charakteristisch für Eukaryoten, z.B., langkettige mehrfach ungesättigte Fettsäuren wie Arachidonsäure (20:4ω6). Beim Einsatz von Hefe als Diät handelt es sich um Pilzbefall Markierungen wie Linolsäure (18:2ω6). Eine gute Wahl ist der so genannte FAME Mix, bestehend aus 37 verschiedenen Fettsäuren häufig in Tier, Pilz und Pflanzenmaterial und die bakterielle Acid Methylester (BAME) mischen (siehe Tabelle der Materialien).
- Zum Ausführen der Beispiele auf der GC-FID, richten Sie eine Sequenz mit der entsprechenden Software des Gerätes. Anweisungen finden Sie im Handbuch des Herstellers. Die Sequenz beginnt mit der externen standard Mischungen (z.B. Ruhm und BAME Mix), gefolgt von den Proben. Beachten Sie, dass es eine Retention Time-Shift, d. h. eine leichte Verzögerung rechtzeitig Elution von Fettsäuren aus dem GC-Säulen mit jedem Lauf, während der Ausführung von Proben! Entweder enthalten einen Standard jeder 10th laufen nacheinander Probe oder Retentionszeit Sperren für Palmitinsäure (16:0).
- GC-Einstellungen abhängig von dem Instrument anzupassen. Das folgende Programm ist für eine hohe Leistung (HP) Kapillare Spalte vorgeschlagen (25 m x 0,2 mm i.d., film Dicke 0,33 µm. Set Injektionsvolumen, 1 µL splitless Modus und Wasserstoff als Trägergas verwenden. Beschäftigen Sie einen Temperatur-Programm Anfang bei 50 ° C (statt für 1 min) und Erhöhung um 25 ° C min-1 bis 175 ° C, gefolgt von 3 ° C min-1 bis 230 ° C (statt 5,7 min).
- Berechnen Sie die Fettsäure Nmol pro Gramm Frischgewicht (trocken) von Organismen mit der Reaktion von FID, für jeden Ruhm Anwendung der bekannten Beträge für die jeweiligen Fettsäure mithilfe der folgenden Formel:
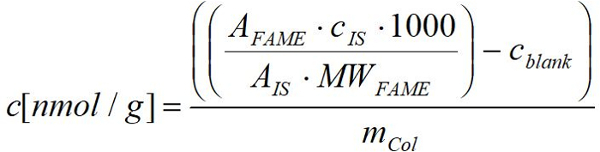
EineBerühmtheit: Peakfläche der jeweiligen Ruhm in der Probe
MWFM: Molekulargewicht des jeweiligen FAME in µg/µmol
CIS: Konzentration des internen Standards in µg
EineIS: Peakfläche des internen Standards
mCol: frische (Trockengewicht) der jeweiligen Collembolen Probe in g
1000: Umrechnungsfaktor von µmol, Nmol
CBW: Konzentration der jeweiligen Ruhm in den Mittelwert der entsprechenden leeren Werte in Nmol
5. 13C Analyse durch Isotopologue Profilierung
- Verwenden Sie ein GC-System gekoppelt, eine Masse selektive Detektor (MS) mit einer Elektronenquelle Ionisation (EI) zur Bestimmung der Isotopologue geliefert.
- Verwenden Sie eine polar Kapillare Spalte (z. B. DB 23, CP-Sil 88), da dies weitere die Trennung von ungesättigten Fettsäuren sogar mit der gleichen Anzahl von Doppelbindungen ermöglicht. Die Wahl des GC-Säulen ist entscheidend für die Ergebnisse, da es die gute Darstellung der Molekül-Ion in der Fettsäuren bestimmt.
- Für eine DB 23-Spalte (60 m x 0,25 mm i.d., Film Dicke 0,15 µm) und starten Sie Ofentemperatur bei 130 ° C erhöhen, indem Sie 6,5 ° C/min auf 170 ° C. Folgen Sie mit einem Plus von 3 ° C/min bis 203 ° C und halten für 1,9 min. folgen mit einem Zuwachs von 40 ° C/min bis 230 ° C und halten Sie für 8,3 min. Set Transfer Temperatur bis 280 ° C. GC-Methode, um das Instrument wieder einstellen
- Verwenden Sie quantitative Standards bestehend aus bekannten Mengen von FAMEs für alle Fettsäuren für 13C Aufnahme untersucht werden. Setzen Sie diese Standards am Anfang und Ende jeder Probe Sequenz ausgeführt werden. Nehmen Sie die Retentionszeiten der Fettsäuren von Interesse von diesen Normen.
- Proben aus den Experimenten immer beginnend mit der unbeschrifteten Proben und die markierten Sonden danach zu messen. Gelten Sie eine Split-Verhältnis angebracht, die Probe Konzentrationen, z.B. 1:12.5.If für das Instrument zur Verfügung, eine Rückspülung mit Helium nach jeder Probe laufen, um ab Spalte vom restlichen Analyten zu löschen.
- Gelten Sie Verweilzeit auf der GC-MS-Methode sperren, so dass der SIM Erwerb nicht verschieben leidet und es reproduzierbare Analyten Retentionszeiten gibt.
- 13C Einbau in der molekularen Ionen von Fettsäuren durch die GC/EI-MS mit der ausgewählten Ion monitoring (SIM)-Modus des Gerätes zu bestimmen. Betrieb im SIM-Modus ermöglicht, um bestimmte Analyten mit erhöhter Empfindlichkeit gegenüber full-Scan-Modus zu erkennen.
- Führen Sie eine erste Überprüfung zuerst zu sehen, was vorhanden ist, und führen Sie dann SIM auf entsprechenden Ionen. Erfassen Sie Daten an molekularen Massen von Interesse durch die Auswahl von m/Z-Scan-Fenster (SIM-Gruppen) umfasst die chromatographische Spitzenzeit der jeweiligen Fettsäure. In der Regel zwei bis vier Ionen pro Analyt und Zeit Fenster zu überwachen.
- Um die Empfindlichkeit zu erhöhen, stellen Sie die Masse Abtastrate und Zeiten (die Zeit, Blick auf jeder Messe) wohnen. Die beste Qualitätsdaten werden mit der niedrigsten Geschwindigkeit, gewonnen und eine Regel in SIM ist in der Regel 8 bis 12 Scans über den Analyten Höhepunkt. Ein Proxy für Geräteeinstellungen ist eine durchschnittliche Verweilzeit pro Masse von 9 ms, eine Taktzeit von 6 s und eine Scanzeit von 175 ms Cycle-1.
- Erkennen des molekularen ions (M+) von der jeweiligen Fettsäure und seine Isotopologues (M+ 1, M+ 2 und So weiter). Zum Beispiel sehen Sie Vertreter Ergebnisse.
- Notieren Sie die Fülle der einzelnen Ionen-Fragment (Isotopologue). Beachten Sie, dass die Fülle der Molekül-Ion und seine Isotopologues relativ gering ist und die Qualität der Quantifizierung stark von der Leistung der MS-Systems hängt. Führen Sie eine Melodie vor Beginn einer großen Stichprobe Sequenz (Experiment) und reinigen Sie die Ionenquelle ggf..
Hinweis: Erstens Ertragsdaten dieser insgesamt 13C Bereicherungen der einzelnen Fettsäuren durch die verbrauchten Vorläufer (hier 16:0). - Verwenden Sie den Anteil der Isotopenzusammensetzung von markierten Fettsäuren aus unmarkierten Gegenstücke, die trophischen kohlenstofffluss zuzuweisen. Verwenden Sie die Atom % Formel in Schritt 6.1 zur Berechnung des Anteils der markierten Kohlenstoff (Prozent Atom, Atom %) in der jeweiligen Fettsäure. Vergleichen Sie den Prozentsatz von 13C in Fettsäuren der Collembolen zwischen gekennzeichnet (Tag 1 und höher) und unbeschriftete Tiere (Tag 0) als relative Indikation für den Fluss von 13C aus Nahrung in Verbraucher.
- Ordnen Sie die Position des 13C Eingliederung in die Fettsäure-Kette. Basierend auf der Verteilung von den Isotopologues zu entwirren das diätetische routing der gesamte markierte Tracer Fettsäure (hier 16:0) von Längung der Kette durch Lipidstoffwechsel. Während Assimilation des gesamten Marker Moleküls die Fülle der Isotopologues am weitesten an das übergeordnete Ion (M+), z. B. erhöhtgekennzeichnet M+ 15, M+ 16, mit Kette Dehnung durch den Einsatz von 13C C2 Fragmente (13C Acetyl-CoA) die Isotopologues in der Nähe der molekularen Ion bekommen häufiger.
6. Berechnung der Anreicherung von 13C
- Entsprechend der Verteilung der Isotopologues, der Gesamtanteil der markierten Kohlenstoff (in Atom %) in die jeweiligen Fettsäure unter Zugrundelegung der folgenden Beziehung berechnen: Atom % (Verhältnis 13C Isotopologue integriert) = x (Frequenz jeweiligen Isotopologue)
Dieser berechnet sich nach Kuppardt Et al. 31 als: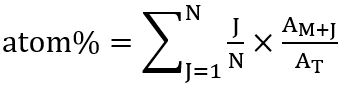
wobei N die Anzahl der Kohlenstoffatome in der Fettsäure ist J ist die Zahl von 13C Isotopen, einM + J ist die Fülle der jeweiligen Isotopologue und einT die gesamte Fülle an alle Isotopologues. - Für die Berechnung Summe der Gegend Spitzenwerte von molekularen Ion (M+) von der jeweiligen FA und alle Isotopologues (M+ 1M+ 2und So weiter), von SIM-MS-Analyse erkannt, und legen Sie auf 100 % relative Häufigkeit. Berechnen Sie die Portion jeder erkannten Isotopologue leicht nach der Regel von drei.
- Subtrahieren der radioaktiv markierten Tag 0 Kontrollwert (natürliche 13C Hintergrund) von den experimentellen Werten um endgültige Daten zu den durchgeführten externen 13C-Kennzeichnung nur zurückverfolgt zu erhalten.
Ergebnisse
Frische Gewicht und Lipid Inhalt der Collembolen
Im Laufe des beschriebenen Versuchs änderte der Inhalt in NLFAs und PLFAs deutlich im Laufe der Zeit sich nicht während das Frischgewicht der Proben leicht aber nicht signifikant24erhöht. Beide Parameter zeigen ein hohes Maß an körperlicher Fitness der Collembolen Exemplare. Achten Sie darauf, zu untersuchen, Collembolens frische Gewicht und Lipid Inhalt während des Experiments, die Probenah...
Diskussion
Isotopologue Profilierung
Eine detaillierte Analyse der quantitativen Aspekte bei 13C Verteilung in FAs braucht Spitzentechnologie, Kohlenstoff in Nahrungsketten Partitionierung zuzuweisen. Die vorliegende Arbeit beschäftigt Isotopologue Profilierung zur Beurteilung der 13C /12C-Verhältnisse in gemeinsamen FA Biomarker für tropische Interaktionen. Diese Methode ist gut etabliert für Aminosäureanalyse von Flüssigchromatographie (LC-MS) und...
Danksagungen
Die finanzielle Unterstützung von R. Menzel und L. Ruess von der Deutschen Forschungsgemeinschaft (RU RU780/11-1) ist dankbar anerkannt. R. Nehring wurde von RU 780/10-1 finanziert. Schließlich sind wir sehr dankbar, dass Dr. Hazel Ruvimbo Maboreke für das Korrekturlesen unserer Handschrift.
Materialien
| Name | Company | Catalog Number | Comments |
| neoLab-Round jars | neoLab | 2-1506 | 69 x 40 mm, 10 pacs/pack |
| Charcoal activated | Carl Roth | X865.1 | p.a., powder, CAS No. 7440-44-0 |
| Alabaster Dental | RÖHRICH-GIPSE | --- | http://www.roehrich-gipse.de/dentalgipse.php |
| Chloroform | Carl Roth | 7331.1 | HPLC ≥ 99,9 % |
| Methanol | Carl Roth | P717.1 | HPLC ≥ 99,9 % |
| Hexan | Carl Roth | 7339.1 | HPLC ≥ 98 % |
| tert-Butyl methyl ether (MTBE) | Carl Roth | T175.1 | HPLC ≥ 99,5 % |
| Aceton | Carl Roth | 7328.2 | HPLC ≥ 99,9 % |
| NaOH | Carl Roth | 6771.1 | p.a. ≥99 %, in pellets |
| di-Natriumhydrogenphosphat | Carl Roth | P030.1 | p.a. ≥99 % , water free |
| Na-dihydrogenphosphat Dihydrat | Carl Roth | T879.1 | p.a. ≥99 % |
| Hypochloric acid (6 N) | VWR International | 26,115,000 | AVS TITRINORM vol. solution |
| Bond Elut (Columns) | Agilent Tech. | 14102037 | HF Bond Elut-SI, 500 mg, 3 mL, 50/PK |
| Präparatengläser Duran | Glasgerätebau Ochs | 135215 | Ø 16 x 100 mm, plus screw cap with handy knurl and integrated PTFE/silicone gasket |
| Supelco 37 Component FAME Mix | Sigma-Aldrich | 47885-U Supelco | 10 mg/mL in methylene chloride, analytical standard |
| FlowMesh | Carl Roth | 2796.1 | Polypropylene mesh, approximately 0.3 mm thick, with 1 mm strand spacing |
| Bacterial Acid Methyl Ester (BAME) Mix | Sigma-Aldrich | 47080-U Supelco | 10 mg/mL in methyl caproate, analytical standard |
| Methyl nonadecanoate | Sigma-Aldrich | 74208 | analytical standard ≥ 98.0 % |
| Hexadecanoic acid-1-13C (Palmitic) | Larodan Fine Chemicals | 78-1600 | GC ≥ 98.0 % (13C: 99.0 %) |
| RVC 2-25 CDplus | Martin Christ Gefrier-trocknungsanlagen | Compact benchtop midi concentrator | |
| Alpha 2-4 LDplus | Martin Christ Gefrier-trocknungsanlagen | Drying manifold | |
| MZ 2C NT | Vacuubrand GMBH | Vacuum pump | |
| Roto-Shake Genie | Scientific Industries | Combined rocking and rotating device | |
| XP64 Micro Comparator | Mettler Toledo | Super high precision balance | |
| GC-System 7890A | Agilent Tech. | Gas chromatograph | |
| 7000 GC/MS Triple Quad | Agilent Tech. | Triple Quad mass spectrometer | |
| 7683B Series Injector | Agilent Tech. | Sample injector | |
| Heraeus Multifuge 3SR+ | Thermo Scientific | Centrifuge with 10 ml tube rotor |
Referenzen
- Ruess, L., et al. Application of lipid analysis to understand trophic interactions in soil. Ecology. 86 (8), 2075-2082 (2005).
- Ruess, L., et al. Lipid composition of Collembola and their food resources in deciduous forest stands - Implications for feeding strategies. Soil Biology and Biochemistry. 39 (8), 1990-2000 (1990).
- Chamberlain, P. M., Bull, I. D., Black, H. I. J., Ineson, P., Evershed, R. P. Fatty acid composition and change in Collembola fed differing diets: identification of trophic biomarkers. Soil Biology and Biochemistry. 37 (9), 1608-1624 (2005).
- Stott, A. W., Davies, E., Evershed, R. P., Tuross, N. Monitoring the routing of dietary and biosynthesised lipids through compound-specific stable isotope (delta C-13) measurements at natural abundance. Naturwissenschaften. 84 (2), 82-86 (1997).
- Ruess, L., Haggblom, M. M., Langel, R., Scheu, S. Nitrogen isotope ratios and fatty acid composition as indicators of animal diets in belowground systems. Oecologia. 139 (3), 336-346 (2004).
- Pollierer, M. M., Scheu, S., Haubert, D. Taking it to the next level: Trophic transfer of marker fatty acids from basal resource to predators. Soil Biology and Biochemistry. 42 (6), 919-925 (2010).
- Ferlian, O., Scheu, S., Pollierer, M. M. Trophic interactions in centipedes (Chilopoda, Myriapoda) as indicated by fatty acid patterns: Variations with life stage, forest age and season. Soil Biology and Biochemistry. 52, 33-42 (2012).
- Ruess, L., Chamberlain, P. M. The fat that matters: Soil food web analysis using fatty acids and their carbon stable isotope signature. Soil Biology and Biochemistry. 42 (11), 1898-1910 (2010).
- Traugott, M., Kamenova, S., Ruess, L., Seeber, J., Plantegenest, M. Empirically characterising trophic networks: What emerging DNA-based methods, stable isotope and fatty acid analyses can offer. Adv Ecol Res. 49, 177-224 (2013).
- Hammer, B. T., Fogel, M. L., Hoering, T. C. Stable carbon isotope ratios of fatty acids in seagrass and redhead ducks. Chemical Geology. 152 (1-2), 29-41 (1998).
- Budge, S. M., Iverson, S. J., Koopman, H. N. Studying trophic ecology in marine ecosystems using fatty acids: A primer on analysis and interpretation. Marine Mammal Science. 22 (4), 759-801 (2006).
- Haubert, D., et al. Trophic structure and major trophic links in conventional versus organic farming systems as indicated by carbon stable isotope ratios of fatty acids. Oikos. 118 (10), 1579-1589 (2009).
- Ngosong, C., Raupp, J., Richnow, H. H., Ruess, L. Tracking Collembola feeding strategies by the natural 13C signal of fatty acids in an arable soil with different fertilizer regimes. Pedobiologia. 54 (4), 225-233 (2011).
- Bec, A., et al. Assessing the reliability of fatty acid-specific stable isotope analysis for trophic studies. Methods in Ecology and Evolution. 2 (6), 651-659 (2011).
- Gladyshev, M. I., Makhutova, O. N., Kravchuk, E. S., Anishchenko, O. V., Sushchik, N. N. Stable isotope fractionation of fatty acids of Daphnia fed laboratory cultures of microalgae. Limnologica. 56 (Supplement C. 56 (Supplement C), 23-29 (2016).
- Gladyshev, M. I., Sushchik, N. N., Kalachova, G. S., Makhutova, O. N. Stable isotope composition of fatty acids in organisms of different trophic levels in the Yenisei river. PLoS One. 7 (3), e34059 (2012).
- Eisenreich, W., Dandekar, T., Heesemann, J., Goebel, W. Carbon metabolism of intracellular bacterial pathogens and possible links to virulence. Nature Reviews Microbiology. 8 (6), 401-412 (2010).
- Eylert, E., Bacher, A., Eisenreich, W. NMR-based isotopologue profiling of microbial carotenoids. Methods Mol Biol. 892, 315-333 (2012).
- Garton, N. J., O'Hare, H. M. Tuberculosis: feeding the enemy. Chemical Biology. 20 (8), 971-972 (2013).
- Rosenblatt, J., Chinkes, D., Wolfe, M., Wolfe, R. R. Stable isotope tracer analysis by GC-MS, including quantification of isotopomer effects. Am J Physiol. 263 (3), E584-E596 (1992).
- Fernandez, C. A., Des Rosiers, C., Previs, S. F., David, F., Brunengraber, H. Correction of 13C mass isotopomer distributions for natural stable isotope abundance. J Mass Spectrom. 31 (3), 255-262 (1996).
- Heuner, K., Eisenreich, W. The intracellular metabolism of legionella by isotopologue profiling. Methods Mol Biol. 954, 163-181 (2013).
- Willenborg, J., et al. Characterization of the pivotal carbon metabolism of Streptococcus suis serotype 2 under ex vivo and chemically defined in vitro conditions by isotopologue profiling. J Biol Chem. 290 (9), 5840-5854 (2015).
- Menzel, R., Ngosong, C., Ruess, L. Isotopologue profiling enables insights into dietary routing and metabolism of trophic biomarker fatty acids. Chemoecology. 27 (3), 101-114 (2017).
- Buse, T., Ruess, L., Filser, J. New trophic biomarkers for Collembola reared on algal diets. Pedobiologia. 56 (3), 153-159 (2013).
- Hutson, B. R. Effects of variations of the plaster-charcoal culture method on a Collembolan, Folsomia candida. Pedobiologia. 18, 138-144 (1978).
- Fountain, M. T., Hopkin, S. P. Folsomia candida (Collembola): a "standard" soil arthropod. Annu Rev Entomol. 50, 201-222 (2005).
- ISO, I. O. f. S. . Soil Quality-Inhibition of reproduction of Collembola (Folsomia candida) by soil pollutants. , (1999).
- Welch, D. F. Applications of cellular fatty acid analysis. Clin Microbiol Rev. 4 (4), 422-438 (1991).
- Dodds, E. D., McCoy, M. R., Rea, L. D., Kennish, J. M. Gas chromatographic quantification of fatty acid methyl esters: flame ionization detection vs. electron impact mass spectrometry. Lipids. 40 (4), 419-428 (2005).
- Kuppardt, S., Chatzinotas, A., Kastner, M. Development of a fatty acid and RNA stable isotope probing-based method for tracking protist grazing on bacteria in wastewater. Appl Environ Microbiol. 76 (24), 8222-8230 (2010).
- Zhang, X., He, H., Amelung, W. A GC/MS method for the assessment of 15N and 13C incorporation into soil amino acid enantiomers. Soil Biology and Biochemistry. 39 (11), 2785-2796 (2007).
- Vetter, W., Thurnhofer, S. Analysis of fatty acids by mass spectrometry in the selected ion monitoring mode. Lipid Technol. 19 (8), 184-186 (2007).
- Thurnhofer, S., Vetter, W. A gas chromatography/electron ionization-mass spectrometry-selected ion monitoring method for determining the fatty acid pattern in food after formation of fatty acid methyl esters. J Agric Food Chem. 53 (23), 8896-8903 (2005).
- Haubert, D., Haggblom, M. M., Scheu, S., Ruess, L. Effects of fungal food quality and starvation on the fatty acid composition of Protaphorura fimata (Collembola). Comparative Biochemistry and Physiology B-Biochemistry & Molecular Biology. 138 (1), 41-52 (2004).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten