Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Profilage de l’acide gras 13C Isotopologue donne un aperçu de transfert trophique de carbone et le métabolisme des lipides des consommateurs d’invertébrés
Dans cet article
Résumé
L’approche de marqueur trophique acide gras, c'est-à-dire l’assimilation des acides gras comme molécule entière et le transfert dans les tissus de consommateurs mineures ou sans aucune modification, est entravée par les lacunes dans les connaissances dans le métabolisme des acides gras des invertébrés du sol petite. Profilage de Isotopologue est fourni comme un outil précieux pour démêler les interactions trophiques.
Résumé
Acides gras (FAs) sont des biomarqueurs utiles en écologie trophique car ils sont généralement assimilés comme une molécule complète et transférés dans les tissus de consommateurs avec le mineur ou sans modification, ce qui permet le routage alimentaires entre différents niveaux trophiques. Toutefois, l’approche de marqueur trophique FA est encore entravée par la connaissance limitée dans le métabolisme des lipides de la faune du sol. Cette étude a utilisé entièrement marqué l’acide palmitique (C16 : 013, 99 % d’atomes) comme traceur dans les voies de métabolisme d’acide gras de deux sols généralisée collemboles, Protaphorura fimata et Heteromurus nitidus. Afin d’enquêter sur le sort et les modifications métaboliques de ce précurseur, on présente une méthode de profilage d’isotopologue, interprété par spectrométrie de masse avec détection d’ions simples. En outre, l’alimentation expérience en laboratoire en amont est décrite, ainsi que l’extraction et la méthylation des fractions lipidiques dominante (lipides neutres, phospholipides) et la formule connexe et les calculs. Profilage de Isotopologue n’est pas seulement le rendement l’enrichissement global de 13C dans les acides gras provenant de 13C marqué précurseur mais produit également le patron des isotopomères supérieure à la masse de l’ion parent (par exemple, l’ion moléculaire de FA M+) de chacun marqué FA par une ou plusieurs unités de masse (M+ 1M+ 2M+ 3, etc.). Cette connaissance permet des conclusions sur le rapport de routage diététique d’une FA entièrement consommée par rapport à la biosynthèse de novo . Le profilage d’isotopologue est proposé comme un outil utile pour l’évaluation du métabolisme des acides gras chez les animaux de la terre à démêler les interactions trophiques.
Introduction
Dans un habitat cryptique comme sol, relations trophiques sont difficiles à aborder et sont plus limitées par la petite taille de la faune. La dernière décennie a vu des avances en écologie biochimique, en particulier dans l’utilisation des acides gras comme biomarqueurs pour définir les stratégies d’alimentation de la faune du sol sous des conditions de champ1,2,3. C’est basé sur le fait que les acides gras provenant de ressources peut être incorporés dans les tissus des consommateurs comme des molécules entières, un processus appelé routage alimentaires4. Transfert des acides gras a été signalé au cours de trois niveaux trophiques, c'est-à-dire des champignons aux nématodes à collemboles5. Récemment, la faune prédatrice était considéré comme6,7 , et les premiers examens des acides gras comme marqueurs trophiques dans les réseaux trophiques du sol ont été publiées8,9.
Plus d’informations sur les interactions trophiques sont atteint par les isotopes stables de l’acide gras sonder (FA-SIP). La détermination de 13C /12C les ratios en acides gras dans les régimes alimentaires et les consommateurs peuvent attribuent des liens binaire et estiment le débit de carbone associé et a été employés en terrestre, eau douce et les réseaux trophiques marins10,11 ,12,13. L’hypothèse de base est que les acides gras alimentaires routés ne sont pas soumis à des processus enzymatiques ; par conséquent, leur 13C signal, c'est-à-dire le 13C /12C ratio de l’acide gras, le consommateur est similaire à celle de l' alimentation1. Toutefois, une diminution progressive de la signature de 13C vers le haut de la chaîne alimentaire a été signalée dans les systèmes aquatiques, en diminuant une application large de FA-SIP dans études trophique14,15,16. En outre, les connaissances dans le métabolisme des lipides dans la plupart des invertébrés dans les réseaux alimentaires terrestres sont encore limitée.
Comprendre les voies de métabolisme lipidique chez les consommateurs est essentielle pour l’utilisation des acides gras de marqueur trophique comme moyen pour la détermination du flux de carbone quantitatives en écologie trophique. Dans cette optique, 13C-isotopologue de profilage, qui en principe peut être appliqué pour l’enquête sur le métabolisme du carbone de tout système biologique17, est une méthode prometteuse. Suite à l’introduction d’un substrat de carbone marqué 13, la répartition des 13C du réseau métabolique est traçable depuis les produits métaboliques générés dans le spectacle de consommateurs une distribution spécifique isotopologue. Cela peut être évaluée par quantitative résonance métabolique spectroscopie18,19 ou20,spectrométrie de masse à21, avec les échantillons biologiques favorisées dans cette dernière avec faible biomasse en raison de son supérieur sensibilité.
Bien que le profilage d’isotopologue a été avec succès appliqué aux acides aminés et fourni la perspicacité dans le métabolisme de carbone en vivo de bactéries pathogènes17,22,23, sa mise en œuvre en gras les acides a pris du retard. La première analyse détaillée sur le sort d’un isotope stable étiquetés acides gras précurseurs, sa diététique routage ou dégradation par β-oxydation, chez les consommateurs d’invertébrés du sol, a été récemment réalisée par Menzel et al. 24. ici, les bases méthodologiques pour des expériences d’incorporation avec 13C marqué suivis d’isotopologue analyse des principaux descendants en invertébrés du sol fréquents, les collemboles, les acides gras sont fournis. Ces microarthropodes sont un groupe de bons modèles puisqu’elles constituent des éléments importants du réseau alimentaire du sol et sont bien étudiés pour leur marqueur trophique acides gras8,25.
Comprendre les voies de métabolisme lipidique chez les consommateurs est essentielle pour l’utilisation des acides gras de marqueur trophique comme moyen pour la détermination du flux de carbone quantitatives en écologie trophique. Le présent protocole donne la conception et la mise en place d’un laboratoire alimentation expérience ainsi que les processus biochimiques d’extraction et de méthylation des fractions de lipides dominante (lipides neutres, phospholipides) de collemboles. Il montre comment la composition d’isotopologue des acides gras est analysée par spectrométrie de masse et décrit la formule connexe et les calculs. Cette procédure se traduit par : (i) les ratios des isotopomères dépassant la masse de l’ion parent (par exemple, l’ion moléculaire de l’acide gras M+) par un ou plusieurs masse unités (M+ 1M+ 2M+ 3, etc.) et (ii) l’ensemble 13 C un enrichissement en acides gras dérivé du précurseur marqué 13C. Bien qu’utilisé pour les collemboles, cette approche peut généralement être appliquée de toutes les autres relations prédateur-proie sur l’hypothèse que ceux-ci sont cultivables en quantité suffisante dans des conditions contrôlées afin d’assurer une absorption étiquette réussie et suivantes vérification.
Protocole
Le protocole décrit ne relève pas de la compétence de l’éthique de l’Animal. Toutefois, lorsque les gens s’adaptent les protocoles décrits aux animaux supérieurs, prendre soin que la Commission d’éthique de l’Animal institutionnelle a approuvé le protocole pour la gestion des animaux.
1. culture des animaux
Remarque : Tout expliqué les étapes expérimentales sont basés sur des protocoles bien établis de27,26,28. Biologiques en laboratoire ont besoin d’un apport continu d’organismes facilement cultivables. Ici, les espèces de collemboles Protaphorura fimata (Gisin, 1952) et Hetermurus nitidus (Templeton, 1835) ont été utilisées. Les deux espèces sont faciles à maintenir comme cultures productives de laboratoire nourries avec de la levure de boulanger.
- Dans des microcosmes en plastique avec couvercle hermétique (diamètre 7 cm, hauteur 4,5 cm), ajouter un mélange de charbon actif, de plâtre et distillée pour fournir un substrat de reproduction très humide (Figure 1 a).
- Lors de la préparation de mélange de microcosmes assez substrat, par exemple, pour 10 microcosmes dans un lot. Mix 9 parties de plâtre de Paris (225 g) et 1 partie de sec au charbon actif (25 g) ensemble dans un pot de plâtre, ajouter avec précaution environ 10 parties d’eau distillée (250 mL) et laisser pour reposer pendant 5 minutes sans remuer, à température ambiante.
- Remuez avec une cuillère de laboratoire à vitesse modérée dans le sens horaire pour éviter les bulles d’air jusqu'à l’obtention d’une consistance épaisse soupy. Verser immédiatement dans des microcosmes à une hauteur d’environ 1 cm.
- Lisser le plâtre en douceur en tapant sur le banc et tourbillonnant. Notez que trous (produits au hasard des bulles d’air) et sillons (activement ajoutés avec une spatule stérile) peuvent encourager les collemboles fertile à pondre il. Cette étude évite les trous et les sillons en faveur d’avoir les mêmes conditions reproductibles. Toutefois, à des fins de démonstration Figure 1 b présente quelques trous.
- Laisser sécher pendant environ 1 à 2 jours à température ambiante ; incubation à 60 ° C peut réduire ce temps 1-2 h.
- Humidifier les microcosmes avant utilisation en ajoutant de l’eau du robinet avec une pipette jusqu'à ce que le substrat soit légèrement humide. Microcosme garder humide ajoutant régulièrement de l’eau distillée comme les collemboles ont une cuticule souple et est sensible à la dessiccation.
- Transférez facilement des collemboles à la base de plâtre à l’aide d’un tube d’aspiration simple, c'est-à-dire un tube de silicone long environ 25 cm de long avec une pointe de pipette équipé d’un petit maillage pour empêcher d’aspiration d’animaux dans le tube. Vous pouvez également transférer animaux en leur permettant d’adhérer sur les poils d’une petite brosse.
- Transfert (Voir l’étape 1.2) 30 fraîchement éclos collemboles dans des microcosmes nouvelles et fournir granulés secs levure alimentaire (sur une pointe de couteau) (Figure 1 b) ; renouveler au moins deux fois par semaine. Indépendant du plan trois répétitions par jour d’échantillonnage ; dans cette étude jours 0 - 7 et 14. Incuber à 15 ° C dans l’obscurité. Maintenir qu'une température constante est indispensable, que les acides gras sont altérées par le métabolisme des animaux pour satisfaire aux exigences pour la fluidité membranaire.
- Nourrir les collemboles avec levure de boulanger pendant quatre semaines avant de commencer les expériences d’exposition pour obtenir un homogène 13C /12C signal et le motif en acides gras. Utiliser un tube d’aspiration ou une brosse pour enlever tous les oeufs (Figure 1), les pelotes fécales, et exuvies régulièrement que les animaux peuvent se nourrir sur les altérant ainsi leur profil lipidique.

Figure 1 : culture de collemboles. (A) microcosme rempli de substrat, un mélange séché de plâtre de Paris, le charbon et l’eau distillée de reproduction. (B) et (C) échantillon représentatif d’une culture de la Protaphorura fimata ; noter les petites pépites de levure de boulanger sèche utilisée comme source de nourriture et aussi sous forme de trous dans le substrat de reproduction (flèche noire) (B) ainsi que deux œufs (flèche blanche) (C). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
2. l’étiquetage alimentaire, la récolte et la manipulation des échantillons
- Étiquetage
- Quatre semaines après avoir établi les cultures de collemboles, 30 personnes chacun dans des microcosmes nouvelles et incuber à 15 ° C dans l’obscurité.
- Introduire l’étiquette de l’impulsion de leur alimentation des levures de boulangerie contenant un entièrement 13C marqué l’acide palmitique (13C16 : 0, 99 % d’atomes) en mélangeant de l’acide palmitique marqué C 13avec levure de Boulanger avec une spatule à un ratio 0.5:1, par exemple, 5 g 13 C16 : 0 et 10 g levure de boulanger à sec. Placer sur une pointe de couteau sur chaque microcosme.
- Après 6 h, remplacez ceci marqué alimentaire avec complètement sans étiquette de levure de boulanger.
-
Autre culture
- Au cours de l’expérience renouveler l’alimentation levure tous les trois jours et ajouter des montants plus élevés que ce qui consommera collemboles dans ce délai. Plus important encore, utiliser un tuyau d’aspiration ou une brosse pour enlever les œufs de collemboles, pelotes fécales et exuvies régulièrement pour assurer une alimentation exclusive par les animaux sur la nourriture fournie.
- Moisson
- Échantillon des microcosmes destructive tous les jours jusqu’au jour 7. Puis, le jour 14, recueille trois répétitions indépendantes à chaque période d’échantillonnage ; temps d’échantillonnage différentes sont possibles.
- Préparer les tubes de verre de 10 mL avec bouchons à vis téflon, un pour chaque échantillon. Au préalable nettoyer ces tubes dans une rondelle de verrerie et rincer deux fois avec de l’eau désionisée. Enfin, pour enlever toute trace de hydrophobe polluants laver deux fois en ajoutant 2 mL de chloroforme (qualité CLHP), vortex grossièrement et jeter le solvant.
- Pour les échantillons de contrôle (jour 0), prendre 3 30 non-exposés collemboles issus des cultures pré comme échantillons jour 0.
- Pour les échantillons exposés (jour 1 et au-delà), prise pour les échantillons quotidiennes dans chaque cas 30 3 exposé des collemboles issus des cultures moyennement nourris avec le mélange de l’acide palmitique marqué C 13et levure de boulanger.
- Enregistrer les collemboles poids frais par un ultra-microbalance. Transférer les animaux à l’aide d’un aspirateur ou une brosse à échelle appropriées-casseroles. Pour assurer une manipulation facile des collemboles au cours de la pesée dans les balance-casseroles, animaux choc-cool avant (-80 ° C pendant 2 h). Sinon, un flux de CO2 pendant 10 min peut étourdir en toute sécurité les animaux.
- Directement après la pesée des animaux mis de l’échelle-casseroles soigneusement dans 10 mL en verre tubes à essai. Remplir les tubes de 1 mL de méthanol (qualité CLHP) et les conserver à-20 ° C jusqu'à l’analyse.
NOTE : De partir de cette étape, évitez d’échantillon manipulant avec équipement en plastique comme les solvants organiques sont impliqués ; Utilisez plutôt des distributeurs et des pipettes qui conviennent aux solvants ainsi que les récipients en verre.
3. lipides Extraction à partir de tissus d’origine animale et méthanolyse
- Préparer trois verre-tubes à essai (équipés avec bouchons à vis téflon) par lot contenant seulement 1 mL de méthanol comme valeurs vides. Ce qui est important, ajoutez ou transférer n’importe quel solvant utilisé dans le présent protocole que par pipettes en verre ou distributeurs résistant aux solvants chloroforme/méthanol rincé.
- Au début du processus d’extraction de lipides, réduire le méthanol appliqué pour le stockage (ou masquer) par évaporation ; un banc compact vide rotation concentrateur (RVC) équipé d’une pompe à vide et un piège à froid est recommandé. Transférer le tube ouvert dans la TVR et évaporer à sec à 50 ° C et sous vide pression 200 hPa pendant 20 min.
- Ajouter 5 mL de solvant d’extraction monophasés (chloroform/methanol/0.05 M 1:2:0.8 de tampon phosphate, pH 7,4) pour chaque échantillon (y compris les espaces) et extraire les lipides des collemboles à température ambiante en agitant pendant la nuit (~ 200 tr/min).
- Transférer le solvant dans de nouveaux tubes et réextraire les échantillons en agitant pendant 3 h avec 2,5 mL de solvant d’extraction supplémentaire. Après cela, combiner des extraits de ces deux étapes ; l’utilisation du verre, que pipettes Pasteur est recommandée. Ajouter 0,8 mL de chloroforme et 0,8 mL d’eau distillée, puis mélanger et centrifuger à 2 000 g à 20 ° C pendant 5 min. Enfin, laisser des échantillons reposer pendant 5 min pour la séparation de l’humeur aqueuse et chloroforme phases.
- Pour l’analyse de profil des acides gras, diviser les lipides cellulaires totaux de collemboles en lipides neutres, glycolipides et fractions phospholipidiques.
- Pour chaque échantillon, préparer une colonne de silice acide (colonne commercial avec 0,5 g d’acide silicique, maille taille 100-200 µm, voir Table des matières) en ajoutant 1 mL de chloroforme (préconditionnement). Pour accélérer ce processus, monter les colonnes sur un bloc vide couramment utilisé en chromatographie en extraction en phase solide. Ne pas utiliser les aiguilles en nylon ; utiliser au-dessus des tubes en acier inoxydable.
- Après le chloroforme utilisé pour préconditionnement a traversé le transfert de la colonne la phase complète de chloroforme inférieure de chaque échantillon d’une colonne individuelle. Pour simplifier cette procédure, la phase aqueuse supérieure peut être retirée au préalable. Utiliser des pipettes Pasteur de verre, cependant, veiller à ce que les colonnes ne s’assèchent pas.
- Successivement éluer les fractions lipidiques avec 5 mL de chloroforme (y compris neutre acides gras des lipides, NLFA), 10 mL d’acétone (glycolipides - ne pas analysés dans ce projet) et 5 mL de méthanol (y compris les acides gras des phospholipides, AGPL). Collecter chaque fraction dans les récipients en verre individuel.
- À la fin de l’extraction, réduire le chloroforme (NLFA) et le méthanol (AGPL) par évaporation dans une TVR. Transférer le tube ouvert à la TVR et évaporer à sec, ~ 90 min à 60 ° C et un vide de 24 hPa.
- Commencer de saponification des lipides (fractions de la NLFA et AGPL) suivant le protocole de Welch (1991),29 , avec addition de 1 mL de solution d’hydroxyde de sodium-méthanol (45 g d’hydroxyde de sodium, 150 mL de méthanol et 150 mL d’eau distillée) et incuber à 100 ° C pendant 30 min dans un bain d’eau. Refroidir l’échantillon dans l’eau glacée pendant 2 min, puis remettre les échantillons sur le banc et continuer à travailler à température ambiante.
- Ajouter l’étalon interne à chaque échantillon, y compris les blancs. Choisissez un acide gras pas commun chez les organismes expérimentaux ; également utiliser un acide gras saturé pour minimiser la perte de clivage et de sélectionner une molécule avec la longueur de la chaîne intermédiaire. De nombreuses utilisations, l’impair nonadecanoic acide (0:19) fonctionne bien. Alors, ajoutez 30 µL d’une solution de 0,74 mM dans l’isooctane. La quantité exacte est très importante - n’oubliez pas de faire vérifier la précision de votre pipette avec une microbalance à l’avance.
- Ajouter 2 mL d’acide chlorhydrique-méthanol (mix 325 mL de l’acide chlorhydrique N 6.0 avec 275 mL de méthanol), incuber à 80 ° C pendant 10 min dans un bain d’eau et laisser refroidir rapidement sur la glace pendant 2 min. Cette étape est la durée et la température sensible ; utiliser 80 ± 1 ° C et 10 ± 1 min. vérifier avec combien d’échantillons peut aller dans le bain d’eau à la fois de garder les 80 ° C.
Remarque : Cette procédure se traduit par les esters méthyliques d’acides gras (FAMEs), c'est-à-dire les analytes acides gras stabilisés pour vaporisation en chromatographie en phase gazeuse. - Enfin, ajoutez 1,25 mL d’hexane/méthyl tertio-butyl éther (1:1) et Roche doucement pendant 10 min, puis centrifuger à 2 000 g pendant 5 min. Retirer la phase de bas et de conserver la phase supérieure composée de FAMEs ; utiliser le verre que pipettes Pasteur. Ajouter 3 mL d’hydroxyde de sodium aqueux (10,8 g de NaOH dissous dans 900 mL d’eau distillée) pour une étape de lavage.
- Rock et centrifuger à nouveau.
- Prendre la phase supérieure complète de contenant des lipides à l’aide d’une pipette Pasteur en verre et le transfert d’un flacon d’échantillonnage de chromatographie en phase gazeuse équipé d’un septum de téflon. Évitez d’inclure même de petites quantités de la phase aqueuse, car cela provoquerait des problèmes avec la mesure de la GC. Encapsuler le flacon et conserver à-20 ° C jusqu'à l’analyse.
4. quantification des acides gras par CG-DIF
- Chromatographie en phase gazeuse permet d’identifier et de quantifier les renommées dans les fractions NFLA et AGPL des lipides de collemboles (animal). Un chromatographe en phase gazeuse (GC) équipé de détecteur à ionisation de flamme (FID) est un équipement établies et éprouvées de30.
- Pour l’identification des FAMEs, comparer les temps de rétention des pics de l’échantillon à ceux dans une norme FAME. Voici les mélange standard qualitatif ou quantitatif de clé FAMEs couvrant une variété de produits alimentaires et les organismes.
- Employer des mélanges étalons FAME comprenant FAs de représentant pour le groupe de l’organisme étudié et la diète expérimentale. Collemboles, ce sont des acides gras caractéristique des eucaryotes, par exemple, la longue chaîne d’acides gras polyinsaturés tels que l’acide arachidonique (20 : 4ω6). Lorsque vous utilisez la levure comme diète Voici les marqueurs fongiques tels que l’acide linoléique (18 : 2ω6). Un bon choix est le soi-disant mélange FAME, comprenant 37 différents acides gras fréquentes chez les animaux, champignon et végétaux et l’ester méthylique de l’acide bactérienne (BAME) mélangent (voir Table des matières).
- Pour exécuter les exemples sur le GC-FID, mis en place une séquence avec le logiciel respectif de l’instrument. Pour obtenir des instructions, reportez-vous au manuel du fabricant. La séquence commence par les mélanges étalons externes (p. ex. FAME et BAME mix), suivies par les échantillons. Notez qu’il existe un décalage de temps de rétention, c'est-à-dire, un léger retard dans le temps d’élution des acides gras de la colonne GC avec chaque course, lors de l’exécution des échantillons ! Soit inclut une norme chaque 10th exécuter dans la séquence de l’échantillon ou utiliser des temps de rétention de verrouillage pour l’acide palmitique (16:0).
- Adapter les réglages de GC dépendants de l’instrument. Le programme suivant est proposé pour une colonne capillaire de haute performance (HP) (25 m x 0,2 mm i.d., film d’épaisseur 0.33 µm. volume d’injection réglée à 1 µL en mode splitless et utiliser l’hydrogène comme gaz vecteur. Employer un début de programme de température à 50 ° C (qui s’est tenue pendant 1 min) et augmentant de 25 ° C min-1 à 175 ° C, suivie de 3 ° C min-1 à 230 ° C (qui s’est tenue pour 5,7 min).
- Calculer l’acide gras nmol par gramme de poids frais (sec) des organismes à l’aide de la réponse obtenue par FID pour chaque célébrité appliquant les quantités connues de l’acide gras respective en utilisant la formule suivante :
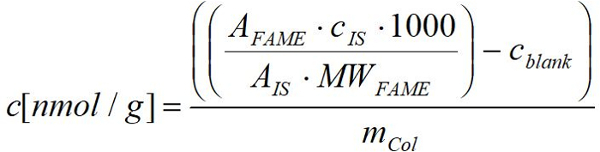
UneFAME: aire du pic de la notoriété respectif dans l’échantillon
MWFM: poids moléculaire de la notoriété respectif en µg/µmol
CIS: Concentration de l’étalon interne en µg
UnIS: aire du pic de l’étalon interne
mCol: frais (sec) poids d’échantillon de collemboles respectif en g
1000 : facteur de Conversion de µmol en nmol
cBW: Concentration de la notoriété respectif dans la moyenne des valeurs vides correspondantes en nmol
5. analyse de 13C par profilage Isotopologue
- Utiliser un système de GC couplé à un détecteur sélectif de masse (MS), fourni avec une source d’ionisation (EI) électrons pour la détermination de l’isotopologue.
- Utilisez une colonne capillaire polaire (par exemple, 23 DB, CP-Sil 88) car cela permet de plus la séparation des acides gras insaturés même avec le même nombre de doubles liaisons. Le choix de la colonne GC est critique pour les résultats, car elle détermine la bonne représentation de l’ion de la molécule dans les acides gras.
- Pour une colonne de 23 DB (60 m x 0,25 mm diamètre intérieur, film épaisseur 0,15 µm), démarrez la température du four à 130 ° C et augmenter de 6,5 ° C/min à 170 ° C. Suivre avec une augmentation de 3 ° C/min jusqu'à 203 ° C et maintenez pour 1,9 min. Suivez avec une augmentation de 40 ° C/min à 230 ° C et maintenez pour 8,3 min. température de ligne transfert Set à 280 ° C. Encore une fois, ajuster la méthode CG à l’instrument.
- Utiliser des normes quantitatives comprenant des quantités connues de FAMEs pour tous les acides gras à investiguer pour incorporation de 13C. Mettre ces normes en début et en fin de chaque séquence exemple exécuter. Prenez les temps de rétention des acides gras d’intérêt de ces normes.
- Mesurer les échantillons des expériences toujours commencer par les échantillons non étiquetés et les sondes marquées par la suite. Appliquer un ratio de partage approprié pour les concentrations de l’échantillon, par exemple 1:12.5.If disponibles pour l’instrument, appliquer un rincez avec de l’hélium après chaque échantillon à dégager outre colonne d’analytes restants.
- Appliquer les temps de rétention de blocage à la méthode GC-MS pour que l’acquisition de la carte SIM ne souffre pas de se déplacer et il est temps de rétention analyte reproductible.
- Déterminer 13C incorporation dans l’ion moléculaire des acides gras par le GC/EI-MS en utilisant l’ion sélectionnée mode (SIM) de l’instrument de surveillance. Fonctionnement en mode SIM permet de détecter des analytes spécifique avec une sensibilité accrue par rapport au mode de balayage complet.
- Exécuter une analyse initiale tout d’abord pour voir ce qui est présent et puis exécutez SIM sur les ions appropriées. Acquérir des données à des masses moléculaires d’intérêt en sélectionnant windows de scan de m/z (groupes SIM) qui englobe le temps pic chromatographique de l’acide gras respectif. En règle générale, suivre deux à quatre ions par fenêtre de l’analyte et le temps.
- Afin d’augmenter la sensibilité, régler la vitesse de numérisation massive et dwell temps (le temps passé en regardant chaque Messe). La meilleure qualité des données sont obtenues à la vitesse la plus basse possible et une règle dans SIM est généralement 8 à 12 analyses sur la crête de l’analyte. Une procuration pour les paramètres de l’instrument est un temps d’arrêt moyen par masse de 9 ms, un temps de cycle de 6 s et un temps de scan de cyle de 175 ms-1.
- Détecter l’ion moléculaire (M+) de l’acide gras respectif et tous ses isotopomères (M+ 1, M+ 2 et ainsi de suite). Pour obtenir des exemples, consultez résultats de représentant.
- Enregistrer l’abondance de chaque fragment d’ion (isotopologue). Notez que l’abondance de l’ion de la molécule et son isotopologues est relativement faible et la qualité de la quantification dépend beaucoup de la performance du système MS. Exécuter un air avant de démarrer une séquence de grands échantillons (expérience) et nettoyer la source d’ions si nécessaire.
Remarque : Tout d’abord, ces données fournissent l' ensemble 13C des enrichissements chaque acides gras par le précurseur consommé (ici 16:0). - La proportion de la composition isotopique des acides gras marqués de leurs homologues non marqués permet d’affecter le flux de carbone trophique. Utilisez la formule % atom étape 6.1 pour calculer le pourcentage de carbone marqué (% dans l’atome, atome %) dans l’acide gras respectif. Comparer le pourcentage de 13C, en acides gras des collemboles entre étiquetés (jour 1 et versions ultérieur) et les animaux non étiquetés (jour 0) comme indication relative pour l’écoulement de 13C du régime alimentaire dans la consommation.
- Affecter la position de 13C incorporation dans la chaîne des acides gras. Basé sur la distribution des isotopomères démêler le routage diététiques des acides gras entier marqué traceurs (ici 16:0) de l’élongation de la chaîne par le métabolisme des lipides. Alors que l’assimilation de la molécule entière marqueur augmente l’abondance des isotopomères plus éloigné à l’ion parent (M+), par exemple, M+ 15, M+ 16, avec l’élongation de la chaîne par l’utilisation de 13C marqué C2 fragments (13C acétyl-CoA) les isotopologues à proximité de l’ion moléculaire reçois plus fréquemment.
6. calculs d’enrichissement 13C
- Selon la distribution de l’isotopologues, calculer le pourcentage global du carbone marqué (en % de l’atome) dans l’acide gras respective en utilisant la relation suivante : atom % = (ratio 13C isotopologue incorporé) x (fréquence isotopologue respectif)
Elle est calculée après Kuppardt et al. 31 comme :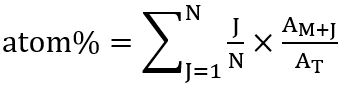
où N est le nombre d’atomes de carbone dans l’acide gras, J est le nombre d’isotopes 13C, unM + J est l’abondance des isotopologue respectif et AT l’abondance totale de tous les isotopologues. - Pour le calcul, somme des valeurs de zone de pic de l’ion moléculaire (M+) de la FA respectif et tous les isotopologues (M+ 1M+ 2et ainsi de suite), détectées par l’analyse de SIM-MS et la valeur 100 % abondance relative. Calculer que la part de chaque isotopologue détecté est facilement en suivant la règle de trois.
- Soustraire la valeur de contrôle de non marquée jour 0 (fond naturel 13C) de valeurs expérimentales pour obtenir les données finales remontées seulement à l' externe effectué 13C-étiquetage.
Résultats
Nouveau contenu poids et lipides de collemboles
Dans le cadre de l’expérience décrite, le contenu en NLFA et AGPL n’a pas changé significativement au fil du temps, alors que le poids frais des spécimens a légèrement augmenté, mais pas de façon significative le24. Les deux paramètres indiquent un bon niveau de forme physique des spécimens collemboles. N’oubliez pas d’étudier le contenu lipidique et de poids frais de collemboles...
Discussion
Profil de Isotopologue
Une analyse détaillée des aspects quantitatifs 13C distribution en FAs a besoin d’une technologie de pointe pour assigner carbone partitionnement dans les réseaux trophiques. Le présent travail employé isotopologue profilage pour évaluer le 13C /12C rapports en communs biomarqueurs de FA pour les interactions tropiques. Cette méthode est bien établie pour l’analyse des acides aminés par chromatographie liquid...
Remerciements
Tient à reconnaître le soutien financier de Menzel de R. et L. Ruess par la Deutsche Forschungsgemeinschaft (RU RU780/11-1). R. Nehring a été financée par la RU 780/10-1. Enfin, nous sommes extrêmement reconnaissants à Mme Hazel Ruvimbo Maboreke pour la relecture de notre manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| neoLab-Round jars | neoLab | 2-1506 | 69 x 40 mm, 10 pacs/pack |
| Charcoal activated | Carl Roth | X865.1 | p.a., powder, CAS No. 7440-44-0 |
| Alabaster Dental | RÖHRICH-GIPSE | --- | http://www.roehrich-gipse.de/dentalgipse.php |
| Chloroform | Carl Roth | 7331.1 | HPLC ≥ 99,9 % |
| Methanol | Carl Roth | P717.1 | HPLC ≥ 99,9 % |
| Hexan | Carl Roth | 7339.1 | HPLC ≥ 98 % |
| tert-Butyl methyl ether (MTBE) | Carl Roth | T175.1 | HPLC ≥ 99,5 % |
| Aceton | Carl Roth | 7328.2 | HPLC ≥ 99,9 % |
| NaOH | Carl Roth | 6771.1 | p.a. ≥99 %, in pellets |
| di-Natriumhydrogenphosphat | Carl Roth | P030.1 | p.a. ≥99 % , water free |
| Na-dihydrogenphosphat Dihydrat | Carl Roth | T879.1 | p.a. ≥99 % |
| Hypochloric acid (6 N) | VWR International | 26,115,000 | AVS TITRINORM vol. solution |
| Bond Elut (Columns) | Agilent Tech. | 14102037 | HF Bond Elut-SI, 500 mg, 3 mL, 50/PK |
| Präparatengläser Duran | Glasgerätebau Ochs | 135215 | Ø 16 x 100 mm, plus screw cap with handy knurl and integrated PTFE/silicone gasket |
| Supelco 37 Component FAME Mix | Sigma-Aldrich | 47885-U Supelco | 10 mg/mL in methylene chloride, analytical standard |
| FlowMesh | Carl Roth | 2796.1 | Polypropylene mesh, approximately 0.3 mm thick, with 1 mm strand spacing |
| Bacterial Acid Methyl Ester (BAME) Mix | Sigma-Aldrich | 47080-U Supelco | 10 mg/mL in methyl caproate, analytical standard |
| Methyl nonadecanoate | Sigma-Aldrich | 74208 | analytical standard ≥ 98.0 % |
| Hexadecanoic acid-1-13C (Palmitic) | Larodan Fine Chemicals | 78-1600 | GC ≥ 98.0 % (13C: 99.0 %) |
| RVC 2-25 CDplus | Martin Christ Gefrier-trocknungsanlagen | Compact benchtop midi concentrator | |
| Alpha 2-4 LDplus | Martin Christ Gefrier-trocknungsanlagen | Drying manifold | |
| MZ 2C NT | Vacuubrand GMBH | Vacuum pump | |
| Roto-Shake Genie | Scientific Industries | Combined rocking and rotating device | |
| XP64 Micro Comparator | Mettler Toledo | Super high precision balance | |
| GC-System 7890A | Agilent Tech. | Gas chromatograph | |
| 7000 GC/MS Triple Quad | Agilent Tech. | Triple Quad mass spectrometer | |
| 7683B Series Injector | Agilent Tech. | Sample injector | |
| Heraeus Multifuge 3SR+ | Thermo Scientific | Centrifuge with 10 ml tube rotor |
Références
- Ruess, L., et al. Application of lipid analysis to understand trophic interactions in soil. Ecology. 86 (8), 2075-2082 (2005).
- Ruess, L., et al. Lipid composition of Collembola and their food resources in deciduous forest stands - Implications for feeding strategies. Soil Biology and Biochemistry. 39 (8), 1990-2000 (1990).
- Chamberlain, P. M., Bull, I. D., Black, H. I. J., Ineson, P., Evershed, R. P. Fatty acid composition and change in Collembola fed differing diets: identification of trophic biomarkers. Soil Biology and Biochemistry. 37 (9), 1608-1624 (2005).
- Stott, A. W., Davies, E., Evershed, R. P., Tuross, N. Monitoring the routing of dietary and biosynthesised lipids through compound-specific stable isotope (delta C-13) measurements at natural abundance. Naturwissenschaften. 84 (2), 82-86 (1997).
- Ruess, L., Haggblom, M. M., Langel, R., Scheu, S. Nitrogen isotope ratios and fatty acid composition as indicators of animal diets in belowground systems. Oecologia. 139 (3), 336-346 (2004).
- Pollierer, M. M., Scheu, S., Haubert, D. Taking it to the next level: Trophic transfer of marker fatty acids from basal resource to predators. Soil Biology and Biochemistry. 42 (6), 919-925 (2010).
- Ferlian, O., Scheu, S., Pollierer, M. M. Trophic interactions in centipedes (Chilopoda, Myriapoda) as indicated by fatty acid patterns: Variations with life stage, forest age and season. Soil Biology and Biochemistry. 52, 33-42 (2012).
- Ruess, L., Chamberlain, P. M. The fat that matters: Soil food web analysis using fatty acids and their carbon stable isotope signature. Soil Biology and Biochemistry. 42 (11), 1898-1910 (2010).
- Traugott, M., Kamenova, S., Ruess, L., Seeber, J., Plantegenest, M. Empirically characterising trophic networks: What emerging DNA-based methods, stable isotope and fatty acid analyses can offer. Adv Ecol Res. 49, 177-224 (2013).
- Hammer, B. T., Fogel, M. L., Hoering, T. C. Stable carbon isotope ratios of fatty acids in seagrass and redhead ducks. Chemical Geology. 152 (1-2), 29-41 (1998).
- Budge, S. M., Iverson, S. J., Koopman, H. N. Studying trophic ecology in marine ecosystems using fatty acids: A primer on analysis and interpretation. Marine Mammal Science. 22 (4), 759-801 (2006).
- Haubert, D., et al. Trophic structure and major trophic links in conventional versus organic farming systems as indicated by carbon stable isotope ratios of fatty acids. Oikos. 118 (10), 1579-1589 (2009).
- Ngosong, C., Raupp, J., Richnow, H. H., Ruess, L. Tracking Collembola feeding strategies by the natural 13C signal of fatty acids in an arable soil with different fertilizer regimes. Pedobiologia. 54 (4), 225-233 (2011).
- Bec, A., et al. Assessing the reliability of fatty acid-specific stable isotope analysis for trophic studies. Methods in Ecology and Evolution. 2 (6), 651-659 (2011).
- Gladyshev, M. I., Makhutova, O. N., Kravchuk, E. S., Anishchenko, O. V., Sushchik, N. N. Stable isotope fractionation of fatty acids of Daphnia fed laboratory cultures of microalgae. Limnologica. 56 (Supplement C. 56 (Supplement C), 23-29 (2016).
- Gladyshev, M. I., Sushchik, N. N., Kalachova, G. S., Makhutova, O. N. Stable isotope composition of fatty acids in organisms of different trophic levels in the Yenisei river. PLoS One. 7 (3), e34059 (2012).
- Eisenreich, W., Dandekar, T., Heesemann, J., Goebel, W. Carbon metabolism of intracellular bacterial pathogens and possible links to virulence. Nature Reviews Microbiology. 8 (6), 401-412 (2010).
- Eylert, E., Bacher, A., Eisenreich, W. NMR-based isotopologue profiling of microbial carotenoids. Methods Mol Biol. 892, 315-333 (2012).
- Garton, N. J., O'Hare, H. M. Tuberculosis: feeding the enemy. Chemical Biology. 20 (8), 971-972 (2013).
- Rosenblatt, J., Chinkes, D., Wolfe, M., Wolfe, R. R. Stable isotope tracer analysis by GC-MS, including quantification of isotopomer effects. Am J Physiol. 263 (3), E584-E596 (1992).
- Fernandez, C. A., Des Rosiers, C., Previs, S. F., David, F., Brunengraber, H. Correction of 13C mass isotopomer distributions for natural stable isotope abundance. J Mass Spectrom. 31 (3), 255-262 (1996).
- Heuner, K., Eisenreich, W. The intracellular metabolism of legionella by isotopologue profiling. Methods Mol Biol. 954, 163-181 (2013).
- Willenborg, J., et al. Characterization of the pivotal carbon metabolism of Streptococcus suis serotype 2 under ex vivo and chemically defined in vitro conditions by isotopologue profiling. J Biol Chem. 290 (9), 5840-5854 (2015).
- Menzel, R., Ngosong, C., Ruess, L. Isotopologue profiling enables insights into dietary routing and metabolism of trophic biomarker fatty acids. Chemoecology. 27 (3), 101-114 (2017).
- Buse, T., Ruess, L., Filser, J. New trophic biomarkers for Collembola reared on algal diets. Pedobiologia. 56 (3), 153-159 (2013).
- Hutson, B. R. Effects of variations of the plaster-charcoal culture method on a Collembolan, Folsomia candida. Pedobiologia. 18, 138-144 (1978).
- Fountain, M. T., Hopkin, S. P. Folsomia candida (Collembola): a "standard" soil arthropod. Annu Rev Entomol. 50, 201-222 (2005).
- ISO, I. O. f. S. . Soil Quality-Inhibition of reproduction of Collembola (Folsomia candida) by soil pollutants. , (1999).
- Welch, D. F. Applications of cellular fatty acid analysis. Clin Microbiol Rev. 4 (4), 422-438 (1991).
- Dodds, E. D., McCoy, M. R., Rea, L. D., Kennish, J. M. Gas chromatographic quantification of fatty acid methyl esters: flame ionization detection vs. electron impact mass spectrometry. Lipids. 40 (4), 419-428 (2005).
- Kuppardt, S., Chatzinotas, A., Kastner, M. Development of a fatty acid and RNA stable isotope probing-based method for tracking protist grazing on bacteria in wastewater. Appl Environ Microbiol. 76 (24), 8222-8230 (2010).
- Zhang, X., He, H., Amelung, W. A GC/MS method for the assessment of 15N and 13C incorporation into soil amino acid enantiomers. Soil Biology and Biochemistry. 39 (11), 2785-2796 (2007).
- Vetter, W., Thurnhofer, S. Analysis of fatty acids by mass spectrometry in the selected ion monitoring mode. Lipid Technol. 19 (8), 184-186 (2007).
- Thurnhofer, S., Vetter, W. A gas chromatography/electron ionization-mass spectrometry-selected ion monitoring method for determining the fatty acid pattern in food after formation of fatty acid methyl esters. J Agric Food Chem. 53 (23), 8896-8903 (2005).
- Haubert, D., Haggblom, M. M., Scheu, S., Ruess, L. Effects of fungal food quality and starvation on the fatty acid composition of Protaphorura fimata (Collembola). Comparative Biochemistry and Physiology B-Biochemistry & Molecular Biology. 138 (1), 41-52 (2004).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon