È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Acido grasso 13C Isotopologue profilatura fornisce approfondimenti trasferimento trofico carbonio e metabolismo dei lipidi dei consumatori invertebrati marini
In questo articolo
Riepilogo
L'approccio di marcatore trofica dell'acido grasso, vale a dire, l'assimilazione degli acidi grassi come intera molecola e trasferimento nel tessuto dei consumatori con no o minori modifiche, è ostacolata da lacune nel metabolismo dell'acido grasso di invertebrati del suolo piccolo. Isotopologue profilatura viene fornito come un prezioso strumento per districarsi tra interazioni trofiche.
Abstract
Gli acidi grassi (FAs) sono biomarcatori utili in ecologia alimentare web perché in genere sono assimilati come una molecola completa e trasferiti nel tessuto dei consumatori con minore o nessuna modifica, che consente il routing dietetico tra diversi livelli trofici. Tuttavia, l'approccio di trofico marcatore FA è ancora ostacolata dalla conoscenza limitata nel metabolismo dei lipidi della fauna del suolo. Questo studio ha utilizzato interamente etichettato acido palmitico (C16: 013, 99% di atomo) come tracciante nelle vie di metabolismo dell'acido grasso del suolo diffuso due Collembola, Protaphorura fimata e Heteromurus nitidus. Al fine di indagare il destino e metaboliche modifiche di questo precursore, è presentato un metodo di profiling isotopologue, eseguita mediante spettrometria di massa utilizzando Monitoraggio singolo ione. Inoltre, il laboratorio a Monte alimentazione esperimento è descritto, così come l'estrazione e la metilazione delle frazioni lipidiche dominante (lipidi neutri, fosfolipidi) e formula correlata e dei calcoli. Profilatura di Isotopologue non solo rendimento complessivo 13C arricchimento in acidi grassi derivato da 13C etichettato precursore, ma produce anche il modello di isotopomeri supera la massa dello ione genitore (vale a dire, lo ione molecolare FA M+) di ciascuna etichetta FA da uno o più unità di massa (M+ 1, M+ 2, M+ 3, ecc.). Questa conoscenza permette di conclusioni sul rapporto di routing dietetica di un FA interamente consumato rispetto biosintesi de novo . La profilatura di isotopologue è suggerita come uno strumento utile per la valutazione del metabolismo dell'acido grasso in animali di suolo per districare le interazioni trofiche.
Introduzione
In un habitat criptico come il suolo, le relazioni trofiche sono difficili da indirizzo e sono ulteriormente limitate dalle ridotte dimensioni della fauna. Nell'ultimo decennio ha visto progressi in ecologia biochimica, specialmente nell'uso degli acidi grassi come biomarcatori per la definizione di strategie d'alimentazione della fauna del suolo sotto campo condizioni1,2,3. Questa è basata sul fatto che gli acidi grassi dalle risorse possono essere incorporati nel tessuto dei consumatori come molecole intero, un processo definito dietetico routing4. Trasferimento degli acidi grassi è stato segnalato oltre tre livelli trofici, cioè, dai funghi ai nematodi a Collembola5. Recentemente, la fauna predatoria era considerata6,7 e le prime recensioni su acidi grassi come marcatori trofici in reti alimentari del suolo sono stati pubblicati8,9.
Informazioni più dettagliate sulle interazioni trofiche sono raggiunto da acido grasso isotopo stabile probing (FA-SIP). La determinazione di 13C /12C rapporti in acidi grassi nelle diete e i consumatori possono attribuiscono binari collegamenti e stimano il flusso di carbonio associati ed sono stati impiegati in terrestre, acqua dolce e marini food webs10,11 ,12,13. Il presupposto di base è che gli acidi grassi dietetici indirizzati non sono soggetti a processi enzimatici; di conseguenza, il loro 13C segnale, vale a dire, 13C /12C rapporto dell'acido grasso, il consumatore è simile a quella di dieta1. Tuttavia, un progressivo esaurimento della firma 13C lungo la catena alimentare è stato segnalato in sistemi acquatici, ostacolando così vasta applicazione di FA-SIP in studi trofiche14,15,16. Inoltre, la conoscenza nel metabolismo dei lipidi nella maggior parte degli invertebrati in reti alimentari terrestri è ancora limitata.
Comprendere le vie del metabolismo dei lipidi nei consumatori è essenziale per l'utilizzo degli acidi grassi trofiche marcatore come mezzi per la determinazione del flusso di carbonio quantitativa in ecologia alimentare web. Con questo in mente, 13C-isotopologue di profilatura, in linea di principio può essere applicato per le indagini del metabolismo del carbonio di qualsiasi sistema biologico17, è un metodo di promessa. A seguito dell'introduzione di un substrato di carbonio C-etichettati 13, la distribuzione di 13C nella rete metabolica è rintracciabile poiché i prodotti metabolici generati nello spettacolo dei consumatori una distribuzione specifica isotopologue. Questo può essere valutato da quantitativi di risonanza nucleare metabolica spettroscopia18,19 o spettrometria di massa20,21, con i campioni biologici favoriti in quest'ultimi con biomassa bassa a causa della sua maggiore sensibilità.
Anche se isotopologue profilatura è stato correttamente applicato agli aminoacidi e fornito la comprensione nel metabolismo del carbonio in vivo di batteri patogeni17,22,23, sua attuazione in grassi acidi è rimasta indietro. La prima analisi dettagliata sul destino di un isotopo stabile etichettati precursore degli acidi grassi, sua dietetico routing o degradazione attraverso β-ossidazione, nei consumatori di invertebrati del suolo, è stata recentemente eseguita da Menzel et al. 24. qui, le basi metodologiche per esperimenti di incorporazione con 13C etichettati acidi grassi seguiti dall'analisi di isotopologue di chiavi discendenti in invertebrati del suolo frequenti, i collemboli, sono fornite. Questi microartropodi sono un gruppo di buon modello formano importanti componenti della rete alimentare del suolo e sono ben studiati per loro marcatore trofiche acidi grassi8,25.
Comprendere le vie del metabolismo dei lipidi nei consumatori è essenziale per l'utilizzo degli acidi grassi trofiche marcatore come mezzi per la determinazione del flusso di carbonio quantitativa in ecologia alimentare web. Il presente protocollo dà la progettazione e la messa a punto per un laboratorio alimentare esperimento e le procedure biochimiche per estrazione e metilazione delle frazioni di lipidi dominante (lipidi neutri, fosfolipidi) da collemboli. Dimostra come la composizione di isotopologue degli acidi grassi è analizzata tramite spettrometria di massa e descrive la relativa formula e calcoli. Questa procedura comporta: (i) i rapporti di isotopomeri supera la massa dello ione genitore (vale a dire, lo ione molecolare dell'acido grasso M+) da uno o più di massa unità (M+ 1, M+ 2, M+ 3, ecc.) e (ii) la complessiva 13 C arricchimento in acidi grassi derivati dal precursore etichettati 13C. Anche se usato per Collembola, questo approccio in genere può essere applicato a qualsiasi altra interazione predatore-preda sulla premessa che questi sono coltivabili in quantità sufficiente in condizioni controllate per garantire un assorbimento efficace delle etichette e le successive Verifica.
Protocollo
Il protocollo descritto non rientra tra le competenze dell'etica animale. Tuttavia, quando persone adatta i protocolli descritti per gli più alti animali, fare attenzione che il Comitato di etica animale istituzionale ha approvato il protocollo per la movimentazione degli animali.
1. coltivazione degli animali
Nota: Tutto spiegato procedure sperimentali sono basate su protocolli ben stabiliti26,27,28. Biotest in laboratorio hanno bisogno di un approvvigionamento continuo di organismi facilmente coltivabili. Qui, le specie di collemboli Protaphorura fimata (Gisin, 1952) e Hetermurus nitidus (Templeton, 1835) sono state utilizzate. Entrambe le specie sono semplici da mantenere come laboratorio produttivo culture alimentate con lievito di birra.
- In microcosmi di plastica con coperchi ermetici (diametro 7 cm, altezza 4,5 cm), aggiungere una miscela di carbone, gesso di Parigi e distillata acqua per fornire un substrato di allevamento altamente umido (Figura 1A).
- Quando si prepara il mix di microcosmi sufficiente substrato, ad esempio, per 10 microcosmi in un batch. Mix 9 parti di gesso di Parigi (225 g) e 1 parte di asciutto carbone attivo (25 g) insieme in una pentola di intonaco, aggiungere con cautela circa 10 parti di acqua distillata (250 mL) e lasciar per riposare per 5 minuti senza agitazione a temperatura ambiente.
- Mescolare con un cucchiaio di laboratorio a velocità moderata in senso orario per evitare bolle d'aria fino ad ottenere una consistenza densa e densa. Versare immediatamente in microcosmi ad un'altezza di circa 1 cm.
- Lisciare l'intonaco delicato toccando in panchina e vorticoso. Si noti che fori (prodotti casuale di bolle d'aria) e solchi (aggiunti attivamente con una spatola sterile) potrebbero incoraggiare Collembola fertile per deporre le uova lì. Questo studio evitata buche e solchi in favore di avere le stesse condizioni riproducibili. Tuttavia, a scopo dimostrativo Figura 1B presenta alcune buche.
- Lasciare asciugare per circa 1-2 giorni a temperatura ambiente; incubazione a 60 ° C può ridurre quel tempo a 1-2 h.
- Inumidire microcosmi prima dell'uso con l'aggiunta di acqua di rubinetto con una pipetta fino a quando il substrato è leggermente umido. Mantenere umido microcosmo aggiungendo regolarmente acqua distillata come Collembola hanno una cuticola morbida ed è suscettibile di essiccazione.
- Trasferire facilmente Collembola alla base gesso utilizzando un semplice tubo di aspirazione, cioè, un tubo di silicone lungo circa 25 cm lungo con un puntale dotato di una piccola rete per impedire l'aspirazione di animali nel tubo. In alternativa, è possibile trasferire gli animali consentendo loro di aderire sulle setole di uno spazzolino.
- Trasferimento (Vedi punto 1.2) 30 appena schiuso Collembola in nuovi microcosmi e fornire granulato lievito secco come alimento (circa una punta di coltello) (Figura 1B); rinnovare almeno due volte a settimana. Piano tre indipendente replica al giorno di campionamento; in questo studio giorni 0 - 7 e 14. Incubare a 15 ° C al buio. Mantenere che una temperatura costante è essenziale, come gli acidi grassi sono alterati dal metabolismo animale per soddisfare i requisiti per la fluidità della membrana.
- Feed Collembola con lievito di birra per quattro settimane prima di iniziare gli esperimenti di esposizione per ottenere un'omogenea 13C /12C segnale e motivo in acidi grassi. Utilizzare un pennello o un tubo di aspirazione per rimuovere tutte le uova (Figura 1), pellet fecali, ed esuvie regolarmente come animali potrebbe alimentare il loro alterando così il loro profilo lipidico.

Figura 1: coltivazione di collemboli. Microcosmo di (A) riempito con allevamento del substrato, una miscela secca di gesso, carbone attivato e acqua distillata. (B) e (C) esemplare rappresentativo di una cultura Protaphorura fimata ; notare le piccole pepite di lievito secco usato come la fonte di cibo e anche come fori nel substrato di allevamento (freccia nera) (B) così come due uova (freccia bianca) (C). Clicca qui per visualizzare una versione più grande di questa figura.
2. etichettatura dieta, raccolta e trattamento dei campioni
- L'etichettatura
- Quattro settimane dopo aver stabilito le culture Collembola, inserire 30 individui ogni nuovi microcosmi e incubare a 15 ° C al buio.
- Introdurre l'etichetta di impulso di nutrirli lieviti di panificazione contenente un interamente 13marcato C acido palmitico (C16: 0 del13, 99% di atomo) mescolando l'acido palmitico 13C-etichetta con lievito di birra con una spatola a un 0.5:1 di rapporto, ad esempio, 5 g 13 C16: 0 e 10 g di lievito del panettiere. Posto di una punta di coltello su ogni microcosmo.
- Dopo 6 h, sostituire questo con etichetta alimentare con lievito completamente senza etichetta.
-
Ulteriore coltivazione
- Nel corso dell'esperimento rinnovare la dieta lievito ogni tre giorni e aggiungere importi superiori a quello che consumerà Collembola entro tale termine. La cosa più importante, utilizzare un tubo di aspirazione o una spazzola per rimuovere le uova Collembolan, pellet fecali ed esuvie regolarmente per assicurare un'alimentazione esclusiva dagli animali sul cibo fornito.
- Vendemmia
- Microcosmi di campione in maniera distruttiva quotidianamente fino al giorno 7. Poi il giorno 14, raccogliere tre replicati indipendenti in ogni momento di campionamento; tempi di campionamento differenti sono possibili.
- Preparare le provette di vetro 10 mL dotate di tappi a vite in Teflon, uno per ogni campione. In anticipo questi tubi in una rondella di cristalleria e sciacquare due volte con acqua deionizzata in seguito. Infine, per rimuovere eventuali tracce di idrofobo inquinanti lavare due volte con l'aggiunta di 2 mL di cloroformio (grado HPLC), vortice approssimativamente ed eliminare il solvente.
- Per campioni di controllo (giorno 0), prendere 3 30 Collembola non esposte dalle pre-culture come campioni di giorno 0.
- Per i campioni esposti (1 ° giorno in poi), prendere per i campioni ogni giorno in ogni caso 3 30 esposti Collembola dalle colture mediamente alimentati con il mix di 13C-etichettati acido palmitico e lievito di birra.
- Peso fresco Collembola record da un ultramicrobilancia. Trasferire gli animali utilizzando un aspiratore o un pennello per piatti di bilancia appropriate. Per garantire maneggevolezza di collemboli durante la pesatura nei piatti di bilancia, shock-cool animali prima (-80 ° C per 2 h). In alternativa, un flusso di CO2 per 10 min in modo sicuro può stordire gli animali.
- Pesatura direttamente dopo mettere animali dai piatti di bilancia attentamente in provette di vetro da 10 mL. Riempire i tubi con 1 mL di metanolo (grado HPLC) e conservare a-20 ° C fino all'analisi.
Nota: Da questo punto in poi, evitare la gestione di campione con apparecchiature in plastica come solventi organici sono coinvolti; invece di utilizzare erogatori e pipette che sono adatti per solventi nonché i contenitori in vetro.
3. lipidi Estrazione da tessuti animali e Methanolysis
- Preparare tre-provette di vetro (dotate di tappi a vite in Teflon) per ogni batch contenente solo 1 mL di metanolo come valori vuoti. Cosa importante, aggiungere o trasferire qualsiasi solvente utilizzato in questo protocollo solo di pipette in vetro o cloroformio/metanolo risciacquato dispenser resistenti ai solventi.
- All'inizio del processo di estrazione dei lipidi, ridurre il metanolo applicato per l'archiviazione (o tranciatura) per evaporazione; è consigliato un compatto benchtop rotazionale vuoto concentratore (RVC) dotato di una pompa a vuoto e una trappola a freddo. Trasferire i tubi aperti nella RVC ed evaporare fino a secco a 50 ° C e vuoto pressione di 200 hPa per 20 min.
- Aggiungere 5 mL di solvente di estrazione monofase (chloroform/methanol/0.05 M 1:2:0.8 di tampone fosfato, pH 7.4) per ogni campione (spazi vuoti compresi) ed estrarre i lipidi Collembolan a temperatura ambiente agitando una notte (~ 200 giri/min).
- Trasferire il solvente in nuove provette e ri-estrarre campioni agitando per 3 h con un ulteriore 2,5 mL di solvente di estrazione. Dopo di che, combinare gli estratti da entrambi i passaggi; è consigliato l'uso del vetro che pipetta Pasteur. Aggiungere 0,8 mL di cloroformio e 0,8 mL di acqua distillata, poi mescolare e centrifugare a 2.000 g a 20 ° C per 5 min. Infine, permetta che i campioni riposare per 5 min per la separazione di acquoso e cloroformio fasi.
- Per analisi di modello dell'acido grasso, dividere i totali lipidi cellulari di collemboli in lipidi neutri, glicolipidi e fosfolipidi frazioni.
- Per ogni campione, preparare una colonna acido di silice (colonna commerciale con 0,5 g acido silicico, maglia dimensione 100-200 µm, Vedi Tabella materiali) aggiungendo 1 mL di cloroformio (condizionamento). Per velocizzare questo processo, è necessario montare le colonne su un blocco di aspirazione come comunemente usato nella cromatografia per estrazione in fase solida. Non utilizzare aghi in nylon; utilizzare acciaio inossidabile sopra i tubi.
- Dopo il cloroformio utilizzato per il precondizionamento è passato attraverso il trasferimento di colonna la fase di cloroformio inferiore completa di ogni campione per una singola colonna. Per semplificare questa procedura, la fase acquosa superiore può essere rimosso in anticipo. Utilizzare pipette Pasteur in vetro, tuttavia, fare attenzione che le colonne non asciugarsi.
- Successivamente eluire frazioni lipidiche con 5 mL di cloroformio (compresi lipidi neutri acidi grassi, NLFAs), 10 mL di acetone (glicolipidi - non analizzati in questo progetto) e 5 mL di metanolo (tra cui gli acidi grassi del fosfolipide, PLFAs). Raccogliere ogni frazione in vasi di vetro individuali.
- Al termine dell'estrazione, ridurre il cloroformio (NLFAs) e metanolo (PLFAs) tramite evaporazione in un RVC. Trasferire i tubi aperti la RVC ed evaporare fino a secco, ~ 90 min a 60 ° C e un vuoto di 24 hPa.
- Avviare la saponificazione dei lipidi (NLFA e PLFA frazioni) seguendo il protocollo da Welch (1991)29 con aggiunta di 1 mL di soluzione di idrossido di sodio-metanolo (45 g di idrossido di sodio, 150 mL di metanolo e 150 mL di acqua distillata) e incubare a 100 ° C per 30 min in un bagno d'acqua. Raffreddare il campione in acqua ghiacciata per 2 min, poi mettere i campioni torna in panchina e continuare a lavorare a temperatura ambiente.
- Aggiungere lo standard interno a ciascun campione compresi gli spazi vuoti. Scegliere un acido grasso non comune negli organismi sperimentali; Inoltre è possibile utilizzare un acido grasso saturo per minimizzare la perdita tramite fenditura e selezionare una molecola con catena-lunghezza intermedia. Per molti scopi, il dispari nonadecanoic acido (19:0) funziona bene. Quindi, aggiungere 30 µ l di una soluzione di 0,74 mM nel isoottano. La quantità esatta è molto importante - essere sicuri di aver verificato la precisione della vostra pipetta con una microbilancia in anticipo.
- Aggiungere 2 mL di acido cloridrico-metanolo (mix 325 mL di soluzione di acido cloridrico N 6.0 con 275 mL di metanolo), incubare a 80 ° C per 10 min in un bagno d'acqua e raffreddare rapidamente in ghiaccio per 2 min. Questo passaggio è tempo e temperatura sensibile; utilizzare 80 ± 1 ° C e 10 ± 1 min Check con quanti campioni possono andare nel bagno di acqua alla volta per mantenere l'80 ° C.
Nota: Questa procedura consente di esteri metilici di acidi grassi (FAMEs), cioè, acido grasso analiti stabilizzato per la vaporizzazione in gascromatografia. - Infine, aggiungere 1,25 mL di esano/metil etere butilico terziario (1:1) e roccia dolcemente per 10 minuti, poi Centrifugare a 2.000 g per 5 min, rimuovere la fase inferiore e tenere la fase superiore è composto da FAMEs; utilizzare vetro che pipetta Pasteur. Aggiungere 3 mL di idrossido di sodio acquoso (10,8 g di NaOH disciolti in 900 mL di acqua distillata) per una fase di lavaggio.
- Roccia e centrifugare nuovamente.
- Prendere la fase superiore completa per contenenti lipidi di usando un vetro pipetta Pasteur ed il trasferimento di una fiala del campione di gas cromatografia dotata di un setto di Teflon. Evitare di includere anche piccole quantità della fase acquosa, come questo può causare problemi con la misurazione di GC. Incapsulare il flaconcino e conservare a-20 ° C fino all'analisi.
4. quantificazione degli acidi grassi mediante GC-FID
- Utilizzare gas cromatografia per identificare e quantificare la FAMEs nelle frazioni di NFLA e PLFA dei lipidi di collemboli (animale). Un gascromatografo (GC) con rivelatore a ionizzazione di fiamma (FID) è un' apparecchiatura stabilita e comprovata30.
- Per l'identificazione di FAMEs, confrontare il tempo di ritenzione dei picchi del campione a quelli in standard FAME. Si tratta di miscela standard qualitativa o quantitativa della chiave FAMEs che coprono una varietà di prodotti alimentari e organismi.
- Impiegare miscele standard di fama composto da FAs rappresentante per il gruppo di esaminatori organismo e la dieta sperimentale. In Collembola, questi sono acidi grassi caratteristici per gli eucarioti, ad es., acidi grassi polinsaturi a catena lunga come l'acido arachidonico (20:4ω6). Quando si impiegano lievito come dieta queste sono marcatori fungini come l'acido linoleico (18:2ω6). Una buona scelta è il cosiddetto mix di fama, che comprende 37 diversi acidi grassi frequenti in animali, fungini e materiale vegetale e l'estere metilico acido batterico (Borgogna) mix (Vedi Tabella materiali).
- Per eseguire gli esempi su GC-FID, è possibile impostare una sequenza con il rispettivo software dello strumento. Per istruzioni, consultare il manuale del produttore. La sequenza inizia con le miscele standard esterne (ad es. FAME e Borgoglio mix), seguite dai campioni. Si noti che c'è uno spostamento di tempo ritenzione, cioè, un leggero ritardo nel tempo di eluizione degli acidi grassi dalla colonna GC con ogni esecuzione, durante l'esecuzione di campioni! O includere uno standard ogni 10th eseguiti nella sequenza del campione o utilizzare il tempo di ritenzione di bloccaggio per l'acido palmitico (16:0).
- Adattare le impostazioni GC dipendente sullo strumento. Il seguente programma è suggerito per una colonna capillare (HP) ad alte prestazioni (25 m x 0,2 mm i.d., spessore 0,33 µm. iniezione Set volume a 1 µ l in modalità splitless della pellicola e utilizzare l'idrogeno come gas di trasporto. Impiegare un inizio di programma di temperatura a 50 ° C (tenuto per 1 min) e l'aumento di 25 ° C min-1 a 175 ° C, seguita da 3 ° C min-1 a 230 ° C (tenuto per 5,7 min).
- Calcolare l'acido grasso nmol per grammo di peso fresco (a secco) organismi con la risposta ottenuta da FID per ogni FAME applicando la quantità note per il rispettivo acido grasso utilizzando la seguente formula:
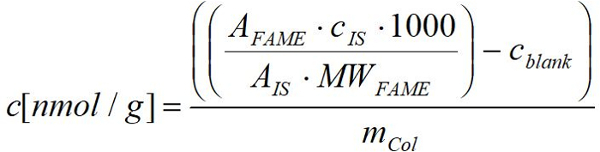
Unafama: area del picco della fama rispettiva nel campione
MW,FM: peso molecolare della rispettiva fama in µ g/µmol
CIS: concentrazione dello standard interno in µ g
UnIS: area del picco dello standard interno
mCol: peso fresco (a secco) di rispettivi Collembola campione in g
1000: fattore di conversione da µmol nmol
cBW: concentrazione della rispettiva fama in media dei corrispondenti valori vuoti in nmol
5. analisi di 13C mediante profilatura, Isotopologue
- Utilizzo un sistema Gascromatografico accoppiato ad un rivelatore selettivo di massa (MS) fornito con un'ionizzazione dell'elettrone sorgente (EI) per la determinazione di isotopologue.
- Usare una colonna capillare polare (ad es., 23 DB, CP-Sil 88) come questo ulteriore permette la separazione di acidi grassi insaturi anche con lo stesso numero di doppi legami. La scelta della colonna GC è fondamentale per i risultati come determina la buona rappresentazione dello ione molecola negli acidi grassi.
- Per una colonna di 23 DB (60 m x 0,25 mm i.d., pellicola spessore 0,15 µm), inizia la temperatura del forno a 130 ° C e aumenta di 6,5 ° C/min a 170 ° C. Seguire con un aumento di 3 ° C/min a 203 ° C premuto per 1,9 minimo seguire con un aumento di 40 ° C/min a 230 ° C e tenere per temperatura della linea di trasferimento Set minimo 8,3 a 280 ° C. Ancora una volta, regolare metodo GC allo strumento.
- Utilizzare degli standard quantitativi composto da quantità note di FAMEs per tutti gli acidi grassi per essere studiati per incorporazione 13C. Mettere questi standard all'inizio e alla fine di ogni sequenza di esempio eseguita. Prendere i tempi di ritenzione degli acidi grassi di interesse da questi standard.
- Campioni di misura dagli esperimenti sempre a partire con i campioni senza etichetta e le sonde etichettate da allora in poi. Applicare un rapporto di divisione appropriato per le concentrazioni di campione, ad esempio 1:12.5.If disponibile per lo strumento, applicare un lavaggio a controcorrente con Elio dopo ogni campione eseguito alla radura fuori colonna da analiti rimanenti.
- Applicare il blocco al metodo GC-MS in modo che l'acquisizione di SIM non soffre di spostamento e c'è i tempi di ritenzione dell'analita riproducibile del tempo di ritenzione.
- Determinare incorporazione di 13C in ione molecolare degli acidi grassi mediante GC/EI-MS utilizzando ioni selezionati (SIM) modalità dello strumento di monitoraggio. Funzionamento in modalità SIM permette di rilevare analiti specifici con maggiore sensibilità relativo alla modalità di scansione completa.
- Eseguire una scansione iniziale prima di vedere ciò che è presente e quindi eseguire SIM sugli ioni appropriati. Acquisire i dati a masse molecolari di interesse selezionando finestre di scansione di m/z (gruppi di SIM) che comprende il tempo di picco cromatografico del rispettivo di acidi grassi. In genere, monitorare gli ioni di due a quattro per ogni analita e tempo finestra.
- Al fine di aumentare la sensibilità, regolare la velocità di scansione di massa e dimorare volte (il tempo trascorso guardando ogni messa). I dati di qualità migliori sono ottenuti alla minima velocità possibile e un generalmente regola in SIM è 8-12 scansioni oltre il picco di analita. Un proxy per le impostazioni dello strumento è un tempo di permanenza medio per massa di 9 ms, un tempo di ciclo di 6 s e un tempo di scansione di 175 ms cyle-1.
- Rilevare lo ione molecolare (M+) l'acido grasso rispettivo e tutti i suoi isotopomeri (M+ 1, M+ 2 e così via). Per esempi, vedere risultati di rappresentante.
- Registrare l'abbondanza di ogni frammento di ioni (isotopologue). Si noti che l'abbondanza dello ione molecola e la sua isotopomeri è relativamente basso e la qualità di quantificazione dipende notevolmente le prestazioni del sistema MS. Eseguire un regolare prima di iniziare una sequenza di grande campione (esperimento) e se necessario, pulire la sorgente di ioni.
Nota: In primo luogo, questi dati resa complessiva 13C arricchimenti di ogni acidi grassi dal precursore consumato (qui 16:0). - Usare la proporzione tra la composizione isotopica dei coniugati acidi grassi dalle loro controparti contrassegnati per assegnare il flusso di carbonio trofiche. Utilizzare la formula di % di atomo nel passaggio 6.1 per calcolare la percentuale di carbonio etichettato (atomo per cento, atomo %) nell'acido grasso rispettivi. Confrontare la percentuale di 13C in acidi grassi di collemboli tra etichettato (giorno 1 e versioni successivo) e animali senza etichetta (giorno 0) come indicazione relativa per il flusso di 13C dalla dieta nel consumatore.
- Assegnare la posizione di 13C incorporazione nella catena dell'acido grasso. Basato sulla distribuzione di districare gli isotopomeri il routing dietetico dell'acido grasso intero tracciante marcato (qui 16:0) dall'allungamento della catena di metabolismo dei lipidi. Mentre l'assimilazione della molecola intero marcatore aumenta l'abbondanza di isotopomeri più lontana allo ione genitore (M+), ad esempio, M+ 15M+ 16con l'allungamento della catena mediante l'uso di 13C etichettati C2 frammenti (13C acetil-CoA) isotopomeri vicino lo ione molecolare fatti più frequenti.
6. calcoli di arricchimento 13C
- Secondo la ripartizione dell'isotopomeri, calcolare la percentuale globale del carbonio etichettato (in % di atomo) rispettivi nell'acido grasso utilizzando la seguente relazione: atomo % = (rapporto 13C isotopologue incorporato) x (frequenza rispettivi isotopologue)
Questo viene calcolato dopo Kuppardt et al. 31 come: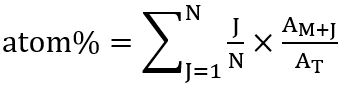
dove N è il numero di atomi di carbonio nell'acido grasso, J è il numero di isotopi 13C, unaM + J è l'abbondanza dei rispettivi isotopologue e unT l'abbondanza totale di tutti gli isotopomeri. - Per il calcolo, sommare i valori di zona di picco dello ione molecolare (M+) della rispettiva FA e tutti i isotopomeri (M+ 1, M+ 2e così via), rilevato da analisi SIM-MS e impostato su 100% relativa abbondanza. Calcolare che la porzione di ogni isotopologue rilevato è facilmente seguendo la regola del tre.
- Sottrarre il valore del controllo non etichettati giorno 0 (sfondo naturale 13C) dai valori sperimentali per ottenere dati definitivi solo risalire al esterni eseguiti 13C-etichettatura.
Risultati
Contenuti freschi di peso e del lipido di collemboli
Nel corso dell'esperimento descritto, il contenuto in NLFAs e PLFAs non è cambiato significativamente nel corso del tempo, mentre il peso fresco degli esemplari è aumentato leggermente ma non significativamente24. Entrambi i parametri indicano un buon livello di forma fisica degli esemplari collemboli. Essere consapevoli di indagare del lipido e peso contenuti freschi di collemboli in tutto ...
Discussione
Profilo di Isotopologue
Un'analisi dettagliata degli aspetti quantitativi nella distribuzione di 13C in FAs ha bisogno di una tecnologia all'avanguardia per assegnare carbonio partizionamento in reti alimentari. Il presente lavoro impiegato isotopologue profilatura per valutare il 13C /12C rapporti in comune biomarcatori FA per interazioni tropici. Questo metodo è affermato per l'analisi dell'amminoacido mediante cromatografia liquida (LC-MS) ...
Riconoscimenti
Si ringraziano il sostegno finanziario di Menzel R. e L. Ruess dalla Deutsche Forschungsgemeinschaft (RU RU780/11-1). R. Nehring è stato finanziato da RU 780/10-1. Infine, siamo estremamente grati a Dr. Hazel Ruvimbo Maboreke per la correzione del nostro manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| neoLab-Round jars | neoLab | 2-1506 | 69 x 40 mm, 10 pacs/pack |
| Charcoal activated | Carl Roth | X865.1 | p.a., powder, CAS No. 7440-44-0 |
| Alabaster Dental | RÖHRICH-GIPSE | --- | http://www.roehrich-gipse.de/dentalgipse.php |
| Chloroform | Carl Roth | 7331.1 | HPLC ≥ 99,9 % |
| Methanol | Carl Roth | P717.1 | HPLC ≥ 99,9 % |
| Hexan | Carl Roth | 7339.1 | HPLC ≥ 98 % |
| tert-Butyl methyl ether (MTBE) | Carl Roth | T175.1 | HPLC ≥ 99,5 % |
| Aceton | Carl Roth | 7328.2 | HPLC ≥ 99,9 % |
| NaOH | Carl Roth | 6771.1 | p.a. ≥99 %, in pellets |
| di-Natriumhydrogenphosphat | Carl Roth | P030.1 | p.a. ≥99 % , water free |
| Na-dihydrogenphosphat Dihydrat | Carl Roth | T879.1 | p.a. ≥99 % |
| Hypochloric acid (6 N) | VWR International | 26,115,000 | AVS TITRINORM vol. solution |
| Bond Elut (Columns) | Agilent Tech. | 14102037 | HF Bond Elut-SI, 500 mg, 3 mL, 50/PK |
| Präparatengläser Duran | Glasgerätebau Ochs | 135215 | Ø 16 x 100 mm, plus screw cap with handy knurl and integrated PTFE/silicone gasket |
| Supelco 37 Component FAME Mix | Sigma-Aldrich | 47885-U Supelco | 10 mg/mL in methylene chloride, analytical standard |
| FlowMesh | Carl Roth | 2796.1 | Polypropylene mesh, approximately 0.3 mm thick, with 1 mm strand spacing |
| Bacterial Acid Methyl Ester (BAME) Mix | Sigma-Aldrich | 47080-U Supelco | 10 mg/mL in methyl caproate, analytical standard |
| Methyl nonadecanoate | Sigma-Aldrich | 74208 | analytical standard ≥ 98.0 % |
| Hexadecanoic acid-1-13C (Palmitic) | Larodan Fine Chemicals | 78-1600 | GC ≥ 98.0 % (13C: 99.0 %) |
| RVC 2-25 CDplus | Martin Christ Gefrier-trocknungsanlagen | Compact benchtop midi concentrator | |
| Alpha 2-4 LDplus | Martin Christ Gefrier-trocknungsanlagen | Drying manifold | |
| MZ 2C NT | Vacuubrand GMBH | Vacuum pump | |
| Roto-Shake Genie | Scientific Industries | Combined rocking and rotating device | |
| XP64 Micro Comparator | Mettler Toledo | Super high precision balance | |
| GC-System 7890A | Agilent Tech. | Gas chromatograph | |
| 7000 GC/MS Triple Quad | Agilent Tech. | Triple Quad mass spectrometer | |
| 7683B Series Injector | Agilent Tech. | Sample injector | |
| Heraeus Multifuge 3SR+ | Thermo Scientific | Centrifuge with 10 ml tube rotor |
Riferimenti
- Ruess, L., et al. Application of lipid analysis to understand trophic interactions in soil. Ecology. 86 (8), 2075-2082 (2005).
- Ruess, L., et al. Lipid composition of Collembola and their food resources in deciduous forest stands - Implications for feeding strategies. Soil Biology and Biochemistry. 39 (8), 1990-2000 (1990).
- Chamberlain, P. M., Bull, I. D., Black, H. I. J., Ineson, P., Evershed, R. P. Fatty acid composition and change in Collembola fed differing diets: identification of trophic biomarkers. Soil Biology and Biochemistry. 37 (9), 1608-1624 (2005).
- Stott, A. W., Davies, E., Evershed, R. P., Tuross, N. Monitoring the routing of dietary and biosynthesised lipids through compound-specific stable isotope (delta C-13) measurements at natural abundance. Naturwissenschaften. 84 (2), 82-86 (1997).
- Ruess, L., Haggblom, M. M., Langel, R., Scheu, S. Nitrogen isotope ratios and fatty acid composition as indicators of animal diets in belowground systems. Oecologia. 139 (3), 336-346 (2004).
- Pollierer, M. M., Scheu, S., Haubert, D. Taking it to the next level: Trophic transfer of marker fatty acids from basal resource to predators. Soil Biology and Biochemistry. 42 (6), 919-925 (2010).
- Ferlian, O., Scheu, S., Pollierer, M. M. Trophic interactions in centipedes (Chilopoda, Myriapoda) as indicated by fatty acid patterns: Variations with life stage, forest age and season. Soil Biology and Biochemistry. 52, 33-42 (2012).
- Ruess, L., Chamberlain, P. M. The fat that matters: Soil food web analysis using fatty acids and their carbon stable isotope signature. Soil Biology and Biochemistry. 42 (11), 1898-1910 (2010).
- Traugott, M., Kamenova, S., Ruess, L., Seeber, J., Plantegenest, M. Empirically characterising trophic networks: What emerging DNA-based methods, stable isotope and fatty acid analyses can offer. Adv Ecol Res. 49, 177-224 (2013).
- Hammer, B. T., Fogel, M. L., Hoering, T. C. Stable carbon isotope ratios of fatty acids in seagrass and redhead ducks. Chemical Geology. 152 (1-2), 29-41 (1998).
- Budge, S. M., Iverson, S. J., Koopman, H. N. Studying trophic ecology in marine ecosystems using fatty acids: A primer on analysis and interpretation. Marine Mammal Science. 22 (4), 759-801 (2006).
- Haubert, D., et al. Trophic structure and major trophic links in conventional versus organic farming systems as indicated by carbon stable isotope ratios of fatty acids. Oikos. 118 (10), 1579-1589 (2009).
- Ngosong, C., Raupp, J., Richnow, H. H., Ruess, L. Tracking Collembola feeding strategies by the natural 13C signal of fatty acids in an arable soil with different fertilizer regimes. Pedobiologia. 54 (4), 225-233 (2011).
- Bec, A., et al. Assessing the reliability of fatty acid-specific stable isotope analysis for trophic studies. Methods in Ecology and Evolution. 2 (6), 651-659 (2011).
- Gladyshev, M. I., Makhutova, O. N., Kravchuk, E. S., Anishchenko, O. V., Sushchik, N. N. Stable isotope fractionation of fatty acids of Daphnia fed laboratory cultures of microalgae. Limnologica. 56 (Supplement C. 56 (Supplement C), 23-29 (2016).
- Gladyshev, M. I., Sushchik, N. N., Kalachova, G. S., Makhutova, O. N. Stable isotope composition of fatty acids in organisms of different trophic levels in the Yenisei river. PLoS One. 7 (3), e34059 (2012).
- Eisenreich, W., Dandekar, T., Heesemann, J., Goebel, W. Carbon metabolism of intracellular bacterial pathogens and possible links to virulence. Nature Reviews Microbiology. 8 (6), 401-412 (2010).
- Eylert, E., Bacher, A., Eisenreich, W. NMR-based isotopologue profiling of microbial carotenoids. Methods Mol Biol. 892, 315-333 (2012).
- Garton, N. J., O'Hare, H. M. Tuberculosis: feeding the enemy. Chemical Biology. 20 (8), 971-972 (2013).
- Rosenblatt, J., Chinkes, D., Wolfe, M., Wolfe, R. R. Stable isotope tracer analysis by GC-MS, including quantification of isotopomer effects. Am J Physiol. 263 (3), E584-E596 (1992).
- Fernandez, C. A., Des Rosiers, C., Previs, S. F., David, F., Brunengraber, H. Correction of 13C mass isotopomer distributions for natural stable isotope abundance. J Mass Spectrom. 31 (3), 255-262 (1996).
- Heuner, K., Eisenreich, W. The intracellular metabolism of legionella by isotopologue profiling. Methods Mol Biol. 954, 163-181 (2013).
- Willenborg, J., et al. Characterization of the pivotal carbon metabolism of Streptococcus suis serotype 2 under ex vivo and chemically defined in vitro conditions by isotopologue profiling. J Biol Chem. 290 (9), 5840-5854 (2015).
- Menzel, R., Ngosong, C., Ruess, L. Isotopologue profiling enables insights into dietary routing and metabolism of trophic biomarker fatty acids. Chemoecology. 27 (3), 101-114 (2017).
- Buse, T., Ruess, L., Filser, J. New trophic biomarkers for Collembola reared on algal diets. Pedobiologia. 56 (3), 153-159 (2013).
- Hutson, B. R. Effects of variations of the plaster-charcoal culture method on a Collembolan, Folsomia candida. Pedobiologia. 18, 138-144 (1978).
- Fountain, M. T., Hopkin, S. P. Folsomia candida (Collembola): a "standard" soil arthropod. Annu Rev Entomol. 50, 201-222 (2005).
- ISO, I. O. f. S. . Soil Quality-Inhibition of reproduction of Collembola (Folsomia candida) by soil pollutants. , (1999).
- Welch, D. F. Applications of cellular fatty acid analysis. Clin Microbiol Rev. 4 (4), 422-438 (1991).
- Dodds, E. D., McCoy, M. R., Rea, L. D., Kennish, J. M. Gas chromatographic quantification of fatty acid methyl esters: flame ionization detection vs. electron impact mass spectrometry. Lipids. 40 (4), 419-428 (2005).
- Kuppardt, S., Chatzinotas, A., Kastner, M. Development of a fatty acid and RNA stable isotope probing-based method for tracking protist grazing on bacteria in wastewater. Appl Environ Microbiol. 76 (24), 8222-8230 (2010).
- Zhang, X., He, H., Amelung, W. A GC/MS method for the assessment of 15N and 13C incorporation into soil amino acid enantiomers. Soil Biology and Biochemistry. 39 (11), 2785-2796 (2007).
- Vetter, W., Thurnhofer, S. Analysis of fatty acids by mass spectrometry in the selected ion monitoring mode. Lipid Technol. 19 (8), 184-186 (2007).
- Thurnhofer, S., Vetter, W. A gas chromatography/electron ionization-mass spectrometry-selected ion monitoring method for determining the fatty acid pattern in food after formation of fatty acid methyl esters. J Agric Food Chem. 53 (23), 8896-8903 (2005).
- Haubert, D., Haggblom, M. M., Scheu, S., Ruess, L. Effects of fungal food quality and starvation on the fatty acid composition of Protaphorura fimata (Collembola). Comparative Biochemistry and Physiology B-Biochemistry & Molecular Biology. 138 (1), 41-52 (2004).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon