Method Article
大鼠甲基 Seq 平台识别与应力接触相关的表观变化
摘要
在这里, 我们描述了同道平台甲基 Seq 的协议和实现, 使用老鼠模型来识别与慢性应力暴露相关的表观变化。结果表明, 大鼠甲基序列平台能够检测大鼠应力暴露引起的甲基化差异。
摘要
随着各种动物的基因组变得可用, 越来越多的工具需要能够捕捉这些动物模型中的动态表观变化。鼠是一种特殊的模型动物, 表观遗传学工具可以补充许多药理学和行为研究, 提供有见地的机械信息。为此, 我们对大鼠的用水靶捕获系统 (称为甲基序列) 进行了调整, 可以评估整个大鼠基因组的 DNA 甲基化水平。大鼠设计有针对性的促进者、CpG 岛、海岛海岸和所有磁铁基因的富 GC 区域。
为实现大鼠实验平台, 雄性杜勒大鼠在3周内暴露于慢性变应激中, 采集血液标本进行基因组 DNA 提取。采用剪切、转接头、靶向富集、亚硫酸氢盐转化、多路复用等方法, 建立了大鼠 DNA 样品的甲基序列文库。在下一代测序平台上对库进行了排序, 并对测序读数进行了分析, 以确定压力和非重读大鼠 DNA 之间的 DMRs。采用亚硫酸氢盐磷酸测序对候选 DMRs 进行了独立验证, 以确认平台的鲁棒性。
结果表明, 大鼠甲基 Seq 平台是一种有用的表观遗传工具, 可捕获受应力影响的甲基化变化。
引言
高通量测序的进展导致了模型和非模型生物体的大量基因组序列。这些序列的可用性促进了遗传学、比较基因组学和转录组学的研究。例如, 可用的基因组序列对于将序列数据与基于其与组蛋白修饰1或亚硫酸氢盐测序相结合的芯片 Seq 实验进行排列, 是非常有用的, 后者可测量 dna 甲基化未甲基化胞嘧啶2的亚硫酸氢盐转化形成的尿嘧啶检测。然而, 由于缺乏可影响基因功能的特定物种调控序列的注解数据, 在同道平台的实施中出现了延迟, 将可用的基因组测序数据纳入其设计中。
特别是, dna 甲基化是对 dna 的最广泛研究的表观遗传修饰之一, 可以利用可用的基因组数据构建 methylomic 平台。一个这样的例子是人类甲基化3的基于阵列的平台, 它已广泛应用于各种学科, 从肿瘤学到精神病学4,5。不幸的是, 与非人类动物模型相似的平台是稀缺的, 因为几乎没有广泛使用的平台在最初的设计中利用了基因组序列。
评估非人类动物模型 methylomic 景观的常用方法是降低亚硫酸氢盐测序 (RRBS)6。这种方法克服了全基因组亚硫酸氢盐测序的成本, 在提供全面的 methylomic 景观的同时, 在基因组的大基因贫乏区域中, 由于成本和有限的功能信息, 提供了较低的读深覆盖率2.RRBS 涉及限制消化和基因组 DNA 的大小选择, 以丰富高 GC 丰富的序列, 如 CpG 岛, 通常在基因促进者附近发现, 并认为在基因调控7中发挥作用。虽然 RRBS 方法已经在许多重要研究中得到了应用, 但它对限制性酶的依赖并不是没有显著的挑战和局限性。例如, RRBS 中富含 GC 的序列的富集完全依赖于由限制性酶识别的特定序列的存在和电泳后的大小选择。这意味着在选择大小时, 不包含这些限制站点的任何基因组区域都将被排除在外。此外, 跨物种比较是具有挑战性的, 除非相同的限制地点存在于不同物种之间相同的位置。
克服 RRBS 局限性的一种方法是使用在平台设计中利用已发布的基因组序列的富集方法。基于阵列的人类平台使用专为特定 CpGs 设计的引物探针, 用于在亚硫酸氢盐转化后进行靶向退火和底漆扩展。它的设计不仅反映了现有的人类基因组序列, 而且还体现了经过实验验证的监管区域从多行查询中获得, 例如编码和 ENSEMBL8。尽管在人类 methylomic 调查中广泛使用, 但模型动物并不存在类似的平台。此外, 基于阵列的格式在可用于探测放置的表面区域上放置了显著的约束。在过去的几年中, 我们已经努力将捕获探针设计所提供的目标特异性与下一代测序的高通量特性相结合。这种努力导致了基于序列的小鼠基因组 (小鼠甲基 Seq) 的靶向浓缩系统, 用于识别脑特异性或糖皮质激素诱导的甲基化9、10的差异。对于其他模型和非模型动物, 需要类似的平台来促进这些动物的同道研究。
在这里, 我们演示了这个新平台的实施, 对大鼠进行 methylomic 分析。鼠是药理学、代谢、神经内分泌学和行为的重要动物模型。例如, 越来越需要了解引起药物毒性、肥胖、压力反应或吸毒成瘾的潜在机制。能够捕获与这些条件相关的 methylomic 变化的高通量平台将增加我们对这些机制的理解。由于大鼠基因组仍缺乏对监管区域的注释, 我们将非冗余启动子、CpG 岛、海岛海岸11和以前确定的富 GC 序列纳入大鼠甲基 Seq 平台12。
为了评估成功的设计和实施用水目标浓缩 (一般称为甲基 Seq) 平台的大鼠基因组, 我们采用了大鼠慢性变应激 (CVS)13模型, 以确定差异甲基化不重读和强调动物之间的区域。我们的平台设计、协议和实施对于那些可能希望对基因组序列已经可用但仍未标注注释的有机体进行全面、无偏见的表观遗传学调查的调查人员可能很有用。
研究方案
所有的实验都是按照所有相关的监管和机构准则完成的, 包括约翰·霍普金斯医学院的机构动物护理和使用委员会。
1. 动物
- 在4周内获得男性青春期杜勒大鼠。在一个温度和湿度控制的房间, 在一个12小时的光, 12 小时的黑暗周期, 在0600小时的光起始, 在聚碳酸酯鼠笼中的动物房子. 为动物提供随意水的广告。
- 允许老鼠适应1周来减少与交通有关的压力。配对动物 (N=16), 以排除隔离应力, 并在5周的年龄, 开始慢性可变应力 (CVS) 方案3周。
2. 慢性变应激
- 在早上 (9-11 AM) 和下午 (在上午) 一次在不规则的时间管理 CVS 方案, 以保持常规不可预知。合并过夜轻度压力源。CVS 方案包括: 1) 3 h 在一个约束缸;2) 10 分钟游泳;3) 3 h 笼型倾斜 4) 1 h 慢震动平台;和 5) 1 小时在4摄氏度冷室。
注意: 过夜压力包括社交拥挤 (每笼 5)、社会隔离、湿寝具、食品限制和照明。表 1提供了一个典型的压力方案每周时间表。
3. 内分泌检测
-
使用尾血 (约50毫升) 采样收集在同一时间 (上午9点) 的皮质酮 (CORT) 的水平在整个实验中, 在 cvs 方案建立基线激素水平 (天 0) 之前, 在每周 cvs 的中间 (天411和 18), 每7天的 cvs (天7和 14), 并在 cvs 的结论 (天 21)。在每日压力方案之前收集血样。
- 在安乐死 (25 天) 中收集一个最终的树干血液样本, 用于 RIA 和基因组 DNA 提取。
- 离心所有血液样本 (600 x g, 4 摄氏度, 10 分钟), 将血浆与血细胞分离。取出血浆 (上清液), 并将样品储存在-80 摄氏度。
- 解冻和使用血浆通过放射免疫测定 (RIA) 确定 CORT 水平。确保压力动物的3周血浆 CORT 水平升高, 以验证应力方案的鲁棒性。
4. 行为
- 在 CVS 方案 (天 23–24) 后, 评估每个动物在高加迷宫 (EPM)14的焦虑样行为。
- 使用摄像机, 在 EPM 设备上记录300s 的动物, 并在中心、闭合的武器和张开的双臂中得分。
5. 大鼠甲基 Seq 的设计
- 使用 UCSC 基因组浏览器, 为 cpg 群岛和岛屿海岸 (1 kb 侧翼 cpg 群岛)、每个磁铁基因的启动器 (± 1 kb) 和其他可能可供使用的序列获取非冗余基因组坐标 (大鼠 2004年11月 rn4 组件)。相关文献。
注: 对于大鼠甲基 Seq, 添加了来自先前基于阵列的甲基化平台的额外的富 GC 序列12。对于大于 5 kbps 的区域, 500 bps 的交替区域采样后跟随 1 kbps 跳过。最终的大鼠甲基序列设计包括 111 Mbps, 230万 CpGs;和平均区域大小为 594 bps。它针对228800独特的位点。 - 输入一个已编译的基因组坐标列表到一个商业可用的目标捕获设计软件, 以进行适当的探头设计。
6. 从基因组 DNA 中构建大鼠甲基序列文库
注意: 要消除批次效果, 请同时处理多个样本, 并相应地放大主混音。使用商业上可用的 dna 提取试剂盒提取 dna。基于柱或沉淀的方法都产生高质量的基因组 DNA (260/280 比值 ~ 1.8)。不建议使用基于苯酚的方法。在低 TE 缓冲液中洗脱或重悬 DNA (10 毫米 te, 0.1 毫米 EDTA, pH 8.0)。
- 样品制备
注: 每步使用 DNA 结合磁珠, 确保珠子在室温下适应至少30分钟, 并在使用前混合好。- 剪切 DNA
- 使用荧光计确定每个样本的初始双链 DNA 浓度。稀释 > 1 µg gDNA 至50µL, 低 TE 缓冲液 (10 毫米 te, 0.1 毫米 EDTA, pH 8.0), 低 DNA 结合微量离心管。
- 使用等温超声仪的剪切样品 (10% 占空比, 5 强度, 每爆裂200次, 6 个周期六十年代, 频率扫描, 4 °c)。
- 使用基于电泳的系统测量 dna 的大小和数量来评估 dna 的质量。
注: 推荐的 DNA 量为1µg 或3µg。如果起始材料有限, 最低输入量应为 > 500 ng, 因为较低的数量将对生成的库的数量和质量产生不利影响。
- 修复 DNA 末端。
- 使用大鼠甲基 Seq 试剂盒来准备冰上的末端修复大师组合。在每个样品中加入52µL 混合物, 在没有加热盖的热循环仪中孵育 (20 摄氏度, 30 分钟, 4 摄氏度保持)。
端修复主混音 (每样):
35.2 µL 水
10µL 末端修复缓冲器 (10x)
1.6 µL dNTP 混合
1µL T4 DNA 聚合酶
2µL 克列诺 DNA 聚合酶
2.2 µL T4 多核苷酸激酶 - 使用180µL 的 DNA 结合磁珠和400µL 新鲜制备的70% 乙醇样品纯化样品。每样添加180µL 的珠子, 室温下孵育5分钟。球团珠, 除去上清和重悬颗粒在200µL 70% 乙醇。去除乙醇, 重复洗涤一次。
- 使用磁性板颗粒珠和去除尽可能多的乙醇。在37摄氏度 heatblock 干燥, 直到珠颗粒完全干燥。在44µL 无核酸酶的水中重悬, 收集大约42µL 的上清液。
停止点: 修复 DNA 末端后, 样品可以密封并储存在-20 摄氏度。
- 使用大鼠甲基 Seq 试剂盒来准备冰上的末端修复大师组合。在每个样品中加入52µL 混合物, 在没有加热盖的热循环仪中孵育 (20 摄氏度, 30 分钟, 4 摄氏度保持)。
- 腺苷酸 3 ' 末端。
- 在冰上准备 Adenylation 大师组合。在每个样品中加入9µL 混合物, 在没有加热盖子的热循环仪中孵育 (37 摄氏度, 30 分钟, 4 摄氏度保持)。
Adenylation 主混音 (每样):
5µL 克列诺缓冲器
1µL 储存
3µL 克列诺 DNA 聚合酶 - 使用90µL 的 DNA 结合磁珠和400µL 新鲜制备的70% 乙醇样品纯化样品。每样添加90µL 的珠子, 室温下孵育5分钟。球团珠, 除去上清和重悬颗粒在200µL 70% 乙醇。去除乙醇, 重复洗涤一次。
- 使用磁性板颗粒珠和去除尽可能多的乙醇。在37摄氏度 heatblock 干燥, 直到珠颗粒完全干燥。在35µL 无核酸酶的水中重悬, 收集大约33.5 µL 的上清液。
- 在冰上准备 Adenylation 大师组合。在每个样品中加入9µL 混合物, 在没有加热盖子的热循环仪中孵育 (37 摄氏度, 30 分钟, 4 摄氏度保持)。
- 结扎甲基化适配器。
- 准备在冰上的结扎大师组合, 并添加16.5 µL 混合到每个样本。在没有加热盖的热循环仪中孵育 (20 摄氏度, 15 分钟, 4 摄氏度保持)。
结扎母料混合 (每样):
2.5 µL 水
2.5 µL 甲基序列甲基化适配器
10µL T4 DNA 连接酶缓冲液 (5x)
1.5 µL T4 DNA 连接酶 - 使用90µL 的 DNA 结合磁珠和400µL 新鲜制备的70% 乙醇样品纯化样品。每样添加90µL 的珠子, 室温下孵育5分钟。颗粒状珠子, 除去上清液, 在200µL 70% 乙醇中重悬颗粒。去除乙醇, 重复洗涤一次。
- 使用磁性板颗粒珠和去除尽可能多的乙醇。在37摄氏度 heatblock 干燥, 直到珠颗粒完全干燥。在22µL 无核酸酶的水中重悬, 收集大约22µL 的上清液。使用 bioanalyzer 评估质量。
注: 如果 dna 总量小于 500 ng, 则在后续步骤之前进行剪切和处理附加 dna。如果平均 dna 大小不增加超过 30 bps, 检查以确保试剂是新的, 因为 T4 DNA 聚合酶, 克列诺和/或 T4 连接可能是旧的。
停止点: 结扎甲基化适配器后, 样品可以密封并储存在-20 摄氏度。
- 准备在冰上的结扎大师组合, 并添加16.5 µL 混合到每个样本。在没有加热盖的热循环仪中孵育 (20 摄氏度, 15 分钟, 4 摄氏度保持)。
- 剪切 DNA
- 杂交
- 将样品转移到低 DNA 结合微量离心管, 并使用加热真空浓缩器将样品体积降至小于3.4 µL. 将样品重组至3.4 µL。
注: 将样品浓缩至约3µL, 确保在所有液体蒸发前将样品从真空浓缩器中除去。 - 在室温下制备杂交缓冲液, 在冰上进行甲基序块混合。在每个样品中加入5.6 µL 的甲基 Seq 块混合物, 在热循环仪中孵育 (95 摄氏度5分钟, 65 摄氏度, 2 分钟, 65 摄氏度保持)。
杂交缓冲液 (每个样本):
6.63 µL 甲基 Hyb 1
0.27 µL 甲基 Hyb 2
2.65 µL 甲基 Hyb 3
3.45 µL 甲基 Hyb 4
甲基 Seq 块混合 (每个样本):
2.5 µL 甲基 Seq 索引块1
2.5 µL 甲基序块2
0.6 µL 甲基序块3 - 准备核糖核酸块混合和捕获库杂交组合。添加20µL 的捕获库杂交混合到每个样本和孵育在65摄氏度至少16小时。
核糖核酸块混合 (每个样本):
0.5 µL 核糖核酸块
1.5 µL 水
捕获库杂交组合 (每个样本):
13µL 杂交缓冲液
2µL 核糖核酸块混合
5µL 大鼠甲基 Seq 捕获库
注: 添加杂交组合以防止非特定绑定时,请保持65摄氏度的反应。 - 等分50µL 链霉素磁珠每样品成一个新的8孔带管。用200µL 的甲基 Seq 结合缓冲液洗涤珠子。使用磁性板颗粒珠和删除上清每个洗涤之间共3洗涤。最后洗涤后, 重悬链霉素珠在200µL 的甲基 Seq 结合缓冲液。
- 使用旋转搅拌机将样品添加到200µL 水洗链霉素磁珠, 室温下孵育30分钟。在混合时, 等分200µL 的甲基 Seq 洗涤缓冲液2到每样96孔板的三份井中, 并放置在热循环仪中预热至65摄氏度。
- 孵化后, 颗粒链霉亲和素磁珠使用磁性板和重悬珠在200µL 甲基 Seq 洗涤缓冲液1。室温下孵育15分钟。使用磁性板颗粒和丢弃上清液。
- 用甲基 Seq 洗涤缓冲液洗涤珠子3次 2: 在200µL 的洗涤缓冲液中重悬珠颗粒 2 (在步骤6.2.5 中预热), 在热循环仪 (65 摄氏度, 10 分钟) 和颗粒珠子中孵育珠子。使用磁性板每次洗涤后, 丢弃上清液。
注: 添加洗涤缓冲液2以防止非特定绑定时, 保持65摄氏度的杂交反应。 - 加入20µL 的甲基序列洗脱缓冲液, 在室温下孵育20分钟. 使用磁性板颗粒珠和转移上清到一个新的条管。扔掉珠子。
注: 在孵化过程中, 准备亚硫酸氢盐转化试剂。
- 将样品转移到低 DNA 结合微量离心管, 并使用加热真空浓缩器将样品体积降至小于3.4 µL. 将样品重组至3.4 µL。
- 亚硫酸氢盐转化
注: 使用适当的试剂和从商业上可用的亚硫酸氢盐转化试剂盒中的说明, 执行洗脱 ssDNA 的亚硫酸氢盐转化。- 将130µL 制备的亚硫酸氢盐转化试剂添加到上一步的上清液中。将150µL 反应分成两个井。在热循环仪中孵育 (64 摄氏度, 2.5 小时, 4 摄氏度保持)。
注: 150 µL 反应均匀地分为两个单独的井, 以确保均匀的温度。孵育2.5 小时后, 立即进行下一步。 - 通过添加600µL 的绑定缓冲区并使用100µL 洗涤缓冲液洗涤一次, 将样本绑定到旋转列。在所有亚硫酸氢盐转化步骤和丢弃流之间的离心柱 (1.5万 x g, 1 分钟)。
- Desulphonate 通过将 Desulphonation 缓冲区的200µL 添加到列中来进行采样。在室温下孵育 15-20 分钟. 重复离心和丢弃流过。
- 用200µL 洗涤缓冲液冲洗两次柱。洗脱每个样品, 加入10µL 的洗脱缓冲液到柱, 在室温下孵育3分钟, 离心 (1.5万 x g, 1 分钟)。重复洗脱步骤共20µL。
- 在冰上制备 PCR 反应主混合物1。添加82µL 混合到每个样本。使用以下程序在热循环仪中孵育。
PCR 反应主混合物 1 (每个样本):
30µL 水
50µL 甲基序列 PCR 主混合物
1µL 甲基 PCR1 底漆 F
1µL 甲基 PCR1 底漆 R
热循环仪程序:
阶段 1, 1 周期:95 °c 2 分钟
阶段 2, 8 周期:95 °c 三十年代, 60 摄氏度三十年代, 72 摄氏度三十年代
阶段 3, 1 周期:72 °c 7 分钟
阶段 4, 1 周期: 4 °c 保持 - 使用180µL 的 DNA 结合磁珠和400µL 新鲜制备的70% 乙醇样品纯化样品。每样添加180µL 的珠子, 室温下孵育5分钟。球团珠, 除去上清和重悬颗粒在200µL 70% 乙醇。去除乙醇, 重复洗涤一次。
- 使用磁性板颗粒珠和去除尽可能多的乙醇。在37摄氏度 heatblock 干燥, 直到珠颗粒完全干燥。在21µL 无核酸酶的水中重悬, 收集大约19.5 µL 的上清液。
- 将130µL 制备的亚硫酸氢盐转化试剂添加到上一步的上清液中。将150µL 反应分成两个井。在热循环仪中孵育 (64 摄氏度, 2.5 小时, 4 摄氏度保持)。
- 索引
- 在冰上制备 PCR 反应主混合物2。将25.5 µL 主混音2添加到每个样本中。将5µL 商业索引引物添加到单个样品中, 并在热循环仪中孵育。
PCR 反应主混合物 2 (每个样本):
25µL 甲基 Seq PCR 主混料
0.5 µL 甲基 Seq 共标引物
热循环仪程序:
阶段 1, 1 周期:95 °c 2 分钟
阶段 2, 6 周期:95 °c 三十年代, 60 摄氏度三十年代, 72 摄氏度三十年代
阶段 3, 1 周期:72 °c 7 分钟
阶段 4, 1 周期: 4 °c 保持
注: 如果起始 DNA 浓度低于推荐值, 则可能需要额外的周期 (2-3)。 - 使用90µL 的 DNA 结合磁珠和400µL 新鲜制备的70% 乙醇样品纯化样品。每样添加90µL 的珠子, 室温下孵育5分钟。球团珠, 除去上清和重悬颗粒在200µL 70% 乙醇。去除乙醇, 重复洗涤一次。
- 使用磁性板颗粒珠和去除尽可能多的乙醇。在37摄氏度 heatblock 干燥, 直到珠颗粒完全干燥。在24µL 无核酸酶的水中重悬, 收集大约24µL 的上清液。
- 使用 bioanalyzer 上的高灵敏度 DNA 检测试剂评估浓度和 bp 的大小。
注意: 如果 bioanalyzer 无法检测到库 DNA 的存在, 请使用额外的 dna 重复制备步骤。
停止点: 纯化后, 可将索引样品密封并存储在-20 摄氏度。 - 为所使用的合适的下一代测序平台汇集样本。
- 使用 bioanalyzer 的浓度数据, 根据给定体积中的库大小和数量确定 DNA 摩尔浓度, 用低 TE 缓冲液 (6.1.1.1) 稀释, 并将所有样品合并到下午15点的最后浓度。
注意: 对库进行量化的一种更灵敏的方法是使用针对结扎适配器的引物定量实时 PCR。 - 在下一代音序器上, 对每条车道4个样本的通道数运行共用样本。
注意: 例如, 如果16个库样本的索引和组合是唯一的, 则运行库超过4个车道, 相当于每条车道4个样本。
- 使用 bioanalyzer 的浓度数据, 根据给定体积中的库大小和数量确定 DNA 摩尔浓度, 用低 TE 缓冲液 (6.1.1.1) 稀释, 并将所有样品合并到下午15点的最后浓度。
- 在冰上制备 PCR 反应主混合物2。将25.5 µL 主混音2添加到每个样本中。将5µL 商业索引引物添加到单个样品中, 并在热循环仪中孵育。
7. 下一代音序器的测序
- 将样本发送到机构排序核心, 以实现甲基 Seq 库的聚类, 其次是在下一代测序机上进行测序。
8. 分析识别 DMRs
- 实现俾斯麦15, 它调用领结2.0 作为内部序列对准器16,17, 以使原始输入读取与亚硫酸氢盐转换, 加链基因组对齐。按照对齐方式, 使用 Bismark_methylation_extractor 执行质量控制, 并将估计的甲基化值分配给每个 CpG。
- 使用 Bioconductor 中的 BS Seq 包18生成 DMRs 列表。根据大于3的连续 CpGs 和 P 值 < 0.05 对 DMRs 进行过滤。
注: 生成包含基因组坐标、距离最近的磁铁基因、每个 dmr 中的 CpGs 数、average% CpG 甲基化值 (例如,应力 vs. 非重音)、P 值和罗斯福 (错误发现率) 值。使用 DMR 列表, i. e. 基因组坐标, 设计磷酸测序底漆进行验证。
9. 亚硫酸氢盐磷酸测序的验证
-
底漆设计
- 亚硫酸氢盐 PCR 和磷酸测序的设计引物。设计两组 pcr 引物 (外部和嵌套), 以便嵌套 pcr 将放大 150–400 bps。
注意: 一般情况下, 设计的引物至少有24个碱基, 至少4–5非连续 G (C 的反向底漆), 以减少退火温度, 从而降低序列复杂度的损失。其中一个嵌套引物将被生物素标记和 HPLC 纯化。但是, 应首先订购标准底漆, 通过解决琼脂糖凝胶上的反应来优化 PCR 步骤。- 设计磷酸测序测定底漆, 使其靶向补充生物素化的链只是 CpGs 的上游基地的检测。根据需要设计多个磷酸测序引物, 因为每个磷酸测序底漆可以可靠地检测下游 30 bps。
- 对于 Rt1-m4, 请使用以下内容:
rRT1M4 外– F TGTAYGATTTTGGTTATYGTAAAT
rRT1M4 外– R AACTTACAAATTTCACCAACTCA
rRT1M4 嵌套-F GTGGGTTAYGTGGATAATATATAG
rRT1M4 嵌套-R AATCACTTACCATTCTCTCTCTAACTA
rRT1M4 Pyro1 TAYGTGGATAATATATAGAT
rRT1M4 Pyro2 GATAGTTATTTGGYGAGTTAG
rRT1M4 Pyro3 GAGTATTTGGAGGAGTTGAT
rRT1M4 Pyro4 GGATTTTAATATTTGGT
- 亚硫酸氢盐 PCR 和磷酸测序的设计引物。设计两组 pcr 引物 (外部和嵌套), 以便嵌套 pcr 将放大 150–400 bps。
-
使用一个商业可用的试剂盒, 以亚硫酸氢盐转换大鼠血液 gDNA。
注: 亚硫酸氢盐转化步骤已从商业化的试剂盒中进行了调整, 并进行了以下修改: 在步骤1中, 添加 50–100 ng 的血液 gDNA, 用水稀释至20µL。在步骤9中, 洗脱20µL 每个样本。- 根据制造商的协议制备亚硫酸氢盐转化试剂, 并与稀释的 gDNA 相结合。在热循环仪中孵育 (64 摄氏度, 2.5 小时, 4 摄氏度保持)。
- 在旋转柱和离心机 (1.5万 x g, 1 分钟) 中添加绑定缓冲区到转换后的 gDNA。洗涤列一次, 然后添加 Desulphonation 缓冲区到列和孵育15分钟在室温下。离心机 (1.5万 x g, 1 分钟)。
- 洗涤塔与洗涤缓冲液和离心机 (1.5万 x g, 1 分钟)。重复洗涤步骤与离心 (1.5万 x g, 2 分钟)。将20µL 洗脱缓冲液和离心机 (1.5万 x g, 1 分钟) 添加到洗脱。
-
PCR 扩增
- 准备外部 PCR 主混合物。将21.5 µL 的主混料添加到3.5 µL 亚硫酸氢盐转换 gDNA 并运行热循环仪程序。
外部 PCR 主混合物:
16.25 µL 水
2.5 µL 聚合酶缓冲液 [10x]
0.5 µL 的 dNTP [10 毫米]
1µL 前底漆 [0.1 µM]
1µL 反向底漆 [0.1 µM]
0.25 µL Taq DNA 聚合酶 [5000 U/毫升]。
热循环仪程序:
阶段 1, 1 周期:94 °c 4 分钟
阶段 2, 47 循环:94 °c 1 分钟, 53 摄氏度三十年代, 72 摄氏度1分钟
阶段 3, 1 周期:72 °c 8 分钟, 4 摄氏度保持 - 准备嵌套 PCR 主混料。将23µL 的主混料添加到2µL 外 pcr 样品中, 然后重复外部 pcr 热循环仪程序。通过凝胶电泳 (1x 泰缓冲液, 1% 琼脂糖凝胶) 评估 PCR 产品质量。
嵌套 PCR 主混料:
17.75 µL 水
2.5 µL 聚合酶缓冲液 [10x]
0.5 µL 的 dNTP [10 毫米]
1µL 前底漆 [0.1 µM]
1µL 反向底漆 [0.1 µM]
0.25 µL Taq DNA 聚合酶 [5000 U/毫升]
注: 对于嵌套 PCR, 前向或反向底漆必须进行生物素化。
- 准备外部 PCR 主混合物。将21.5 µL 的主混料添加到3.5 µL 亚硫酸氢盐转换 gDNA 并运行热循环仪程序。
-
磷酸测序
- 使一个主混合物包含38µL 的结合缓冲液, 35 µL 水, 和2µL 链霉素涂层琼脂糖珠每样品。在96孔板中, 添加75µL 的主混音和5µL 的嵌套 PCR 产物。摇动 15-60 分钟的板式振动筛。
- 摇晃时, 将12µL 的底漆 (0.5 µM, 稀释在退火缓冲液中) 放入磷酸测序测定板的井中。
- 摇动后, 使用结合反应洗涤缓冲液执行洗涤步骤。将真空工具放入水槽中, 然后从板材中收集样品。在半填充槽内浸没真空工具, 含70% 乙醇、氢氧化钠 (0.2 M) 和三醋酸盐缓冲液 (10 mM, pH 7.4)。断开与真空的连接, 将真空工具放在 HS 检测板上转移珠子。
- 把盘子放在热块上, 在80摄氏度孵育2分钟. 允许板冷却5分钟然后开始火焰兵计划。
结果
大鼠甲基 Seq 平台的成功实施取决于几个标准。图 1显示了研究的整体工作流程, 并重点介绍了在前进前所需的特定质量控制 (QC) 步骤。首先要考虑的因素之一是动物模型和压力方案的健壮性, 它决定了甲基化发生的表观遗传变化的大小。由于我们的动物工作是基于我们以前的观察, 皮质酮 (CORT) 暴露可能导致 DNA 甲基化19,20的变化, 我们的慢性变应激 (CVS) 方案需要足够的严谨性来产生具有高血浆 CORT 水平的大鼠。典型的每周 CVS 方案如表 1所示, 每天早上、下午和夜间都由每日压力源组成, 以防止习惯性化和减轻压力反应。在整个3周的疗程中, 受压力的动物表现出显著升高的平均血浆 CORT [天 4–21, 控制: 32.7 3.7 ng/毫升, 压力: 103.0 11.9 ng/毫升 (平均 SEM), P = 2.2 x 10-4,图 2A] 在那些非重音,控制动物。同样, 这些动物在高加迷宫 (epm) 上也表现出更大的焦虑行为, 这表明在 EPM 的闭合臂上花费的时间大大增加, 张开双臂的时间更少 (图 2B)。这些结果表明, CVS 暴露导致了重大的内分泌和行为变化, 导致我们调查这些变化是否与特定的 DNA 甲基化特征相关。
我们强调几个检查站, 这对于成功地建设甲基 Seq 图书馆至关重要。从足够数量的 dna 开始是必要的, 因为超声、多重洗涤/纯化、目标浓缩和亚硫酸氢盐转化步骤相继减少了成品库中 dna 的数量。虽然几个 pcr 扩增步骤减轻了 DNA 模板的损失, 但过量的 pcr 周期数可能会带来更高的重复读取。对于目前的大鼠甲基序列研究, 采用了每鼠2克血 gDNA。我们注意到, 甲基 Seq 文库可以由起始 DNA 量低至 500 ng。较小的起始材料允许用户通过流化管 (荧光活化细胞分类) 或针冲分离出的 DNA 生成库, 尽管在后续测序中产生的库数量不足的风险增加。QC 是通过电泳 1 L 的样品在 bioanalyzer, 提供 DNA 分子量, 数量和摩尔浓度。三需要使用 bioanalyzer 的关键步骤是: 1) 遵循超声步骤, 以确保足够的 DNA 剪切 (~ 170 bp, 红色,图 3);2) 以下适配器结扎步骤指示由剪切的 DNA 的平均大小的移位 (~ 200 bp, 蓝色,图 3), 以确保其随后通过 PCR 扩增;和 3) 遵循最终的库纯化步骤, 确保库的数量和大小进行测序。
Bioconductor 中的 R 包 BSSeq 和 BSmooth 用于分析亚硫酸氢盐测序数据18。它们包括对齐序列读取、执行质量控制和识别差异甲基化区域 (DMRs) 的工具和方法。BSmooth 软件调用领结 2.016、17作为内部序列定位仪, 通过将原始输入读数与亚硫酸氢盐转换的基因组序列对齐来获得 CpG 级测量摘要。然后, 通过严格的质量控制程序对对齐的读取进行筛选, 以找出可能会扭曲下游分析的系统排序和基调用误差。在这一过滤过程中, 会生成一系列图解来帮助视觉辅助。还会生成排序指标来记录相关信息, 如对齐读取数、目标百分比和每个 CpG 覆盖率 (表 2)。筛选数据后, 将执行平滑/规范化算法, 其中每个 CpG 都根据每个样本的所有 QC 读数和邻近 CpGs 的估计值分配估计甲基化数值, 以确保更准确地调用甲基即使在序列覆盖率较低的情况下也是如此。此值提供了对每个 CpG 站点甲基化概率的平滑估计。通过比较两个治疗组之间每个样本的平滑甲基化估计值和将基因组区域从最显著的不同到最小的排名, 生成 DMRs 的列表 (表 3)。
在大鼠主要组织相容性基因Rt1-m4的启动子中, 压力和非重读组之间的顶部的 DMR 位于, 有压力的动物在所有 CpGs 的甲基化水平高于不重读动物 (图 4A)。为了确认甲基 seq 平台的成功实施和数据分析, 引物是针对 DMR 进行设计的, 在整个队列中的压力和非重读动物 (8 按甲基序列和8未测序测序) 的血液 DNA 甲基化水平由亚硫酸氢盐磷酸测序评估。结果表明, 在 12 CpGs 测定的10中, DNA 甲基化显著增加 (甲基化5.1–10.4 变化, P < 0.037,图 4B)。对所有名义上重要的 DMRs 进行 KEGG 通路分析, 以确定与应力相关的通路。与慢性应激接触相关的疾病, 如糖尿病、心血管疾病和癌症 (表 4) 一直是与 DMR 相关的通路。21,22,23为了证明表观遗传学数据与压力接触程度之间的关联, CpG-10 的甲基化水平与每种动物平均3周 CORT 水平相比较。结果表明, 内分泌和甲基化数据之间存在适度的相关性 (R2= 0.54, P = 0.001,图 5)。
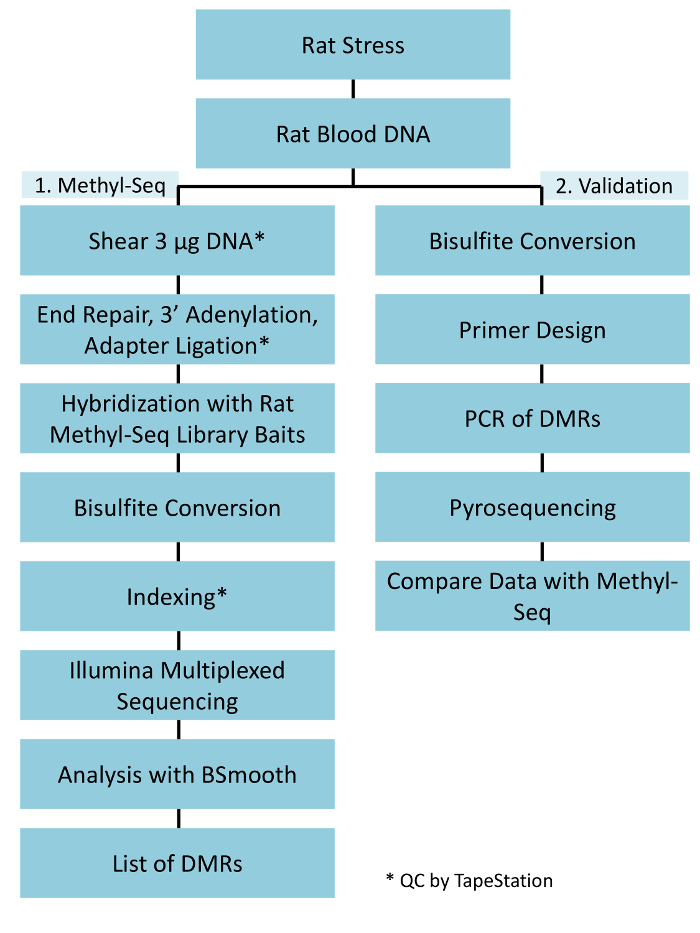
图 1: 大鼠甲基 Seq 平台的总体示意图工作流程.首先处理从受压和控制大鼠血液中提取的基因组 DNA 的一 g, 用于构造用于测序、分析和目标识别的甲基 Seq 库。另有 100 ng DNA 用于独立验证亚硫酸氢盐磷酸测序确定的表观遗传靶点。请点击这里查看这个数字的更大版本.
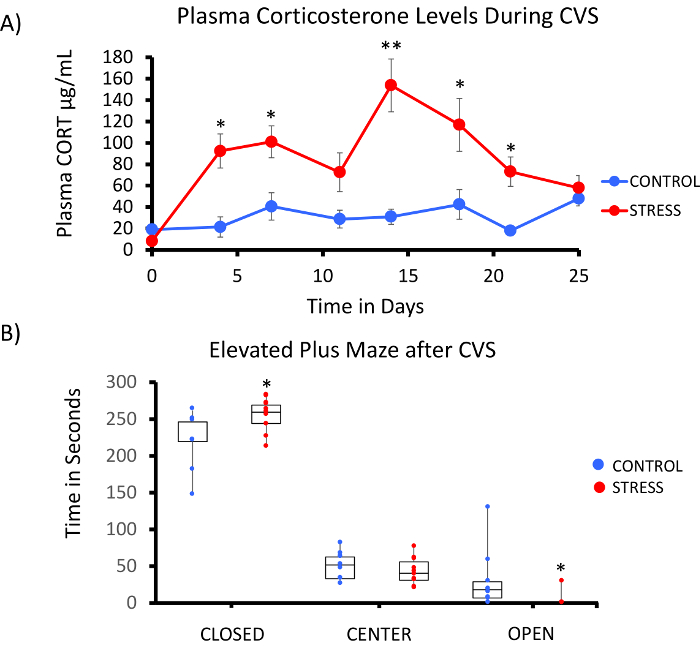
图 2: 接触慢性变应激 (CVS) 会导致大鼠的内分泌和行为改变.(A) 多采样的皮质酮 (CORT) 证明了3周 CVS 方案的稳健性。在每日压力方案之前, 早晨收集血样。(B) 受压力的动物在紧闭的双臂中花费更多的时间, 在高架加迷宫 (EPM) 的张开臂上的时间更少。当显示每个动物的数据点。对学生进行 t-检验以获得统计学意义。* p < 0.05, *** p < 0.01, 和 *** p < 0.001。请点击这里查看这个数字的更大版本.
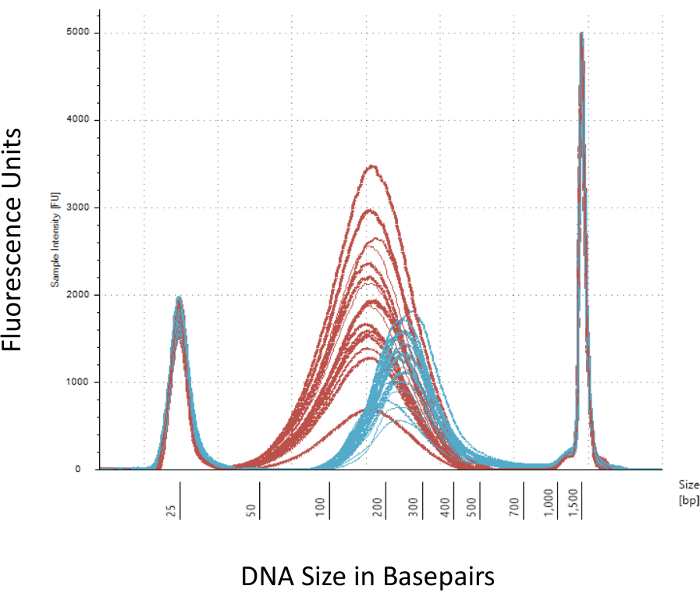
图 3: bioanalyzer 上剪切和适配器结扎大鼠 DNA 的定量.红色和蓝色曲线分别显示了在等温超声仪和适配器结扎中剪切后基因组 DNA (红色) 的数量和大小。每条线代表一个样本, 红色和蓝色曲线反映了在几个步骤 (末端修复、3 '-adenylation 和样本清理) 期间的 DNA 损失, 以及由于连接适配器而增加的 bp 大小。25 bp 和 1500 bp 的尖峰是已添加到加载缓冲区的标准标记。请点击这里查看这个数字的更大版本.
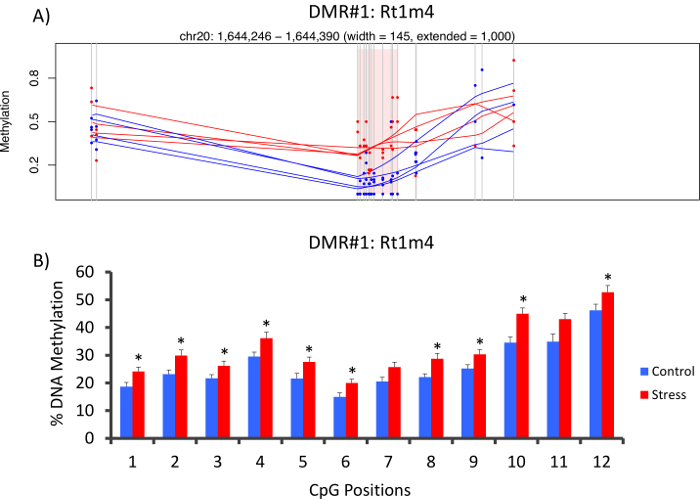
图 4: CVS 诱导的表观遗传变化由大鼠甲基序列检测.(A) 对大鼠甲基序列数据的分析, Rt1m4基因的启动子与受压 (红) 和对照 (蓝色) 大鼠之间的差异甲基化区域 (DMR) 有关。Rt1m4 DMR (粉红色阴影区域) 的图形输出显示每个 CpG (垂直灰色线)、每个组中的四个样本 (红色或蓝色线), 以及每个动物的 the% 甲基化水平 (红色或蓝色点)。(B) 在 CpGs 内, 有十二的磷酸测序是由亚硫酸氢盐所证实的。条形图表示为平均扫描电镜, 并对学生进行 T 检验以获得统计学意义。* P < 0.05。请点击这里查看这个数字的更大版本.
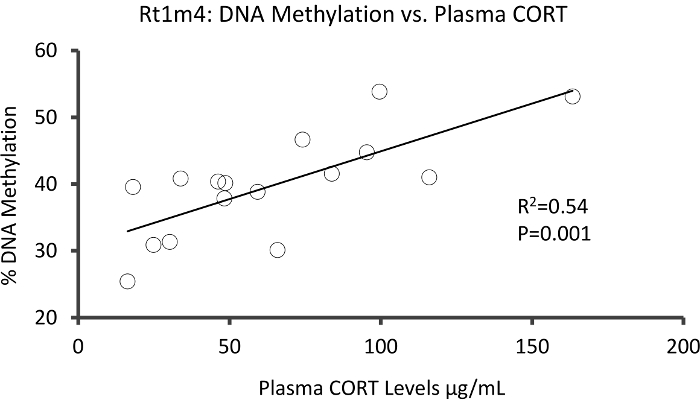
图 5: 线性回归分析显示Rt1m4 CpG-10 的 DNA 甲基化与3周平均血浆 CORT 水平 (N=16) 之间存在着适度的相关性。受压力动物的数据由红圈代表.请点击这里查看这个数字的更大版本.
| 周 | 1天 | 2天 | 3天 | 4天 | 5天 | 6天 | 7天 |
| 是 | 约束 | 游泳 | 冷室 | 游泳 | 约束 | 振动筛 | 游泳 |
| 下午 | 振动筛 | 保持架倾斜 | 约束 | 振动筛 | 冷室 | 约束 | 冷室 |
| 一 夜 之间 | 食品限制 | 湿式床上用品 | 分离 | 亮起 | 拥挤 | 亮起 | 湿式床上用品 |
表 1: 慢性变应激方案 (CVS) 的典型每周计划。
| 测序指标 | 压力1 | 控制1 |
| (n = 4) | (n = 4) | |
| 配对的结束读取 (每) | 89290397 | 80165674 |
| 唯一映射成对的结束读取 (UMPER) | 39200255 | 35013406 |
| 对齐速率/制图效率 (UMPER/PER) | 44% | 44% |
| 重复读取 (占 UMPER 的%) | 73% | 65% |
| 重复 UMPER | 10481031 | 12306018 |
| 平均读取深度覆盖率 (x) (ARDC) | 6x | 6x |
| CpGs (N) | 12056878 | 12056878 |
| CpGs 的 ARDC (x) | 2x | 2x |
| CpGs 至少10次读取 (N) | 481383 | 595850 |
| ARDC (X) 的 CpGs 至少有10次读取 | 19 | 19 |
| 目标 CpGs (与探针目标区域完全重叠) | 1923872 | 2007638 |
| 关于 CpGs 的靶 ARDC (x) | 7x | 8x |
| 在目标 CpGs 至少有10次读取 (N) | 428249 | 531419 |
| 在目标 ARDC (x) 的 CpGs 至少10次读取 | 18x | 18x |
| 在目标 (每个与探测目标区域的1或更多基对重叠) (UMPER) | 8277715 | 9369523 |
| 目标百分比 (重复 UMPER) | 78% | 77% |
| 在目标 (映射的总基数) Mb | 125兆 | 128兆 |
| 目标平均读取深度覆盖率 (x) (ARDC) | 9x | 10x |
| 1根据每个组中各科目的平均值排序指标 |
表 2: 从大鼠甲基 Seq 平台获得的测序指标。
| chr | 开始 | 结束 | 基因 | 距离 | areaStat | meanDiff | 应力 | 控制 | 方向 |
| chr20 | 1644246 | 1644390 | RT1-M4 | in_gene | 93.03 | 0.22 | 0.33 | 0.11 | 获得 |
| chr5 | 160361352 | 160361564 | LOC690911 | in_gene | -70.75 | -0.19 | 0.72 | 0.91 | 损失 |
| chr3 | 61138281 | 61138330 | RGD1564319 | 265569 | 61.79 | 0.21 | 0.94 | 0.72 | 获得 |
| chr2 | 143064811 | 143065010 | Ufm1 | 8569 | -59.48 | -0.11 | 0.13 | 0.24 | 损失 |
| chr7 | 30764111 | 30764284 | Ntn4 | in_gene | 57.04 | 0.21 | 0.94 | 0.73 | 获得 |
| chr17 | 12469112 | 12469218 | Idnk | 41996 | -50.91 | -0.13 | 0.74 | 0.88 | 损失 |
| chr7 | 47101725 | 47101930 | Pawr | in_gene | -50.54 | -0.12 | 0.64 | 0.76 | 损失 |
| chr5 | 76111248 | 76111822 | Txndc8 | 151703 | -50.38 | -0.11 | 0.85 | 0.96 | 损失 |
| chr11 | 80640132 | 80640356 | Dgkg | in_gene | -50.07 | -0.16 | 0.73 | 0.89 | 损失 |
| chr8 | 71759248 | 71759411 | Mir190 | 210226 | -47.84 | -0.17 | 0.58 | 0.75 | 损失 |
表 3: 前10差异化的甲基化区域.对于每个 DMR, 输出表显示从左到右列: 染色体位置 (chr), 坐标 (开始/结束), 基因名称, 距离转录起始点, 应力和对照组之间的差区统计 (areaStat), 平均值差甲基化 (meanDiff), 平均甲基化水平在每个 DMR 为压力和控制组 (应力/控制), 和从控制甲基变化的方向。
| KEGG 通路术语 | 基因计数 | % | P 值 | Benjamini |
| 糖尿病 | ||||
| II 型糖尿病 | 12 | 0。1 | 3.6 x 10-4 | 9.8 x 10-3 |
| 心血管疾病 | ||||
| 血管平滑肌收缩 | 18 | 0。1 | 1.6 x 10-3 | 3.6 x 10-2 |
| 致心律失常右心室心肌病 (ARVC) | 13 | 0。1 | 4.0 x 10-3 | 7.1 x 10-2 |
| 扩张型心肌病 | 14 | 0。1 | 7.6 x 10-3 | 1.2 x 10-1 |
| 神经元功能 | ||||
| 长期程增强 | 11 | 0。1 | 1.5 x 10-2 | 1.4 x 10-1 |
| 信号 | ||||
| 信号通路 | 35 | 0。2 | 2.4 x 10-4 | 9.9 x 10-3 |
| 钙信号通路 | 22 | 0。1 | 1.2 x 10-2 | 1.4 x 10-1 |
| 趋化因子信号通路 | 21 | 0。1 | 1.2 x 10-2 | 1.3 x 10-1 |
| 癌症 | ||||
| 癌症的通路 | 42 | 0。3 | 4.1 x 10-5 | 3.4 x 10-3 |
| 胶质 瘤 | 15 | 0。1 | 4.4 x 10-5 | 2.4 x 10-3 |
| 非小细胞肺癌 | 10 | 0。1 | 7.9 x 10-3 | 1.1 x 10-1 |
| 直肠癌 | 13 | 0。1 | 8.4 x 10-3 | 1.1 x 10-1 |
| 慢性髓系白血病 | 12 | 0。1 | 1.2 x 10-2 | 1.3 x 10-1 |
表 4: 从大鼠甲基序列中鉴定的 DMRs 的 KEGG 通路分析。
讨论
在本研究中, 我们设计并实现了大鼠基因组的甲基 Seq 平台。通过对大鼠应力模型的研究表明, 实验和分析管道可以在两个比较组之间提供不同的甲基化区域。
为了确保平台的成功实施, 需要遵守几个关键步骤。首先, 初始 DNA 质量和数量对最终的甲基序列库的质量和数量有显著的影响。我们使用荧光计, 而不是分光光度计, 以确保我们的 dna 测量反映了双链 dna 存在的数量。bioanalyzer 用于测量剪切后和适配器结扎后 DNA 的分子大小和数量。验证这些步骤之间的分子尺寸 "移位" 对于确认在后续步骤中将接受适配器介导的 PCR 的每个 DNA 片段的末端是否存在适配器至关重要。在适配器结扎步骤结束时剩余的 DNA 数量也很重要, 因为此步骤需要至少 100 ng 的库产品, 以确保在目标富集和亚硫酸氢盐转化步骤之后有足够数量。在构造的甲基 Seq 库上进行了最后的高灵敏度测量, 以便在下一代音序器上对库进行适当稀释以进行后续聚类。最后, 采用亚硫酸氢盐磷酸测序作为一种高定量、独立的方法来评估分析管道的精度。使用原始样本和使用其他动物进行复制的最终验证是确保实验能够检测 DNA 甲基化的生物显著变化的关键步骤。
我们还包括在偏离协议或遇到问题的情况下的若干准则。首先, 在末端修复、适配器结扎或磁珠纯化步骤中, 可能会丢失过多的 DNA。或者, 由于组织/DNA 的可用性有限或各种浓缩方法 (如荧光活化细胞分类) 的实施, DNA 的起始量可能小 (< 200 ng)。在两个库的放大步骤中增加循环数可能能够弥补整个图书馆建设协议中 dna 的过量损失或低起始 dna 量。但是, 建议不要超过一个额外的的, 因为过多的模板放大可能会导致序列化重复读取的数量增加。在对齐步骤中排除这些重复项, 以防止甲基化百分比计算中的偏差。其次, 如果平均 DNA 大小不增加超过 30 bps, 检查以确保试剂是新的, 因为 T4 DNA 聚合酶, 克列诺和/或 T4 连接可能是旧的。可使用市面上可用的替代试剂。
此外, 预测的 DMRs 可能无法通过磷酸测序验证, 其中 DNA 甲基化差异不存在或显著低于分析预测的。对候选区域的验证较差是许多基因组分析的一个非常普遍的问题, 例如当磷酸测序结果不确认差异甲基化或效果大小比分析预测的要小得多时。BSmooth 是一个分析包, "平滑" 在多个 CpGs 窗口的甲基化水平。对于目前的实验, BSmooth 牵连到一个 DMR, 其甲基化水平由亚硫酸氢盐磷酸测序验证。但是, BSmooth 和磷酸测序验证的甲基化水平之间可能存在差异。差异产生的平滑函数, 估计在一个 DMR 中的所有 CpGs 的平均甲基化值, 包括连续 CpGs, 可能不同的 DNA 甲基化超过50% 或 CpGs 其甲基化值被排除由于子阈值读取深度。R 封装 (如 MethylKit24 ) 可用于识别 CpGs 或甚至单个 CpGs 的较小窗口, 其甲基化水平与磷酸测序验证的那些更紧密相关。通过磷酸测序实现不同的包并测试其预测区域或 CpGs 差异甲基化, 将确保数据的鲁棒性。或者, 可以用原始的甲基 Seq 库, 并将其添加到读取文件中, 以增加读取深度。由于甲基化水平的测定是半定量的, 由读取次数 [(CpGs)/(TpGs + CpGs 的 #)] 决定, 增加给定 CpG 的读取深度将提高其甲基化值的准确度。在本研究中, 我们只考虑了 CpGs, 其甲基化值由至少十次读数确定, 并达到每个 CpG 的总读覆盖率19x。
大鼠甲基 Seq 平台是没有它的局限性。虽然它比全基因组亚硫酸氢盐测序更具成本效益, 但它比其他方法昂贵得多。然而, 大部分的成本是在音序器上购买车道, 而不是捕获系统。根据所需的读取深度, 在 DNA 甲基化中, 由于大 (25–70%) 差异12而需要较少的跨组织比较, 可以通过多路采样和使用更高容量的平台来减少成本。此外, 样品制备比其他方法更耗时。虽然类似于其他下拉方法, 包括下一代测序, 添加亚硫酸氢盐转化和纯化步骤增加工作负荷。总的来说, 甲基 Seq 平台是一种经济高效的替代全基因组测序的方法, 可提供超过 230万 CpGs 的基对分辨率, 比基于微阵列的平台所测定的多。到目前为止, 商业上可用的人类和小鼠甲基序列平台已经被用来记录在猕猴大脑中的酒精依赖性变化25,26, 神经发育基因在小鼠脑9和血脑糖皮质激素10的目标。此外, 无论通过限制性酶的顺序识别, 定位特定区域的能力都使其成为跨物种比较的理想平台。在这项研究中, 我们设计了大鼠的甲基 Seq 平台, 在没有基因组范围的 methylomic 工具的情况下, 进行了许多药理学、代谢和行为实验。我们的数据表明, 它可用于检测大鼠应力模型中的 DMRs, 并关联其他生理参数, 如整体血浆 CORT 水平。
甲基 Seq 平台是动物的表观遗传学实验的理想选择, 有序列的基因组, 可能没有足够的实验证据记录监管区域。当这些区域可用时, 其他区域可以自定义设计并附加到当前版本。此外, 该平台是比较基因组学的理想选择, 因为目标富集不受限制性酶识别的约束。例如, 任何兴趣基因的启动子区域都可以被捕获, 无论它是否有一个特定的限制站点。同样, 任何监管区域, 如在鼠标或人类中发现的, 在感兴趣的基因组中保存的, 都可以被捕获。
披露声明
这份手稿是安捷伦科技竞赛奖的一部分。
致谢
这项研究由 NIH 赠款 MH101392 (RSL) 资助, 并得到以下奖项和基金会的支持: NARSAD 年轻研究员奖, 玛格丽特 Ann 价格调查基金, 詹姆斯华情绪障碍学者基金通过查尔斯 t. 鲍尔基金会, 贝克基础和项目匹配基础 (RSL)。
材料
| Name | Company | Catalog Number | Comments |
| Radioimmuno assay (RIA) | MP Biomedicals | 7120126 | Corticosterone, 125I labeled |
| Master Pure DNA Purification Kit | Epicentre/Illumina | MC85200 | |
| Thermal-LOK 2-Position Dry Heat Bath | USA Scientific | 2510-1102 | Used with 1.5 mL tubes |
| Vortex Genie 2 | Fisher | 12-812 | Vortex Mixer |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023 | 100% Ethanol, molecular grade |
| Centrifuge 5424 R | Eppendorf | - | Must be capable of 20,000 x g |
| Qubit 2.0 | ThermoFisher Scientific | Q32866 | Fluorometer |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32850 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32851 | High sensitivity DNA detection reagents |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| SureSelectXT Rat Methyl-Seq Reagent Kit | Agilent Technologies | G9651A | Reagents for preparing the Methyl-Seq library |
| SureSelect Rat Methyl-Seq Capture Library | Agilent Technologies | 931143 | RNA baits for enrichment of rat targets |
| IDTE, pH 8.0 | IDT DNA | 11-05-01-09 | 10 mM TE, 0.1 mM EDTA |
| DNA LoBind Tube 1.5 mL | Eppendorf | 22431021 | |
| Covaris E-series or S-series | Covaris | - | Isothermal sonicator |
| microTUBE AFA Fiber Pre-Slit Snap-Cap 6x16mm (25) | Covaris | 520045 | |
| Water, Ultra Pure (Molecular Biology Grade) | Quality Biological | 351-029-721 | |
| Veriti 96 Well-Thermal Cycler | Applied Biosystems | 4375786 | |
| AMPure XP Beads | Beckman Coulter | A63880 | DNA-Binding magnetic beads |
| 96S Super Magnet | ALPAQUA | A001322 | Magnetic plate for purification steps |
| 2200 TapeStation | Agilent Technologies | G2965AA | Electrophresis-based bioanalyzer |
| D1000 ScreenTape | Agilent Technologies | 5067-5582 | |
| D1000 ScreenTape High Sensitivity | Agilent Technologies | 5067-5584 | |
| D1000 Reagents | Agilent Technologies | 5067-5583 | |
| D1000 Reagents High Sensitivity | Agilent Technologies | 5067-5585 | |
| DNA110 SpeedVac | ThermoFisher Scientific | - | Vacuum Concentrator |
| Dynabeads MyOne Streptavidin T1 magnetic beads | Invitrogen | 65601 | Streptavidin magnetic beads |
| Labquake Tube Rotator | ThermoFisher Scientific | 415110Q | Nutator Mixer is also acceptable |
| EZ DNA Methylation-Gold Kit | Zymo Research | D5006 | Bisulfite conversion kit. Contains Binding, Wash, Desulphonation, and Elution buffers |
| Illumina Hi-Seq 2500 | Illumina | - | Next-generation sequencing machine |
| PCR and Pyrosequencing Primers | IDT DNA | Variable | |
| Taq DNA Polymerase with ThermoPol Buffer - 2,000 units | New England BioLabs | M0267L | |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | |
| Pyromark MD96 | QIAGEN | - | Pyrosequencing machine |
| Ethyl Alcohol 200 Proof | Pharmco-Aaper | 111000200 | 70% Ethanol solution |
| Sodium Hydroxide Pellets | Sigma-Aldrich | 221465 | 0.2 M NaOH denature buffer solution |
| Tris (Base) from J.T. Baker | Fisher Scientific | 02-004-508 | 10 mM Tris Acetate Buffer wash buffer solution |
| PyroMark Gold Q96 Reagents (50x96) | QIAGEN | 972807 | Reagents required for pyrosequencing |
| PyroMark Annealing Buffer | QIAGEN | 979009 | |
| PyroMark Binding Buffer (200 mL) | QIAGEN | 979006 | |
| Streptavidin Sepharose High Performance Beads | GE Healthcare | 17-5113-01 | Streptavidin-coated sepharose beads |
| PyroMark Q96 HS Plate | QIAGEN | 979101 | Pyrosequencing assay plate |
| Eppendorf Thermomixer R | Fisher Scientific | 05-400-205 | Plate mixer. 96-well block sold separately (cat. No 05-400-207) |
| SureDesign Website | Agilent Technologies | - | Target capture design software (https://earray.chem.agilent.com/suredesign/) |
| UCSC Genome Browser | University of California Santa Cruz | - | rat Nov 2004 rn4 assembly |
| Agilent Methyl-Seq Protocol | Agilent Technologies | - | https://www.agilent.com/cs/library/usermanuals/public/G7530-90002.pdf |
参考文献
- Barski, A., et al. High-resolution profiling of histone methylations in the human genome. Cell. 129 (4), 823-837 (2007).
- Meissner, A., et al. Genome-scale DNA methylation maps of pluripotent and differentiated cells. Nature. 454 (7205), 766-770 (2008).
- Bibikova, M., et al. High density DNA methylation array with single CpG site resolution. Genomics. 98 (4), 288-295 (2011).
- Naumov, V. A., et al. Genome-scale analysis of DNA methylation in colorectal cancer using Infinium HumanMethylation450 BeadChips. Epigenetics. 8 (9), 921-934 (2013).
- Wockner, L. F., et al. Genome-wide DNA methylation analysis of human brain tissue from schizophrenia patients. Translational Psychiatry. 4, e339 (2014).
- Meissner, A., et al. Reduced representation bisulfite sequencing for comparative high-resolution DNA methylation analysis. Nucleic Acids Research. 33 (18), 5868-5877 (2005).
- Smith, Z. D., Gu, H., Bock, C., Gnirke, A., Meissner, A. High-throughput bisulfite sequencing in mammalian genomes. Methods. 48 (3), 226-232 (2009).
- Slieker, R. C., et al. Identification and systematic annotation of tissue-specific differentially methylated regions using the Illumina 450k array. Epigenetics Chromatin. 6 (1), 26 (2013).
- Hing, B., et al. Adaptation of the targeted capture Methyl-Seq platform for the mouse genome identifies novel tissue-specific DNA methylation patterns of genes involved in neurodevelopment. Epigenetics. 10 (7), 581-596 (2015).
- Seifuddin, F., et al. Genome-wide Methyl-Seq analysis of blood-brain targets of glucocorticoid exposure. Epigenetics. 12 (8), 637-652 (2017).
- Irizarry, R. A., et al. The human colon cancer methylome shows similar hypo- and hypermethylation at conserved tissue-specific CpG island shores. Nature Genetics. 41 (2), 178-186 (2009).
- Lee, R. S., et al. Adaptation of the CHARM DNA methylation platform for the rat genome reveals novel brain region-specific differences. Epigenetics. 6 (11), 1378-1390 (2011).
- Jankord, R., et al. Stress vulnerability during adolescent development in rats. Endocrinology. 152 (2), 629-638 (2011).
- Pellow, S., Chopin, P., File, S. E., Briley, M. Validation of open:closed arm entries in an elevated plus-maze as a measure of anxiety in the rat. Journal of Neuroscience Methods. 14 (3), 149-167 (1985).
- Krueger, F., Andrews, S. R. Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications. Bioinformatics. 27 (11), 1571-1572 (2011).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), R25 (2009).
- Hansen, K. D., Langmead, B., Irizarry, R. A. BSmooth: from whole genome bisulfite sequencing reads to differentially methylated regions. Genome Biology. 13 (10), R83 (2012).
- Lee, R. S., et al. Chronic corticosterone exposure increases expression and decreases deoxyribonucleic acid methylation of Fkbp5 in mice. Endocrinology. 151 (9), 4332-4343 (2010).
- Lee, R. S., et al. A measure of glucocorticoid load provided by DNA methylation of Fkbp5 in mice. Psychopharmacology. , (2011).
- Bose, M., Olivan, B., Laferrere, B. Stress and obesity: the role of the hypothalamic-pituitary-adrenal axis in metabolic disease. Current Opinion in Endocrinology, Diabetes, and Obesity. 16 (5), 340-346 (2009).
- Brydon, L., Magid, K., Steptoe, A. Platelets, coronary heart disease, and stress. Brain, Behavior, and Immunity. 20 (2), 113-119 (2006).
- McKlveen, J. M., et al. Chronic Stress Increases Prefrontal Inhibition: A Mechanism for Stress-Induced Prefrontal Dysfunction. Biological Psychiatry. 80 (10), 754-764 (2016).
- Akalin, A., et al. methylKit: a comprehensive R package for the analysis of genome-wide DNA methylation profiles. Genome Biology. 13 (10), R87 (2012).
- Cervera-Juanes, R., Wilhelm, L. J., Park, B., Grant, K. A., Ferguson, B. Alcohol-dose-dependent DNA methylation and expression in the nucleus accumbens identifies coordinated regulation of synaptic genes. Translational Psychiatry. 7 (1), e994 (2017).
- Cervera-Juanes, R., Wilhelm, L. J., Park, B., Grant, K. A., Ferguson, B. Genome-wide analysis of the nucleus accumbens identifies DNA methylation signals differentiating low/binge from heavy alcohol drinking. Alcohol. 60, 103-113 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。