需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
利用体外荧光共振能量转移研究毫秒时间尺度蛋白质配合物的动力学
摘要
蛋白质-蛋白质相互作用对生物系统至关重要, 对结合动力学的研究为蛋白质复合物的动力学和功能提供了见解。我们描述了一种利用荧光共振能量转移和停止流动技术量化蛋白质复合物动力学参数的方法。
摘要
蛋白质是生物系统的主要操作人员, 它们通常与其他大分子或小分子相互作用, 以发挥其生物功能。这种相互作用可以是高度动态的, 这意味着相互作用的子单位以一定的速度不断地联系和分离。虽然使用定量下拉式等技术测量结合亲和力可以揭示相互作用的强度, 但研究结合动力学可以提供有关相互作用发生的速度以及每个复合物存在多长时间的见解。此外, 在存在蛋白质交换因子或药物等附加因素的情况下测量相互作用的动力学, 有助于揭示相互作用受另一个因素调节的机制, 为生物和医学研究的进步。在这里, 我们描述了一个协议, 用于测量具有高内在关联率并可由另一种蛋白质快速分离的蛋白质复合体的结合动力学。该方法利用荧光共振能量转移报告蛋白质复合物的体外形成, 并能实时监测停止流动的荧光仪上的蛋白质复合物的快速关联和离解。利用该方法, 对蛋白质复合物的关联和离解速率常数进行了定量。
引言
生物活动最终是由蛋白质进行的, 其中大部分蛋白质与其他蛋白质相互作用, 以获得适当的生物功能。使用计算方法, 人类蛋白质相互作用的总量估计为约 650, 000,1, 而这些相互作用的中断往往导致疾病2。由于其在控制细胞和组织过程中的重要作用, 研究蛋白质-蛋白质相互作用的方法已被开发出来, 如酵母-双杂交, 双分子荧光互补, 分裂荧光酶互补, 联合免疫沉淀检测3。虽然这些方法善于发现和确认蛋白质-蛋白质相互作用, 但它们通常是非定量的, 因此提供的相互作用的蛋白质伙伴之间的亲和力有限。定量拉下可以用来测量结合亲和力 (例如, 离解常数kd), 但它不测量结合的动力学, 也不能在 k d 由于不足而非常低的情况下应用信噪比4。表面等离子体共振 (spr) 光谱量化了结合动力学, 但它需要一个特定的表面和在表面的一个反应物的固定, 这可能会改变反应物5的结合性能。此外, spr 很难测量快速关联和离解率5, 使用 spr 来表征蛋白质复合体中交换蛋白质亚基的事件是不合适的。在这里, 我们描述了一种方法, 允许测量在毫秒时间尺度上的蛋白质复杂组装和拆卸速率。该方法对于确定 c ullin-a ssociated-edd8-d 同体蛋白 1 (cand1) 作为 f- 盒蛋白交换因子6,7的作用至关重要.
cand1 调节 Skp1•Cul1•F 盒蛋白 (scf) e3 脂类的动态, e3 配体属于库林-岭泛素脂质的大家族。scfs 由结合 ing 域蛋白 rbx1 的 cullin cullin 和一种可互换的 f-箱蛋白组成, 后者通过适配器蛋白 skp18 招募基质并将 cullin 结合。作为 e3 连接酶, scf 催化泛素与底物的结合, 当底物被 f-盒蛋白吸收, 当 cul1 被泛素样蛋白 nedd89 修饰时, 它就会被激活。cand1 结合未经修饰的 cul1, 在结合时, 它破坏了 Skp1•F 盒蛋白与 cul1 的联系, 以及 ned8 与 cul1、11、12、13的结合。因此, cand1 似乎是体外 scf 活性的抑制剂, 但体内的 cand1 缺乏引起了缺陷, 表明 cand1 在调节体内 scf 活性方面发挥了积极作用 14、15、16,17. 这一悖论最终通过一项定量研究得到了解释, 该研究揭示了 cul1、cand1 和 Skp1•F 盒蛋白之间的动态相互作用。利用荧光共振能量传递 (fret) 检测 scf 和 Cul1•Cand1 配合物的形成, 分别检测出关联和离解速率常数 (分别为 kon 和k)。单独测量。测量结果表明, cand1 和 Skp1•F 盒蛋白与 cul1 的复合体极其紧密, 但 scf 的k含量因 cand1 而急剧增加, Cul1•Cand1 的k含量大幅增加由 Skp1•F 盒蛋白6,7。这些结果为定义 cand1 作为蛋白质交换因子的作用提供了初步和关键的支持, 该因子通过从旧的 scf 复合物中回收 cul1 来催化新的 scf 复合物的形成。
本文介绍了利用 fret 法研究 Cul1•Cand1 复合物7动力学的方法, 并将同样的原理应用于各种生物分子的动力学研究。当供体被适当的波长激发, 而具有激发谱重叠供体发射光谱的受体存在于10-100 的距离内时, 就会发生 fret。激发态转移到受体, 从而降低供体强度, 增加受体强度18。fret (e) 的效率取决于 förster 半径 (r0) 和供体与受体荧光 (r) 之间的距离, 定义为: e = r0 6/(r0 )6 + r6)。förster 半径 (r0) 取决于几个因素, 包括偶极子角方向、供体受体对的光谱重叠和使用的解 19.为了将 fret 检测应用于停止流式荧光仪上, 该方法实时监测供体排放的变化, 并能够测量快速k开和关,有必要建立高效的 fret, 以实现从而显著减少了捐助方的排放。因此, 通过选择合适的对荧光染料和在目标蛋白上的位点来连接染料来设计高效的 fret 是非常重要的, 将在本方案中进行讨论。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 设计 fret 检测。
- 从蛋白质数据库 (文件 1u6g) 下载 Cul1•Cand1 复合体的结构文件。
- 查看 pymol 中 Cul1•Cand1 复合体的结构。
- 使用 pymol 向导菜单下的测量功能来估计 cand1 的第一个氨基酸和 cul1 的最后一个氨基酸之间的距离 (图 1)。
- 加载在线光谱查看器 (见材料表), 同时查看 7-氨基-4-甲基香豆素 (amc) 和 flash 的激发和发射光谱 (图 2)。请注意, amc 是 fret 供体, flash 是 fret 受体。
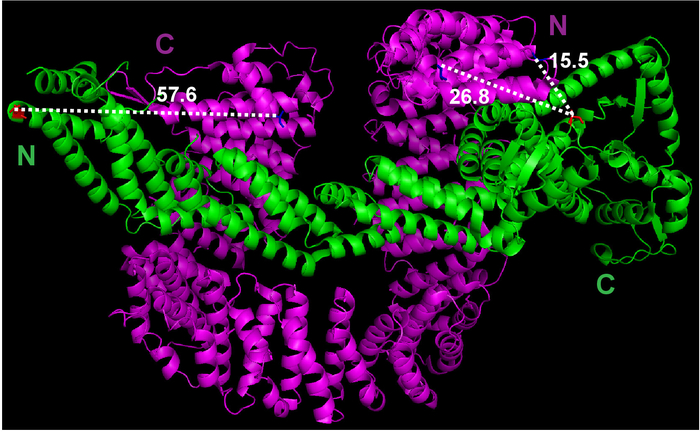
图 1: Cul1•Cand1 的晶体结构和测量潜在标记位点之间的距离.晶体结构文件是从蛋白质数据库 (文件 1u6g) 下载的, 并在 pymol 中查看。选定原子之间的测量是用 pymol 进行的。请点击这里查看此图的较大版本.
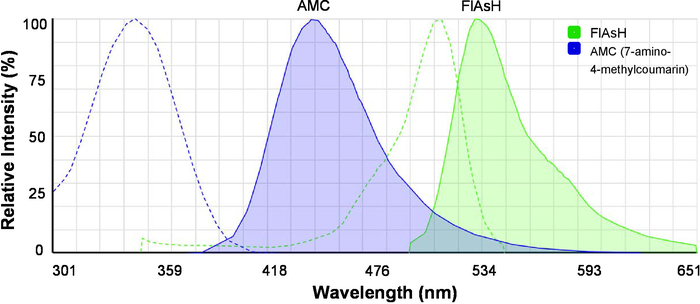
图 2: fret 用荧光染料的激发和发射光谱.显示了 amc (7-氨基-4-甲基香豆素) 和 flash 的光谱。虚线表示激发谱, 实线表示发射光谱。该图像最初是由荧光光谱查看器生成的, 并进行了修改以提高清晰度。请点击这里查看此图的较大版本.
2. camc·rbx1 培养细胞的制备, fret 供体蛋白
- 构建质粒, 表达大肠杆菌细胞中的人的能力1山梨·rbx1。请注意, 在大肠杆菌细胞中共同表达人类 Cul1•Rbx1 的两个质粒在上一份报告20中进行了详细描述。
- 通过标准 pcr 和克隆方法 21, 22, 在 cul1 编码序列的 3 ' 结尾添加 dna 序列编码 "lpetghhhhhhh" (sorase-he-他的6个标记)。
- 序列新的质粒, 以确认基因插入是准确的。
- 大肠杆菌细胞中的聚合酶1 肌胶酶· rbx1. 该方法来自以前的报告20。
- 使用热冲击法23将两种质粒中的每一种混合 100 ng 与 bl21 (de3) 化学辅助细胞进行共转化。在37°c 隔夜生长在 lb 琼脂板上, 含有100μgml 氨匹西林和34μgml 氯霉素。
- 用新鲜转化的菌落接种50毫升, 在37°c 下生长, 每分钟晃动250转。这就给了一个首发文化。
- 接种6个烧瓶, 每个瓶有1升 lb 介质, 每个5毫升发酵剂培养, 在37°c 生长, 250 转分晃动, 直到 od600是 ~ 1.0。将培养物冷却至 16°c, 并将异丙基β-d-硫代唑酮苷 (iptg) 添加至 0.4 mm。保持培养在16°c 过夜, 每小时250转。
- 通过离心收集大肠杆菌细胞, 以 5, 000 x 克的速度 15分钟, 并在50毫升锥形管中收集细胞颗粒。
注: 细胞颗粒可以进行蛋白质纯化加工, 也可以在-80°c 下冷冻, 然后再进入蛋白质纯化步骤。
- 纯化培养 1的肌酸酶·rbx1 复合物。此方法来自以前的报告20。
- 在表达 cle1 肌酶的大肠杆菌细胞颗粒中加入50毫升的裂解缓冲液 (30 mm Tris-HCl、200 mm nocl、5 mL sortase、10% 甘油、1片蛋白酶抑制剂鸡尾酒、ph 7.6 ).
- 将细胞在冰上以50% 的振幅进行超声检查。在1秒开到1秒关闭之间交替, 运行3分钟。
- 重复步骤 2.3.2 2-3x。
- 将细胞裂解液转移到50毫升的离心管中, 以 25, 000 x 克的离心去除细胞碎片, 时间为45分钟。
- 在4°c 条件下, 用5毫升谷胱甘肽珠培养透明细胞裂解液2小时。
- 在4°c 条件下, 以 1, 500 x 克离心珠状裂解混合物2分钟。取出上清液。
- 用5毫升的裂解缓冲液 (无蛋白酶抑制剂) 清洗珠子, 在4°c 离心后在 1, 500 x 克下取出上清液2分钟。
- 重复步骤 2.3.7 2x。
- 在洗涤的珠子中加入3毫升的裂解缓冲液, 并将珠子浆料转移到空柱中。
- 在色谱柱中加入5毫升洗脱缓冲液 (50 mm Tris-HCl, 200 mm ncl, 10 mm 还原谷胱甘肽, ph 8.0)。孵化 10分钟, 收集洗脱液。
- 重复步骤 2.3.10 3-4 x。
- 在谷胱甘肽珠的洗脱液中加入200μl 的 5 mgml 凝血酶 (见材料表), 并在4°c 下孵育过夜。
注: 协议可以在此处暂停。 - 用缓冲液 a 稀释蛋白质样品 (25 mm hepes, 1 mm dtt, 5% 甘油, ph 6.5) 三倍。
- 在带有缓冲区 a 的 fplc 系统上建立阳离子交换色谱柱 (见材料表)。
- 以 0.5 mL/min 的流速将蛋白质样品加载到平衡的阳离子交换色谱柱上。
- 在 40 ml 中, 通过混合缓冲液 a 和0至50% 缓冲 b (25 mm hepes, 1 m ncl, 1 mL dtt, 5% 甘油, ph 6.5), 以 1 ml/min 的流速排出氯化钠梯度的蛋白质。
- 使用 sds-page24检查不同组分的洗脱蛋白。
注: 协议可以在此处暂停。 - 将含有 cul1山梨酸甲的洗脱分数池。
- 通过超滤膜 (30 kda 截止) 将浓缩的cul1 山梨糖层酶·rbx1 样品浓缩至 2.5 ml。
- 通过排序酶介导的转位 21,22,将amc 添加到 cl-终止物中。
- 使用脱盐柱将 c4:rbx1样品中的缓冲液改为色氨酸酶缓冲液 (50 mm Tris-HCl、150 mm nacl、10 mm cacl2、ph 7.5) (见材料表)。
- 平衡具有25毫升的沙质酶缓冲液的脱盐柱。
- 将2.5 毫升的cul1 肌酸酶·rbx1 样品加载到色谱柱中。放弃流通。
- 用3.5 毫升的山梨酶缓冲液对样品进行洗脱。收集流经。
- 在100μl 的cul1 山梨酶中, 在酶缓冲液中加入100μl 的600μm 纯化山梨酶 a 溶液和10μl 的 25mm gggggggac 肽.在黑暗中隔夜将反应混合物在30°c 下培养。请注意, 此步骤将生成 cul1 amc·rbx1.
注意: 荧光染料对光敏感, 因此在蛋白质和样品制备过程中, 避免将其暴露在环境光中。
注: 协议可以在此处暂停。 - 在反应混合物中加入50μl 的 ni-nta 琼脂糖珠, 在室温下孵育30分钟。
- 将 ni-nta 琼脂糖珠颗粒在 5, 000 x 克离心 2分钟, 并收集上清液。
- 在 fplc 系统上平衡带有缓冲液 (30 mm tris-hcl, 100 mm 氯化萘, 1 mm dtt, 10% 甘油) 的尺寸排除色谱柱 (见材料表)。
- 将所有 cul1 amc·rbx1 样品加载到尺寸排除色谱柱上.具有1.5倍柱体积的缓冲液。
- 用 sds-page24检查洗脱分数。
- 将包含 cul1amc·rbx1 的洗脱分数集中起来.
- 用分光光度计测量蛋白质浓度, 使用其在280纳米的吸收率。
- 在-80°c 下, 将蛋白质溶液倾斜。
注: 协议可以在此处暂停。
- 使用脱盐柱将 c4:rbx1样品中的缓冲液改为色氨酸酶缓冲液 (50 mm Tris-HCl、150 mm nacl、10 mm cacl2、ph 7.5) (见材料表)。
3. flashcand1 的制备, fret 受体蛋白
注: 此部分中的大多数步骤与步骤2相同。下面将详细介绍不同的条件。
- 构建质粒, 表达大肠杆菌细胞中的人的四联坎托1。
- 通过常规 pcr25 (引物序列) 在 cand1的第15氨基酸之前添加编码为 "ccpgccggsg" (四胞氨酸标记) 的 dna 序列: tgtggctgcgcgcagcacacacaccaccagttag;(ccatgtccattgattccag)。
- 将 pcr 产物插入 pgex-4t-2 载体中。序列质粒, 以确认基因插入是准确的, 在框架内。
- 以与步骤2.2 相同的方式表达四氯化器cand1 e. e. e. e. e. e. e. e. e。
- 从大肠杆菌细胞中纯化四氯化萘蜡烛1。
- 用谷胱甘肽珠裂解大肠杆菌和提取四氯化枝蜡烛。这些步骤与 2.3.1 2.3.12 的步骤相同。
- 用缓冲液 c (50 mm tris-hcl, 1 mm dtt, 5% 甘油, ph 7.5) 稀释谷胱甘肽珠中的蛋白质洗脱。在带有缓冲区 c 的 fplc 系统上平衡阴离子交换色谱柱 (见材料表), 并以 0.5 mL/min 流速加载稀释的蛋白质样品。
- 通过混合缓冲液 c 和0至50% 缓冲 d (50 mm Tris-HCl, 1 m 氯化碳, 1 mL dtt, 5% 甘油, ph 7.5), 以 1 mlmin 流量在 40 ml 中使用氯化碳梯度 (50 mm Tris-HCl, 1 mm 氯化萘, 1% dtt, 5% 甘油, ph 7.5), 以氯化钠梯度排出蛋白。使用 sds-page24检查不同分数中的洗脱蛋白, 并将含有ttrecyscand1 的组分相列。请注意,与免费商品及服务税相比, 电话产品的保留量更大。
- 通过超滤膜 (30 kda 截止) 传递缓冲液, 将浓缩的四氯化萘cand1 样品。
- 在 fplc 系统上平衡带有标记缓冲液 (20 mm tris-hcl、100 mm 氯化钠、2 mm tcep、1 mm edta、5% 甘油) 的尺寸排除色谱柱。加载四氯化萘蜡烛1样品 (每次 500μl), 并通过 sds-page24检查洗脱分数。
- 通过超滤膜 (30 kda 截止) 将含有t龙奇坎德1 的所有组分集中在 ~ 40μm 中。使用其吸收度为280纳米来估计蛋白质浓度。将蛋白质作为50μl 的脂肪储存在-80°c。
注: 协议可以在此处暂停。
- flashcand1 的制备。
- 在50μl 条约蜡烛1溶液中加入 1μl的 flash溶液 (见材料表)。
- 在室温下在室温下混合, 孵育 1-2小时, 得到flashcand1。
注: 协议可以在此处暂停。
4. 蜡烛1的制备, fret 追逐蛋白
注: 蛋白质制备协议类似于步骤 3, 但有以下修改。
- 将全长 cand1 的编码序列插入 pgex-4t-2 矢量。
- 将步骤3.3.5 中使用的缓冲液改为含有 30 mm Tris-HCl、100 mm 氯化钠、1毫米 dtt、10% 甘油的缓冲液。
- 消除3.3.7 和3.3.8 的步骤。
5. 测试并确认 fret 检测
- 制备含有 30 mm Tris-HCl、100 mm 氯化钠、0.5 mm dtt、1 mg/ml 卵白蛋白、ph 7.6 的 fret 缓冲液, 并在室温下使用。
- 在荧光仪上测试 camc·rbx1 和flashcand1 之间的 fret.
- 在300μl 的 fret 缓冲液中, 将camc·rbx1 (fret 供体) 添加到最终浓度为 70 nm 的浓度中。将解决方案转换为 cuvette。
- 将立方体放入荧光灯的样品支架中。用350纳米激发光激发样品, 并以1纳米增量扫描400纳米至 600 nm 的发射信号。
- 重复步骤 5.2.1,但将 camc·rbx1 改为flashcand1 (fret 受体)。使用与步骤5.2.2 相同的方法扫描 flash cand1 样本。
- (可选)用 510 nm 激发光激发 flashcand1, 并扫描从500纳米到 650 nm 的发射信号。
- 在 300μl fret 缓冲液中, 将 cul1 amc·rbx1 和flashcand1 添加到 70 nm 的最终浓度中.以与步骤5.2.2 相同的方式分析示例。
- 通过添加追逐 (未标记的cand1) 蛋白质 (图 3c), 确认 camc·rbx1 和flashcand1 之间的 fret。
- 在300μl 的 fret 缓冲液中, 加入 70 nmcul1amc·rbx1 和 700 nm cand1。扫描样品发射, 如步骤5.2.2。
- 在300μl 的 fret 缓冲液中, 加入 70 nm flash cand1 和 700 nm cand1。扫描样品发射, 如步骤5.2.2。
- 在300μl 的 fret 缓冲液中, 加入 70 nmcul1 amc·rbx1 和 70 nm flashcand1, 并在室温下孵育样品5分钟。然后添加 700 nm cand1, 并在添加后立即扫描样品发射, 如步骤 5.2.2)。请注意, 此步骤类似于步骤5.2.5。
- 在 300μl fret 缓冲液中, 依次添加 70 nm cul1 amc·rbx1、700 nm cand1 和 70 nm flash cand1.在室温下对样品进行5分钟的孵化, 并按照5.2.2 的步骤扫描样品的排放。请注意, 这是追逐示例 (图 3c中的绿线)。
6. 测量 Cul1•Cand1 的关联速率常数 (k打开)
注: 操作停止流式荧光灯的详细信息已在上一份报告26中说明。
- 准备用于测量的塞流荧光灯。
- 根据制造商的说明打开停止流荧光灯。
- 将激发光设置为 350 nm, 并使用带通滤波器, 允许 450 nm 发射光通过, 并阻止 500-650 nm 发射光。
- 将样品阀放在填充位置, 并连接装满水的3毫升注射器。用水清洗两个样品注射器 (a 和 b), 方法是将样品注射器驱动器上下移动几次。丢弃此步骤中使用的所有水。
- 将样品阀保持在填充位置, 并连接装有 fret 缓冲器的3毫升注射器。用 fret 缓冲液清洗两个样品注射器, 方法是多次上下移动样品注射器驱动器。放弃此步骤中使用的所有 fret 缓冲区。
- 进行控制测量 (图 4c)。
- 连接3毫升注射器, 并在 fret 缓冲液中加载带有 100 nmcul1amc·rbx1 的注射器 a。将样品阀转到驱动位置。
- 连接3毫升注射器, 并使用 fret 缓冲液加载注射器 b。将样品阀转到驱动位置。
- 使用 "在软件中获取" 下的 "控制面板" 在停止流荧光仪上拍摄五张照片 (混合相同数量的注射器 a 和注射器 b 样本), 而无需记录结果。
- 打开 "在软件中获取" 下的 "控制面板", 并通过程序记录6零60秒以上的 c法1 amc 排放量.然后打一枪。
- 重复步骤 6.2.4 2x。
- 将样品阀转到填充位置。清空注射器 b, 用 fret 缓冲液清洗。
- 测量观察到的 Cul1•Cand1 关联速率常数 (kbs) (图 4b)。
- 将注射器 a 中的样品保持在步骤6.2.1 相同的地方。
- 连接一个3毫升注射器, 并加载注射器 b与100 nm flash cand1 在 fret 缓冲液。将样品阀转到驱动位置。
- 使用 "在软件中获取" 下的"控制面板" 拍摄五张照片, 而无需记录结果。
- 打开 "在软件中获取" 下的 "控制面板", 并通过程序记录6零60秒以上的 c法1 amc 排放量.然后打一枪。
- 重复步骤 6.3.4 2x。
- 清空注射器 b, 用 fret 缓冲液清洗。
- 重复步骤 6.3.1-6.3.6 几次, 在 fret 缓冲液中的 flashcand1 浓度不断增加。
- 将随着时间的推移, 从每次拍摄测量到的荧光信号的变化 (减少) 调整为一个指数曲线。这将在每次测量中给出k 点, 并且单位为s-1。请注意, 上一份报告27已很好地讨论了这一计算的基础。
- 计算所使用的每个flashcand1 浓度的 k 形的平均值和标准偏差。绘制平均k个小块对cand1 浓度 (图 4d), 线的斜率表示 Cul1•Cand1 的k, 单位为m-1 s-1。
7. 在存在 Skp1•F 盒蛋白的情况下测量 Cul1•Cand1 的离解率常数 (k关)。
注: 此步骤类似于步骤 6, 但进行了以下修改。
- 在注射器 a 中, 在填充位置下, 在 fret 缓冲区中加载 100 nm cul1amc·rbx1 和 100 nm flash cand1的解决方案。将样品阀转到 "驱动" 位置。
- 在注射器 b 中, 在"填充"位置下加载 Skp1•Skp2 的解决方案 (在上一次报告20之后准备)。将样品阀转到 "驱动" 位置。
- 打开 "在软件中获取" 下的 "控制面板", 并通过程序记录 cal1 amc 超过30秒的排放.然后打一枪。混合注射器 a 和注射器 b 溶液后, 荧光信号会随着时间的推移而增加 (图 5)。
Access restricted. Please log in or start a trial to view this content.
结果
为了测试 cul1amc 和 flashcand1 之间的 fret, 我们首先分别确定了 70 nm cul1 amc (供体) 和 70 nm flashcand1 (受体) 的排放强度 (图 3a-c, 蓝线)。在每次分析中, 只存在一个发射峰值,而 flashcand1 (受体) 的排放量较低。当每个 cul1amc 和 flashcand1 混合 70 nm 生成 fret 时, 发射光谱中存在两个发射峰, cul1...
Access restricted. Please log in or start a trial to view this content.
讨论
fret 是一种物理现象, 对研究和理解生物系统非常感兴趣。在这里, 我们提出了一个测试和使用 fret 研究两种相互作用的蛋白质的结合动力学的方案。在设计 fret 时, 我们考虑了三个主要因素: 供体发射和受体激励之间的光谱重叠、两个荧光体之间的距离以及荧光的偶极子方向 28.为了选择 fret 的荧光体, 我们对荧光的激发和发射光谱进行了叠加, 并寻找了其发射峰?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
我们感谢加州理工学院 (shu-ou shan) 就 fret 检测的发展进行了富有见地的讨论。硕士、y. z. 和 x. l. 由普渡大学向 y. z. 提供的创业资金资助 X.L.This 工作部分由普渡大学植物生物学中心的种子赠款提供。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
参考文献
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37(2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62(2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。