Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Protein kompleksleri milisaniyelik zaman ölçekte dinamikleri çalışmaya vitro Floresans rezonans enerji transferi içinde kullanma
Bu Makalede
Özet
Protein-protein etkileşimleri biyolojik sistemleri için kritik olan ve bağlama Kinetik çalışmalar dynamics ve protein kompleksleri fonksiyonu sağlar. Floresans rezonans enerji transferi ve durdu-akış tekniği kullanarak karmaşık bir proteinin Kinetik parametrelerin quantifies bir yöntemi açıklanmaktadır.
Özet
Proteinler biyolojik sistemleri Birincil operatörler, ve genellikle diğer makro veya onların biyolojik iþlevlerini küçük moleküller ile etkileşim. Bu tür etkileşimler son derece dinamik, etkileşen alt birimleri sürekli ilişkili ve belirli oranlarda ayrışmış anlamı olabilir. Nicel açılan bağlama Kinetik eğitim etkileşimin gücünü ortaya çıkarır gibi teknikleri kullanarak bağlama benzeşme ölçme anlayışlar üzerinde nasıl hızlı sağlarken etkileşim oluşur ve ne kadar her karmaşık bulunabilir. Ayrıca, bir etkileşim bir protein Döviz Çarpanı veya bir ilaç gibi ek bir faktör huzurunda Kinetik ölçme hangi etkileşim için önemli bilgi sağlayan diğer bir faktör tarafından düzenlenmiş mekanizması ortaya çıkarmak yardımcı olur biyolojik ve tıbbi araştırma ilerlemesi. Burada, biz yüksek iç Derneği oranına sahip ve hızlı bir şekilde başka bir protein tarafından ayrışmış bir protein kompleksi bağlama Kinetik ölçmek için bir iletişim kuralı tanımlamak. Protein kompleksi tüp bebek oluşumu raporlamak için Floresans rezonans enerji transferi yöntemi kullanır ve hızlı Derneği ve gerçek zamanlı olarak durdu-akış fluorimeter kompleks ayrılma izleme sağlar. Bu tahlil kullanarak, protein kompleksi Derneği ve ayrılma hızı sabitler sayısal.
Giriş
Biyolojik etkinlikler sonuçta proteinler, en hangisinin uygun biyolojik fonksiyonlar için başkalarıyla etkileşim tarafından yapılmaktadır. Hesaplamalı bir yaklaşım kullanarak, insan protein-protein etkileşimler toplam miktarı ~ 650.0001olduğu tahmin edilmektedir ve bozulma bu etkileşimlerin kez hastalıklar2' ye yol açar. Hücresel ve organizma işlemlerin denetlenmesinde önemli rolleri nedeniyle, Maya-iki-hibrid, bimolecular floresan uluslara, Böl-luciferase gibi protein-protein etkileşimleri çalışmak için çeşitli yöntemler geliştirilmiştir uluslara ve co-immunoprecipitation tahlil3. Bu yöntemler keşfetmek ve protein-protein etkileşimleri onaylayarak, iyi olmakla birlikte, genellikle sigara nicel ve böylece etkileşen protein ortakları arasında benzeşme hakkında sınırlı bilgi sağlar. Nicel çekme-çıkışlar bağlama benzeşim (örneğin, ayrışma sabiti Kd) ölçmek için kullanılabilir ama bağlama Kinetik ölçmek değil, ne de Kd bir yetersiz nedeniyle çok düşük olduğunda uygulanabilir Sinyal-gürültü oranı4. Yüzey plasmon rezonans (SPR) spektroskopisi bağlama Kinetik quantifies ama bir belirli yüzey ve potansiyel olarak reaksiyona5bağlama özelliğini değiştirebilirsiniz yüzeyi bir reaksiyona immobilizasyon gerektirir. Ayrıca, SPR hızlı Derneği ve ayrılma oranları5ölçmek zor ve SPR bir protein kompleksi protein alt alışverişi olay karakterize etmek için kullanmak için uygun değildir. Burada, protein karmaşık montaj ve demontaj, bir milisaniye zaman ölçeği oranları ölçme sağlayan bir yöntem açıklanmaktadır. Bu yöntem F-box protein Satım faktörü6,7 Cullin - ssociated -Nedd8 -dissociated protein 1 (Cand1)rolünü belirlemek için gerekliydi.
Cand1 Cullin-RING Ubikuitin ligazlar büyük ailesine ait Skp1•Cul1•F-box protein (SCF) E3 ligazlar dinamikleri düzenlemektedir. Yüzük etki alanı proteini Rbx1 bağlar, Cul1 ve yüzeylerde acemi ve Cul1 adaptör protein Skp18ile bağlar değiştirilebilir bir F-box protein, cullin ve SCFs oluşur. Bir E3 ligaz olarak SCF Ubikuitin, substrat için konjugasyon tromboksan ve ne zaman belgili tanımlık substrate F-box protein tarafından oluşturulmuştur ve Cul1 gibi Ubikuitin protein Nedd89tarafından değiştirildiğinde etkinleştirilir. Cand1 değiştirilmemiş Cul1 bağlar ve bağlayıcı, hem Skp1•F-box protein Cul1 ile dernek ve Nedd8 fiil Cul110,11,12,13bozan. Sonuç olarak, Cand1 bir inhibitörü SCF faaliyet tüp bebek olduğu ortaya çıktı, ama Cand1 eksikliği organizmalarda in vivo14,15,16 SCF faaliyetleri düzenleyen Cand1 olumlu bir rol göstermektedir kusur neden , 17. Bu paradoksun sonunda Cul1, Cand1 ve Skp1•F-box protein arasındaki dinamik etkileşimler ortaya bir nicel araştırma tarafından açıklandı. SCF ve Cul1•Cand1 komplekslerinin oluşumu tespit Floresans rezonans enerji transferi (FRET) deneyleri kullanarak, dernek ve ayrılma oranı sabitleri (kTarih ve kkapalı, sırasıyla) olduğunu tek tek ölçülür. Cand1 ve Skp1•F-box protein formu son derece sıkı kompleks Cul1, ama kkapalı SCF ile Cand1 tarafından önemli ölçüde artar ve kkapalı Cul1•Cand1 önemli ölçüde artırılır ölçümleri ortaya tarafından Skp1•F-box protein6,7. Bu sonuçlar Cul1 eski SCF kompleksleri geri dönüşüm yoluyla yeni SCF kompleksleri tromboksan protein Satım faktör olarak Cand1 rolü tanımlamak için ilk ve kritik destek sağlar.
Burada, biz geliştirme ve Cul1•Cand1 karmaşık7dinamiklerini incelemek için FRET tahlil kullanma prosedürü mevcut ve çeşitli biomolecules dinamiklerini incelemek için aynı prensip uygulanabilir. FRET oluşur uygun dalga boyu ile heyecan verici ve bir alıcısı donör emisyon spektrum örtüşen uyarma spektrum ile 10-100 bir mesafe içinde varsa Å. Heyecan durumu böylece donör yoğunluğu azalan ve artan alıcısı yoğunluğu18alıcısı için aktarılır. PERDE (E) verimliliğini Förster RADIUS (R0) ve donör ve alıcısı fluorophores (r) arasındaki mesafe bağlıdır ve tanımı: E = R06/ (R0 6 + r6) Donör-alıcısı çifti ve çözüm spektral çakışma19kullanılan, Förster RADIUS (R0) dipol açısal yönlendirme gibi birkaç etkene bağlıdır. Donör emisyon gerçek zamanlı olarak değişiklik izler ve hızlı kTarih ve kkapalı, bir durdu-akış fluorimeter perde tahlil uygulamak için verimli FRET kurmak gereklidir bu donör emisyon önemli bir azalma sonuçlarında. Bu nedenle, boyalar takmak için uygun çift floresan boyalar ve hedef proteinler sitelerinde seçerek verimli perde tasarımı önemlidir ve bu protokol için ele alınacak.
Protokol
1. tasarım perde tahlil.
- Cul1•Cand1 karmaşık yapısı dosyayı Protein veri bankasından (dosya 1U6G) yükleyin.
- Cul1•Cand1 PyMOL içinde karmaşık yapısını görüntüleyin.
- Cand1 ilk amino asit ve Cul1 son amino asit arasındaki uzaklık PyMOL Sihirbazı menüsünden Ölçüm işlevini kullanın (şekil 1).
- Online spectra Görüntüleyici yükler ( Tablo malzemelerigörmek) ve uyarma ve emisyon spectra 7-amino-4-methylcoumarin (AMC) ve FlAsH aynı anda görüntüleyebilirsiniz (Şekil 2). AMC FRET donör ve flaş perde alıcısı olduğunu unutmayın.
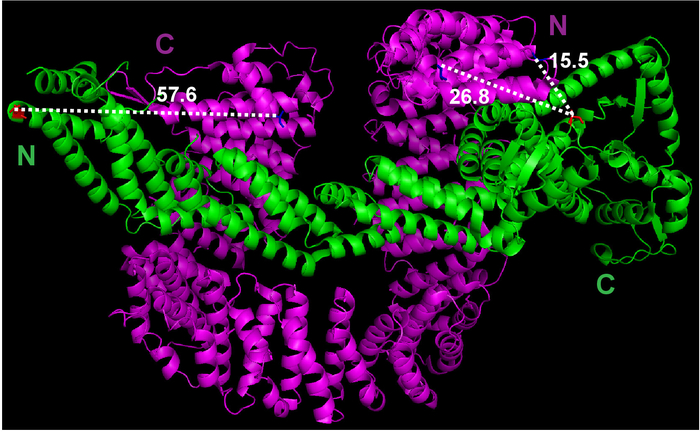
Şekil 1: Cul1•Cand1 ve etiketleme siteleri potansiyel arasındaki mesafe ölçümü kristal yapısını. Kristal yapısı dosya Protein veri bankasından (dosya 1U6G) indirilen ve PyMOL içinde görüntülenebilir. Seçili atomlar arasındaki ölçümleri PyMOL tarafından yapıldı. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
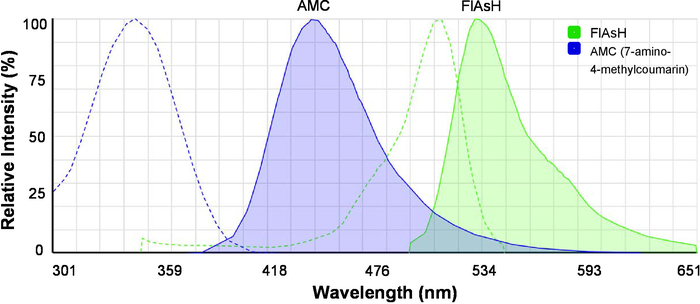
Şekil 2: uyarma ve floresan boyalar için FRET emisyon spectra. Spectra AMC (7-amino-4-methylcoumarin) ve FlAsH gösterilir. Kesik çizgiler uyarma spectra gösterir ve düz çizgiler emisyon spectra gösterir. Görüntü aslında floresan SpectraViewer tarafından oluşturulan ve daha iyi anlaşılır olması için güncellenmiştir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
2. Cul1 hazırlanmasıAMC•Rbx1, FRET donör protein
- Plazmid E. coli hücrelerdeki insan Cul1sortase•Rbx1 ifade etmek için inşa. E. coli hücrelerdeki insan Cul1•Rbx1 ortak ifade iki plazmid ayrıntılı bir önceki rapor20olarak açıklanan unutmayın.
- "LPETGGHHHHHH" (sortase-O'nun6 etiketi) kodlama bir DNA dizisi ile standart PCR ve kopyalama yöntemleri21,22Cul1 kodlama dizisi 3' sonuna ekleyin.
- Gen ekleme doğru olduğunu onaylamak için yeni plazmid sıra.
- Cul1sortase•Rbx1 E. coli hücrelerdeki co hızlı. Yöntem bir önceki rapor20türetilmiştir.
- Mix 100 ng her iki Plasmid'ler ile BL21 in (DE3) kimyasal olarak yetkili hücreleri ortak dönüşüm kullanarak ısı şok yöntemi23. Hücreleri 100 µg/mL Ampisilin ve 34 µg/mL kloramfenikol 37 ° C'de gecede içeren LB agar plaka üzerinde büyür.
- 50 mL taze dönüştürülmüş koloniler ile LB kültürünün aşılamak ve gecede 37 ° C'de 250 d/d sallayarak ile büyür. Bu bir starter kültür verir.
- 6 şişeler, her biri 1 L LB orta, 5 mL starter kültür her ile aşılamak ve 37 ° C'de 250 OD600 ~ 1.0 olana sallayarak rpm ile büyür. 16 ° c kültür serin ve izopropil-β-D-thiogalactoside (IPTG) eklemek için 0.4 mM. Kültür 16 ° C'de gecede 250 d/d sallayarak ile devam et.
- Santrifüjü 5000 x g 15dk için de ile E. coli hücreleri hasat ve hücre granül 50 mL konik tüpler toplamak.
Not: Hücre topakları protein arıtma işlenebilir veya protein saflaştırma adımlara geçmeden önce-80 ° C'de donmuş olabilir.
- Cul1sortase•Rbx1 karmaşık arıtma. Bu yöntem bir önceki rapor20türetilmiştir.
- 50 mL lizis arabelleği (30 mM Tris-HCl, 200 mM NaCl, 5 mM DTT, % 10 gliserol, 1 tablet proteaz inhibitörü kokteyl, pH 7,6) Cul1sortase•Rbx1 ifade E. coli hücreleri Pelet için ekleyin.
- Sonication % 50 genlik, buzla sayfasındaki hücreleri parçalayıcı. 1-ikinci açık ve 1 saniye kapalı arasında geçiş ve 3 dakikadır çalıştırın.
- 2.3.2 x 2-3 arasındaki adımları yineleyin.
- Hücre lysate 50 mL aralıklarla tüp içine aktarmak ve hücre artıkları Santrifüjü 25.000 x g için 45 dk de kaldırmak.
- Açik Hücre lysate ile 5 mL glutatyon boncuk 4 ° c 2 h için kuluçkaya.
- 1.500 x g 4 ° C'de 2 min için boncuk lysate karisimin santrifüj kapasitesi Süpernatant kaldırın.
- Boncuk 5 mL lizis arabelleği (ile hiçbir proteaz inhibitörleri) ile yıkama ve süpernatant Santrifüjü 1.500 x g 4 ° C'de 2 min için de sonra kaldırın
- 2.3.7 adımı yineleyin 2 x.
- 3 mL lizis arabellek yıkanmış boncuk ekleyin ve boncuk Bulamaç boş bir sütuna aktarmak.
- Elüsyon arabellek (50 mM Tris-HCl, 200 mM NaCl, 10 mM indirgenmiş glutatyon, faz 8.0) 5 mL sütuna ekleyin. 10 dakikadır kuluçkaya ve eluate toplamak.
- 2.3.10 x 3-4 arasındaki adımları yineleyin.
- 5 mg/mL Trombin 200 µL ekleyin ( Tablo malzemelerigörmek) glutatyon boncuk üzerinden eluate için ve bir gecede 4 ° C'de kuluçkaya
Not: Protokol burada duraklatılmış. - Protein örnek (25 mM HEPES, 1 mM DTT, %5 gliserol, pH 6,5) arabelleği A ile üç katı oranında seyreltin.
- Bir ba * exchange Kromatografi sütun equilibrate ( Tablo malzemelerigörmek) arabelleği A. ile bir FPLC sistemde
- Dengelenmiş katyon değişim Kromatografi sütun bir 0.5 mL/dk akış hızı, protein örneğe yükleyin.
- Protein ile 40 mL NaCl Gradyanda arabellek A ve 1 mL/dk akış hızı % arabellek B (25 mM HEPES, 1 M NaCl, 1 mM DTT, %5 gliserol, pH 6,5) 0-50 karıştırarak elute.
- Eluted protein farklı kesirler SDS-sayfa24kullanarak kontrol edin.
Not: Protokol burada duraklatılmış. - Cul1sortase•Rbx1 içeren eluate kesirler havuz.
- Arabelleğin bir Ultrafiltrasyon membran (30 kDa kesme) üzerinden geçerek havuza alınan Cul1sortase•Rbx1 örnek 2.5 ml konsantre.
- AMC Cul1 C-terminus sortase-aracılı transpeptidation21,22ekleyin.
- Sortase arabellek (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7.5) Cul1sortase•Rbx1 örnek arabellekte değiştirmek desalting sütun kullanarak ( Tablo malzemelerigörmek).
- Sortase arabellek 25 mL desalting bir sütun equilibrate.
- Cul1sortase•Rbx1 örnek 2.5 mL sütuna yerleştirin. Akışı aracılığıyla atın.
- Sortase arabellek 3,5 mL ile örnek elute. Akışı aracılığıyla toplamak.
- İçinde 900 µL Cul1sortase•Rbx1 çözüm sortase arabellek 600 saf µM sortase bir çözüm 100 µL ve 25 mM GGGGAMC peptid 10 µL ekleyin. Reaksiyon karışımı 30 ° C'de karanlık geceleme kuluçkaya. Not Bu adımda Cul1 çıkarılırAMC•Rbx1.
Dikkat: Floresan boyalar ışık, bu yüzden onları mümkün olduğunca protein ve örnek hazırlama sırasında ortam ışığına kullanılmasını önlemek için duyarlıdır.
Not: Protokol burada duraklatılmış. - Ni-NTA özel boncuk 50 µL tepki karışıma ekleyin ve 30 dk için oda sıcaklığında kuluçkaya.
- Ni-NTA özel boncuk tarafından Santrifüjü 5000 x g 2 min için de cips ve süpernatant toplamak.
- Bir boyut dışlama Kromatografi sütun equilibrate ( Tablo malzemelerigörmek) arabelleği (30 mM Tris-HCl, 100 mM NaCl, 1 mM DTT, % 10 gliserol) FPLC sistemi ile.
- Tüm Cul1 yükAMC•Rbx1 örnek boyutu dışlama Kromatografi sütun. 1.5 x sütun hacmi arabellek ile elute.
- Eluate kesirler SDS-sayfa24' kontrol edin.
- Cul1 içeren eluate kesirler havuzAMC•Rbx1.
- Protein konsantrasyonu 280 onun Absorbans kullanarak ölçmek nm bir Spektrofotometre ile.
- Aliquot protein çözüm ve mağaza-80 ° C'de
Not: Protokol burada duraklatılmış.
- Sortase arabellek (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7.5) Cul1sortase•Rbx1 örnek arabellekte değiştirmek desalting sütun kullanarak ( Tablo malzemelerigörmek).
3. FlAsHCand1, FRET alıcısı protein hazırlanması
Not: Çoğu adımları bu bölümünde, adım 2 ile aynıdır. Farklı koşullar aşağıda ayrıntılı olarak açıklanmıştır.
- İnsan TetraCysCand1 E. coli hücrelerdeki ifade etmek için plazmid oluşturmak.
- "CCPGCCGSG" (tetracysteine/TetraCys etiketi) kodlama DNA dizisi 15inci amino asit Cand1 ile düzenli PCR25 önce Ekle (astar sıraları: TGCTGTCCGGGCTGCTGCGGCAGCGGCATGACATCCAGCGACAAGGACTTTAG; CTAACTAGTGTCCATTGATTCCAAG).
- PCR ürünü pGEX-4T-2 vektör takın. Doğru ve çerçevede gen ekleme onaylamak için plazmid sıra.
- Plazmid Rosetta yetkili hücrelere dönüştürülür dışında olduğunu TetraCysCand1in E. coli hücreleri adım 2.2, aynı şekilde ifade eder.
- E. coli hücrelerden TetraCysCand1 arıtma.
- E. coli hücre topakları parçalayıcı ve TetraCysCand1using glutatyon boncuk ayıklayın. Aşağıdaki adımları adımları 2.3.1–2.3.12 aynıdır.
- Protein eluate arabellek C (50 mM Tris-HCl, 1 mM DTT, %5 gliserol, pH 7.5) ile glutatyon boncuk üç folds tarafından gelen sulandırmak. Bir anyon Satım Kromatografi sütun equilibrate ( Tablo malzemelerigörmek) arabellek C ve yük seyreltilmiş protein örneği bir 0.5 mL/dk akış hızı'ile FPLC sistemde.
- Protein ile 40 mL NaCl Gradyanda arabellek C ve 1 mL/dk akış hızı % arabellek D (50 mM Tris-HCl, 1 M NaCl, 1 mM DTT, %5 gliserol, pH 7.5) 0-50 karıştırarak elute. SDS-sayfa24kullanarak farklı kesirler eluted protein denetleyin ve TetraCysCand1 içeren kesirleri havuz. TetraCysCand1 ücretsiz GST daha büyük bir saklama hacim olduğuna dikkat edin.
- Havuza alınan TetraCysCand1 örnek arabellek Ultrafiltrasyon membran (30 kDa kesme) üzerinden geçerek konsantre.
- Boyutu dışlama Kromatografi sütun etiketleme arabelleği (20 mM Tris-HCl, 100 mM NaCl, 2 mM TCEP, 1 mM EDTA, %5 gliserol) ile FPLC sistemde equilibrate. TetraCysCand1 örnek (500 µL her zaman) yük ve eluate kesirler SDS-sayfa24' kontrol edin.
- TetraCysCand1 içeren tüm kesirler havuz ve arabelleğin bir Ultrafiltrasyon membran (30 kDa kesme) üzerinden geçerek ~ 40 µM için protein konsantre. Onun Absorbans 280 kullanarak protein konsantrasyonu tahmin nm. Protein 50 µL aliquots-80 ° C'de olarak saklamak
Not: Protokol burada duraklatılmış.
- FlAsHCand1 hazırlanması.
- FlAsH çözüm 1 µL ekleyin ( Tablo malzemelerigörmek) 50 µL TetraCysCand1 çözüm için.
- İyice karıştırın ve 1-2 h FlAsHCand1 almak için karanlık oda sıcaklığında karisimin kuluçkaya.
Not: Protokol burada duraklatılmış.
4. Cand1, FRET chase protein hazırlanması
Not: Protein hazırlık protokolü aşağıdaki değişiklikleri ile adım 3'e benzer.
- Tam uzunlukta Cand1 kodlama dizisi pGEX-4T-2 vektör yerleştirin.
- 30 mM Tris-HCl, 100 mM NaCl, 1 mM DTT, % 10 gliserol içeren bir arabellek 3.3.5. adımda kullanılan arabellek değiştirin.
- Adım 3.3.7 ve 3.3.8 ortadan kaldırmak.
5. test ve perde tahlil onaylayın
- 30 mM Tris-HCl, 100 mM NaCl, 0,5 mM DTT, 1 mg/mL ovalbumin, pH 7,6, içeren FRET arabellek hazırlamak ve oda sıcaklığında kullanın.
- Cul1 arasında perde testAMC•Rbx1 ve FlAsHCand1 üzerinde bir fluorimeter.
- İçinde 300 µL FRET arabelleği Cul1 eklemekAMC•Rbx1 (FRET donör) son konsantrasyonu 70 nM. Çözüm bir küvet aktarın.
- Küvet yer bir fluorimeter örnek tutucu. 350 nm uyarma ışık ile örnek heyecanlandıracak ve emisyon sinyal--dan 400 tarama 600 nm 1 nm aralıklarla nm'de.
- 5.2.1 adımı yineleyin ama değişiklik Cul1AMC•Rbx1 FlAsHCand1 için (FRET alıcısı). Adım 5.2.2 olduğu gibi aynı yöntemi kullanarak FlAsHCand1 örnek inceden inceye gözden geçirmek.
- (İsteğe bağlı) 510 nm uyarma ışık ile FlAsHCand1 heyecanlandıracak ve emisyon sinyal 500 üzerinden tarama nm 650 nm.
- Her iki Cul1 300 µL FRET arabellekte eklemekAMC•Rbx1 ve FlAsHCand1 son konsantrasyonu 70 nM. Adım 5.2.2 olduğu gibi aynı şekilde örnek analiz.
- Cul1 arasında perde onaylamakAMC•Rbx1 ve chase (etiketsiz Cand1) protein (şekil 3 c) ekleyerek FlAsHCand1.
- 70 300 µL FRET arabelleği eklemek nM Cul1AMC•Rbx1 ve 700 nM Cand1. Adım 5.2.2 olduğu gibi örnek emisyon inceden inceye gözden geçirmek.
- 70 300 µL FRET arabelleği eklemek nM FlAsHCand1 ve 700 nM Cand1. Adım 5.2.2 olduğu gibi örnek emisyon inceden inceye gözden geçirmek.
- 70 300 µL FRET arabelleği eklemek nM Cul1AMC•Rbx1 ve 70 nM Cand1, FlAsHve 5 min için oda sıcaklığında örnek kuluçkaya. Sonra ekleyin 700 nM Cand1 ve hemen eklenmesinden sonra adım 5.2.2 olduğu gibi örnek emisyon tarama). Bu adım adım 5.2.5 benzer olduğunu unutmayın.
- 300 µL FRET arabellekte sırayla ekleyin 70 nM Cul1AMC•Rbx1, 700 nM Cand1 ve 70 nM FlAsHCand1. Örnek 5 min için oda sıcaklığında kuluçkaya ve adım 5.2.2 olduğu gibi örnek emisyon tarayın. Bu chase örnek ( şekil 3 ciçinde yeşil hat) olduğuna dikkat edin.
6. Cul1•Cand1 (küzerinde) Derneği hızı sabiti ölçmek
Not: durdu-akış fluorimeter faaliyet detayları bir önceki rapor26yılında tarif edilmiştir.
- Durdu-akış fluorimeter ölçüm için hazırlayın.
- Üreticinin yönerge göre durdu-akış fluorimeter açın.
- Işık uyarma 350 nm ve 450 nm emisyon olanak tanıyan bir bant geçiren Filtre geçmesine ışık ve 500-650 nm emisyon ışık engeller kullanım ayarlamak.
- Örnek vanalar DOLDURMAK konumda tutmak ve su 3 mL şırınga bağlayın. İki örnek şırınga (A ve B) örnek şırınga sürücü birkaç kez yukarı ve aşağı hareket ettirerek su ile yıkayın. Bu adımda kullanılan su atın.
- Örnek vanalar DOLDURMAK konumda tutmak ve perde tampon ile dolu bir 3 mL şırınga bağlayın. İki örnek şırınga FRET arabelleği ile örnek şırınga drive birkaç saat boyunca taşıyarak yıkayın. Bu adımda kullanılan tüm perde tampon atmak.
- Bir denetim ölçüm (şekil 4 c) alın.
- 3 mL şırınga bağlanmak ve şırınga A 100 nM Cul1AMC•Rbx1 perde tampon yükleyin. Örnek Vana sürücü konumuna dön.
- 3 mL şırınga bağlanmak ve şırınga B ile perde tampon yüklemek. Örnek Vana sürücü konumuna dön.
- Edinme altında Denetim Masası (şırınga A ve şırınga B örnekleri eşit miktarda karıştırın) beş üzerinde durdu-akış fluorimeter sonuçları kayıt olmadan çekim için yazılımı kullanın.
- Edinme altında Denetim Masası yazılımı açın ve programı Cul1 emisyon kayıt içinAMC 60 s. O zaman tek bir atış yap.
- 6.2.4 adımı yineleyin 2 x.
- Örnek kapak DOLDURMAK pozisyonuna dön. Şırınga B boş ve perde tampon ile yıkayın.
- Ölçü Derneği hızı sabitler (kobs) Cul1•Cand1 görülmektedir (şekil 4B).
- Örnek A şırıngada tutmak adım 6.2.1 aynıdır.
- 3 mL şırınga bağlanmak ve şırınga B 100 ile yük nM FlAsHCand1 perde tampon. Örnek Vana sürücü konumuna dön.
- Edinme altında Denetim Masası sonuçları kayıt olmadan beş çekim için yazılımı kullanın.
- Edinme altında Denetim Masası yazılımı açın ve programı Cul1 emisyon kayıt içinAMC 60 s. O zaman tek bir atış yap.
- 6.3.4 adımı yineleyin 2 x.
- Şırınga B boş ve perde tampon ile yıkayın.
- Adımları 6.3.1–6.3.6 birkaç kez FlAsHCand1 perde tampon konsantrasyonları artan ile yineleyin.
- Değiştir (azalış) floresan sinyallerini tek bir üstel eğriyi kareden, her zaman içinde ölçülen uygun. Bu her ölçüm kobsverecektir ve s-1birimdir. Not Bu hesaplama temeli bir önceki rapor27' de tartışıldı.
- Ortalama ve kobsher FlAsHCand1 yoğunlaşması kullanılan standart sapmasını hesaplar. Cand1 konsantrasyonu (şekil 4 d) karşı ortalama kobsarsa ve çizgisinin eğimini küzerinde Cul1•Cand1, M-1 s-1birimi ile temsil eder.
7. ayrılma hızı sabiti (kkapalı) Cul1•Cand1 Skp1•F-box protein huzurunda ölçmek.
Not: Bu adım adım 6 için aşağıdaki değişiklikleri ile benzerdir.
- Yük DOLDURMAK konum'un altında şırınga A, içinde 100 nM Cul1AMC•Rbx1 bir çözüm ve 100 nM FlAsHCand1 perde tampon. Örnek Vana "Sürücü" pozisyonuna dön.
- Şırınga B DOLDURMAK konumu'nun altında Skp1•Skp2 (bir önceki rapor20hazırlanan) bir çözüm yükleyin. Örnek Vana "Sürücü" pozisyonuna dön.
- Edinme altında Denetim Masası yazılımı açın ve programı Cul1 emisyon kayıt içinAMC 30 s. O zaman tek bir atış yap. Floresan sinyallerini şırınga A ve şırınga B (şekil 5) çözümler karıştırma sonra zamanla artırın.
Sonuçlar
PERDE arasında Cul1 test etmek içinAMC ve FlAsHCand1, biz ilk 70 emisyon yoğunluğu tespit nM Cul1AMC (donör) ve 70 nM FlAsHCand1 (alıcısı), sırasıyla (şekil 3A-C, mavi çizgiler). Sadece bugünü ve FlAsHCand1 emisyon bir emisyon zirve oldu her analizde, (alıcısı) düşük. 70 zaman her Cul1 nM FRET oluşturmak için karışıkAMC ve FlAsHCand1, iki emi...
Tartışmalar
PERDE eğitim ve biyolojik sistemleri19anlamak için büyük ilgi olduğunu fiziksel bir olgudur. Burada, test etme ve iki etkileşen protein bağlayıcı Kinetik çalışmaya FRET kullanma için bir iletişim kuralı mevcut. PERDE tasarımı yaparken, biz üç önemli faktörler olarak kabul: donör emisyon ve alıcısı uyarma, iki fluorophores ve fluorophores28dipol yönünü arasındaki mesafe arasında spektral örtüşme. Fluorophores perde için seçmek için uyarma ...
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Shu-Ou Shan (Kaliforniya Teknoloji Enstitüsü) perde tahlil gelişimi üzerinde anlayışlı tartışma için teşekkür ediyoruz. M.G., Y.Z. ve X.L. Y.Z. Purdue Üniversitesi'nden başlangıç fonlar tarafından finanse edilmiştir ve X.L.This iş kısmen bitki Biyoloji için Purdue Üniversitesi Merkezi bir tohum hibe tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
Referanslar
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37 (2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62 (2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -. C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır